非洲猪瘟病毒解旋酶D1133L基因序列分析、蛋白结构预测及亚细胞定位
2021-07-26申超超张大俊史喜绢崔卉梅袁兴国赵登率陈学辉张克山郑海学刘湘涛
侯 景,申超超,张大俊,杨 博,史喜绢,张 婷,崔卉梅,袁兴国,赵登率,陈学辉,张克山,郑海学,刘湘涛
(中国农业科学院兰州兽医研究所 家畜疫病病原生物学国家重点实验室 农业农村部畜禽病毒学重点开放实验室 国家口蹄疫参考实验室,兰州 730046)
1921年,非洲猪瘟(African swine fever,ASF)首次在肯尼亚报道[1],2018年,首次传入中国,由于其高死亡率引起生猪大面积的死亡,给我国生猪养殖业和经济发展带来严峻挑战[2-3]。非洲猪瘟病毒(African swine fever virus,ASFV)是ASFV科的唯一成员,是线性双链DNA病毒,呈直径约为200 nm的正二十面体,其含有直径70~100 nm的DNA核心,外周是直径172~191 nm的衣壳和含类脂的囊膜,整个基因组大约含有151个开放阅读框(open reading frame,ORF),可以编码150~200种蛋白[4-10]。
ASFV基因组编码5个解旋酶,分别被命名为D1133L、B962L、QP509L、Q706L、A859L,均属于解旋酶超家族II家族成员。ASFV的5个解旋酶中,QP509L和Q706L属于解旋酶超家族II DEXH/D-box家族中DEAD-box家族成员,通过基因序列分析间接证明D1133L为该家族成员[11]。DEAD-box家族解旋酶的最主要特征为含有D-E-A-D(天冬氨酸-谷氨酸-甘氨酸-天冬氨酸)特定的氨基酸序列[12-13],该家族Elf4A和Elf4B解旋酶发挥功能的氨基酸是谷氨酸,因谷氨酸为酸性氨基酸在R基团的碳环上含有极键[14],因此谷氨酸在DEAD-box家族中更加稳定保守。在天然免疫领域中的DEAD-box家族解旋酶主要作用是促进双链DNA、双链RNA解螺旋,与mRNA 结合抑制转录水平进而抑制翻译,在蛋白水平上抑制宿主天然免疫应答进而促进肿瘤发生和炎症形成[12]。研究证明D1133L和B962L位于ASFV的病毒基因组中心,两者被40 kb碱基分开。D1133L和B962L类似于酵母“DEAH”前期mRNA 处理牛痘病毒ATP酶的D11L和D6R蛋白[15]。
本团队研究发现D1133L蛋白明显抑制宿主天然免疫应答(数据未发表),已报道单独敲除D1133L、QP509L和Q706L后ASFV无法复制,说明解旋酶是ASFV的关键基因[11]。为进一步明确ASFV D1133L基因结构和功能的关系,本研究使用Mega6.0软件绘制了ASFV编码的5个解旋酶基因系统进化树,对D1133L蛋白进行了二、三级结构预测,合成了D1133L基因,构建了D1133L真核表达质粒,研究了D1133L蛋白在宿主细胞的亚细胞定位。
1 材料与方法
1.1 材料
pCMV-N-flag真核表达载体、293T人源肾上皮细胞、PK-15猪源肾上皮细胞均由兰州兽医研究所口蹄疫与新发病流行性病创新团队保存;pCMV-N-flag-D1133L基因的真核表达质粒由武汉金开瑞合成;大肠杆菌Trans5α 感受态细胞购自全式金公司(CD201-02);EcoR Ⅰ内切酶(1040s)、BamH Ⅰ内切酶(1010s)、PrimeSTAR®HS (Premix)(R040A)购于TaKaRa公司;LipofectamineTM2000转染试剂购于Invitrogen公司(11668019);胶回收试剂盒(D2500-2)、质粒小量提取试剂盒(D6943-02)均购于Omega公司;DMEM培养基(C11965500BT)、Opti-MEM(31985070)和0.25% EDTA胰酶(25200072)均购于Gibco公司;PBS溶液(pH7.4)购于Hyclone公司(SH30256.01);鼠抗FLAG单抗(80010-1-RR)、HRP标记Alexa山羊抗鼠IgG二抗(SA00001-1)均购于Proteintech公司;鼠抗β-Actin单抗(MA1-140)购于Thermo Scientific公司。3 cm玻底共聚焦专用小皿(801001)购自NEST公司。ASFV阳性灭活血清由中国农业科学院兰州兽医研究所口蹄疫与新发病流行病学团队提供。
1.2 D1133L基因序列分析及结构预测
从NCBI数据库中下载ASFV 5个解旋酶的基因序列和NCBI数据库中公布的7株不同基因型的D1133L基因序列,利用在线工具PRABI、GOR4、SWISS-MODEL、cNLS Mapper、EXPASY、Psort等和SnapGene 4.1.2、Mega 6、RasMol2.7、DNAman分析软件对7株不同基因型的D1133L基因序列多基因序列比对、基因系统进化树构建、同源性结构分析,并对5个解旋酶多基因序列比对、基因系统进化树构建、同源性结构分析、跨膜区、NLS序列和二、三级结构预测等。
1.3 D1133L真核表达质粒构建
以GenBank公布的Georgia 2007/1(LR743116.1)基因组数据中D1133L基因序列为参考,全基因合成D1133L的基因序列,经序列测定鉴定正确,将该基因正确插入到pCMV-N-flag表达质粒中。
1.4 重组质粒表达验证
以1×106293T细胞量铺于6孔板,5%CO2,37 ℃,放置5~8 h,待细胞长至80%~90%时,用脂质体2000转染293T细胞(参考说明书),转染量为1 μg·孔-1(DNA∶脂质体2000=1 μg∶2 μL)。制备细胞蛋白样品:转染24 h后,收集转染重组质粒pCMV-N-flag-D1133L的细胞以及转染空载的对照细胞,加入RAPI裂解液(含1 mmol·L-1PMSF),充分裂解后,12 000 r·min-1离心10 min,收集上清,加入5 ×SDS 蛋白上样缓冲液,100 ℃煮样10 min。配制5%的浓缩胶和10%的分离胶,以鼠抗flag单抗为一抗,HRP标记的山羊抗鼠IgG抗体为二抗,Western blot验证pCMV-N-flag-D1133L的表达。
1.5 亚细胞定位观察
以1×106PK-15细胞铺于3 cm玻底培养皿,用脂质体2000转染0.5 μg pCMV-N-flag-D1133L重组质粒(参考说明书),转染12 h后,弃掉上清,PBS(pH 7.4)洗涤3次,每次10 min,再用4%多聚甲醛固定24 h,PBST(pH 7.4,含0.5%Tween 20)洗涤3次,Triton-X100处理10 min,2%BSA封闭1 h,PBST(同上)洗涤3次,加入ASFV阳性灭活血清(1∶100),孵育18 h,PBS(pH 7.4)洗涤3次,再孵育偶联荧光Alexa山羊抗鼠IgG二抗(1∶500)5 h,PBS(同上)洗涤3次,加入4,6-二脒基-2-苯基吲哚(4,6-diamidino-2-phenylindole,DAPI)对细胞核染色10 min,再用PBS(pH 7.4)洗涤3次,每次2 min,激光共聚焦显微镜63×油镜下观察荧光蛋白的亚细胞定位分布。此外,通过Image J软件分别检测了D1133L阳性蛋白在细胞核与细胞质的分布,并统计总分布(重复3次)。
1.6 统计分析
将Image J检测的3次数据求取平均值,以总分布作为对照,通过对照/对照,平均值/对照计算D1133L蛋白在PK-15细胞的分布比例,并利用Graph pad prism 8.0.1绘图。
2 结 果
2.1 ASFV 编码的解旋酶基因序列比较分析
根据NCBI在线数据库中公布的7株不同基因型的D1133L基因;以Georgia 2007/1(LR743116.1)序列,获得5个解旋酶基因,各个解旋酶详情见表1。7株不同基因型的D1133L核苷酸序列完全一致(图1A)。在5个解旋酶基因中,D1133L与其他4个 解旋酶核苷酸同源性较低,且5个解旋酶均相对保守独立,应用Mega6构建基因系统进化树,发现D1133L与Q706L在全基因组中距离较近(图1B)。
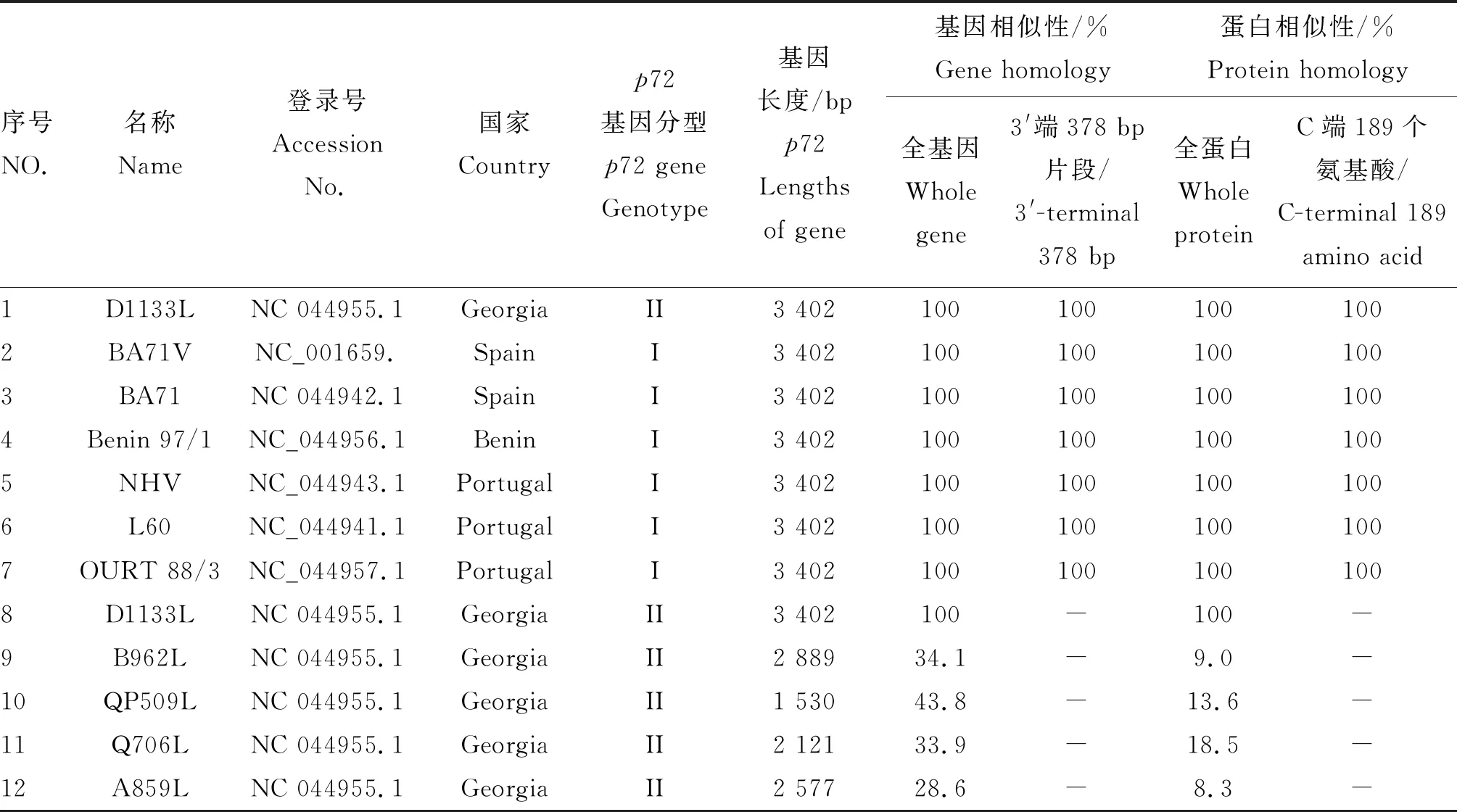
表1 解旋酶基因相似性比较Table 1 Gene homology comparison of helicase genes
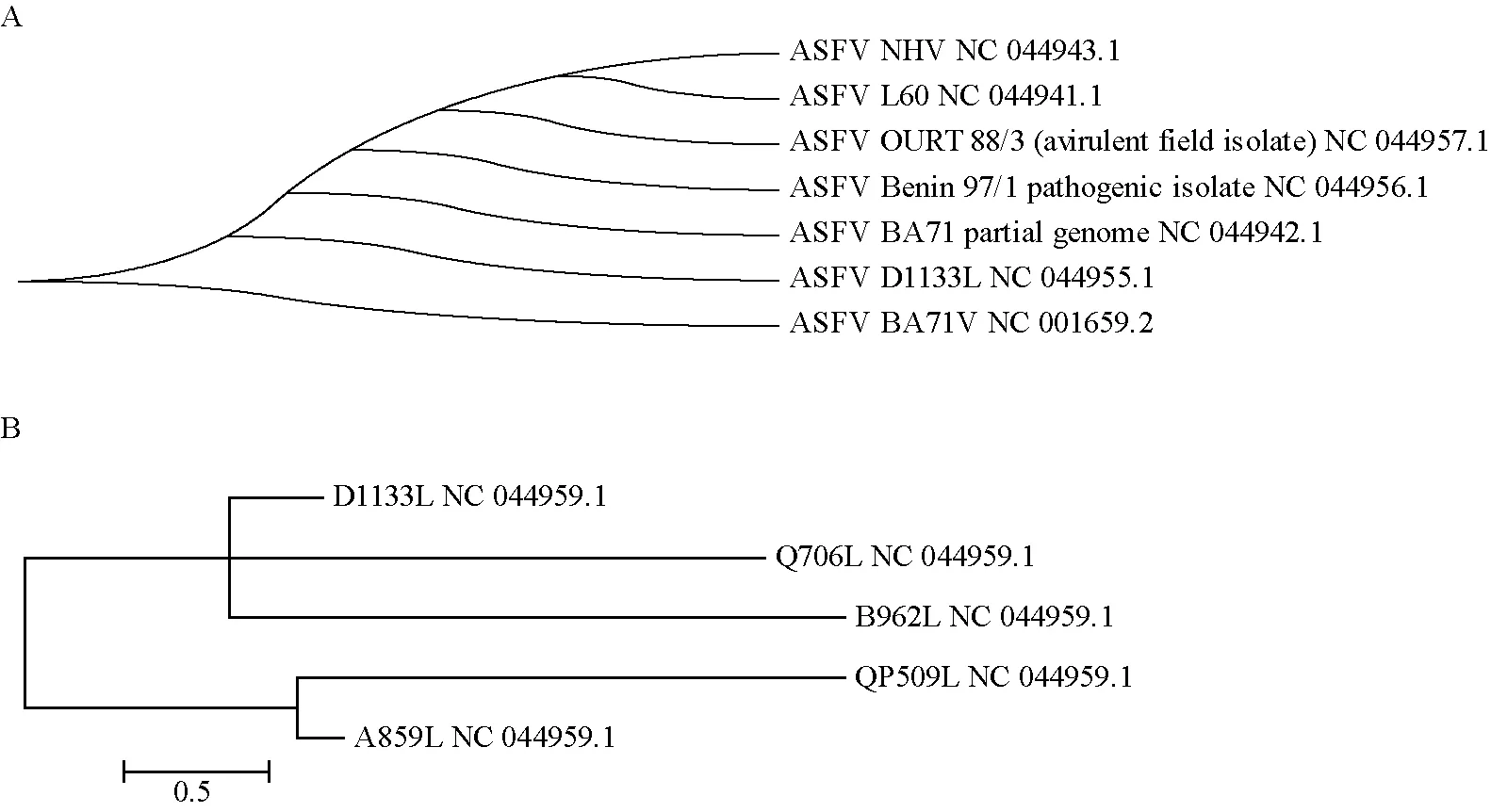
A.7株不同基因型的D1133L基因;B.获得的5个解旋酶基因A.D1133L genes of 7 different genotypes;B.5 helicase genes obtained图1 ASFV解旋酶基因系统进化树Fig.1 Phylogenetic tree of helicase genes
Georgia 2007/1为Ⅱ型强毒株,通过DNAMAN序列比对,7个不同的D1133L氨基酸序列[D1133L(NC_044959.1)、BA71V(NC_001659.)、BA71(NC044942.1)、Benin 97/1(NC_044956.1)、NHV(NC_044943.1)、L60(NC_044941.1)、OURT 88/3(NC_044957.1)]完全一致(结果略)。5个解旋酶基因在Georgia 2007/1毒株中仅有7个同源位点,5个解旋酶高度保守,通过相似性分析发现D1133L与QP509L在5个解旋酶当中距离较近,详见表2。

表2 5个解旋酶氨基酸同源性分析Table 2 Homology analysis of 5 helicase proteins
2.2 D1133L蛋白结构预测
7株D1133L基因一级结构表明该基因全长均为3 402 bp,G+C含量为6.1%,A+T含量为10.6%(图2A);表达的蛋白为124.63 ku,通过GOR4网络预测该蛋白二级结构结果显示α螺旋占48.90%,无规卷曲为33.27%,扩展链占13.50% (图2B),氨基酸相似域曲线分析结果表明D1133L蛋白主要为组氨酸,其次是谷氨酸、半胱氨酸、苏氨酸、甘氨酸和天冬氨酸(图2C)。

A.一级结构;B.二级结构;C.氨基酸序列分析A.Primary structure;B.Secondary structure;C.Amino acid sequence analysis图2 GOR4软件分析并预测D1133L蛋白二级结构Fig.2 Analysis and prediction of secondary structure of D1133L protein
将D1133L蛋白氨基酸序列提交在线工具Swiss-Model,获得了三维结构模型,结果显示预测的D1133L蛋白三级结构与Ankyrin-1、Ankyrin-2结构的QMQE为0.20,覆盖率84%;证明了D1133L蛋白的高级结构是α螺旋为主(图3)。

A.骨架结构;B.带状结构;C.球形结构A.Backbone structure;B.Ribbon structure;C.Spherical structure图3 D1133L蛋白三级结构预测Fig.3 Prediction of tertiary structure of D1133L protein
将D1133L蛋白序列输入在线工具NLS-Mapper中,预测得到D1133L蛋白可能包括28段NLS双分型序列(无NLS单分型序列),其Cut-off分数均大于2.0,根据得分从高到低排序(表3)。基于SVM算法的预测方法LOCSVMPSI和基于PSI-BLAST数据的预测方法HSLpred均显示D1133L蛋白可能定位于线粒体的概率为4.3%,分泌系统囊泡的概率为4.3%,真核细胞核内的概率为43.5%,真核细胞质的概率为43.5%。

表3 PCMV-D1133L蛋白序列的双分型NLS预测Table 3 Prediction of the bimorphic NLS of PCMV-D1133L protein sequence
2.3 pCMV-N-flag-D1133L重组质粒构建和表达验证
转染细胞24 h后,按照“1.4”对细胞样品处理,WB验证pCMV-N-flag-D1133L真核质粒的表达大小为124.63 ku,与预期一致。内参为细胞骨架微丝肌动蛋白β-actin蛋白(42 ku)。结果证明,构建的pCMV-N-flag-D1133L真核表达质粒可以在293T人源肾上皮细胞中表达(图4 A),且β-actin正常表达 (图4B)。

A.pCMV-N-flag-D1133L表达验证(-.EV过表达产物;+.D1133L过表达产物);B.内参(-.EV过表达产物内参;+.D1133L过表达产物内参)A.pCMV-N-flag-d1133l expression verification (-.293T cell lysis product;+.D1133L overexpression product);B.Internal reference (-.Internal reference of lysis product of 293T cells;+.Internal reference of D1133L overexpressed product)图4 293T细胞Western blot验证pCMV-N-flag-D1133L重组质粒的表达Fig.4 Expression verification of pCMV-N-flag-D1133L recombinant plasmid by Western blot
2.4 PCMV-D1133L蛋白的亚细胞定位观察
按照“1.5”对细胞样品处理,转染细胞24 h后,通过3 cm玻底小皿观察DAPI染色细胞核可见呈蓝色荧光(图5A),pCMV-N-flag-D1133L蛋白是红色荧光(Alexa)(图5B)定位在细胞核与细胞质中 (图5C)。Graph pad prism 8.0.1统计结果表明,D1133L蛋白在PK-15细胞的细胞核中占1.58%,细胞质中占7.5% (图5D)。

A.细胞核(DAPI);B.pCMV-N-flag-D1133L蛋白(Alexa);C.A、B合并;D.D1133L蛋白在细胞核与细胞质的分布比例A.Nucleus (DAPI);B.pCMV-N-flag-D1133L (Alexa);C.Merge of A,B;D.D1133L protein ratio图5 pCMV-N-flag-D1133L蛋白亚细胞定位观察(PK-15细胞)Fig.5 Observation of subcellular localization of pCMV-N-flag-D1133L protein (in PK-15 cells)
3 讨 论
根据NCBI已公布的ASFV序列数据,其基因组长度为170 ~190 kb,能够编码大约100多种非结构蛋白和超过50种的结构蛋白,ASFV入侵宿主后能够快速激活天然免疫反应、与先天性宿主天然免疫拮抗竞争,活化诱导淋巴细胞、产生免疫抗体,在病毒不断刺激下进而击溃天然免疫系统,最急性可在未发病期间直接导致生猪突然死亡[9,16-19],推测可能由于病毒繁殖速度快,病毒核酸在细胞质中包装完整ASFV,部分蛋白介入细胞核对核内IFN-β或NF-κB产生抑制作用,导致天然免疫系统不能抵抗病毒,最终导致机体崩溃,但其机制尚未明确[20-23]。国内外关于ASFV致病机制的研究仍未详尽[24-25]。
解旋酶家族分为SF1、SF2、SF3、SF4、SF5、SF6六类,其SF1主要是原核类,SF2是哺乳动物类,SF3~6是昆虫节肢类,数量相当大,至今解旋酶家族全部成员也没有完全统计出来[26-28]。现已知ASFV的RNA解旋酶包括D1133L、B962L、QP509L、Q706L和A859L,其中D1133L、QP509L、Q706L已经被报道为解旋酶超家族II DEXH/D-box家族中DEAD-box家族成员[11],ASFV复制分为早期、中期和晚期[29],研究发现D1133L在ASFV的晚期复制,D1133L的复制、转录与牛痘病毒的D6R和D11相似[15]。相关研究提出ASFV内pE248R 参与病毒融合,pE248R与痘病毒进入宿主细胞融合复合物的某些成员具有序列相似性[30-34]。在此可以预测LD1133L的复制方式与pE248R具有相似性[35-37]。此外,本研究通过7株不同基因型的D1133L多基因型序列分析及多氨基酸序列分析,结果显示完全一致。ASFV基因组中的5个解旋酶的生物信息学分析、多基因序列相似性分析和基因系统进化树结果显示,ASFV 5个解旋酶同源性较低,氨基酸相似性分析发现,5个解旋酶不仅在基因序列上是相对保守的,在氨基酸水平上也相对保守,相比之下,D1133L与QP509L的氨基酸序列较为相似。通过预测分析和IFA检测,D1133L定位于细胞核与细胞质。因此,D1133L可能是ASFV复制、转录的关键蛋白,本研究明确了ASFV解旋酶D1133L基因序列和氨基酸序列在不同毒株间相似性是100%,进一步阐述了D1133L基因在同一毒株当中与其他4个解旋酶的基因序列和氨基酸序列没有同源性。此外,预测了D1133L蛋白的结构与分布,同时过表达D1133L后通过IFA在PK-15细胞中证实了蛋白分布,在细胞核中占1.58%,细胞质中占7.5%。研究报道,DEAD-box解旋酶或可成为疫苗研发的新途径,在复合佐剂方面有着广阔的开发空间[38-42]。本研究绘制了ASFV编码的5个解旋酶基因的遗传进化图,预测了其D1133L基因的二、三级结构并明确了D1133L基因的亚细胞定位,研究结果为从ASFV编码的解旋酶角度揭示ASFV致病机制积累了资料。
4 结 论
ASFV包含5个解旋酶,均属于DEAD-box家族成员,其中D1133L蛋白在7株不同ASFV毒株中高度保守,其二级结构以α螺旋为主,与ASFV的另一解旋酶QP509L进化关系较近。IFA结果表明,D1133L可同时分布于细胞质与细胞核,预测显示其可能还定位于线粒体、分泌系统囊泡中。D1133L蛋白在真核细胞中的分布比例为细胞核占1.58%,细胞质占7.5%。这将为研究D1133L蛋白在ASFV自有的复制和转录系统中的功能提供参考。
