外周血单个核细胞GPSM2水平可能是肝细胞癌预后判断的新型标志物*
2021-07-14杨永平
于 哲,罗 婧,杨永平
肝癌是一种常见的消化系统恶性肿瘤,全球年新增肝癌病例达84.1万,死亡约78.2万,分别排在恶性肿瘤的第6位和第2位[1]。肝细胞癌(hepatocellular carcinoma, HCC)这一病理学类型占肝癌总体近90%,即使在发达国家其5年生存率也低于20%。因此,对HCC的早期诊治尤为重要。目前,HCC的确诊、复查和预后判断仍依赖血清学、影像学和组织病理学检查。组织病理学检查存在有创性,血清学检查的有效性稍差,反复CT或MRI检查存在着放射性污染和费用贵的问题。因此,亟需更精准的无创性生物标记物用于HCC患者的诊断和预后判断。有研究报道,乳腺癌细胞可将外周血单个核细胞(peripheral blood mononuclear cells,PBMC)募集到肿瘤部位,在肿瘤微环境中将其转化为促进肿瘤发展的免疫细胞,随后激活相关信号通路,改变PBMC的基因表达谱[2]。因此,PBMC基因水平差异的变化往往早于肿瘤周围的正常组织,提示通过检测PBMC基因有可能实现对肿瘤更精准的早期诊断或预后判断。多组学结合生物信息学工具的联合应用使我们有望找到HCC潜在的关键基因和治疗靶点。本研究通过挖掘公共数据库中HCC患者基因和临床数据,探索了HCC患者PBMC与肿瘤组织基因水平变化的相似性,以寻找有潜力的PBMC关键基因,研究关键基因在肿瘤发展过程中可能的作用及其与预后的关系,以期找到诊断HCC的新型标志物,为HCC的早期诊断和预后判断提供新指标。
1 资料与方法
1.1 数据检索 从NCBI-GEO数据库(https://www.ncbi.nlm.nih.gov/geo/)[3]检索数据集,纳入标准如下:①同时包含有HCC患者和健康人的PBMC资料数据;②HCC患者和健康人样本量均≥10例;③数据检测类型为总RNA。最终选择GSE49515数据集进行深入分析。该数据集基于GPL570芯片分析平台的处理,包含26例PBMC样本,其中HCC患者10例、健康人10例、胃癌3例和胰腺癌3例。本研究选择HCC患者和健康人样本进行研究[4]。此外,在TCGA数据库(https://portal.gdc.cancer.gov/)下载HCC患者基因和临床数据,用于分析关键基因在HCC患者肿瘤组织中的表达水平与预后的关系。
1.2 数据分析 本研究所用各类分析工具如下:应用在线分析工具GEO2R(https://www.ncbi.nlm.nih.gov/geo/geo2r/)获取HCC与健康人间的差异表达基因(differentially expressed genes, DEGs)。当一个探针对应多个基因时,说明探针特异性不强,予以删除;当多个探针对应一个基因时,取差异倍数(fold change,FC)绝对值的最大值。应用STRING(https://string-db.org/)[5]完成基因功能与通路的富集分析及蛋白质相互作用(protein-protein interaction, PPI)网络的构建。应用软件Cytoscape 3.7.2[6]及其中的MCODE插件完成PPI网络可视化和基因模块的筛选。应用GEPIA(http://gepia.cancer-pku.cn/)[7]完成关键模块基因的生存分析。GEPIA数据库整合了TCGA和GTEx两大数据库,可有效验证基因水平与生存期的关系。最后,应用IBM SPSS Statistics 25结合TCGA中的临床数据进行单因素和多因素Cox比例风险回归模型分析。
1.3 基因筛选与富集分析 应用GEO2R在线工具筛选HCC患者PBMC中的DEGs,标准为:|logFC|>2,调整后的P值<0.05;将获取的DEGs录入STRING,分析其对应的编码蛋白的相互作用关系。同时,进行基因本体分子功能(molecular function, MF)、生物学过程(biological process, BP)、细胞组分(cellular component, CC)和京都基因与基因组百科全书数据库(Kyoto Encyclopedia of Genes and Genomes, KEGG)通路富集分析,构建PPI网络图,其中数据证据等级设置为最高的0.9[8, 9]。将PPI网络数据文件下载并导入Cytoscape软件,应用其中的MCODE插件寻找关键基因模块。
1.4 验证基因水平与预后的关系 应用GEPIA对关键模块内的DEGs进行生存分析验证,LogrankP值<0.01的基因被列为关键基因,在TCGA数据库下载的HCC数据中提取对应关键基因,综合临床数据对HCC分期进行分层的单因素和多因素Cox比例风险回归模型分析,获取与预后相关的基因,比对肿瘤与PBMC中与预后相关的基因,再确定有预后判断价值的基因。
2 结果
2.1 数据分析结果 由GSE49515分析得到可用DEGs共225个,其中上调基因105个,下调基因120个。为了观察这些DEGs的编码蛋白质间相互作用关系,在STRING中按预定设置上传此225个基因,去除与其他蛋白质无关联的点后,构建完成PPI网络图(图1)。进一步查看DEGs的富集情况,可见:1)DEGs显著富集于细胞死亡的正向调控(positive regulation of cell death)、对有机物的反应(response to organic substance)和细胞死亡的调控(regulation of cell death)等基因本体BP;2)DEGs显著富集于结合(binding)、信号受体结合(signaling receptor binding)和有机环状化合物结合(organic cyclic compound binding)等基因本体MF;3)DEGs显著富集于细胞内(intracellular)、细胞质(cytoplasm)和细胞器(organelle)等基因本体CC;4)DEGs在KEGG通路中显著富集于类风湿性关节炎(rheumatoid arthritis)、甲型流感(influenza A)、疟疾(malaria)(表1)。这些结果充分体现了基因水平变化对细胞增殖的调控作用,在多种感染和免疫相关疾病的信号通路富集中也体现了炎症和免疫反应活跃的特点。

图1 根据DEGs编码蛋白间相互作用关系构建的PPI网络图

表1 差异水平基因功能和通路富集分析
2.2 关键基因的筛选 将PPI网络的数据文件导入Cytoscape中构建可视化网络图,应用软件中的MCODE插件筛选关键模块。该插件可根据各基因所编码蛋白间联系的证据强度来筛选联系最紧密的基因,进而构建出基因模块。将K-core值设置为6,可得到两个关键基因模块。模块1和模块2都包含有8个关键基因(图2)。分别将两个模块的关键基因录入STRING中进行富集分析。模块1主要富集于mRNA的剪接、剪接体、癌组织中的转录失调等功能和通路;模块2主要富集于G蛋白偶联受体信号通路、白细胞迁移的正向调控、趋化因子信号通路、细胞因子与细胞因子受体的相互作用和IL-17信号通路等功能和通路,体现了两个模块在HCC发生发展过程中对细胞转录、免疫和信号转导等方面起着重要作用。
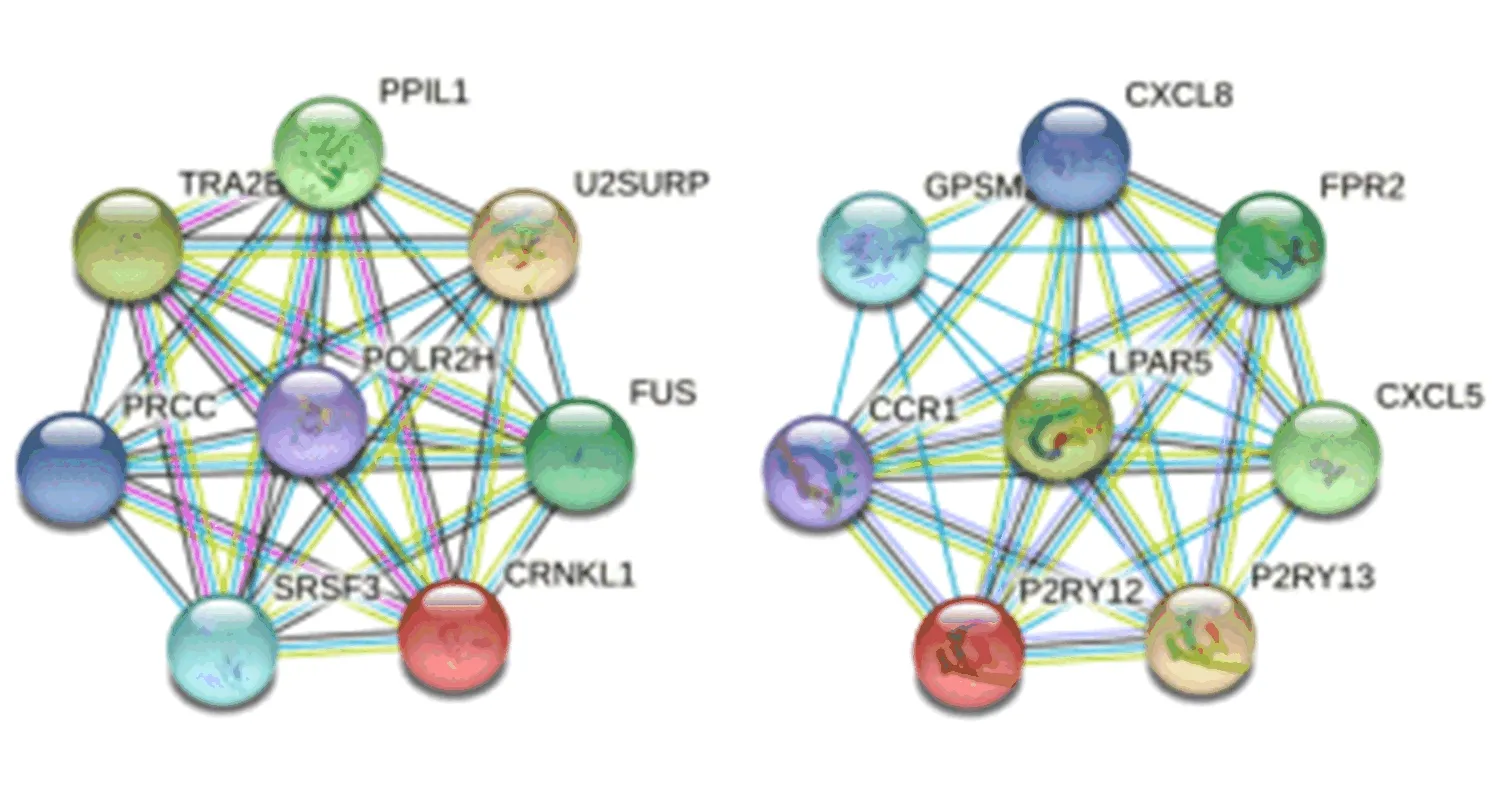
图2 筛选得到的关键基因模块1(左)和模块2(右)
2.3 判断基因水平与预后的关系 为了判断HCC肿瘤组织中来自关键模块的16个关键基因水平与患者总体生存期的关系,我们在GEPIA在线工具上将16个关键基因分别进行了总生存期验证,得出其中6个关键基因的高水平与总生存期缩短呈显著相关,这6个基因名称分别为GPSM2、PPIL1、POLR2H、CRNKL1、U2SURP和TRA2B。随后,我们从TCGA数据库下载到包括371例HCC患者基因水平和临床数据的数据集,在该数据集中,采用射频消融、无水酒精注射、微波聚束热疗和其他治疗者分别为22例、3例、1例和2例,而TNM分期I期、II期、III期和IV期分别为171例、86例、85例和5例。影响肝癌患者生存预后的大样本研究表明,住院时长和是否接受手术治疗是患者预后的独立保护因素,而TNM分期III期和IV期是患者预后的独立危险因素[10]。鉴于上述数据中治疗方法的有效记录缺失过多,因此将HCC患者按TNM分期进行分层,利用这6个基因水平数据并综合患者的临床数据进行影响生存预后的单因素和多因素分析(表3和表4),结果显示PPIL1是影响I期HCC患者生存预后的独立危险因素(HR=1.061,95%CI:1.005~1.121),GPSM2是影响III期HCC患者生存预后的独立危险因素(HR=1.556,95%CI:1.153~2.100),说明GPSM2和PPIL1水平与不同TNM分期的HCC患者生存预后独立相关。最后,通过比对PBMC与肿瘤组织中这6个关键基因的水平,发现其中GPSM2、TRA2B和U2SURP在PBMC和肿瘤组织中基因水平趋势相同。总之,肿瘤组织GPSM2高水平与生存预后缩短相关,且PBMC中GPSM2水平与肿瘤组织趋势相同,预示GPSM2可能是未来无创性判断HCC患者,尤其是III期HCC患者生存预后的关键指标。
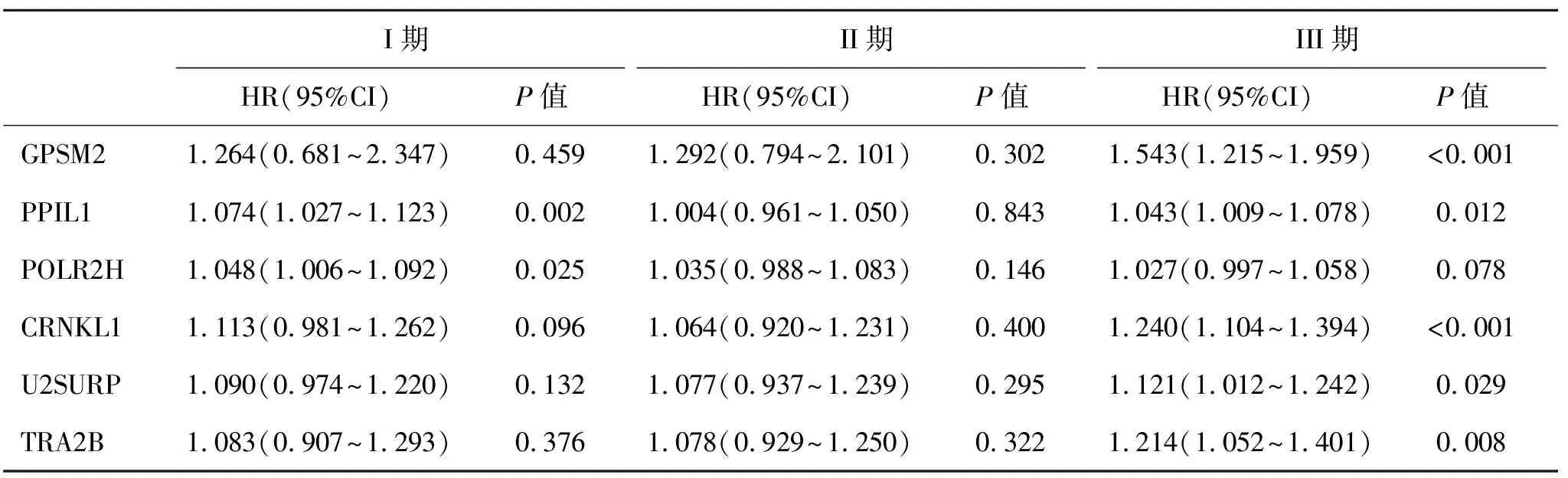
表3 影响HCC患者生存预后的单因素分析

表4 影响HCC患者生存预后的多因素分析
3 讨论
HCC的恶性程度极高,虽然早期治疗效果较好,但确诊时多已错过最佳的治疗时机,疗效和预后差,与其复杂的发病机制密切相关。常用的临床病理学、血清学和影像学等检查仍不能达到满意的诊断敏感度和特异度[11,12]。随着多种组学技术的发展和联合应用,在基因层面的探索可发现致癌基因的过度表达、抑癌基因的低水平表达和基因突变等,尤其对HCC组织DEGs的解析有助于揭示疾病本质并为诊疗新技术的探索指明方向。
本研究通过深入挖掘GEO和TCGA数据库,综合应用多种在线工具和软件,对比了HCC患者与健康人PBMC中的DEGs,探索可能与HCC发生发展及预后判断相关的关键基因。我们结合DEGs的基因本体和通路的富集分析揭示可能参与其中的细胞组分和信号通路,构建PPI网络,识别关键模块,结合GEPIA生存分析工具和TCGA数据的多因素分析验证确定了6个关键基因,即GPSM2、PPIL1、POLR2H、CRNKL1、U2SURP和TRA2B。它们在分子层面与mRNA的剪接、癌症基因转录失调和多个免疫反应信号通路密切相关,且GPSM2和PPIL1与肝癌患者预后独立相关。经过比对确定GPSM2、TRA2B和U2SURP这三个关键基因在PBMC中与肿瘤组织中有相似的上调趋势。因此,我们认为GPSM2、TRA2B和U2SURP可能是诊断HCC的潜在靶基因,尤其是GPSM2与III期HCC患者预后独立相关,更值得我们深入研究。
基因GPSM2编码蛋白为G蛋白信号调节因子2,是在细胞有丝分裂过程中纺锤体定向的决定因素。有体外细胞实验研究报道,基因GPSM2高水平可促进癌细胞的增殖、迁移和侵袭,同时抑制癌细胞凋亡,可能通过PI3K/AKT通路起致癌基因的作用[13]。近年来,也有研究证实GPSM2的过度表达同样是非小细胞肺癌和乳腺癌不良结局的独立预后因素[14, 15]。基因TRA2B编码TRA2β蛋白,属于富含丝氨酸/精氨酸的剪接因子家族成员,能够以浓度依赖的方式调节基因的选择性剪接[16]。TRA2B在多种癌症中过度表达,如女性的乳腺癌、宫颈癌、卵巢癌,男性的前列腺癌和肺癌等[17-20]。TRA2B过度表达是前列腺癌复发和不良预后的重要预测因子[18]。抑制TRA2B过度表达可降低子宫内膜癌细胞增殖和侵袭能力。TRA2B也被认为是骨肉瘤的致癌基因。研究证实来源于骨髓间充质干细胞的外泌体中存在miR-206,通过靶向TRA2B基因实现抑制骨肉瘤进展。
基因U2SURP又称SR140,编码小核糖核蛋白复合体U2亚基。该亚基富含丝氨酸/精氨酸,是构成剪接体的主要部件之一,研究证明其可与钙稳态内质网蛋白协同调节RNA监视因子的表达,进而在结直肠癌的发生过程中起致癌作用。也有研究确定基因U2SURP是与乳腺癌患者生存期密切相关的剪接体相关基因。
GPSM2、TRA2B和U2SURP在PBMC中的高水平有可能成为诊断HCC的新型精准生物标志,尤其GPSM2基因与III期HCC患者生存预后相关,有较大的探索价值。这些关键基因的具体分子机制有待进一步验证,需要更深入的研究明确其在细胞功能中的作用,阐明分子间的相互作用关系和信号传导通路,以期能更好地验证这些基因在HCC早期诊断和预后判断中的价值,甚至在综合治疗中成为新的治疗靶点。
