栽培大豆GRAS转录因子家族基因鉴定及其盐胁迫下表达模式分析
2021-06-30张斌陈丽娟李其华唐满生
张斌 陈丽娟 李其华 唐满生


摘要: 利用生物信息学手段及转录组测序方法对大豆78个GRAS家族基因进行系统分析。染色体定位结果表明78个GRAS基因不均匀地分布在20条染色体上。通过系统进化分析将大豆GRAS家族分为11个亚族。基因结构和保守基序分布分析结果表明GRAS家族成员在进化上具有保守性,尤其是进化关系较近的成员多具有类似的基因结构和蛋白质结构。转录组数据及qRT-PCR结果显示,5个基因受盐胁迫诱导上调,5个基因受盐胁迫诱导下调,其中GmGRAS14、GmGRAS33和GmGRAS69在盐处理12 h时上调倍数最高,而GmGRAS17、GmGRAS54和GmGRAS57在盐处理12 h时表达量下调倍数最高,说明这些基因可能在大豆响应盐胁迫方面发挥重要功能。
关键词: 大豆;GRAS家族;盐胁迫;表达模式
中图分类号: S565.101 文献标识码: A 文章编号: 1000-4440(2021)02-0296-14
Abstract: In this study, 78 genes of GRAS family in soybean were systematically investigated using bioinformatics and RNA-seq. The chromosomal distribution map showed that 78 GRAS genes were randomly located in 20 chromosomes. Phylogenetic analysis showed that the soybean GRAS family could be divided into 11 subfamilies. The gene structure and conserved motif distribution analysis suggested that the GRAS family members were consertive in evolution, and most of the GRAS members with close evolutionary relationship had similar gene and protein structures. Transcriptomal and qRT-PCR results showed that five genes were up-regulated and five genes were down-regulated under salt stress. Among them, GmGRAS14, GmGRAS33 and GmGRAS69 had the highest fold of up-regulation at 12 h of salt treatment. GmGRAS17, GmGRAS54 and GmGRAS57 showed the highest down-regulation multiple at 12 h of salt treatment, suggesting that these genes may play an important role in the response to salt stress of soybean.
Key words: soybean;GRAS family;salt stress;expression pattern
转录因子通过结合到启动子区发挥激活或抑制作用调控特定基因的表达,在植物生长发育以及生物或非生物胁迫响应过程中发挥着重要作用。目前很多转录因子家族基因的功能已被报道,如WRKY、MYB、MADS、bZIP、AP2/ERF、GRAS等[1],其中GRAS是植物特有的转录因子家族。GRAS家族基因的命名是根据最早鉴定出的该家族3个成员的基因名而来,包括GAI、RGA和SCR[2-4]。通常GRAS家族蛋白长度为400~700 aa,并且具有序列非常保守的C端和序列可变的N端[5]。根据系统进化树可将GRAS家族分成多个亚家族,但是不同物种中亚家族的数量也存在差异。如GRAS家族最早在拟南芥中被分成8个亚族:LISCL、PAT1、SCL3、DELLA、SCR、SHR、LAS和HAM[6];在蓖麻中被分成13个亚族:SCR、DLT、OS19、LAS、SCL4/7、OS4、OS43、DELLA、PAT1、SHR、HAM、SCL3和LISCL[7];在棉花和木薯中均可分为14个亚族[8-9]。目前,有多种植物GRAS转录因子家族基因的全基因组已被鉴定,如拟南芥与水稻[10]、烟草[11]、玉米[12]、白杨[13]、甘薯[14]、棉花[8]等。
GRAS转录因子在植物生长发育过程中发挥着多种功能,参与信号转导、调控组织发育、生物或非生物胁迫应答等过程[15]。拟南芥SCL3蛋白屬于SCL3亚族,其在根的内胚层可通过减弱DELLA抑制效应以维持赤霉素(GA)途径正常功能,并在拟南芥生长过程中参与调控根细胞的伸长[16]。水稻SCR亚族基因OsGRAS32基因在赤霉素代谢过程发挥重要调控作用[17]。过表达丹参SmGRAS3基因导致水杨酸和赤霉素的合成以及丹参毛状根的生长均受到抑制[18]。此外,大量研究结果表明GRAS转录因子参与植物对生物和非生物胁迫的响应过程。通过RNAi技术将水稻GRAS基因OsCIGR2沉默后接种稻瘟病菌,发现细胞死亡数目较野生型材料显著增加[19]。在拟南芥中过表达甘蓝型油菜LAS亚族基因BnLAS,则可通过降低过表达材料叶片失水率、减小气孔开度、合成较多的叶表皮蜡质增强转基因材料耐旱性[20]。胡杨SCL4/7亚族基因PeSCL7的表达受干旱和盐胁迫诱导上调,过表达PeSCL7基因可提高转基因拟南芥耐旱和耐盐能力[21]。在盐和甘露醇处理条件下,番茄GRAS家族基因SlGARS7的表达水平显著上调,SlGARS7基因过表达材料相较于野生型材料更加耐旱、耐盐[22]。通过对甘薯、棉花GRAS家族的全基因组学鉴定,也发现大量基因在盐、干旱、热、冷胁迫条件下被诱导表达上调[8,14]。
在很多植物中已对GRAS家族进行了全基因组学鉴定,并且对一些基因功能进行了深入研究。大豆中也有少数GRAS家族基因的功能被报道,例如GmSCL6和GmNSP2(即本研究中的GmGRAS1和GmGRAS12)的表达被miRNA(gma-miR171o和gma-miR171q)调控,进而影响大豆结瘤相关基因NIN、ENOD40和结瘤所需乙烯响应因子基因ERN的表达水平[23]。但是关于大豆GRAS家族的全基因组学分析及在非生物胁迫下的功能研究尚未见报道。本研究中,我们从大豆基因组数据库中鉴定GRAS家族成员,并对其理化性质、染色体定位、系统进化关系、基因结构、蛋白质氨基酸序列保守基序以及在不同组织中的表达模式和盐胁迫下的表达模式进行分析,为进一步研究GRAS家族基因在环境适应方面的功能提供理论基础。
1 材料与方法
1.1 大豆GRAS家族基因的获取和GRAS蛋白理化性质预测
在栽培大豆数据库(http://soykb.org/)检索GRAS转录因子家族成员,并下载检索到的所有基因编码的蛋白质氨基酸序列。将蛋白质氨基酸序列在数据库SMART(http://smart.embl-heidelberg.de/)和NCBI(https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)中检测是否含有GRAS结构域(PF03514),去除不含有此保守结构域的成员后即为最终鉴定出的大豆GRAS转录因子家族成员。根据候选基因在染色体上的位置依次命名。利用在线数据库ExPASy(https://web.expasy.org/protparam/)对GRAS家族成员蛋白质的氨基酸序列长度、相对分子质量、等电点、不稳定系数等理化性质进行预测。
1.2 染色体定位、基因结构和蛋白质保守基序分析
栽培大豆所有染色体的总长度信息下载自NCBI数据库(https://www.ncbi.nlm.nih.gov/genome/?term=Glycine+max),大豆GRAS家族成员基因在染色体上的位置信息从Phytozome数据库(https://phytozome.jgi.doe.gov/)下载整理,利用Map Chart 2.2软件绘制大豆GRAS家族成员基因在染色体上的定位图[24]。根据初步鉴定出的大豆GRAS家族成员的基因ID,在大豆数据库中逐一下载对应的基因碱基序列和CDS序列,利用在线工具GSDS(http://gsds.cbi.pku.edu.cn/index.php)绘制基因结构图[25]。利用在线数据库MEME(http://meme-suite.org/)分析GRAS家族成员蛋白质氨基酸序列上的保守基序[26],参数为:保守基序最小长度为6,最大长度为50,数量设置为10。
1.3 系统进化树的构建
在拟南芥TAIR数据库(http://www.arabidopsis.org/index.jsp)下载拟南芥GRAS家族成员的氨基酸序列(33个),在水稻数据库(http://rice.plantbiology.msu.edu/)下载水稻GRAS家族成员的氨基酸序列(50个)。将大豆、拟南芥和水稻GRAS家族成员的氨基酸序列在软件Clustal X中进行多重比对,在MAGA 7.0软件中利用邻接法(Neighbor-Joining,NJ)绘制系统进化树,参数设置:Bootstrap method 1 000,Poisson model,Pairwise deletion[8]。
1.4 GRAS家族基因在不同组织中的表达模式分析
栽培大豆GRAS家族基因在不同组织的表达数据从Phytozome数据库下载整理,即转录组测序数据中的FPKM值。包括基因在根、根毛、茎、顶端分生组织、叶、花、豆荚、种子、根瘤共9个部位的表达数据,所有的FPKM值经公式ln(X+1)计算后得到的数值用于表示基因的相对表达水平,并利用软件Hem1.0绘制基因表达热图[8]。
1.5 大豆幼苗的培养和盐处理后的转录组测序
试验所用的栽培大豆(Glycine max)品种为天隆1号。将充分吸水的大豆种子播种于湿润的营养土中(丹麦品氏泥炭土),置于生长室生长,生长室条件:恒温25 ℃,湿度为50%~60%,光照为中日照(12 h昼/12 h夜)。大豆生长约10 d后,第1张三出复叶完全长出,将大豆分为2组,一组浇灌普通自来水(对照组),另一组浇等量的200 mmol/L NaCl水溶液,6 h后分别取对照组和处理组大豆幼苗的地上部分(含茎和叶)于10 ml离心管中(均有3组生物重复),并迅速置于液氮中。样品交予上海欧易生物公司进行转录组学测序(RNA-seq)。从转录组数据中先初步筛选出GO注释为GRAS家族的基因,并进一步找出前期通过生物信息手段鉴定的大豆GRAS家族基因在正常生长和盐处理6 h后的基因表达FPKM值,绘制基因表达热图以分析盐胁迫条件下GRAS家族所有基因的表达模式。
1.6 RNA提取和荧光定量PCR检测
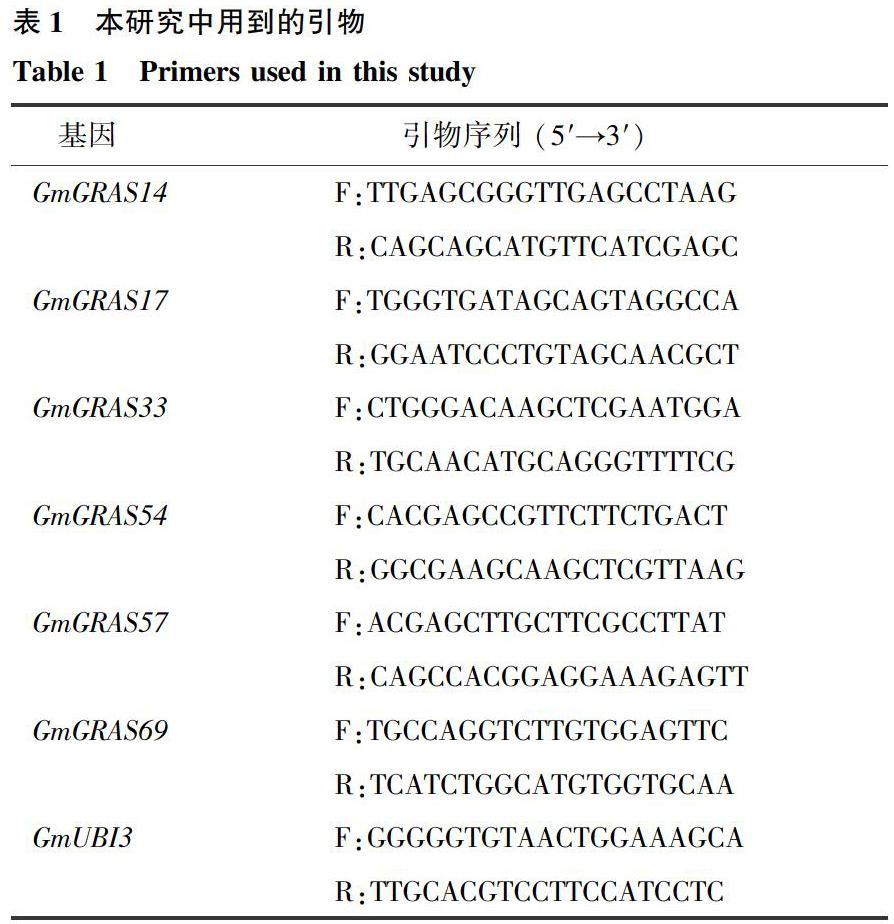
为了验证转录组数据中GRAS家族基因表达模式的准确性,重新培养一批大豆幼苗并进行NaCl处理(幼苗生长条件、生长时间、处理方式和方法同方法1.5)。使用TRIzol法提取大豆幼苗总RNA,使用反转录试剂盒SuperScript III First-Strand Synthesis SuperMix(Thermo)合成cDNA。以合成的cDNA为模板进行qRT-PCR分析,试验操作参照文献[27]。PCR反应总体系为20.0 μl:10.0 μl SYBR Green Mix(Thermo),8.0 μl RNAase-free水,正向和反向引物各0.5 μl,cDNA模板1.0 μl。利用ABI 7500定量PCR仪进行PCR扩增反应,反应程序:94 ℃,预变性32 min;94 ℃變性10 s,60 ℃退火10 s,72 ℃延伸10 s,共40个循环。利用2-△△Ct法计算基因相对表达水平。大豆UBI3基因作为内参,本研究所用引物如表1所示。
2 结果与分析
2.1 大豆GRAS家族成员的理化性质及GRAS家族基因染色体定位
本研究最终在栽培大豆中共鉴定出78个GRAS家族成员,并根据基因在染色体上的位置将这些基因依次命名为GmGRAS1~GmGRAS78。通过数据库ExPASy对大豆GRAS家族成员的理化性质进行分析。结果如表2所示,该家族成员的理化性质差异较大,蛋白质的长度在168 aa(GmGRAS35)至744 aa(GmGRAS1)之间,相对分子质量则介于1.897 584×104(GmGRAS35)至8.238 555×104(GmGRAS9)之间,等电点在4.82(GmGRAS63)至9.21(GmGRAS35)之间,不稳定系数在39.54(GmGRAS26)至61.75(GmGRAS1)之间,其中仅有GmGRAS26和GmGRAS47的不稳定系数小于40,说明这2个蛋白质被预测为稳定蛋白质。染色体定位分析结果表明,大豆GRAS家族78个基因不均匀地分布在20条染色体上,其中第11、12、13、15号染色体上分布较多,分别有12、6、9、7个;其次为第16号染色体,共有5个;第1、10、17号染色体上均包含4个GRAS基因,第2、3、5、9、18号染色体上均为3个GRAS基因,第4、6、7、14、20号染色体上则均有2个GRAS基因;而第8和第19号染色体上含有的GRAS基因数量最少,均为1个(图1)。
2.2 大豆GRAS家族基因的系统进化分析
为了探究GRAS家族基因在大豆和其他物种之间的进化关系,我们将拟南芥、水稻和大豆GRAS家族蛋白质氨基酸序列进行多重比对后,绘制了系统进化树。结果如图2所示,参照GRAS家族基因在拟南芥和水稻中的分组方法,并参考蓖麻中GRAS亚家族的命名方式(其分为PAT1、SHR、SCL3、LISCL、HAM、SCL4/7、SCR、DELLA、DLT、Os43、 Os4、Os19、Rc_GRAS共13个亚族),将大豆GRAS家族分为11个亚家族,分别是PAT1、SHR、SCL3、LISCL、HAM、SCL4/7、SCR、Os24、DELLA、DLT、Gm10,而进化树中LAS亚族不包含大豆GRAS家族成员。分析不同亚家族GRAS基因的数量,发现GRAS基因家族成员不均匀地分布在不同的亚家族。LISCL是最大的亚族,家族成员最多(共29个),包括了14个大豆GmGRAS基因、10个水稻OsGRAS基因和5个拟南芥AtGRAS基因。其次为PAT亚族,共包含27个GRAS基因,其中包括16个大豆GmGRAS基因、7个水稻OsGRAS基因和4个拟南芥AtGRAS基因。而DLT、SCL4/7、Gm10和SCL3则为较小的亚家族,分别包含了6、5、4、8个GRAS基因,其中Gm10亚族包含的4个成员均为大豆GRAS基因,SCL3亚族8个成员中仅有1个是大豆GRAS基因(GmGRAS18)。而在HAM、DELLA、SHR、Os24、SCR亚族中则分别包含10、10、9、5、4个大豆GRAS基因。
2.3 大豆GRAS家族基因结构及GRAS蛋白氨基酸序列上保守基序分布
利用在线软件GSDS构建了GRAS家族基因的外显子-内含子结构图(图3)。结果显示,大部分基因(66个)不含有内含子,仅12个基因有内含子,其中Gm10亚族的GmGRAS62的内含子数量最多(6个),GmGRAS18属于SCL3亚族,其包含4个内含子;GmGRAS24和GmGRAS35均有3个内含子,GmGRAS52含有2个内含子,而GmGRAS10、GmGRAS22、GmGRAS23、GmGRAS31、GmGRAS38、GmGRAS65和GmGRAS76均有1个内含子。大多数大豆GRAS基因(57个)在5′末端和3′末端都含有非翻译区。此外,在78个GRAS蛋白氨基酸序列上共鉴定出10个保守基序(Motif 1-Motif 10)(图4)。所有GRAS蛋白的保守基序主要集中在C端,并且同一亚族蛋白质含有的保守基序类型及位置均非常相似。如DELLA亚族蛋白质均含有除Motif 9以外的其他9个保守基序,SCR亚族蛋白质均含有除Motif 2和Motif 9以外的其他8个保守基序,这也进一步证明了相同亚族的GRAS家族成员进化关系较近。此外,除GmGRAS18仅含有1个保守基序Motif 4、GmGRAS35仅含有1个基序Motif 2、GmGRAS38仅含有2个基序Motif 3和Motif 7以外,其余大豆GRAS蛋白上的保守基序数量在6至10个之间。根据对不同亚族GRAS基因结构和GRAS蛋白保守基序的分析结果可知,同一亚族内的成员相对保守,推测同一亚族的基因功能可能相似。
2.4 大豆GRAS家族基因在不同组织中的表达
利用公共数据库Phytozome中大豆GRAS家族基因在不同组织表达的FPKM值绘制基因表达热图。结果如图5A所示,78个大豆GRAS基因的表达模式呈多样化,其中GmGRAS18、GmGRAS23、GmGRAS25、GmGRAS35、GmGRAS38和GmGRAS62共6个基因在所有组织均未检测到表达。有48个基因在所有组织均检测到不同程度的表达,其中GmGRAS8、GmGRAS15、GmGRAS16、GmGRAS20、GmGRAS22、GmGRAS39和GmGRAS73共7个基因的表达水平在所有组织中均较高,而GmGRAS51在所有组织中表达均较低。此外,有些大豆GRAS基因表现出组织特异性高表达,如GmGRAS16、GmGRAS29、GmGRAS46、GmGRAS55和GmGRAS56在根中的表达水平最高,GmGRAS39、GmGRAS40和GmGRAS57在茎中的表达水平最高,而GmGRAS1、GmGRAS26、GmGRAS73和GmGRAS78在种子中的表达量最高,可以推测这些组织特异性高表达的基因可能在特定的组织发挥特定功能。总而言之,大豆GRAS家族基因在不同组织中的表达模式存在较大的差异,说明该家族基因在不同组织可能发挥其独特的生物学功能。然而,同一亚族的某些基因具有类似的表达模式,如LISCL亚族的GmGRAS53和GmGRAS58,SHR亚族的GmGRAS54和GmGRAS57,DELLA亞族的GmGRAS15和GmGRAS20,且它们在进化树上的进化关系也非常近(图2),说明具有类似表达模式的基因可能也具有类似的功能。
2.5 GRAS家族基因响应盐胁迫的表达模式
通过对正常生长和盐处理(200 mmol/L NaCl,6 h)后的大豆植株进行转录组测序,获得了大豆GRAS家族78个基因在正常和盐胁迫下的表达数据FPKM值,并进一步绘制成表达热图(图5B)。结果表明,大多数基因的表达水平在正常生长和盐处理的大豆中表达水平无显著差异,有5个基因(GmGRAS6、GmGRAS14、GmGRAS15、GmGRAS33、GmGRAS69)的表达水平在盐胁迫下显著上调,5个基因(GmGRAS17、GmGRAS45、GmGRAS54、GmGRAS57、GmGRAS71)在盐胁迫下显著下调。此外,我们还挑选了受盐胁迫表达水平显著变化的一些基因(GmGRAS14、GmGRAS33、GmGRAS69和GmGRAS17、GmGRAS54、GmGRAS57)通过qRT-PCR进行进一步验证。结果见图6,盐处理后这些基因的表达水平变化与转录组测序结果一致,其中GmGRAS14和GmGRAS69在盐处理3 h、6 h和12 h时表达水平均显著上调,且在12 h时上调倍数最高,分别为3.67倍和5.82倍。GmGRAS33在盐处理6 h和12 h时表达水平显著上调,且在12 h时达到极显著水平,上调5.92倍。而GmGRAS17和GmGRAS57在盐处理3 h、6 h和12 h时表达水平均显著下调;GmGRAS54在盐处理3 h时与对照无显著差异,在盐处理6 h和12 h时其表达水平显著下调。以上结果说明表达水平受盐胁迫上调或下调的基因在大豆盐胁迫响应过程中可能发挥调控作用。
3 讨论
GRAS家族基因在植物生长发育和多种生理过程中发挥重要作用,参与赤霉素信号转导、赤霉素等激素的代谢、根的发育、响应生物和非生物胁迫的转录调控等过程。GRAS家族的全基因组学鉴定以及它们的进化关系也已在很多植物中被研究,如模式植物拟南芥[6]和烟草[11]以及常见的农作物水稻[10]、玉米[12]、甘薯[14]、棉花[8]等。并且已有部分GRAS家族基因在植物中的功能及分子机制被报道。但是关于大豆GRAS家族的全基因学鉴定及基因功能的研究却很少。因此,本研究从大豆基因组数据库鉴定出GRAS家族成员并进行全面的生物信息学分析。这将有助于我们研究大豆GRAS家族的进化并推测一些未知功能的大豆GRAS基因的生物学功能。
本研究在大豆基因组中共鉴定出78个大豆GRAS家族基因,比拟南芥(33个)[6]、水稻(50个)[10]、白菜(46个)[28]、甘薯(70个)[14]的GRAS家族成员数量多,但是比棉花GRAS家族成员少(150个)。造成这一现象的原因可能是不同物种间基因组大小的差异,如大豆基因组大小为952.48 Mb,而拟南芥、水稻、白菜和甘薯的基因组大小分别为125.0 Mb、374.0 Mb、283.3 Mb和492.38Mb,棉花的基因组大小则为2 126.03 Mb。此外,需要特别注意的是,Wu等[29]在大豆中鉴定出106个GRAS家族基因,而本研究鉴定出的GRAS基因数量与之有所差异,可能是筛选方式不同所致。本研究鉴定GRAS基因时主要利用公共数据库的数据,但同时参考我们自己测定的转录组结果中的GO注释,或许我们的转录组测序结果中注释的GRAS基因较实际要少一些,但本文研究的大豆GRAS基因均包含于已报道的106个基因中。Hirsch等[6]将拟南芥GRAS家族基因分为8个亚家族,包括LISCL、PAT1、SCL3、DELLA、SCR、SHR、LAS和HAM。而我们构建的拟南芥、水稻和大豆GRAS家族成员的进化树分为12个亚族,但是LAS分支中不含有大豆GRAS基因,因此将大豆GRAS基因家族分为11个亚家族,包括LISCL、PAT1、SCL3、DELLA、SCR、SHR、HAM、SCL4/7、Os24、DLT和Gm10,其中Gm10亞族中的4个成员均来自大豆。说明大多数GRAS家族成员在进化上非常保守,而少数成员则在进化过程中具有物种特异性。此外,我们发现蛋白质上保守基序数量及分布位置相同的GRAS成员,其理化性质也类似,推测其基因也具有类似的功能,如GmGRAS27和GmGRAS77。
通常认为在系统进化树上聚集在同一亚族的基因可能具有类似的功能[14,30-32],因此我们分析了拟南芥、水稻和大豆GRAS基因家族的系统进化关系,为大豆GRAS家族基因功能的预测提供可靠的依据。进一步分析了大豆GRAS家族基因在不同组织中的表达模式,发现大豆GRAS家族基因在不同组织的表达模式存在差异,与之前报道的其他物种(如甘薯、白菜)中的表达情况类似[14]。有7个大豆GRAS基因(GmGRAS8、GmGRAS15、GmGRAS16、GmGRAS20、GmGRAS22、GmGRAS39和GmGRAS73)的表达水平在所有组织中均较高,说明这些基因可能在大豆生长发育的多个环节都具有重要功能。尽管Wu等[29]的研究已包含大豆GRAS家族全基因组学鉴定方面的工作,但是其未关注GRAS家族基因在盐胁迫下表达模式分析。本研究通过对正常生长和NaCl处理的大豆进行转录组测序,分析了大豆GRAS家族基因在盐胁迫后的表达情况,结果表明,有5个基因(GmGRAS6、GmGRAS14、GmGRAS15、GmGRAS33、GmGRAS69)的表达水平在盐胁迫下显著上调,而GmGRAS17、GmGRAS45、GmGRAS54、GmGRAS57和GmGRAS71的表达水平显著下调。qRT-PCR分析结果显示GmGRAS14、GmGRAS33和GmGRAS69在盐处理6 h时表达水平均显著上调,与转录组测序结果一致,且3个基因均在盐处理12 h时上调倍数最高,分别为3.67倍、5.92倍和5.82倍;而GmGRAS17、GmGRAS54和GmGRAS57的表达水平在盐处理6 h、12 h时均显著下调,与转录组结果一致。通过系统进化分析、盐胁迫下的表达模式分析以及qRT-PCR检测,可初步推测大豆GRAS家族基因在生长发育过程和盐胁迫响应方面具有重要功能。本研究也为大豆GRAS基因功能的进一步研究及利用该家族基因培育大豆耐盐新品种提供了理论依据。
參考文献:
[1] HOANG X L T, NHI D N H, THU N B A, et al. Transcription factors and their roles in signal transduction in plants under abiotic stresses[J]. Current Genomics, 2017, 18(6): 483-497.
[2] DI LL, WYSOCKADILLER J, MALAMY JE, et al. The SCARECROW gene regulates an asymmetric cell division that is essential for generating the radial organization of the Arabidopsis root[J]. Cell, 1996, 86(3): 423-433.
[3] PENG J, CAROL P, RICHARDS D E, et al. The Arabidopsis GAI gene defines a signaling pathway that negatively regulates gibberellin responses[J]. Genes & Development, 1997, 11(23):3194.
[4] SILVERSTONE A L, CIAMPAGLIO C N, SUN T. The Arabidopsis RGA gene encodes a transcriptional regulator repressing the gibberellin signal transduction pathway[J]. Plant Cell, 1998, 10(2): 155-169.
[5] SUN X, XUE B, JONES W T, et al. A functionally required unfoldome from the plant kingdom: intrinsically disordered Nterminal domains of GRAS proteins are involved in molecular recognition during plant development[J]. Plant Molecular Biology, 2011, 77(3): 205-223.
[6] HISCH S, OLDROYD G E D. GRAS-domain transcription factors that regulate plant development[J]. Plant Signaling & Behavior, 2009, 4(8): 698-700.
[7] XY W, CHEN Z, AHMED N, et al. Genome-wide identification, evolutionary analysis, and stress responses of the GRAS gene family in Castor beans[J]. International Journal of Molecular Sciences, 2016, 17(7):1004.
[8] ZHANG B, LIU J, YANG Z E, et al. Genome-wide analysis of GRAS transcription factor gene family in Gossypium hirsutum L[J]. BMC Genomics, 2018, 19(1): 348.
[9] SHAN Z Y, LUO X L, WU M Y. Genome-wide identification and expression of GRAS gene family members in cassava[J]. BMC Plant Biology, 2020, 20(1): 46.
[10]TIAN C, WAN P, SUN S, et al. Genome-wide analysis of the GRAS gene family in rice and Arabidopsis[J]. Plant Molecular Biology, 2004, 54(4): 519-532.
[11]CHEN Y Q, TAI S S, WANG D W, et al. Homology-based analysis of the GRAS gene family in tobacco[J]. Genetics and Molecular Research, 2015, 14(4): 15188-15200.
[12]GUO Y, WU H, LI X, et al. Identification and expression of GRAS family genes in maize (Zea mays L.)[J]. PLoS One, 2017, 12(9): e0185418.
[13]LIU X, WIDMER A. Genome-wide comparative analysis of the GRAS gene family in populus, Arabidopsis and rice[J]. Plant Molecular Biology Reporter, 2014, 32(6): 1129-1145.
[14]CHEN Y, ZHU P P, WU S Y. Identification and expression analysis of GRAS transcription factors in the wild relative of sweet potato Ipomoea trifida[J]. BMC Genomics, 2019, 20(1): 911.
[15]BOLLE C. The role of GRAS proteins in plant signal transduction and development[J]. Planta, 2004, 218(5): 683-692.
[16]HEO J, CHANG K, KIM I, et al. Funneling of gibberellin signaling by the GRAS transcription regulator SCARECROW-LIKE 3 in the Arabidopsis root[J]. PNAS, 2011, 108(5): 2166-2171.
[17]LI W, WU J, WENG S, et al. Identification and characterization of dwarf 62, a loss-of-function mutation in DLT/OsGRAS-32 affecting gibberellin metabolism in rice[J]. Planta, 2010, 232(6): 1383-1396.
[18]BAI Z, XIA P, WANG R, et al. Molecular cloning and characterization of five SmGRAS genes associated with tanshinone biosynthesis in Salvia miltiorrhiza hairy roots[J]. PLoS One, 2017, 12(9): e0185322.
[19]TANABE S, ONODERA H, HARA N, et al. The elicitor-responsive gene for a GRAS family protein, CIGR2, suppresses cell death in rice inoculated with rice blast fungus via activation of a heat shock transcription factor, OsHsf23[J]. Bioscience, Biotechnology and Biochemistry, 2016, 80(2): 1145-1151.
[20]YANG M, YANG Q, FU T, et al. Overexpression of the Brassica napus BnLAS gene in Arabidopsis affects plant development and increases drought tolerance[J]. Plant Cell Reports, 2011, 30(3): 373-388.
[21]MA HS, LIANG D, SHUAI P, et al. The salt- and drought-inducible poplar GRAS protein SCL7 confers salt and drought tolerance in Arabidopsis thaliana[J]. Journal of Experimental Botany, 2010, 61(14): 4011-4019.
[22]HABIB S, WASEEM M, LI N, et al. Overexpression of SlGRAS7 affects multiple behaviors leading to confer abiotic stresses tolerance and impacts gibberellin and auxin signaling in tomato[J]. International Journal of Genomics, 2019, 2019: 4051981.
[23]HOSSAIN M S, HOANG N T, YAN Z. Characterization of the spatial and temporal expression of two soybean miRNAs identifies SCL6 as a novel regulator of soybean nodulation[J]. Frontiers in Plant Science, 2019, 10: 475.
[24]VOORRIPS R E. MapChart: Software for the graphical presentation of linkage maps and QTLs[J]. Journal of Heredity, 2002, 93(1): 77-78.
[25]HU B, JIN J, GUO A, et al. GSDS 2.0: an upgraded gene feature visualization server[J]. Bioinformatics, 2015, 31(8): 1296.
[26]BAILEY T L, MIKAEL B, BUSKE F A , et al. MEME Suite: tools for motif discovery and searching[J]. Nucleic Acids Research, 2009, 37(S): 202-208.
[27]PI B Y, HE X H, RUAN Y, et al. Genome-wide analysis and stressresponsive expression of CCCH zinc finger family genes in Brassica rapa[J]. BMC Plant Biology, 2018, 18(1): 373.
[28]SONG X, LIU T, DUAN W, et al. Genome-wide analysis of the GRAS gene family in Chinese cabbage (Brassica rapa ssp. pekinensis)[J]. Genomics, 2014, 103(1):135-146.
[29]WU N N, ZHU Y, SONG W L, et al. Unusual tandem expansion and positive selection in subgroups of the plant GRAS transcription factor superfamily[J]. BMC Plant Biology, 2014, 14:373.
[30]李 倩,蓋江涛,白蓓蓓,等.茄科植物中HCT基因家族的鉴定及进化和表达分析[J].江苏农业科学,2019,47(19):65-68.
[31]付瑜华,蒙秋伊,李秀诗,等. 薏苡油脂合成关键基因克隆及其生物信息学分析[J].南方农业学报,2020,51(3):485-495.
[32]陈亨德,褚武英,李玉珑,等. 罗非鱼SIRT1基因的克隆及其表达规律分析[J]. 江苏农业科学,2019,47(21):103-106.
(责任编辑:张震林)
