基于图嵌入与拓扑结构信息的蛋白质复合物识别算法*
2021-06-25徐周波刘华东
徐周波,李 萍,刘华东,李 珍
(桂林电子科技大学广西可信软件重点实验室,广西 桂林 541004)
1 引言
蛋白质复合物作为大分子组装体,在细胞稳态、生长和增殖所必需的多种生化活动中发挥着重要作用[1]。蛋白质复合物是生化体制和细胞结构的研究基础,因此,蛋白质复合物的识别成为近年来的研究热点。
目前蛋白质复合物识别技术主要分为2类:(1)基于实验的蛋白质复合物识别技术;(2) 基于计算方法的蛋白质复合物识别技术。基于实验的蛋白质复合物识别技术主要根据实验结果识别蛋白质复合物,如免疫共沉淀[2,3]和双杂交系统[4,5],但其通常耗时较长且需高水平的专业知识作为基础。为克服基于实验的蛋白质复合物识别技术的缺点,研究者们提出了多种基于计算方法的蛋白质复合物识别技术。基于计算方法的蛋白质复合物识别技术的基本思想是从蛋白质相互作用PPI(Protein-Protein Interaction) 网络中识别呈现蛋白质复合物某些典型特性的簇。因此,PPI网络通常建模为图的形式,其中图的节点表示蛋白质,边表示蛋白质间的相互作用。蛋白质复合物的识别问题可以归结为一个传统的图聚类问题,由此产生的子图聚类被视为感兴趣的蛋白质复合物。Nepusz等[6]提出了聚类算法ClusterONE用于从PPI网络中发现重叠蛋白质复合物。ClusterONE可有效识别重叠蛋白质复合物,并采用最大匹配率MMR(Maximum Matching Rate)来评估算法的复杂度,该算法精准度和敏感度较低。Liu等[7]提出了一种基于最大团的聚类算法CMC (Clustering-based on Maximal Cliques),利用最大团簇从加权PPI网络中发现复合群。CMC使用迭代评分的方法为蛋白质对分配权重,可以改善其他蛋白质复合物预测方法的性能,减少随机噪声的影响。CMC算法提高了识别蛋白质复合物的精准度,但对小规模复合物检测能力较差,且敏感度较低。Wang等[8]提出了一种快速分层聚类算法HC-PIN。HC-PIN对假阳性具有鲁棒性,并且可以发现低密度的功能模块。HC-PIN算法虽然也提高了识别蛋白质复合物的精准度,但其同样存在敏感度低的问题。Wu等[9]提出的COACH算法考虑到蛋白质复合物的拓扑结构,先检测出核心蛋白质,然后将附属蛋白质连接到核心蛋白质上。该算法考虑到了蛋白质结构上的特点,一定程度上提高了预测的准确性。Zhao等[10]用不确定图模型建模PPI网络,提出了一种基于不确定图模型的蛋白质复合物算法DCU (Detecting Complex based on Uncertain graph model),改善了COACH算法,进一步提高了预测的准确性。
由于非监督学习算法的随机特性会在一定程度上影响算法识别结果,因此近年来监督学习算法也逐渐被用于蛋白质复合物的识别。这类算法通过提取样本特征克服非监督的随机性,并将特征放入分类器中训练,最终得出具有一定准确性的分类器。其分类效果的好坏主要依赖于提取的特征能否较好地反映出蛋白质复合物的真实特性。然而,监督学习算法的特征通常都是人为构造的,其准确性和完整性有待考量。
针对传统算法存在敏感度和F-measure低以及现有监督学习算法中特征构造不完备等不足,近年来许多利用图嵌入进行蛋白质复合物识别的方法应运而生。图嵌入的方法将图转换为向量的形式进行处理,并且同时保留了图的局部和全局信息,使得蛋白质复合物的识别更加容易和准确。Xu等[11]提出了一种基于从GO知识库中学习蛋白质复合物向量的复合物识别算法GANE。该算法利用AANE[12]模型来学习复合物的向量表示,基于此向量构造加权邻接矩阵并利用团挖掘的算法来进行复合物的识别。Yao等[13]首先将蛋白质以功能不同的标准分组,利用node2vec[14]方法将蛋白质转换为向量表示,构造相似度矩阵,并利用聚类算法来进行蛋白质复合物的识别。本文提出的graph2vec-SVM与复合物拓扑结构信息相结合的搜索方法,利用graph2vec[15]将图转换为向量并结合SVM分类器来进行蛋白质复合物的识别,不仅克服了非监督学习算法的随机性,还解决了监督学习构造特征不完备等问题,有效弥补了传统算法和监督学习算法的不足。同时,相较于文献[14]利用node2vec将复合物中每个节点转换为向量表示,graph2vec将整个图转换为向量表示的做法更加便于计算。通过实验分析,该算法有较好的敏感度,在准确度和F-measure方面也显示出良好的性能。
2 相关知识介绍
2.1 graph2vec
PPI网络通常建模为图数据模型,图的节点表示蛋白质,边表示蛋白质间的相互作用。图数据模型是一个4元组G=(V,E,W,Lv),其中,V是节点集;E是边集;W:E→[0,1]是权重分配函数,它给每条边赋予一个权重;Lv是节点标签分配函数,它从标签集中选择标签分配给节点。本文以节点度作为图的标签,将PPI网络建模为图数据模型后利用graph2vec将图转换为向量。
graph2vec是由Narayanan等[15]提出的一种图嵌入(将图转换为向量)算法,该算法基于word2vec[16]和doc2vec[17]的思想,将整个图作为文档,图的根子图作为文档中的词,通过训练浅层神经网络后最终得到整个图的向量。其中,根子图为图的子树模式,且子树中允许出现相同的节点。例如,图G(图1a)的最大步长为2的根子图如图1b所示。
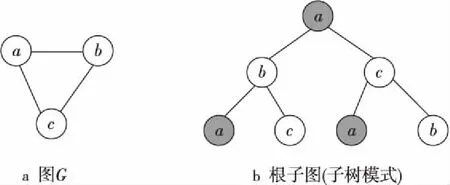
Figure 1 2-rooted subgraph
graph2vec采用skipgram模型来学习图的向量表示,如图2所示。给定一个文档集G={G1,G2,…,Gn}以及从文档Gq∈G(1≤q≤n)中采样的词SG(Gq)={sg1,sg2,…,sgm},skipgram模型通过最大化式(1) 的似然函数得到文档的向量表示。
(1)
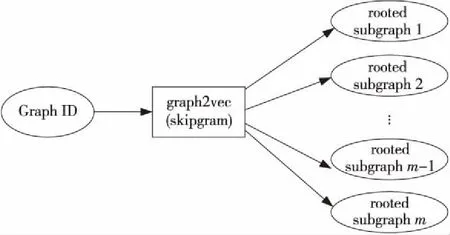
Figure 2 skipgram model
目前现有识别算法通常先将蛋白质复合物建模为图数据结构,再对其进行特征提取,如图的密度、节点个数和节点度统计等,并结合机器学习分类器进行蛋白质复合物的识别。由于这些特征是人为构造的,其构造特征的准确性有待考量。graph2vec利用图本身的特性(每个节点的根子图),通过skipgram模型训练后得到的图的向量表示能够较准确地保留原图的信息,且能够方便地利用机器学习分类器进行后续的蛋白质复合物识别。
2.2 支持向量机
支持向量机SVM(Support Vector Machine)是由Vapnik[18]提出的一种监督学习二分类器。它的基本思想是拟合出一个最大化间隔的划分超平面,使其能够具有准确的分类性能。
SVM的划分超平面可用如式(2)所示的线性方程描述:
wTx+b=0
(2)
其中,x为分类样本矩阵;w为法向量,决定了超平面的方向;b为位移量,决定了超平面与原点的距离。分类样本标签yi为-1或+1,当分类样本xi能够被正确分类时,其满足式(3):
(3)
其中,i表示分类样本xi在分类样本矩阵x中的索引,其取值为[0,M],M为分类样本的总数。式(3)等价于:
yi(wTxi+b)≥1
(4)
满足yi(wTxi+b)=1的样本称为支持向量。2个异类支持向量到划分超平面的距离之和称为间隔,即:
(5)
最大化间隔γ即最小化w,求解
s.t.yi(wTxi+b)≥1,i=1,2,…,M
(6)
求解出参数w和b后可得到最大化间隔超平面。
式(6)可用拉格朗日乘子法转换为对偶问题 ( Dual Problem )的求解,其最后的求解式变为:
(7)

3 基于图嵌入与拓扑结构信息的蛋白质复合物识别算法
3.1 graph2vec-SVM算法
graph2vec-SVM算法将标准库中的蛋白质复合物以及随机生成图(非蛋白质复合物)用graph2vec技术提取出每个节点的根子图后,以式 (1) 作为目标函数,利用skipgram模型将图转换为向量,转换后的向量即为SVM分类器的训练样本集,然后开始训练SVM分类器。其主要过程如算法1所示。
算法1graph2vec-SVM
输入:G={G1,G2,…,Gn},k,N,D,ep,l。
输出:model。
1.T={};
2.foreachGq∈G
3. randomly generateNsubgraphs fromGqwith the same size asGq,regard them as negative samples and insert them intoT;
4.T=T∪G;
5.vectors=graph2vec(T,k,D,ep,l)
6.model=SVM(vectors,labels);
returnmodel
算法1中,G为蛋白质复合物的集合,k为根子图的最大步长,N为对每个蛋白质复合物生成随机子图的个数,D为向量的维度,ep为graph2vec算法的迭代次数,l为学习率。算法1第1~4行根据每个蛋白质复合物随机生成N个子图作为负样本,并将正负样本加入训练集合T中。第5行将训练集T用graph2vec转换为向量,第6行将向量和标签放入SVM分类器中,开始训练分类器。其中labels为样本的标签,正样本的标签为+1,负样本的标签为-1。
以图1a为例,设最大步长k为1,则由算法1对图G提取每个节点的根子图后,训练skipgram模型,最终得到图G的向量表示V(G),如图3所示。

Figure 3 Steps of graph2vec
3.2 构造候选蛋白质复合物
蛋白质复合物被认为是PPI网络中的稠密子图,如何从PPI网络中划分出稠密子图是蛋白质复合物识别的关键。本文利用模块度Q来衡量一个子图c的稠密程度。子图c的模块度Q的定义如式(8)所示:
(8)
其中,Vc为子图c中的节点集,Ec为子图c中的边集。weightin(c)=∑v,u∈VcP(u,v)u,v为子图c中的节点,P(u,v)为边e=(u,v)的权重,e∈Ec。weightout(c)=∑v∈Vc,u∉VcP(u,v),u为子图c中的节点,v不为子图c中的节点,P(u,v)为边e=(u,v)的权重,e∉Ec。δ为模块校正参数,可用于代表所预测复合物中暂未发现的蛋白质,同时也可用于消除噪声。Vapnik[18]通过实验分析,δ取值为PPI网络平均度的一半时效果最佳。由式(8) 计算得到子图c的模块度,若簇边界内的边权值总和大于其边界外的边权值总和,即:
则称子图c为稠密子图。
由于蛋白质复合物是稠密子图,在PPI网络中从度较大的节点开始搜索候选蛋白质复合物,将会更快搜索到稠密子图,因此本文首先考虑选取节点度大于平均度的节点作为种子节点。由种子节点开始,向外扩散搜索构造子图,并计算该子图的模块度,直至其模块度达到最大,将其加入候选集中。获取蛋白质复合物候选集合candidate_set的具体过程如算法2所示。获取候选集合后,将候选集合中的蛋白质复合物转换为向量即可用graph2vec-SVM模型进行识别分类。
算法2getcandidate_set
输入:PPI networkG=(V,E,W,Lv)。
输出:candidate_set。
1.fornodev∈V/*获取种子节点集合,种子节点为度大于平均度的节点*/
2.ifdegree ofvmore than average degree ofG,insertvinto the set seed/*由种子节点开始构造候选蛋白质复合物*/
3.fors∈seed
4.c={s};Q(c)=0;
5.Nv(s)//computing the neighbors ofs
6.foreachnoden∈Nv(s)
7.c′=c∪{n};
8. computeQ(c′);//计算子图模块度
9.ifQ(c′) ≥Q(c)
10.c=c′;
11. insertcintocandidate_set;
12.returncandidate_set
算法2在执行过程中可能会因为复合物高度重合而造成冗余。本文将候选蛋白质复合物间重合度大于0.7[19]的复合物认为是重合的,重合度计算方法如式(9)所示(即重合度为复合物A和复合物B共有节点个数与复合物A节点个数和复合物B节点个数乘积的比值),并剔除模块度小的复合物。
OS(A,B)=|A∩B|2/(|A|×|B|)
(9)
去重算法过程如算法3所示。
算法3get finalcandidate_set
输入:candidate_set。
1.forc∈candidate_set
2.ifSize(c) < 2//丢弃规模小于2的子图
3. removecfromcandidate_set;
4.forA∈candidate_set
5.forB∈candidate_set
//计算蛋白质复合物间的重合度
6.ifOS(A,B) > 0.7
//保留模块度大的蛋白质复合物
7.ifQ(A) ≥Q(B)
8. removeBfromcandidate_set
9.elseremoveAfromcandidate_set
3.3 蛋白质复合物的识别
3.1节利用标准库中的复合物和随机生成子图训练graph2vec-SVM算法并得到具有识别复合物功能的分类器;3.2节利用式(8) 定义的模块度搜索稠密子图,去重后得到待识别的候选蛋白质复合物集合;本节利用3.1节中训练好的graph2vec-SVM算法识别3.2节中去重后得到的候选蛋白质复合物。与算法1相似,在进行蛋白质复合物的识别前,需先利用graph2vec将其转换为向量,具体过程如算法4所示。
算法4Predict protein complex
输入:candidate_set,k,D,ep,l。
输出:predictions。
//用graph2vec将每个候选蛋白质复合物转换为向量
1.c∈candidate_set
2.vectors=graph2vec(candidate_set,k,D,ep,l);
3.model=graph2vec-SVM(G,k,D,ep,l);
//对候选蛋白质复合物进行识别
4.Predictions=model.predict(vectors);
5.returnpredictions
4 实验结果及分析
本文将graph2vec-SVM蛋白质复合物识别算法与目前较为经典的4种算法,包括ClusterOne、CMC、HC-PIN和COACH在酵母菌相互作用网络DIP(Database of Interacting Proteins)[20]上进行比较。蛋白质复合物标准库采用CYC2008[21]和 MIPS[22]标准库。2个标准库分别由408个复合物和428个复合物组成。
4.1 评价指标
本文将所识别的蛋白质复合物与标准库中的蛋白质复合物进行比较以保证蛋白质复合物识别的质量。识别质量的评价指标主要有精准度(Precision)、敏感度(Sensitivity)和F-measure。 精准度为识别的复合物中真实复合物的数量与识别的复合物总数量的比值;敏感度为识别的复合物中真实复合物的数量与总真实复合物数量的比值;F-measure是精准度和敏感度的调和平均值,其计算方法如式(10)所示:
(10)
Precision=TP/(TP+FP)
(11)
Sensitivity=TP/(FN+TP)
(12)
其中,TP为所识别复合物中与标准库中复合物相匹配的复合物数量,其匹配程度通过式(9)计算,OS>R的识别复合物被认为是真正的蛋白质复合物,R为匹配程度的阈值,其值通常设置为0.2[23]。TN为识别结果中真实非蛋白质复合物的数量,FN为真实蛋白质复合物被识别为假蛋白质复合物的数量。
4.2 graph2vec参数设置
本文使用标准库中节点数大于2的蛋白质复合物作为正样本,负样本为随机生成的子图。利用graph2vec将正样本和负样本转换为向量,参数的设置如表1所示,转换后的向量即为分类器的训练集。

Table 1 Setting of graph2vec parameter
表1中,batch_size为一次训练所选取的样本数;epochs为训练样本被整体训练的次数;max_k为根子图的最大步长;embedding_size为图转换为向量的维数,若embedding_size太小会导致图的信息丢失,从而造成识别算法不能很好地识别出蛋白质复合物,若其太大又会包含冗余的信息,从而影响蛋白质复合物的识别。实验过程中发现,当embedding_size=512时其能够较好地表示图的信息。num_negsamples为噪声样本的数量,learning_rate为学习率,最终得到的向量为V(G)=(v1,v2,…,vδ)。
4.3 对比模型的选取
本文在DIP数据集上采用3种机器学习分类器(LR、SVM和XGBoost)进行蛋白质复合物的识别,蛋白质复合物标准库为MIPS,其结果分别如图4和表2所示。
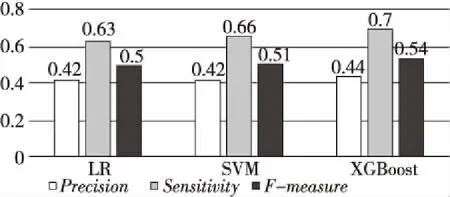
Figure 4 Performance of three classifiers on DIP dataset

Table 2 Identify results of three classifiers on MIPS standard library
由图4可知,LR、SVM和XGBoost在3项指标中都有较好的结果,但从表2可看出,LR和XGBoost正确识别蛋白质复合物数量较高,但正确识别非蛋白质复合物的数量极低,而SVM的综合表现相对较好,所以本文最终选取SVM分类器进行蛋白质复合物的识别。
4.4 与非监督学习算法的对比
graph2vec-SVM与4种非监督学习算法(CMC、COACH、HC-PIN和ClusterOne)在DIP数据集上精准度、敏感度和F-measure的表现如图5所示,其中蛋白质复合物的标准库采用的是CYC2008。从图5可以看出,graph2vec-SVM在3项指标中都取得了良好的效果,在该数据集上的精准度(0.42)有待提高,敏感度(0.66)和F-measure(0.51)均好于其他算法的。

Figure 5 Performance of each algorithm on DIP dataset
为进一步分析实验结果,将CYC2008标准库替换为MIPS标准库后,结果如表3所示。从表3可以看出,graph2vec-SVM识别算法在所有对比算法中识别出正确蛋白质复合物的数量最多,且其F-measure也最高,正确识别非蛋白质复合物的数量比COACH算法次之,但综合来说graph2vec-SVM识别算法相较于对比算法表现较好。
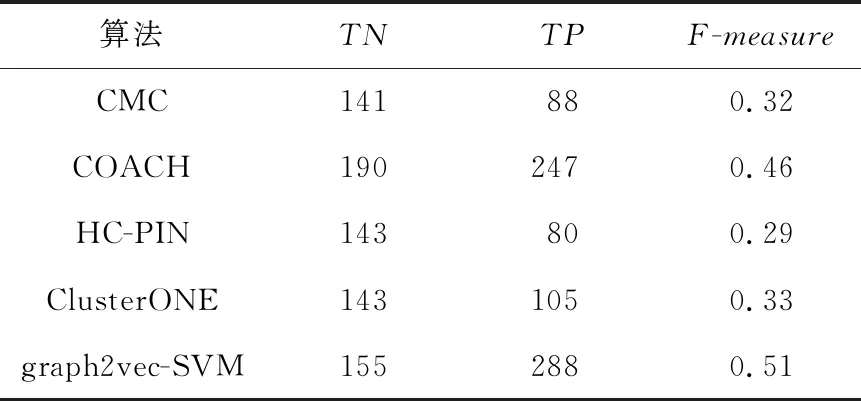
Table 3 Comparison of algorithms on MIPS standard library
4.5 与监督学习算法的对比
本节将graph2vec-SVM识别算法与3种监督学习算法(SCI-BN,SCI-SVM和RM)在DIP数据集上进行对比。4种算法均采用MIPS标准库中的蛋白质复合物作为正样本进行模型训练。3种监督学习算法参数均参照文献[23-25]设置。实验对比结果如表4所示,从表4中可以看出,graph2vec-SVM在DIP数据集上Precision、Sensitivity和F-measure的表现相对其他3种算法都较好。
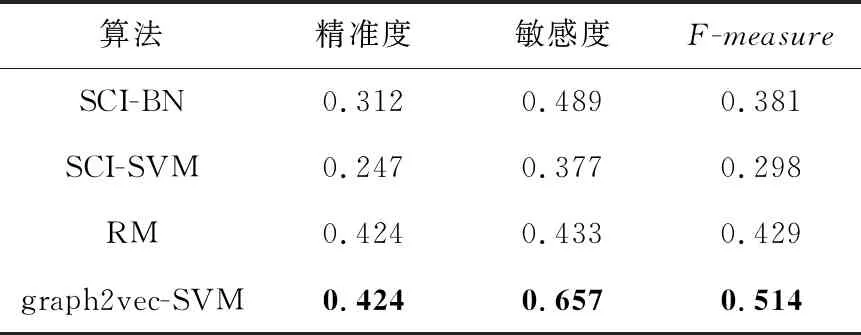
Table 4 Comparison with supervised algorithms on MIPS standard library
5 结束语
本文针对非监督学习识别算法的随机特性会影响复合物的识别准确性,以及监督学习识别算法的人为构造特征不完备等缺陷,提出了graph2vec-SVM蛋白质复合物识别算法。该算法利用grah2vec将图的信息转换为向量,并进一步采用SVM分类器进行蛋白质复合物的识别,实验结果表明,该算法与目前流行的监督学习算法与传统非监督学习算法在敏感度和F-measure上都取得了较好的效果,但由于在生成随机子图时存在离散点而导致精准度不高,未来在完善识别算法时我们将着手克服离散点来尝试提高精准度。
