禾谷炭疽菌内吞相关蛋白找寻及其生物信息学
2021-06-24韩长志
覃 悦,韩长志
(1. 西南林业大学生物多样性保护学院,昆明 650224; 2. 西南林业大学研究生院,昆明 650224; 3. 云南省森林灾害预警与控制重点实验室,昆明 650224)
受体介导的内吞作用(receptor-mediated endocytosis)是生物体中大多数细胞实现物质内吞的重要途径之一[1-2],该作用是由直径约100 nm小泡产生介导的,小泡具有由胞浆蛋白网格蛋白组成的形态特征性被膜[3]。在模式生物真菌研究中,内吞与其细胞的生长、发育以及分化等各个过程密切相关。前人研究表明,模式真菌(酿酒酵母、构巢曲霉等)中含有NPFxD基序的蛋白参与内吞靶向信号过程,该信号在实现含有特征化弗林蛋白酶样蛋白酶Kex2p胞质结构域的嵌合蛋白过程发挥重要作用[4]。同时,真菌的包被网格蛋白囊泡(CCV)广泛存在于细胞中,并形成质膜的区域,该区域可浓缩具有不同受体的大细胞外分子,不同受体则负责配体的低密度脂蛋白、生长因子、转铁蛋白和抗体等受体介导的内吞作用[5],并通过CCV摄取的蛋白质通常具有内吞靶向信号,可直接掺入新囊泡中[6]。
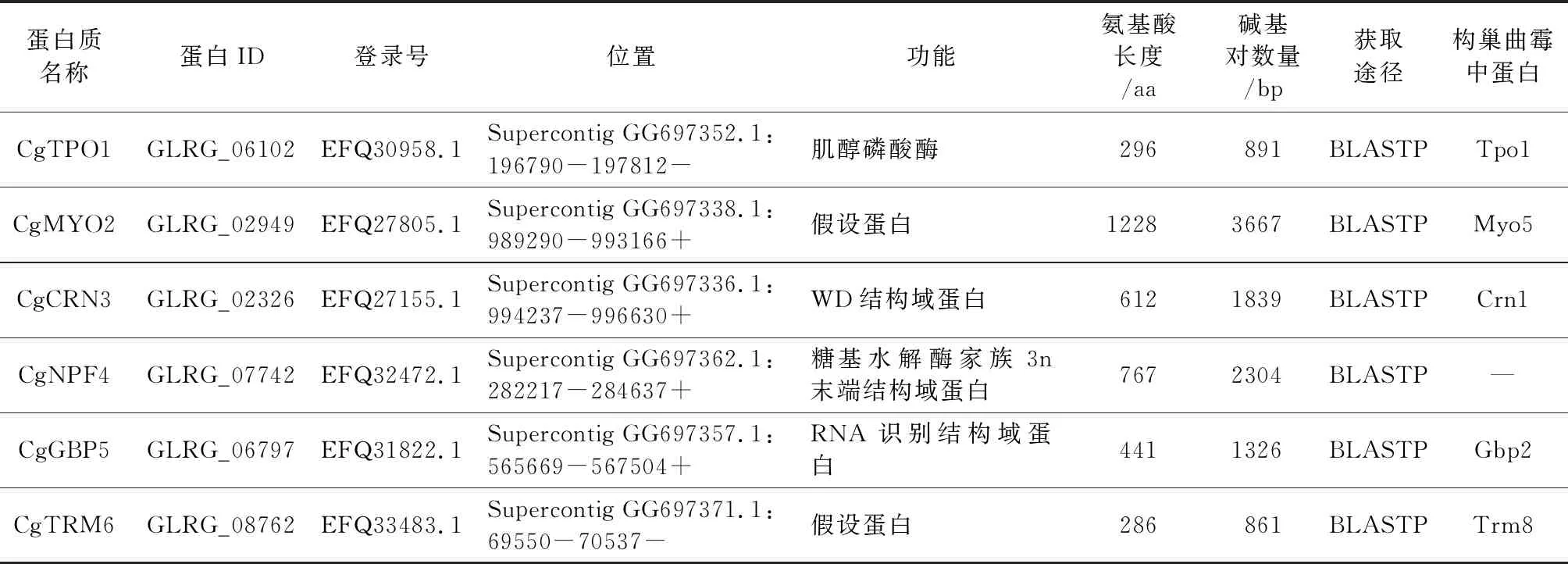
表 1 禾谷炭疽菌中NPFxD基序蛋白的基本信息及获取方法Table1 Basic information and method of obtaining NPFxD motif protein in C. graminicola
禾谷炭疽菌Colletotrichumgraminicola(Cesati)Wilson是一种半活体营养病原菌,主要危害玉米、小麦等农作物[7-9],也可定殖并感染蓝草、黑麦草和羊茅等禾本科植物上,严重危害着禾本科作物的健康生产,影响农业经济的发展。随着该菌全基因组序列的释放[10],前人对其分泌蛋白、G蛋白信号通路蛋白、MAPK(丝裂原活化蛋白激酶)途径蛋白[11]、RGS(G蛋白信号转导调节蛋白)[12]等进行了预测及生物信息学分析,为进一步明确上述蛋白在该病菌致病过程中的功能解析奠定了理论基础。然而,尚未见有关禾谷炭疽菌中存在具有NPFxD基序的蛋白情况及其生物信息学分析的研究报道,严重制约着植物病原丝状真菌中内吞相关蛋白的功能研究。
因此,基于前人报道,现利用模式生物构巢曲霉Aspergillusnidulans中具有NPFxD基序的蛋白氨基酸序列[13],通过在禾谷炭疽菌蛋白质数据库中进行Blastp比对分析,同时,利用关键词在美国国家生物信息中心(NCBI)数据库中进行搜索,明确C.graminicola中NPFxD基序蛋白序列,并对上述序列开展跨膜结构、理化性质、疏水性、二级结构以及信号肽、亚细胞定位等生物信息学分析,以及结合其他植物病原丝状真菌中的NPFxD基序蛋白同源的氨基酸序列,开展遗传关系分析,为进一步开展同属于炭疽菌属但其基因组序列尚未公布的核桃炭疽病菌相关内吞蛋白研究提供重要的理论指导。
1 材料与方法
1.1 禾谷炭疽菌NPFxD蛋白序列获取
通过NCBI数据库下载禾谷炭疽菌的蛋白质序列,利用EMBOSS fuzzpro[14]预测程序获取该菌中具有NPFxD保守基序的蛋白质序列,同时,利用“NPFxD”“NPFxD-mediated endocytosis”等关键词对禾谷炭疽菌蛋白数据库进行搜索,此外,以A.nidulans中具有NPFxD基序的蛋白序列[13]为基础,进行Blastp比对分析,对上述所获得序列进行汇总、去重,最终明确禾谷炭疽菌中NPFxD基序蛋白以及相关信息(图 1)。
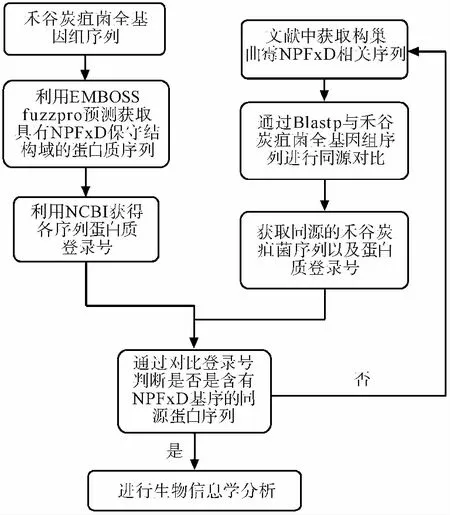
图 1 禾谷炭疽菌NPFxD基序蛋白获取方法Fig.1 Method for obtaining NPFxD motif protein in C. graminicola
1.2 实验方法
1.2.1 跨膜预测
利用在线跨膜区结构预测网站HMMTOP version 2.0[15]和TMHMM Server v. 2.0[16]对NPFxD基序蛋白进行预测。
1.2.2 保守结构域预测
利用在线保守结构域特征分析软件SMART[17]对NPFxD基序蛋白进行预测。
1.2.3 亚细胞定位分析
利用亚细胞定位分析软件ProtComp v9.0[18]对NPFxD基序蛋白进行预测,并绘制定位情况。
1.2.4 理化性质分析
利用理化性质测定程序Protscale[19]对NPFxD基序蛋白进行预测。
1.2.5 转运肽及信号肽预测
利用蛋白质转运肽分析软件TargetP 1.1 Server[20]对NPFxD基序蛋白进行预测;同时,利用蛋白质信号肽软件SignalP 5.0 Server[16]对NPFxD基序蛋白进行预测。
1.2.6 二级结构预测
采用蛋白质二级结构分析软件PHD[21]对NPFxD基序蛋白进行预测。
1.2.7 遗传关系进化树构建
在NCBI中在线进行Blastp同源搜索获取同源序列,并利用ClustalX[22]和MEGA X软件[23]分别对其进行多重比对分析以及构建遗传关系进化树。
2 结果与分析
2.1 禾谷炭疽菌NPFxD基序蛋白找寻及保守结构域预测
前人对构巢曲霉开展了NPFxD基序蛋白的搜索工作[24],利用EMBOSS fuzzpro软件对禾谷炭疽菌全部蛋白质序列进行扫描分析,结果获得了33条含有NPFxD基序的蛋白序列,同时,利用“NPFxD-mediated endocytosis”以及“NPFxD”等关键词进行搜索,未获得相关序列,此外,进一步利用Blastp比对分析,获得了621条同源序列(结果未显示),对上述蛋白序列进行对比筛选、去除重复,结果表明,与A.nidulans同源的C.graminicola中含有NPFxD基序蛋白质序列共6条,其蛋白ID分别为GLRG_06102、GLRG_02949、GLRG_02326、GLRG_07742、GLRG_06797、GLRG_08762,根据同源关系,将上述所获得的的NPFxD蛋白分别命名为CgTPO1、CgMYO2、CgCRN3、CgNPF4、CgGBP5、CgTRM6(表 1)。
基于TMHMM跨膜结构域软件分析,上述6个蛋白均不具有跨膜结构域;同时,进一步利用HMMTOP跨膜结构域软件进行预测,结果表明,除CgNPF4、CgGBP5未发现明显的跨膜结构域外,其他NPFxD基序蛋白均具有跨膜结构域(表 2)。同时,利用SMART在线预测禾谷炭疽菌中6个NPFxD基序蛋白的保守结构域,结果表明,上述NPFxD基序蛋白序列具有Inositol_P、MYSc、DUF100以及RRM、Methyltransf_4等保守结构域,然而,并未存在同种保守结构域(图 2),推测上述不同类型保守结构域在实现禾谷炭疽菌内吞作用过程中发挥着其他特殊作用。
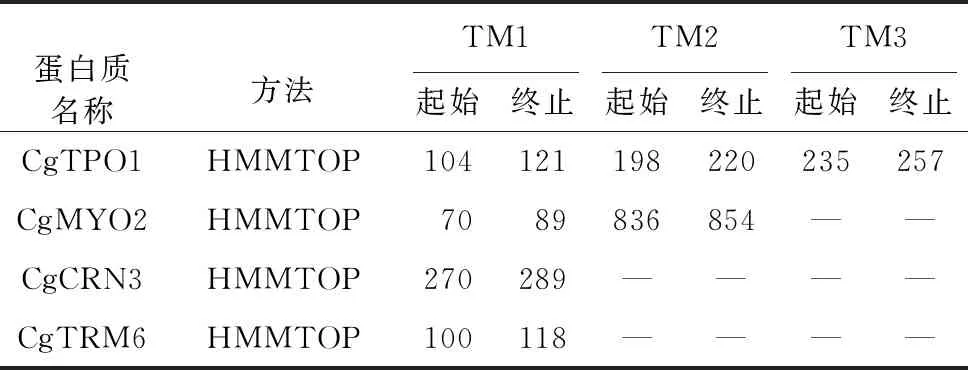
表 2 跨膜情况预测Table2 Prediction of transmembrane
2.2 禾谷炭疽菌中NPFxD基序蛋白序列的亚细胞定位分析
结合S.cerevisiae、P.oryzae以及其他模式生物,特别是参考SNARE蛋白家族、Rab蛋白家族以及Dynamin家族蛋白等有关植物丝状真菌内吞信号的一般途径,结合跨膜结构域分析,绘制出C.graminicola中所含的NPFxD亚细胞定位情况(图 3)。
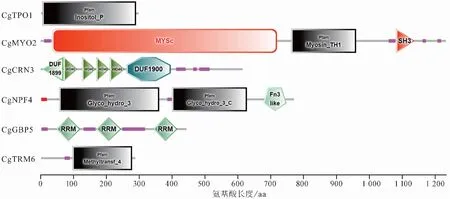
图 2 保守结构域分析Fig.2 Analysis of conserved domains
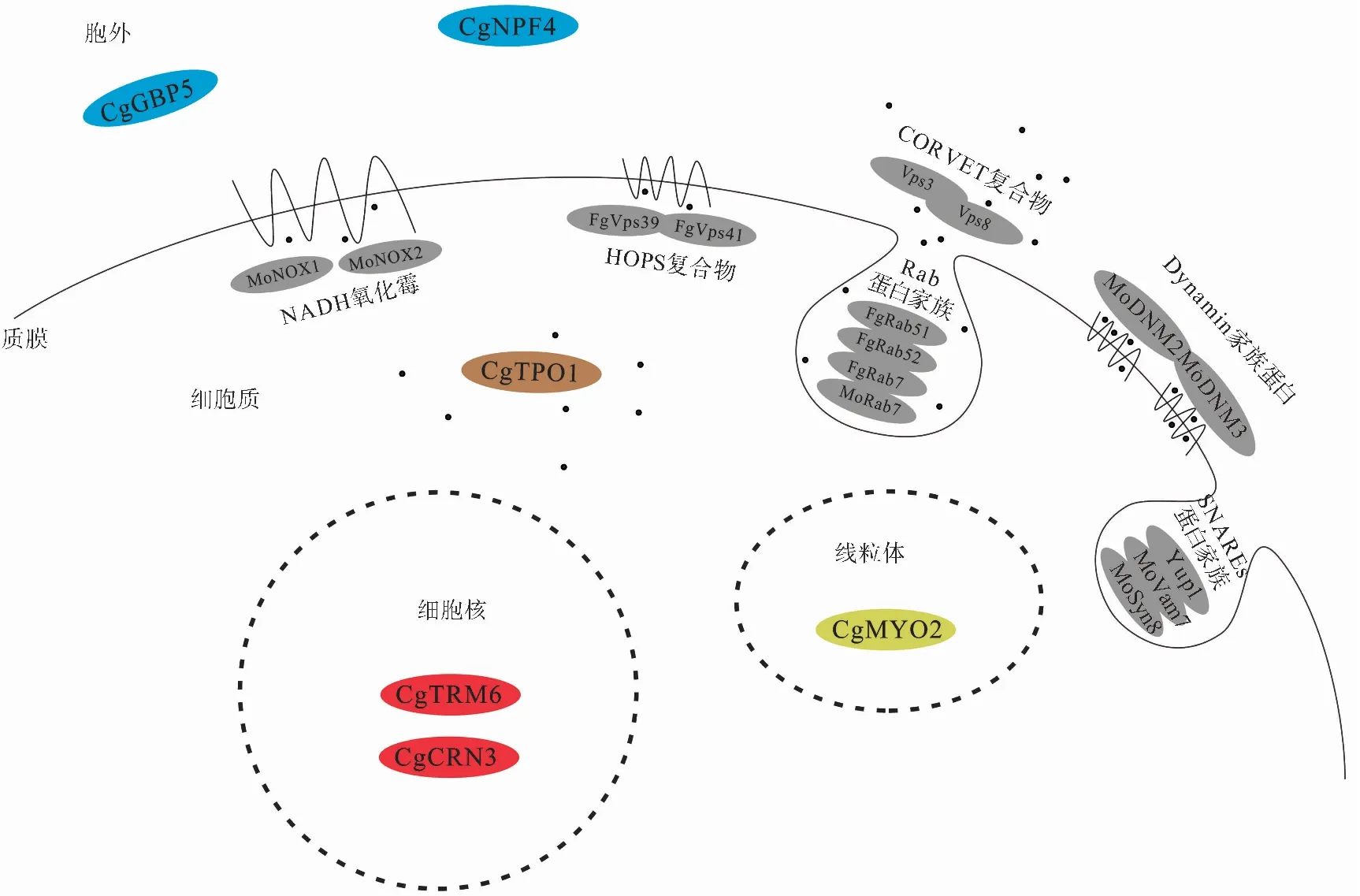
Yup1、MoVam7、MoSyn8、FgRab51、FgRab52、FgRab7、MoRab7、MoDNM2、MoDNM3、Vps3、Vps8、FgVps39、FgVps41、MoNOX1、MoNOX2分别为 真菌Ustilago maydis、Pyricularia oryzae、P. oryzae、F.graminearum、P. oryzae、Aspergillus nidulans、F.graminearum、P. oryzae中已经报道的蛋白图 3 亚细胞定位分析Fig.3 Subcellular localization
2.3 禾谷炭疽菌NPFxD蛋白质理化性质分析
对禾谷炭疽菌中的6个NPFxD基序蛋白开展氨基酸残基组成情况分析,结果表明,CgTPO1、CgGBP5中所含有的G(甘氨酸)氨基酸残基所占比例较高,分别为11.10%、22.20%;而CgMYO2、CgCRN3、CgNPF4、CgTRM6中含有的A(丙氨酸)氨基酸残基所占比例较高,分别为9.00%、11.40%、11.10%以及9.40%(表 3);同时,对上述蛋白中含有较低比例的氨基酸残基进行分析,结果发现,在CgTPO1蛋白中所含有的H(组氨酸)最低,所占比例为1.00%,在CgTRM6蛋白中所含有的W(色氨酸)最低,所占比例为1.00%,在CgMYO2、CgCRN3、CgNPF4、CgGBP5蛋白中所含有的C(半胱氨酸),所占比例分别为0.60%、0.30%、1.40%和0.20%(表 3)。
进一步对上述NPFxD基序蛋白开展理化性质分析,结果表明,在分子式、相对分子质量、理论等电点、正负电荷残基数以及原子总数、脂肪族氨基酸指数等理化性质方面均存在着较大的不同,尽管如此,上述NPFxD基序蛋白总平均亲水性为亲水性蛋白,半衰期均为30h,同时,上述蛋白稳定性不尽相同,其中,CgTPO1、CgNPF4和CgGBP5蛋白属于不稳定蛋白,其余2个蛋白则属于稳定性蛋白(表 4)。
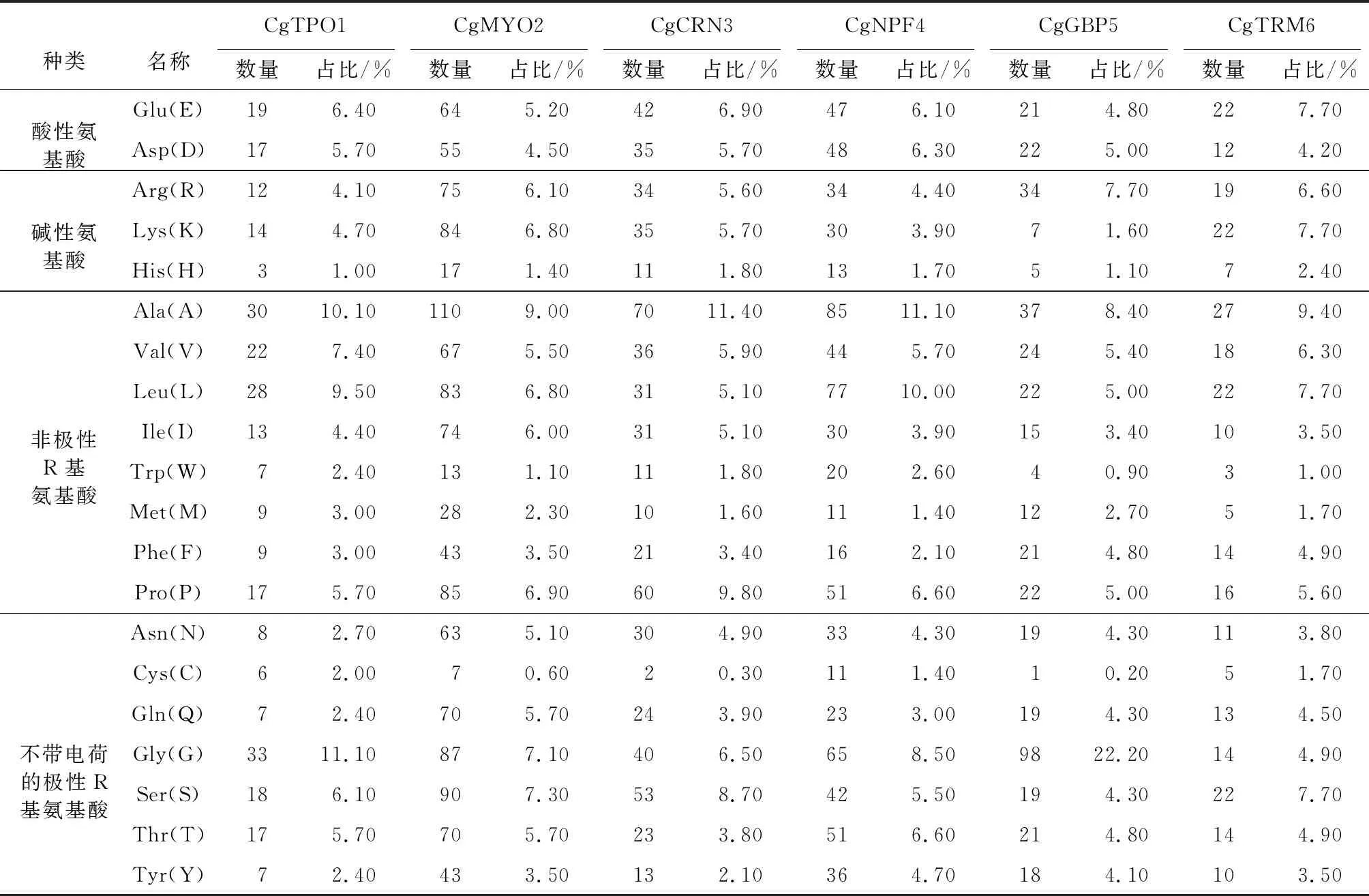
表 3 氨基酸残基组成情况Table3 Amino acid residues composition
为了进一步明确上述6个NPFxD基序蛋白中氨基酸残基的亲(疏)水性情况,利用疏水性预测网站对其进行分析,结果表明,不同蛋白的最强亲(疏)水性残基及其位置情况、最强亲(疏)水性残基数值综合情况均不尽相同,其中,CgTPO1和CgTRM6在最强亲水性残基均为T(苏氨酸),而其在最强疏水性残基的组成上不同;CgTPO1和CgCRN3在最强疏水性残基均为V(缬氨酸),CgMYO2和CgTRM6则均为L(亮氨酸),上述蛋白在最强亲水性残基上不同(表 5)。
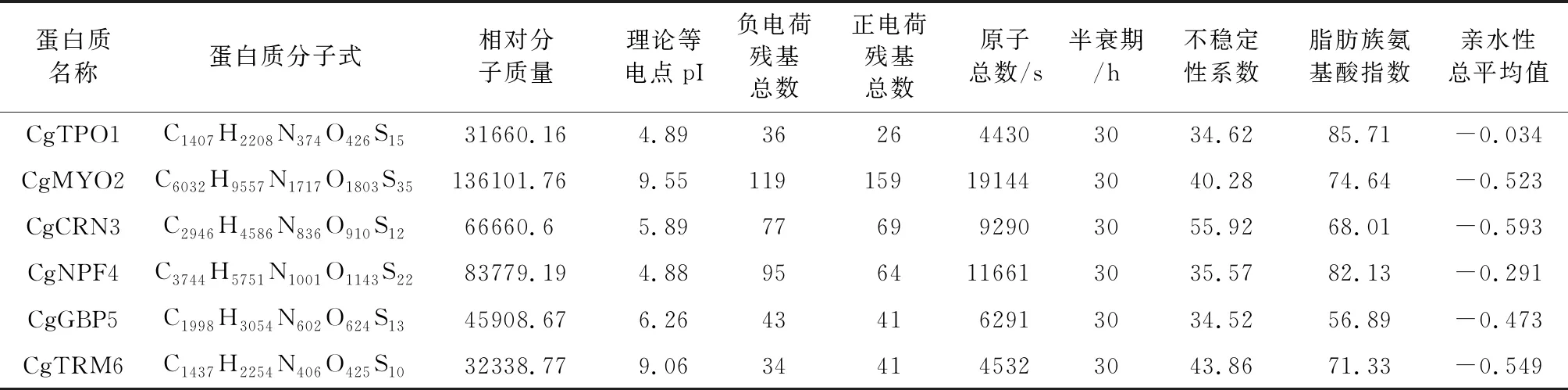
表 4 基本理化性质分析Table4 The physicochemical properties
2.4 禾谷炭疽菌NPFxD蛋白转运肽及信号肽特征
经过TargetP在线分析蛋白的转运肽情况,结果表明,C. graminicola中的NPFxD基序蛋白中的转运肽预测定位情况及结果可靠性情况不尽相同,除CgCRN3和CgNPF4分别定位于线粒体和分泌途径外,结合之前所开展的亚细胞定位分析结果,发现两者结果不一致,同时,其他4个蛋白均未得到有效定位情况(表 6),上述研究结果有待于进一步通过生物学试验进行解析和验证。
由于TMHMM和HMMTOP预测程序对于蛋白跨膜结构域的预测与信号肽的预测之间存在着一定的重叠性,因此,本研究进一步利用信号肽预测软件SignalP 5.0对上述蛋白进行分析,结果表明,上述6条蛋白序列中仅有1条序列(CgNPF4)具有明显的信号肽,其信号肽切割位置在17~18 aa,最大切割率为0.949(表 7)。
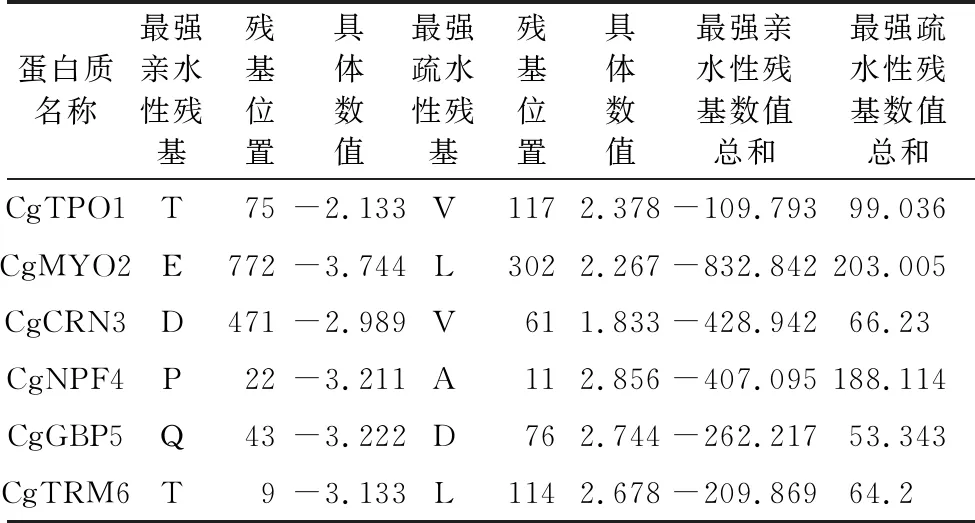
表 5 亲(疏)水性氨基酸残基位置Table5 Hydrophobic and hydrophilic amino acid residue positions
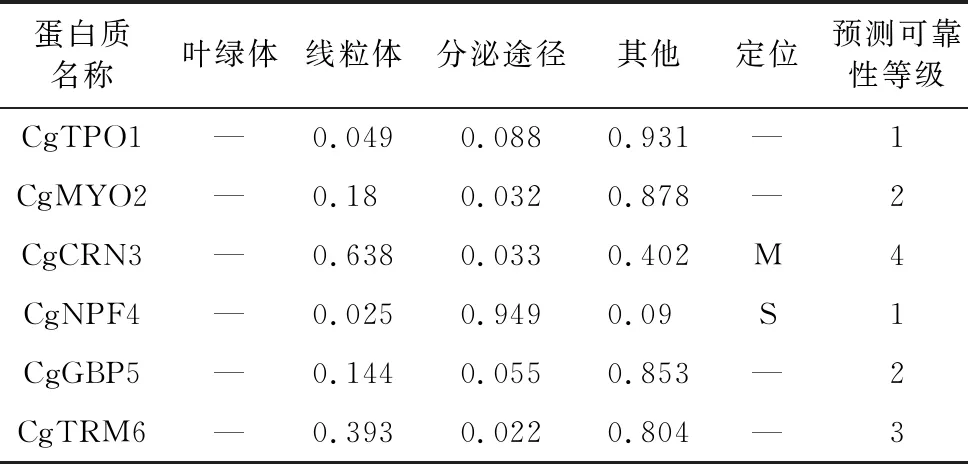
表 6 潜在转运肽的可能性预测Table6 Possibility prediction of transit peptides
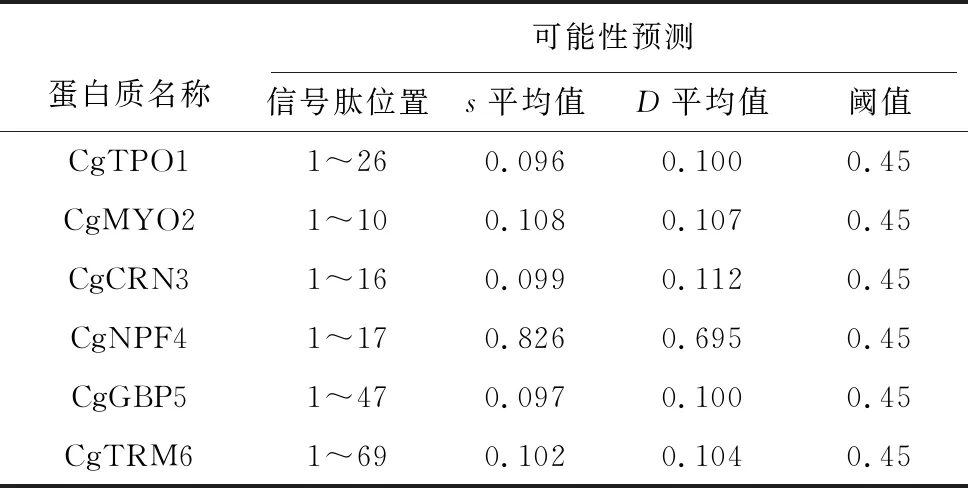
表 7 含有信号肽的可能性预测Table7 Probability prediction of signal peptide
2.5 禾谷炭疽菌NPFxD蛋白二级结构预测
一般而言,蛋白质的基本理化性质由其氨基酸组成情况决定,而其二级结构则较好地反映其生化性质。为了进一步明确禾谷炭疽菌C.graminicola中的NPFxD基序蛋白的二级结构情况,更好地明确其功能情况,对上述6个蛋白开展了二级结构预测工作,结果表明,CgMYO2中所含的α螺旋所占比例较高,为45%;而CgCRN3所含有的β卷曲所占比例较高,为37%;相对而言,CgMYO2中所含有的β卷曲所占比例则较低,为9%,而CgGBP5中所含有的无规则卷曲所占比例较高,为48%(图 4)。
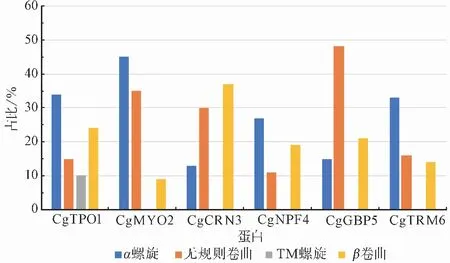
图 4 二级结构分析Fig.4 Secondary structure analysis
2.6 遗传关系
为了进一步明确不同植物病原真菌中NPFxD基序蛋白的遗传关系,以C.graminicola中的CgTPO1、CgMYO2、CgCRN3、CgNPF4、CgGBP5、CgTRM6氨基酸序列为基础,在NCBI数据库中进行同源序列搜索,同时,利用ClustalX[22]和MEGA X软件[23]分别对上述序列开展多重序列比对和系统进化树构建等,结果表明,在所选择的30个蛋白中,分别以C.graminicola中的6个NPFxD基序蛋白为核心蛋白聚类形成了四大类,同时,该菌中的NPFxD基序蛋白又与炭疽菌属真菌(C.sublineola、C.incanum)中相关蛋白有着较近的亲缘关系,此外,禾谷炭疽菌中的CgMYO2和CgTRM6彼此之间亲缘关系最为紧密,其次为CgTPO1和CgNPF4(图 5)。
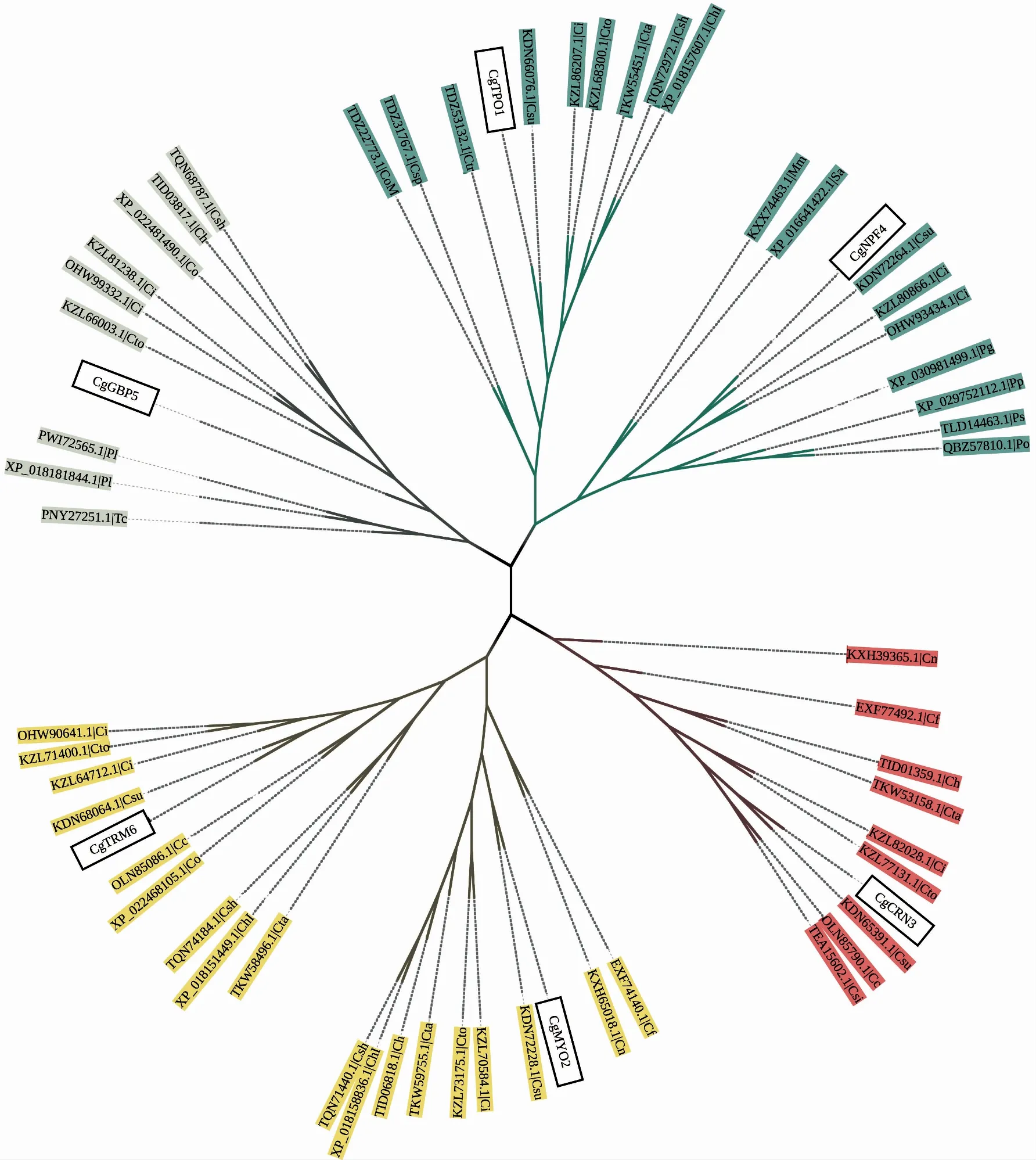
Cc、Ch、ChI、Cf、Ci、Cn、Csh、Csi、Csp、Csu、Cta、Cto、Ctr、Co、CoM、Pg、Pp、Pl、Ps、Po、Sa、Tc、Mm分别为C.higginsianum、C. higginsianum IMI 349063、C. chlorophyti、C. fioriniae PJ7、C. incanum、C. nymphaeae SA-01、C. shisoi、C. sidae、C. spinosum、C. sublineola、C. tanaceti、C. tofieldiae、C. trifolii、C. orchidophilum、C. orbiculare MAFF 240422、P. grisea、P. pennisetigena、Purpureocillium lilacinum、Pyricularia sp. CBS 133598、P. oryzae、Scedosporium apiospermum、Tolypocladium capitatum、Madurella mycetomatis物种的缩写图 5 禾谷炭疽菌NPFxD蛋白与其他物种之间的遗传关系Fig. 5 Genetic relationship between the NPFxD protein in C. graminicola and other species
3 结论与讨论
3.1 结论
通过关键词搜索和Blastp比对分析,明确了C.graminicola中存在6个与构巢曲霉具有高度同源性的NPFxD基序蛋白;并通过在线分析软件SMART、SignalP等,对上述蛋白的保守结构域、信号肽、理化性质及亚细胞定位等进行预测分析,明确上述蛋白在保守结构域方面具有多种不同的保守域,且缺乏较为同一的保守域结构;上述蛋白在信号肽、理化性质、疏水性、亚细胞定位等方面也存在着明显的差异性;进一步利用上述NPFxD基序蛋白开展遗传关系分析,明确该菌中NPFxD基序蛋白与炭疽菌属真菌中的蛋白具有较近的亲缘关系。
3.2 讨论
2012年,禾谷炭疽菌的全基因组序列释放,为进一步开展其致病因子提供了重要的数据支撑[10]。前人基于全基因组序列数据,对该菌中的诸多致病基因开展了基因敲除、蛋白生信分析、功能解析等方面的研究,一些如G蛋白偶联受体GPCR[25]、磷脂酰肌醇特异性磷脂酶PI-PLC[26]、磷脂酰肌醇转移蛋白Pth11[27]、细胞外膜蛋白CFEM[28]等涉及G蛋白信号效应分子及其特征得到进一步明确。上述研究为解析禾谷炭疽菌侵染禾本科植物过程中,蛋白亚细胞定位、功能发挥等方面的深入研究提供了重要的理论基础。然而,作为真菌中重要的细胞活动之一——内吞,尚缺乏在植物病原丝状真菌中的解析工作,特别是对于禾谷炭疽菌中NPFxD基序蛋白的研究尚未见报道。前人对于模式真菌中内吞作用的研究工作已经明确,具有NPFxD基序的蛋白可用于其他正常受体的内化过程,对于缺乏泛素化位点的受体尤为重要[29]。内吞作用可以大量出现在丝状真菌菌丝尖端,并且菌丝尖端的快速延伸可能需要有效的内吞作用[30]。
中外学者对内吞在模式真菌及植物病原丝状真菌的菌丝生长中作用已明确,例如,构巢曲霉中存在着诸多内吞相关蛋白,稻瘟菌中内吞调控蛋白可以有效抑制寄主免疫反应[31]。结合前人关于植物病原丝状真菌特别是稻瘟病菌中内吞机制调控蛋白的研究,有理由推测禾谷炭疽菌在菌丝生长和发育过程中也很可能与内吞机制调控有关。就禾谷炭疽菌与禾本科植物互作而言,当禾本科植物通过各种信号途径激活自身防卫反应后,可以有效地阻止病原真菌在其体内的进一步扩散和传播;同样,当禾谷炭疽菌中相关蛋白感应到植物的防卫反应时,也必将进一步采取和调整相应的攻击方法,如分泌毒素、角质酶、果胶酶和纤维素酶等致病因子,进一步破坏寄主植物的免疫反应,从而实现其与植物之间的此消彼长“军备竞赛”关系。在上述过程中,内吞作用在禾谷炭疽菌侵染植物以及不同营养阶段中的地位和作用是什么?其内吞作用与植物中解毒物质分泌之间的关系如何?是否还存在其他不同基序的内吞蛋白?研究中所获得NPFxD基序蛋白在保守结构域方面具有其他结构发挥何种功能?上述问题均有待于进一步通过生物学试验开展研究和探讨,从而较好地明确禾谷炭疽菌中的内吞作用机制,为进一步防控该病菌提供重要的解决思路。
