乳酸乳球菌乳酸亚种的m6A甲基化差异研究
2021-06-07郑慧娟李伟程赵飞燕蔡虹宇孙志宏张和平
郑慧娟,李伟程,赵飞燕,蔡虹宇,孙志宏,张和平
(内蒙古农业大学 乳品生物技术与工程教育部重点实验室 农业农村部乳制品加工重点实验室内蒙古自治区乳品生物技术与工程重点实验室 呼和浩特010018)
乳酸乳球菌乳酸亚种(Lactococcus lactis subsp.lactis)是兼性厌氧、不产气、不产芽孢且无荚膜的革兰氏阳性菌,是公认安全的食品级微生物[1]。其可将大分子营养物质降解成易被人体消化吸收的小分子营养物质,其中2,3-丁二醇等物质可赋予发酵制品特殊的滋气味,同时产生的乳酸等还可抑制有害菌生长,延长发酵制品的货架期[2]。此外乳酸乳球菌乳酸亚种具有多种益生功能,被作为发酵剂广泛应用于发酵乳制品、发酵果蔬制品与发酵肉制品等[3]。乳酸乳球菌乳酸亚种较高的经济价值使其成为研究热点。随着分子生物学的发展,测序手段与生物信息学分析的成熟,研究者从基因层面了解乳酸乳球菌乳酸亚种愈发便利。越来越多乳酸乳球菌乳酸亚种的全基因组序列完成图被鉴定,为生产提供了遗传学理论指导[4-5]。
随着研究地深入,部分研究发现基因型相似的菌株间表型存在较大的差异性,而这些差异可能由菌株的遗传表观修饰造成的[6-7]。其中,DNA甲基化是一种典型遗传表观修饰,指DNA 碱基在甲基转移酶的作用下增加甲基基团化学修饰的过程,其具有不改变DNA 序列的特点。Zhu 等[6]发现平均核苷酸同源性高于95%的伯克霍尔德氏菌(Burkholderia seminalis)菌株适应于不同生态位,可能是菌株甲基化组的差异导致的。序列相似度高于98%的短双歧杆菌(Bifidobacterium breve)拥有多种可保护自身免受外源DNA 侵染甲基转移酶[7]。Cooper 等[8]通过对不同血清型的大肠杆菌(Escherichia coli)进行基因组测定,发现一部分菌株毒力的产生与其DNA 的甲基化有关。目前许多甲基化相关研究发现细菌中的甲基化修饰位点以m6A(N6-methyladenosine,N6-甲基腺嘌呤)为主[9]。这类研究的关注对象多集中于致病菌,而优良发酵剂相关的研究鲜有报道。
BL19 与IMAU10978 均分离自乳源的乳酸乳球菌乳酸亚种,表型差异明显。其中BL19 具有发酵剂的特点,包括:发酵速度快,蛋白水解彻底,能产生具有特殊香气的挥发性物质等[10]。本文旨在从基因组与甲基化组角度比较BL19 与IMAU10978,探究2 株菌发酵特性差异的原因,表明乳酸乳球菌乳酸亚种功能基因组特征和m6A 基序类型、分布与功能等甲基化组特征,推测发酵特性差异的原因,为发酵工业的菌株选育提供理论依据。
1 材料与方法
1.1 试验菌株与试剂
本研究的实验菌株BL19 与IMAU10978 均由内蒙古农业大学乳品生物技术与工程教育部重点实验室提供。BL19 分离自俄罗斯布里亚特共和国昆库尔的嚼口,IMAU10978 分离自中国内蒙古自治区赤峰市的酸牛奶。同时从NCBI(National Coalition Building Institute,https://www.ncbi.nlm.nih.gov/)下载分离自乳源且被广泛研究的乳酸乳球菌乳酸亚种KLDS 4.0325、Il1403、FM03、S0 和UC06 的序列,以进行比较分析(序列登录号如表1所示)。
M17 培养基,英国Oxoid 公司;RNA 酶,天根生化科技有限公司;Wizard®Promega Genomic DNA Purification Kit,美国Promega 公司;核酸染料,北京全式金生物技术有限公司。
1.2 仪器、设备
Qubit2.0 荧光计,美国Life Technologies 公司;LRH-250 电热恒温培养箱、HWS28 电热恒温水浴锅,上海一恒科技有限公司;5810R 台式高速冷冻离心机,德国Eppendorf 公司;ZHJH-C1214C型智能安全型超净工作台,上海智城分析仪器制造有限公司;PacBio SMRT RSⅡ测序平台,Pacific Biosciences 公司。
1.3 菌株活化与菌泥制备
将BL19 与IMAU10978 通过M17 肉汤培养基活化至3 代,培养条件为37℃,24 h。对第3 代培养液离心以收获菌泥,4 000×g,5 min。之后用PBS 洗涤菌泥2 次,7 000×g 离心2 min。
1.4 DNA 提取与纯化
按照Wizard®Genomic DNA Purification Kit说明书对洁净菌泥进行DNA 提取。
通过0.6×琼脂糖凝胶对提取的DNA 进行电泳,以判定所提取DNA 条带的完整性与纯净性。用0.46×磁珠对DNA 进行吸附。弃废液,并用75%乙醇对吸附DNA 的磁珠进行快速洗涤以纯化DNA。最后用回溶液将纯化后的DNA 从磁珠上回溶。并用Qubit 检测纯化后DNA 的质量浓度,通过所得DNA 体积与质量浓度相乘获得所纯化DNA 的最终质量。
1.5 全基因组测序及组装
上机要求每株菌的DNA 体积在150 μL 以内,质量大于7 ng。通过PacBio RSⅡ测序平台对高质量的乳酸乳球菌乳酸亚种DNA 进行全基因组测序。通过SMRT®Portal(V2.7)软件的方案RS_HGAP_Assembly.3 进行质控和菌株基因组组装。使用circlator(V1.5.5)对下机数据进行环化已得到基因组完成图。
1.6 基因组比较分析
1.6.1 基因组分析 计算试验菌株的平均核苷酸一致性(Average Nucleotide Identity,ANI)与总核苷酸一致性(Total Nucleotide Identity,TNI)[11-12]。
1.6.2 功能基因组分析 将上述组装好的核酸序列文件上传至RAST(Repaid Annotion using Subsystem Technology http://rast.nmpdr.org/rast.cgi)进行注释,并下载对应的氨基酸注释文件。
将BL19 与IMAU10978,以及从NCBI 下载的FM03、Il1403、KLDS 4.0325、S0、UC06 菌株的氨基酸注释序列与蛋白相邻类的聚簇(Cluster of Orthologous Groups of proteins,COG)数据库比对,设置参数:E 值小于10-3。采纳问询序列与数据库序列相似度大于40%且问询序列比对上长度与序列总长度的比值大于80%的比对结果[13]。
将各菌株的蛋白注释序列与碳水化合物活性酶(Carbohydrate-Active enZYmes,CAZy)数据库比对。采纳E 值小于10-3、相似度大于40%且问询序列比对上长度与序列总长度的比值大于80%的比对结果[13]。
1.7 甲基化组分析
使用SMRT®Portal(V2.7)软件中RS_Motification_and_motify_Analysis.1 方案对BL19 与IMAU10978 进行甲基化分析,参考序列选择对应组装效果好的序列。对基因组的甲基化位点进行预测分析,并分析特定基序的分布及其功能。
1.8 数据作图
使用R(V3.5.0)软件中的“pheatmap”程序包绘制热图。使用OriginPro 2015 绘制折线图、柱状图与3D 色带图(3D Ribbons)。
2 试验结果
2.1 基因组概况
菌株BL19 的基因组大小为2.72 Mb,包含5个质粒,GC 含量为35.19%,菌株IMAU10978 的基因组大小为2.82 Mb,包含6 个质粒,GC 含量为35.20%。从NCBI 下载的菌株UC06、S0、KLDS 4.0325、Il1403、FM03 的基因组大小为(2.57 ±0.17)Mb,其GC 含量为(35.26 ±0.07)%。除了Il1403 与S0,所有菌株的基因组均含有多个质粒。
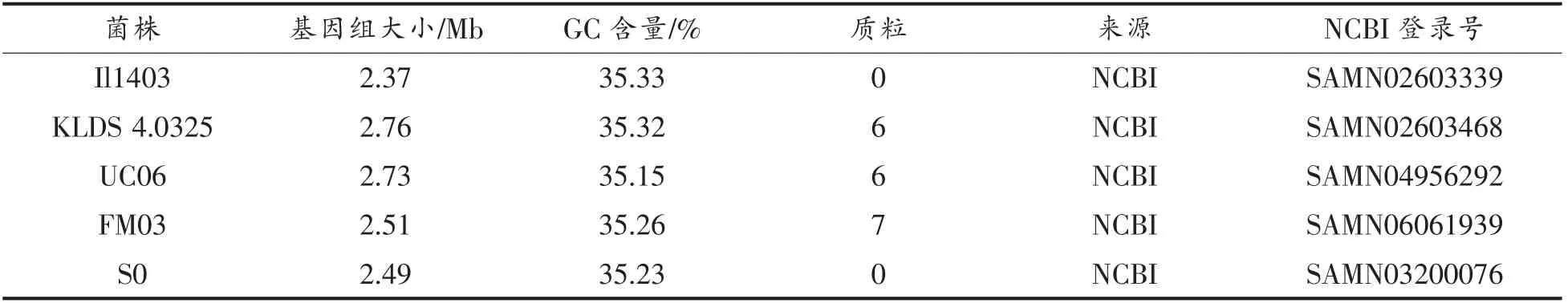
表1 试验菌株的基本信息Table 1 Basic information of experimental strains
ANI 反映基因组间的相似性,比DNA 杂交(DNA-DNA hybridization,DDH)更简单便利,常被用于进化距离的计算[14]。由图1可知,试验菌株之间的ANI 相似度均高达98.24%,TNI 均高达77.63%。这2 个最低值均高于乳酸乳球菌乳酸亚种的平均值,说明试验选取菌株具有较高的基因组相似性[15]。

图1 试验菌株的核苷酸一致性Fig.1 Nucleotide identity of test strains
2.2 功能预测
2.2.1 RAST 注释RAST 注释结果(图2a)显示乳酸乳球菌乳酸亚种基因组以碳水化合物、氨基酸及其衍生物与蛋白代谢相关基因为主。其中BL19 与IMAU10978 蛋白质折叠、蛋白质生物合成、蛋白质加工和改性相关基因数量基本一致。同时2 株菌均包含低聚果糖和棉子糖利用与乳糖和半乳糖的摄取和利用等碳水化合物相关基因,其数量不存在明显差别。
2.2.2 COG 注释 试验选取菌株的基因组约83.60%得到注释。图2b 发现试验菌株具有类似的特点,即E(氨基酸运输和代谢)、J(翻译,核糖体结构和生物合成)与G(碳水化合物转移和代谢)3 种已知功能大类在基因组中占最高比例。其中,氨基酸运输和代谢功能相关的基因数量相同。

图2 试验菌株的功能注释Fig.2 Functional annotations of test strains
2.2.3 CAZy 注释对试验菌株进行CAZy 注释。结果(图2c)表明,7 株乳源乳酸乳球菌乳酸亚种均注释到碳水化合物酯酶家族(Carbohydrate esterase family,CE)、糖苷水解酶家族(Glycoside hydrolase family,GH)、糖基转移酶家族(Glycosyltransferase family,GT)和辅助酶家族(Auxiliary activities family,AA)的基因,包括CE3、CE10、GH2、GT3、GH20、GH125、CE1、GH1、GH77、CE9与GT5 等酶的基因,其中以GH1(包括β-葡萄糖苷酶与β-半乳糖苷酶等)的基因居多。
3 种功能注释结果均显示试验菌株在功能基因组层面表现出极高的相似性。与菌株生长代谢相关的基因在基因组中占更高的比例,包括碳水化合物与氨基酸代谢等。Kelleher 等[16]认为乳酸乳球菌乳酸亚种区别于乳酸乳球菌乳脂亚种的特点为富含丰富的碳水化合物相关基因。其COG 注释显示乳酸乳球菌乳酸亚种的碳水化合物代谢和氨基酸代谢相关基因明显高于乳脂亚种,分别占基因组14%和15%。其结果略高于本研究的结果,这可能是不同的筛选条件导致的,总之2 种结果的整体规律是一致的。Sun 等[17]发现乳酸菌含有丰富多样的GH 与GT 基因,本研究的注释结果相似也发现了类似规律。
2.3 甲基化比较结果
由于试验菌株基因组较为相似,无法很好地解释BL19 与IMAU10978 的发酵特性差异,因而从甲基化组角度对2 株菌进行更深入地分析。由图3可知,2 株菌碱基A 的甲基化质量值(Modification quality value,Mod QV)均明显高于其它碱基,同时碱基A 与C 均高于甲基化检测阈值。甲基化质量值可用来预示位点发生甲基化的概率,甲基化检测阈值通常为Mod QV 30(P=0.001)[18]。同时,Murray 等[19]也认为SMRT 测序技术检测甲基化是方便可靠。
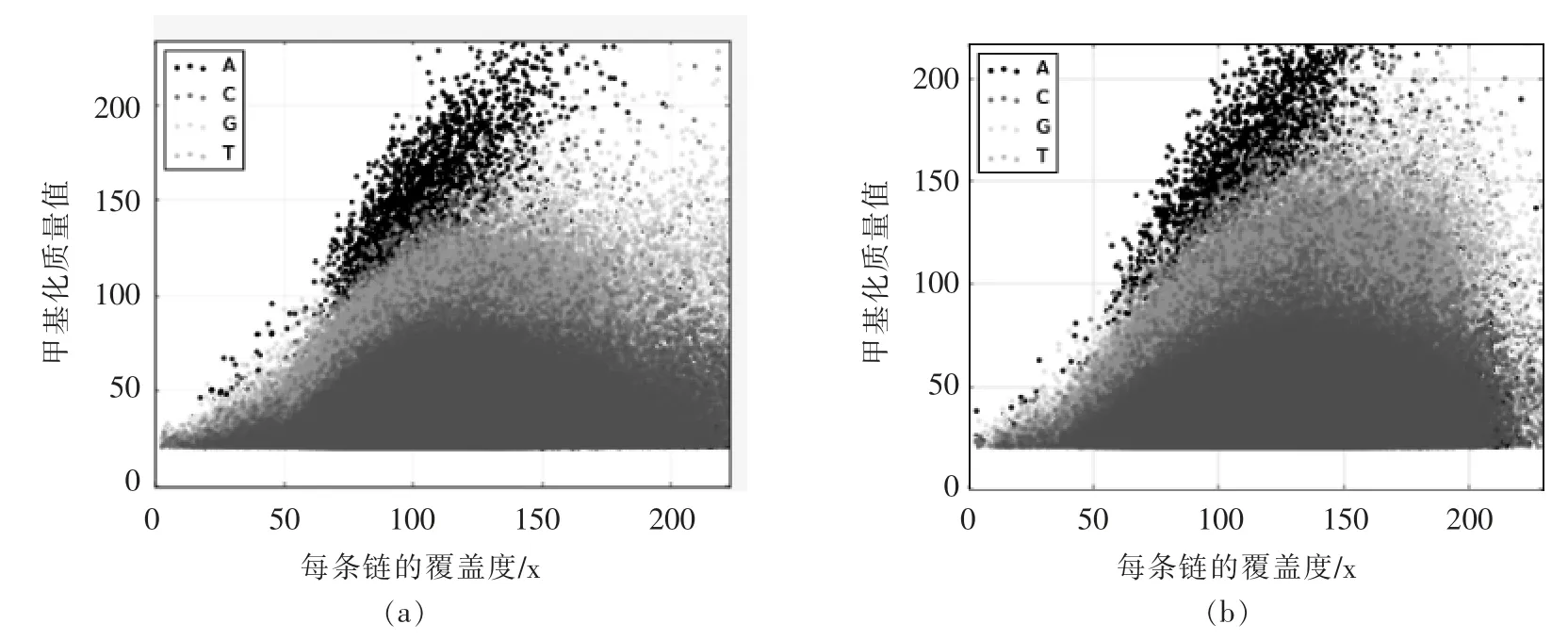
图3 IMAU10978(a)与BL19(b)的甲基化位点动力学检测散点图Fig.3 Scatter plot of kinetic detection of modification sites IMAU10978(a)and BL19(b)
2.3.1m6A 修饰位点及基序鉴定结果IMAU10978与BL19 的基因组中m6A 修饰位点分别有9 377与4 541 个。2 株菌中m6A 修饰的特有基序类型的统计情况如表2。BL19 基因组中甲基化率最高的m6A 甲基化基序为ACAAYC,其甲基化率为99.02%。IMAU10978 基因组中甲基化率最高的m6A 甲基化基序为ATGNNNNNGTTA 和GAAHNNN NNNTCC,其甲基化率分别为98.04%和97.50%。
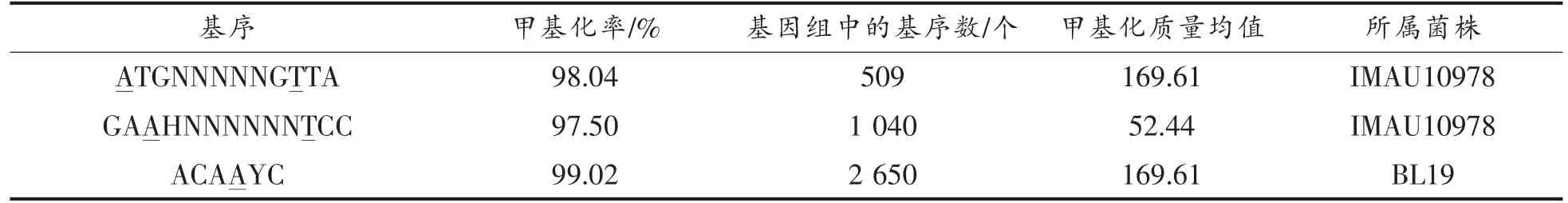
表2 IMAU10978 与BL19 的甲基化基序信息Table 2 Methylation motif information of IMAU10978 and BL19
2.3.2m6A 修饰基序的分布对上述m6A 修饰基序进行分布统计,发现3 种m6A 修饰基序在基因间区与基因区分布如图4a所示,3 种基序均大量分布于基因间区,推测m6A 修饰位点呈一种随机分布。与分布于基因间区的m6A 修饰位点相比,分布于功能基因组的m6A 修饰位点可能对菌株产生更严重的影响,从而导致发生这类变化的菌被淘汰的更多[20]。
对3 种m6A 修饰基序的分布情况进行近一步的分析。由图4b~d所示,BL19 的ACAAYC 与IMAU10978 的ATGNNNNNGTTA 均在染色体基因组中随机分布,而GAAHNNNNNNTCC 则在染色体基因组的起始处分布频次出现极短暂的小高峰,随后迅速达到平稳。3 种m6A 修饰基序的分布趋势整体表现为随机分布,在一定程度证实了基序随机分布的推论。Zhu 等[21]在结核杆菌的甲基化基序分布趋势中发现相似规律。
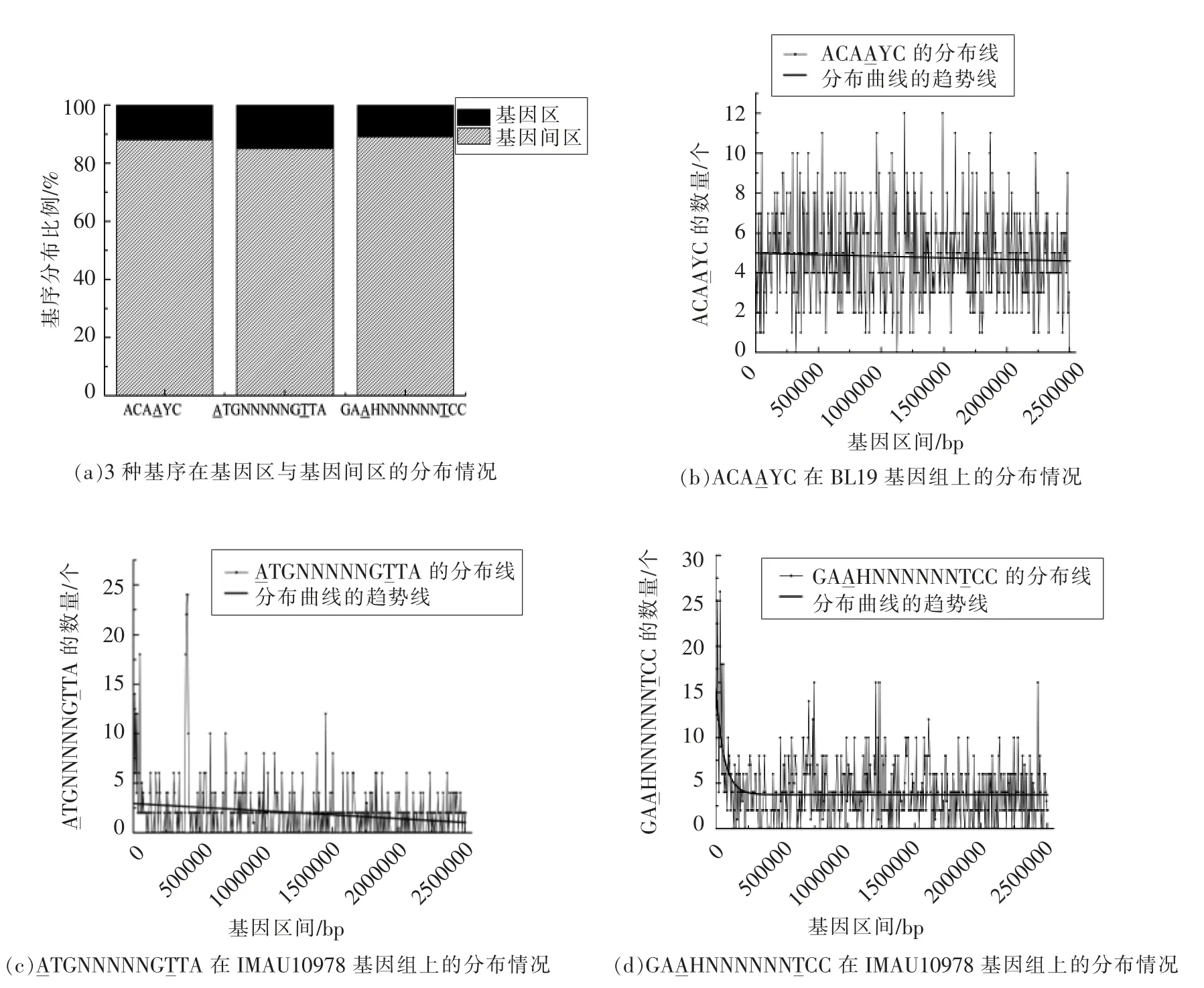
图4 3 种基序的分布Fig.4 Distribution of three motifs
2.3.3m6A 修饰基因的注释对分别含有3 种m6A修饰基序的基因进行功能注释。由图5可知,3 种基序中m6A 修饰位点的分布规律存在差异。BL19特有基序ACAAYC 主要分布于可移动元件蛋白(Mobile element protein)、复制蛋白(Replication protein)、噬菌体蛋白(Phage protein)、DNA 原酶(DNA primase,EC:2.7.7.-)等功能基因上。而IMAU11978 的基序ATGNNNNNGTTA 和GAAHNNNNNNTCC 则主要分布于流动元素蛋白(Mobile element protein)、位点特异性重组酶/DNA 转化酶相关蛋白(Site-specific recombinase DNA invertase Pin related protein)、精氨酸/鸟氨酸反转运子ArcD(Arginine/ornithine antiporter ArcD)、PTS 系统甘露醇特异性IIC 组分(PTS system mannitol-specific IIC component)与非特征化MFS 型转运体(Uncharacterized MFS-type transporter)等功能基因上。
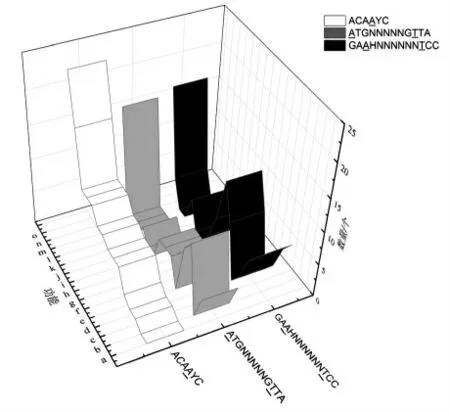
图5 3 种特有基序的功能注释结果Fig.5 Functional annotations of three specific motifs
与BL19 相比,IMAU11978 的基因组上含有较多m6A 修饰的肽转运载体基因,这可能是2 株菌蛋白水解特性存在差异的一个原因。同时,2 株菌m6A 修饰基序在PTS 系统相关基因上的分布存在差异,IMAU11978 中m6A 修饰基因包括:甘露醇特异性IIC 组分(10 个)、细胞二糖特异性IIB 成分(Cellobiose-specific IIB component,EC:2.7.1.205,4 个)、海藻糖特异性IIA 成分(Trehalose-specific IIA component,EC:2.7.1.201,2 个)、纤维二糖特异性IIC 组分(Cellobiose-specific IIC component,1 个)与甘露醇特异性IIA 成分(Mannitolspecific IIA component,EC:2.7.1.197,1 个),共计18 个。BL19 中m6A 修饰基因有PTS 系统的甘露醇特异性IIC 组分(5 个)与乳糖特异性IIA 成分(Lactose-specific IIA component,EC:2.7.1.207,1个)。推测m6A 修饰位点在PTS 系统相关基因的差异分布可能导致了菌株能量代谢的差异[22]。
值得注意的是,BL19 中发生m6A 修饰基因有乙偶姻特异性的2,3-丁二醇脱氢酶(EC:1.1.1.4),推测这种酶可能在NAD+协助下,将2,3-丁二醇转化为乙偶姻。GC-MS(Gas Chromatography-Mass Spectrometer)检测显示BL19 含有丰富的2,3-丁二醇[10]。因此推测该基因的m6A 修饰可能导致其表达活性降低,致使BL19 的发酵乳中2,3-丁二醇得以积累。Ferrocino 等[23]也发现香肠微生物基因组中的2,3-丁二醇脱氢酶主导的通路与乙偶姻含量呈正相关。
此外,在2 株菌中发生m6A 修饰的基因都注释到了丰富的微量元素相关基因与限制修饰系统相关基因。IMAU11978 中HsdS 以及I 型限制修饰系统的限制子单元R(Restriction subunit R,EC:3.1.21.3)与特异性亚单位S(Specificity subunit S)的基因发生了m6A 修饰,BL19 含有I 型限制性修饰系统特异性亚单位S 的m6A 修饰基因。限制修饰系统(Restriction modification system)由甲基转移酶与限制性内切酶组成,其作用是保护细菌免受外源DNA 的侵染[24]。有研究发现结核杆菌与双歧杆菌等原核生物均包含丰富的限制修饰系统[7,21]。其中HsdS 是I 型DNA 甲基转移酶中负责识别特定DNA 序列的特异性亚基[25]。
综上所述,BL19 的ACAAYC 和IMAU11978的ATGNNNNNGTTA 和GAAHNNNNNNTCC 基序对功能基因的影响,包括:肽转运载体、PTS 系统与2,3-丁二醇脱氢酶等相关基因,可能是导致2株菌表型差异的重要原因。
3 结论
本研究对发酵特性存在明显差异的乳酸乳球菌乳脂亚种IMAU10978 与BL19 进行了基因组与甲基化组分析,基因组分析发现2 株菌的基因组相似性高,ANI 值高达98.24%。甲基化组分析结果显示IMAU10978 与BL19 的m6A 甲基化差异巨大。BL19 的基序以m6A 修饰的ACAAYC 为主,其甲基化比率高达99.06%,主要分布于可移动元件蛋白、复制蛋白和噬菌体蛋白等功能基因上。I MAU10978 的基序以m6A 修饰的AT GNNNNNGTTA 与GAAHNNNNNNTCC 为主,其甲基化比率均高于97.02%,主要分布于流动元素蛋白、位点特异性重组酶/DNA 转化酶相关蛋白、精氨酸/鸟氨酸反转运子ArcD 与PTS 系统甘露醇特异性IIC 组分等功能基因上。2 株菌的肽转运载体、PTS 系统与2,3-丁二醇脱氢酶相关基因的甲基化差异巨大。这可能是导致BL19 与IMAU10978 在发酵速度、蛋白水解与产生特殊香气物质等方面存在差异的原因。本研究展现了乳酸乳球菌乳脂亚种的基因组、m6A 甲基化特征及其功能,从遗传学和表观遗传学2 个角度探究了菌株表型产生差异可能的原因,为理解遗传表观修饰对乳酸乳球菌乳脂亚种的影响及优良发酵剂的筛选提供坚实的理论基础和数据支持。
