基于宏转录组学解析不同贮藏时期冷鲜滩羊肉微生物代谢过程
2021-06-04赵珺怡罗瑞明张赫宇苏春霞胡倩倩赵晓策
赵珺怡,杨 波,罗瑞明,*,张赫宇,苏春霞,胡倩倩,赵晓策
(1.宁夏大学农学院,宁夏 银川 750021;2.宁夏大学生命科学学院,宁夏 银川 750021)
冷鲜滩羊肉是生鲜滩羊肉的一种重要产品,在常规0~4 ℃冷藏过程中,冷鲜滩羊肉会被外源性腐败微生物[1-2]污染,从而导致肉的腐败变质,甚至爆发食源性疾病乃至食物中毒[3]。有研究表明,微生物的生长繁殖不仅取决于贮藏环境中氧气含量、温度高低,也与生长基质的pH值有关[4]。不同屠宰、分割工序[5]与不同包装形式[6]所含有的微生物有一定的差异。目前,针对冷鲜肉微生物的研究热点多集中于采用选择性培养基、聚合酶链式反应(polymerase chain reaction,PCR)技术、酶联免疫法等分析冷鲜肉的生物多样性,通过致腐微生物生长值与模型参数拟合,结合肉品质感官指标与理化指标,建立货架期预测模型等[7-11]。但对于冷鲜肉微生物与微环境之间的分子生态关系与群落结构动态演替的分子机制却鲜有报道。
随着后基因组时代的来临,组学技术在食品微生物领域也得到广泛关注。Dugat-Bony等[12]应用宏基因组和宏转录组学技术研究了由9 种微生物组成的奶酪表皮在4 周成熟期中的变化。通过对其转录本表达量的分析,发现氨基酸代谢与脂肪代谢途径是最显著的代谢途径,相关差异基因源于假丝地霉酵母、干酪棒杆菌和蜂房哈夫尼亚菌。Jeong等[13]运用宏转录组学技术研究了韩国泡菜发酵水中的微生物群落演替,发现发酵泡菜水中初期微生物群落中含有明串珠菌、乳酸菌、假单胞菌属和泛菌属,而发酵第3天直至发酵结束明串珠菌占主导地位。在冷鲜肉微生物领域,张赫宇等[14]采用高通量测序技术对不同冷藏阶段滩羊肉微生物的16S rRNA基因V3区进行测序,研究发现假单胞菌、肠杆菌、热死环丝菌和乳酸菌为冷藏过程中的优势细菌,而优势真菌主要为酵母菌。张铁华等[15]研究了冷鲜牛肉微生物菌相变化,结果表明在低温贮藏过程中,G-小杆菌系是导致无包装鲜牛肉腐败变质的主要表面菌系,而G+球菌系是导致真空包装鲜牛肉腐败变质的主要菌系。
宏转录组学兴起于宏基因组之后,以微生物群落的总RNA为研究对象[16],探索特定时期下群体全部基因组转录情况及转录规律。宏转录组学技术能从转录水平研究复杂微生物群落变化,能更好的挖掘潜在的新基因和差异表达基因功能,探究各微生物成员对整体环境的贡献以及微生物功能与功能之间相互影响调控的作用机制。
本研究以冷鲜滩羊肉表面微生物为研究对象,采用宏转录组学技术对0、4、8 d微生物基因组差异转录、代谢途径与3 个贮藏时期微生物群落演替的关系进行探究,通过比较3 个贮藏时期的变化推测微生物优势菌优势生长的原因,为冷鲜肉保鲜分子生态学理论构建打下基础,为冷鲜滩羊肉的贮藏加工提供理论指导。
1 材料与方法
1.1 材料与试剂
滩羊后腿肉(当天屠宰),取自宁夏盐池县大夏牧场食品有限公司(滩羊饲养期间不做任何生物性防疫工作,且定期进行清粪、消毒处理),托盘密封包装。
TRIzol试剂盒 美国ThermoFisher Scientific公司;GeneReadTM试剂盒 德国Qiagen公司;三磷酸脱氧核糖核苷酸(dNTPs)、fragmentation buffer、核糖核酸酶H、DNA聚合酶I 美国TaKaRa Bio公司;AMPure XP纯化磁珠 英国Beckman Coultier公司。
1.2 仪器与设备
SW-CJ-1G超净工作台 上海博讯实业公司;Sorvall Legend Micro 17常温离心机、Forma 994 -80 ℃超低温冰箱 美国Thermo公司;HiSeq高通量测序仪 中国Novogene公司;Tanon-2500凝胶成像系统 中国天能公司;vortex-genie2 G-560E旋涡混合仪 美国SI公司;Qubit 2.0荧光计 上海Life Technologies公司;ScanSpeed MiniVac Alpha冷冻离心机 美国Scan Speed公司;GeneAmp PCR system 9700普通PCR仪 美国Life公司;Implen GmbH纳米光度计 德国Schatzbogen公司。
1.3 方法
1.3.1 样品采集
选用宁夏盐池县大夏牧区的滩羊作为实验材料,在0~4 ℃条件下贮藏的冷鲜滩羊肉上采集样品。采集样品前,用体积分数70%乙醇溶液对冷冻收集管、镊子、刀等取样器进行消毒,干燥后密封于无菌密封袋。用消毒刀分别在贮藏时间为0、4 d和8 d共9 个滩羊腿(每个贮藏时间段取3 个羊腿)上进行刮擦,得到刮屑。采集的样本包括肌肉、结缔组织、血液和脂肪等。
在第0天时,快速刮取3 个羊腿上的同一位置获得3 个平行性样本(≥150 mg),分别放入3 个冷冻管并标记为C0_1、C0_2和C0_3;然后将其表面刮擦物含菌落、血液、肉汁液等放置在-80 ℃的液氮中冷冻1 h。冷鲜贮藏4 d的3 个羊腿与0 d取样相同位置以同样方式获得样本,分别标记为C4_1、C4_2、C4_3;冷鲜贮藏8 d的3 个羊腿也以同样的方式取样分别标记为C8_1、C8_2和C8_3。将所有样品放入冰箱(0~4 ℃)中冷藏。每组样本分别进行3 个生物学重复实验。
1.3.2 RNA的提取
将上述样本封装于灭菌容器,在无菌条件玻璃球振荡分散,制备蒸馏水悬浮液,质量浓度1 g/100 mL。取1.0×109(1 mL菌液OD600nm为1~1.5)的细菌培养物放入2 mL离心管中,室温12 000×g离心30 s,除去上清液。使用TRIzol试剂盒,按照试剂盒说明分别提取样本的总RNA,通过琼脂糖凝胶电泳检测RNA降解情况(图1)。确保RNA的纯度和浓度符合后续实验要求,即RNA完整值(RNA integrity number,RIN)大于等于8.0。
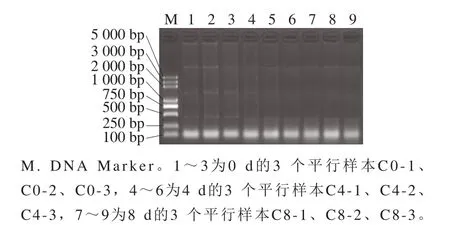
图1 琼脂糖凝胶电泳的总RNA质量分析Fig.1 Agarose gel electrophoresis for quality analysis of total bacterial RNA
1.3.3 cDNA文库的构建及Illumina HiSeq测序
使用评估污染物试剂盒对1.3.2节所得RNA进行评估,确定样品未发生污染后,使用GeneReadTM试剂盒除去rRNA。并加入碎片缓冲液使mRNA分解成小片段。
使用随机六聚体引物以上述mRNA为模板合成单链cDNA,使用TaKaRa试剂盒以单链cDNA为模板合成双链cDNA。使用AMPure XP纯化磁珠纯化双链cDNA后,加入A尾,修复末端。通过PCR扩增修饰的双链cDNA,用AMPure XP磁珠浓缩回收PCR产物,构建基因文库。
使用Qubit2.0荧光计初步定量每个文库。将每个文库质量浓度稀释至2 ng/μL。使用Q-PCR方法确定文库的有效浓度(确定为>2 nmol/L)。
使用Agilent Bioanalyzer 2100系统检测并确定DNA插入物的大小是否符合预期。在Illumina HiSeq平台进行高通量测序。
1.3.4 数据预处理
测序产生的原始数据存在一定比例低质量数据,为了保证后续分析的结果准确可靠,首先对原始的测序数据进行预处理,获取用于后续分析的有效数据。预处理方法参见方法。处理步骤如下:1)去除质量值≤5的碱基数达到一定比例的reads(默认reads长度的40%,设置为40);2)去除含N的碱基数目达到一定比例的reads(默认reads长度的10%,设置为10);3)去除Adapter污染(默认Adapter序列与reads序列有15 bp的overlap,设置为15);4)在有宿主污染可能性的前提下,需与宿主数据库进行比对,过滤掉可能是宿主污染的reads(默认设置比对一致性大于等于90%的reads为宿主污染)。
1.3.5 De novo组装
针对每个样品经预处理得到的clean reads,先使用NCBI的rRNA、tRNA以及SILVA数据库进行比对分离出来宏基因组中rRNA序列,剩下的mRNA序列则使用拼接软件Trinity(version: r20140413p1)分别进行从头组装,然后对所有样品的序列整合并使用CD-HIT-EST去冗余(设定序列一致性阈值为0.95),得到unigene集合。
1.3.6 生物信息学分析
将得到的unigene集合进行一系列差异表达基因分析:差异基因的基因本体(gene ontology,GO)注释、差异基因京都基因与基因组百科全书(Kyoto Encyclopedia of Genes and Genomes,KEGG)注释、GO显著性富集分析及KEGG显著性富集分析。
2 结果与分析
2.1 数据预处理结果
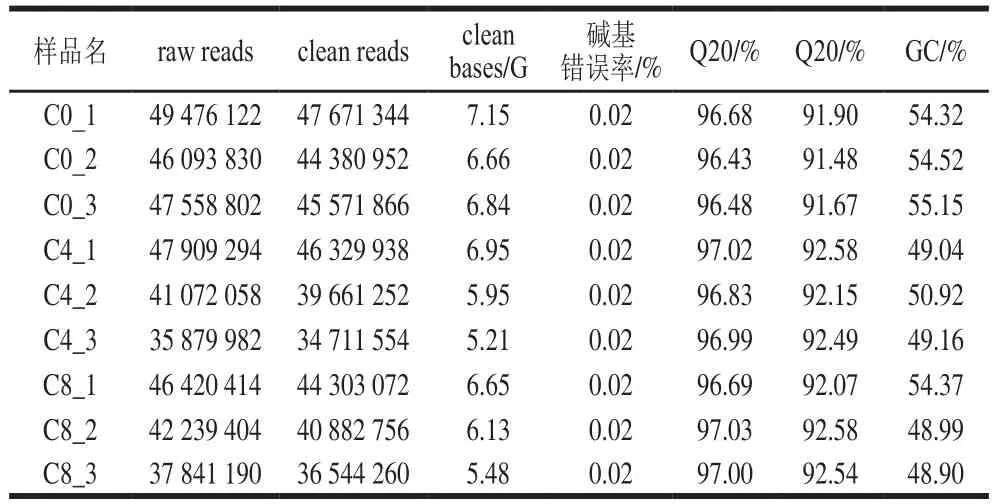
表1 数据预处理统计Table 1 Preprocessing results of metatranscriptomic data
限定序列质量后,共获得有效数据57.02 GB。由表1可知,每个样品的Q30碱基的占比不小于91.48%,此结果表明序列数据可靠,可用于下一步分析。此外,整个基因组的GC含量(鸟嘌呤和胞嘧啶的比例)在48.9%到55.15%之间,这表明在基因组测序过程中,覆盖的整个区域没有偏差,保证了后续拼接和注释的可靠性。
2.2 De novo组装结果
单基因长度区间分布在301~500 bp之间,单基因总数达77 709 个,占所有单基因长度区间的60.25%。统计结果如图2所示,4 746 个单基因长度大于2 000 bp,占所有单基因长度区间的0.04%,平均长度643 bp,连续N50长度为672 bp。164 836 个转录序列来自序列重叠组,平均长度733 bp,连续N50长度为871 bp。单基因长度主要分布在300~2 000 bp之间,占所有单基因长度区间的99.94%,8 829 个基因长度大于2 000 bp,占所有单基因长度区间的0.06%。总之,单基因数目丰富,转录长度分布合理,可以满足后续实验的需要。

图2 单基因序列和转录序列长度分布图Fig.2 Unigene and transcript length distribution
2.3 物种注释结果
通过与Nr(NCBI non-redundant protein sequences)库进行BLAST比对(e-value≤10-5),由于每一条序列可能会有多个比对结果,得到多个不同的分类级别,为了保证其生物意义,采取LCA算法(应用MEGAN软件的系统分类)[17],将出现第一个分支前的分类级别,作为该序列的物种注释信息。从门、属和种水平上的相对丰度表出发,选取出在各样品中的最大相对丰度排名前10的门类,并将其余的物种设置为其他,绘制出各样品对应的物种注释结果在其水平的统计图,如图3所示。

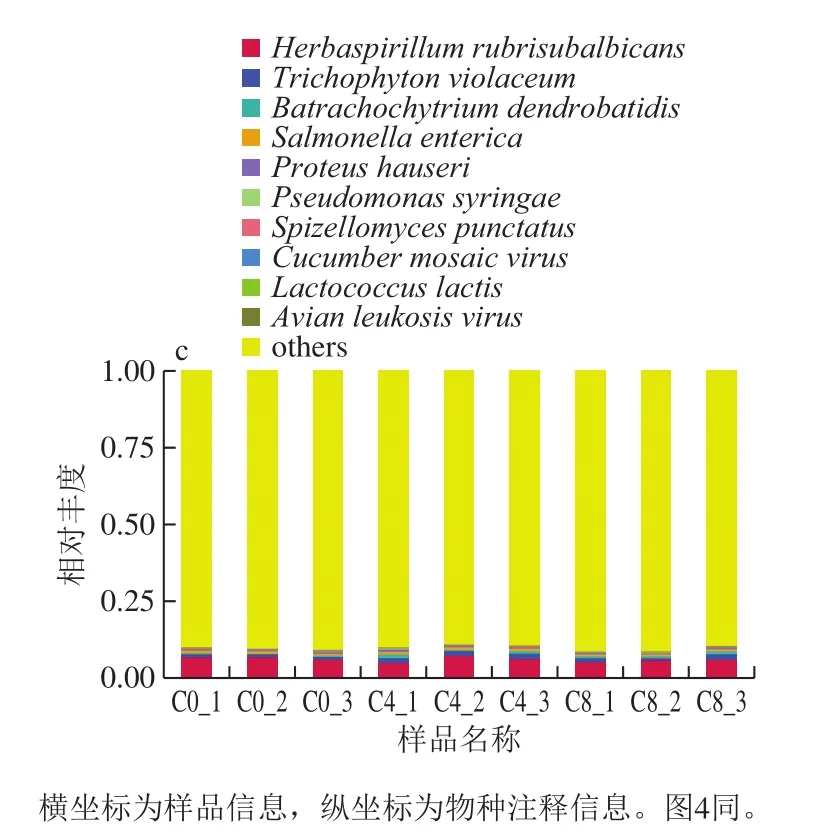
图3 物种注释在门(a)、属(b)和种(c)水平的结果Fig.3 Results of species annotation at the levels of phylum (a),genus (b) and species (c)
根据所有样品在各水平的物种注释及丰度信息,选取丰度排名前35 名的属及其在每个样品中的丰度信息绘制热图,并从分类信息和样品间差异两个层面进行聚类,找出研究样品中聚集较多的物种或样品,结果展示见图4。
由图3与图4所知,Proteus hauseri(变形杆菌)、Macrococcus caseolyticus(巨大球菌属)、Pseudomonas(假单胞菌)、Aspergillus flavus(黄曲霉)、Terrabacter(肿大地杆菌属)、Aeromonas hydrophila(嗜水气单胞菌)在C4_1样品中富集,C4_3样品中的优势菌株是Trichodesmium erythraeum(红色毛癣菌)。C8_1样品中的优势菌是Lactococcus lactis(乳酸菌),而在C8_2样品中占主导地位是Gammaproteobacteria bacterium(伽马蛋白细菌)和L. lactis。在4 d的样品中发现了3 组在不同样品中具有相似丰度变化的生物,第1组是P. hauseri和M. caseolyticus;第2组是A. flavus;第3组是Terrabactersp.28和A. hydrophila(嗜水气单胞菌)。由此可以看出,贮藏至第8天时,冷鲜滩滩羊肉的主要优势微生物为变形菌门、乳酸菌属等。其中,变形菌门中的假单胞菌和肠杆菌是造成冷鲜肉腐败变质的主要微生物,这与已有研究结果一致[18-19]。
滩羊肉冷藏第8天的优势菌株为G. bacterium(变形菌)和L. lactis(乳酸菌)。在滩羊肉冷藏初期,革兰氏阴性细菌如P. hauseri、N. spumigena、M. caseolyticus、G. prolifera、A. flavus、A. phagocytophilum、Terrabactersp. 28、A. hydrophila、G. bacterium占据主导地位。


图4 物种丰度聚类图在门(a)、属(b)和种(c)水平的结果Fig.4 Clustering maps showing species abundance at the levels of phylum (a), genus (b) and species (c)
假单胞菌是好氧微生物,生长几乎不受其他微生物影响[20]。在滩羊肉的冷藏过程中,假单胞菌优先利用贮藏环境中的氧气,当氧气消耗殆尽时,滩羊肉成熟过程中产生的葡萄糖成为其生长所需的能量来源[21]。假单胞菌在繁殖和新陈代谢的过程中会产生具有特殊气味的硫化物,酯、酸和其他变质代谢产物[22]会导致滩羊肉的腐败变质。与假单胞菌相比,肠杆菌的增长速度较缓慢。肠杆菌为兼性厌氧菌,可以发酵葡萄糖并产酸产气。在冷藏环境下,滩羊肉中的葡萄糖与中间代谢产物6-磷酸葡萄糖作为碳源被肠杆菌利用,产生胺类代谢物质导致肉的腐败[23-24]。随着贮藏时间的延长,滩羊肉细胞逐渐衰老死亡,可提供的能量和氧气逐渐变少,革兰氏阳性菌特别是乳酸菌(L. lactis)逐渐成为优势微生物。乳酸菌分泌一种名为nisin的抗菌肽,该物质能与脂II分子结合在革兰氏阴性菌细胞膜上形成孔洞,致其细胞死亡[25],从而确保自己的优势地位。
2.4 差异基因的筛选结果
通过对0~4、4~8、0~8 d的冷鲜滩羊肉中微生物群落的基因表达情况检测,采用基于负二项分布的DESeq[26]进行分析。统计显著性(signature,S)以q-value小于0.005和|log2(fold change)|大于1为筛选条件判断基因是否为差异基因[27],TRUE:为差异基因,FALSE意为不是差异基因。将筛选出来的差异表达基因用火山图展示。横坐标代表基因在不同实验组中表达倍数的变化;纵坐标代表基因表达量变化的统计学显著程度,q-value越小,-lg(q-value)越大,则差异越显著,结果如图5所示。
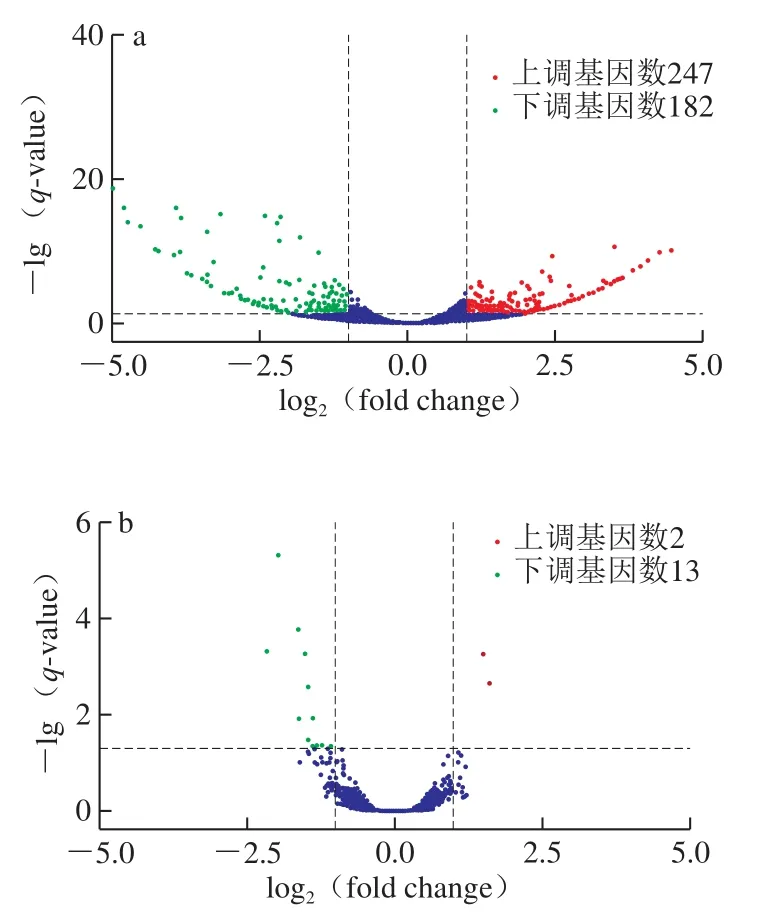

图5 差异表达基因的火山图Fig.5 Volcano plots of differentially expressed genes
筛选结果显示,从贮藏时间为4 d与0 d、8 d与4 d、8 d与0 d的冷鲜滩羊肉中测得的差异表达基因数分别为429、15、1 529 个。在C4与C0中,上调基因为247 个,下调基因为182 个;在C8与C4中,上调基因为2 个,下调基因为13 个;在C8与C0中,上调基因为727 个,下调基因为802 个。上调基因分别占基因总数的42.42%、13.33%和47.54%。下调基因分别占基因总数的57.58%、86.67%和52.46%。
2.5 差异基因的GO富集分析

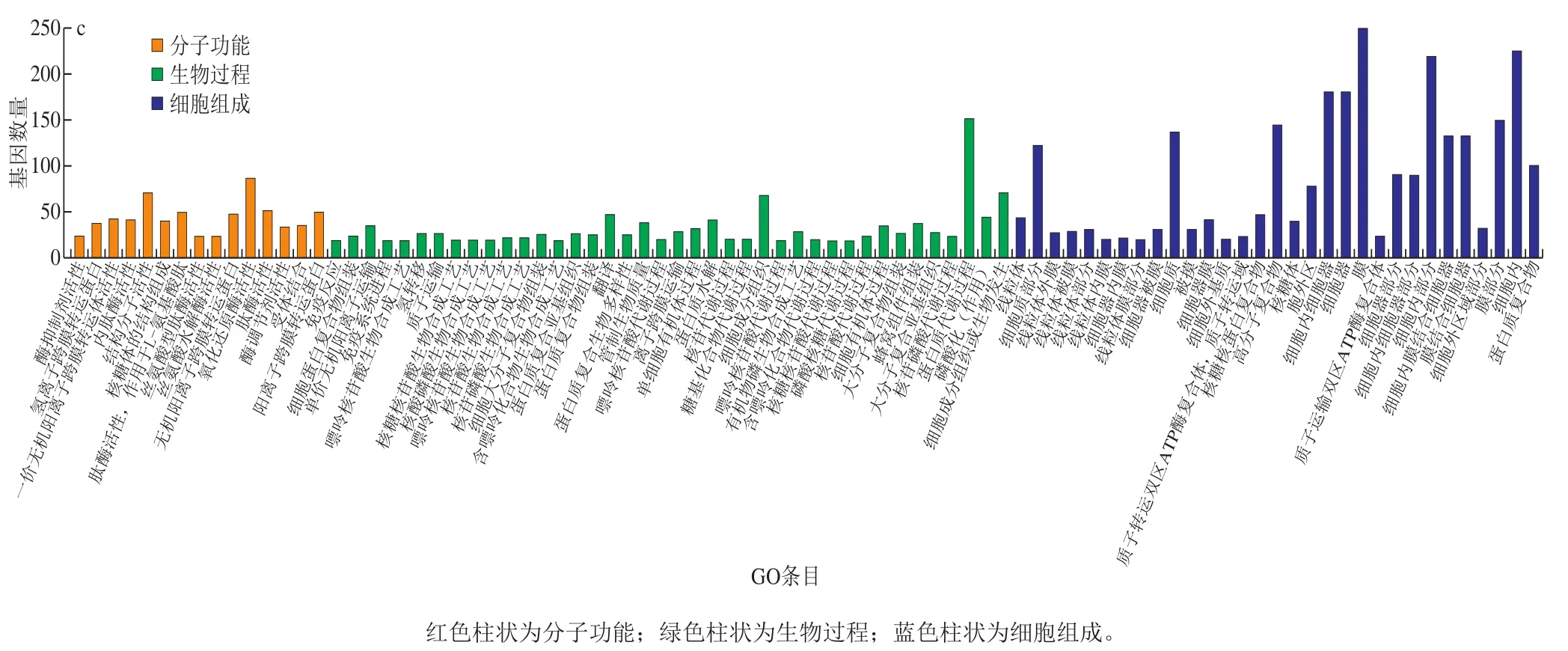
图6 差异基因GO显著富集图Fig.6 Significantly enriched GO terms of differentially expressed genes
采用非中央超几何分布的GOseq[28]方法将P<0.05作为阈值筛选GO条目,如图6所示,具体地显示了拥有较多基因富集的GO富集分析的条目。其中,在C0与C4对比组内的60 个分子功能、74 个生物过程和27 个细胞组成分别于329、838、277 个unigenes有关;在C4与C8对比组内8 个分子功能、35 个生物过程和17 个细胞组成分别与10、55、18 个unigenes有关;在C0与C8对比组内的70 个分子功能、121 个生物过程和57 个细胞组成分别与981、1 811、2 860 个unigenes相关。除去细胞结构的差异基因,C0与C4对比组,富集差异基因较多的分子功能是受体、蛋白质、GTP和核苷酸的结合,肽酶的活性以及结构分子的活性;富集差异基因较多的生物过程有信号转导、蛋白质聚合与水解和光合作用等;C4与C8对比组,富集差异基因较多的生物过程有离子转运和线粒体转运;C0与C8对比组中,富集差异基因较多的分子功能是不同氨基酸酶的活性,富集差异基因较多的生物过程主要是核苷酸代谢、蛋白质水解与组装、蛋白质代谢、磷酸化作用和离子转运等。
2.6 差异表达基因的EggNOG/COG注释
通过EggNOG分析将25 类unigenes簇进行分类,如图7所示。其中包含最多基因的功能基因簇分别为R—一般功能预测(8 150 个匹配基因)、S—功能未知(7 900 个基因)和T—信号转导机制(6 000 个基因)。此外,同源基因簇内的基因具有相似的结构和生物学功能,被认为来自同一祖先。而E—氨基酸转运和代谢、F—核苷酸转运和代谢、G—碳水化合物的运输和代谢、H—辅酶运输和代谢与I—脂质运输和代谢也占有一定份额,这与GO富集分析得到的结果较为一致。

图7 差异基因单基因EggNOG 注释结果的统计图Fig.7 EggNOG annotation results of differentially expressed unigenes
2.7 差异基因的KEGG富集分析
在生物体内,不同基因相互协调行使其生物学功能,通过Pathway显著性富集能确定差异表达基因参与的最主要生化代谢途径和信号转导途径。KEGG是有关Pathway的主要公共数据库[29]。将生物代谢通路划分为6 类,分别为:细胞过程、环境信息处理、遗传信息处理、人类疾病、新陈代谢、生物体系统,将3 个不同贮藏时期的微生物所富集的差异基因进行KEGG注释,如图8所示。
由图8可知,富集基因较多的代谢通路有核苷酸代谢、脂质代谢、能量代谢、碳水化合物代谢以及氨基酸代谢等。作为微生物的基础代谢,微生物普遍存在的核苷酸水解酶对核苷酸代谢分解具有促进作用。核苷酸水解酶先将核苷酸水解,生成核苷,再通过酶的催化作用下生成碱基和磷酸核糖[30],磷酸核糖参与磷酸戊糖途径生成6-磷酸果糖并且参与糖酵解,最终产物丙酮酸进入三羧酸循环途径进行反应,将能量代谢与物质代谢联系在一起,增加了微生物可利用的碳源,增强了微生物的增殖能力。

图8 不同贮藏时期差异基因表达的KEGG Pathway富集结果Fig.8 KEGG pathway enrichment analysis of differentially expressed genes at different chilled storage period
氨基酸代谢在微生物演替中的作用主要包括2 个方面,一方面主要用于合成自身独特的蛋白质、多肽和其他含氮物质,以产生相应的氮源为微生物利用;另一方面可通过脱氨、转氨、联合脱氨的方式分解为α-酮酸、胺类及二氧化碳[31]。其中,α-酮酸可通过胺化反应转化为糖、脂质或非必需氨基酸,也可参与三羧酸循环完全氧化成二氧化碳和水,并释放出大量能量[32]。乳酸菌以游离氨基酸作为底物,通过氨基酸的脱羧反应产生相应的碱性生物胺,维持自身的生长繁殖[33]。且氨基酸代谢产生的大量二氧化碳会降低贮藏环境的氧气浓度,正好满足了兼性厌氧的乳酸菌的生长条件,使得乳酸菌成为冷鲜滩羊肉贮藏期内的优势微生物之一。
能量代谢属于新陈代谢的一种,碳水化合物代谢也是能量代谢的过程。碳水化合物代谢过程中会形成磷酸盐化合物,在连接磷酸盐基团和其他分子的化学键中存储大量能量[34]。随着能量代谢与碳水化合物代谢的显著富集表明冷鲜滩羊肉表面微生物的代谢活跃,贮藏后期的微生物种类和数量相比于原始菌相产生了较大的增长,与能量代谢有关的差异基因表达富集较为显著。经过不同时间的冷鲜贮藏,滩羊肉表面微生物也呈现出一定的差异,但是就总体3 个贮藏时间段而言,因为原始菌相存在的基础使微生物群落结构的相似性更大,差异性则相对较小,而差异性主要是由于冷鲜肉微生物的生长代谢导致的。
在0~4 ℃的贮藏条件下,微生物的生命活动显著降低,一些嗜冷菌可以正常增殖,假单胞菌和肠杆菌都可以在这个温度条件下进行生长繁殖。宰前活体滩羊肉的pH值为中性,但在宰后成熟阶段,滩羊肉肌肉组织中的糖原在糖酵解酶作用下生成乳酸。同时,呼吸作用并没有停止,产生了大量能量和磷酸,在乳酸和磷酸的共同作用下,滩羊肉的pH值下降,造成胴体僵硬。此时钙激活酶作用于僵硬的胴体使其自溶,pH值降低至5.6~6.0[35],这种酸性环境最适合乳酸菌的生长。在贮藏过程中,冷鲜滩羊肉中的脂肪成分被附着在表面的优势微生物脂肪酶催化水解成脂肪酸和甘油[36],由甘油氧化形成的三碳醇酸是丝氨酸降解的中间产物,经磷酸化后生成3-磷酸甘油酯,可进一步参与糖异生或糖酵解反应。1-磷酸葡萄糖是糖异生途径产物,在储藏前期,假单胞菌和肠杆菌先利用氧气进行生长繁殖,在贮藏后期时,假单胞菌和肠杆菌则利用之前反应产生的葡萄糖为底物继续繁殖生长,成为主要优势微生物。而在贮藏中期丰度较高的不动杆菌属为专性需氧菌,在氧气含量较少的贮藏后期大幅减少;微小杆菌属和放线菌属为非嗜冷菌,在低温贮藏环境下的生长繁殖相对收到抑制,且乳酸菌在生长代谢中产生的乳酸也会抑制这些微生物的生长繁殖,使它们随着贮藏时间的延长,逐渐变为劣势微生物。
3 结 论
为系统揭示冷鲜肉微生物群落的变化,本研究利用宏转录组学技术对0、4、8 d三个冷鲜贮藏时间段滩羊肉表面微生物群落的结构进行了解析,共获得57.02 GB的转录本有效数据。研究表明在3 个贮藏时期中,C4与C0组的上调基因为247 个,下调基因为182 个;C8与C4组的上调基因为2 个,下调基因为13 个;而在C8与C0组中,上调基因和下调基因分别为727 个和802 个。这些差异表达基因主要富集在核苷酸代谢、脂质代谢、能量代谢、碳水化合物代谢以及氨基酸代谢等代谢途径。通过利用这些代谢途径所产生的代谢物和能量作为微生物生长繁殖的底物,假单胞菌和肠杆菌成为贮藏期间冷鲜滩羊肉表面的优势微生物。在贮藏后期,由于贮藏环境中残存的少量氧气与滩羊肉较低的pH值,乳酸菌丰度涨幅较大,也成为优势微生物之一。本研究对贮藏过程中的冷鲜滩羊肉微生物进行分析,可为冷鲜滩羊肉贮藏期间微生物及肉品质量预报、控制技术研发提供理论参考。
