丝氨酸(S13和S14)残基侧链位置特性对模型蛋白Trp⁃cage折叠的影响
2021-04-29张孝春黄卓然徐成振张兴旺吴晓敏
张孝春,汪 萌,黄卓然,徐成振,张兴旺,张 强,吴晓敏
(1.淮北师范大学生命科学学院污染物敏感材料与环境修复安徽省重点实验室,淮北235000;2.伊犁师范大学生物与地理科学学院,伊宁835000;3.淮北师范大学计算机科学与技术学院,淮北235000)
蛋白质天然态结构的稳定形成是发挥其生物功能的生物物理基础,研究蛋白质残基间相互作用与其结构折叠形成的关系一直是结构生物学研究重点。蛋白质折叠、稳定以及功能的发挥均与蛋白质结构中关键氨基酸残基密切相关,而这些残基的缺失或突变替换等都会对蛋白质结构和功能产生显著破坏。如镰刀型贫血症就是由于血红蛋白(HbA)结构中β 链第6 位氨基酸谷氨酸突变为缬氨酸形成了异常的血红蛋白S(HbS)[1]。p53 是突变频率最高的一种肿瘤抑制蛋白,其结构中第220 位酪氨酸残基突变为半胱氨酸会引起结构亲水性空腔变大,最终导致p53 蛋白变性且抑癌功能丧失[2]。因此,研究蛋白质结构中哪些残基在其折叠和功能中发挥重要作用对多肽设计、蛋白质结构优化、药物靶点预测等研究都具有重要的指导意义。残基点突变被看作是探测所突变残基在其结构中是否发挥重要作用的最直接有效方法[3],但是通过常规实验突变来构建突变体,一方面工作量大成本高,另一方面观察到的蛋白结构折叠转变时间和空间分辨率均受到限制。分子动力学(Molecular dynamics,MD)模拟计算随着近年来计算机软硬件的迅速发展,模拟计算的精度和时间尺度都迅速提升,已成为研究蛋白结构折叠和药物筛选设计的重要工具[4]。通过MD 模拟可为常规实验提供理论指导,大大降低实验的工作量,提高实验突变的准确率。在蛋白质受体⁃配体的模拟计算中,可通过结合自由能分解确定残基是否在结合过程中发挥重要作用[5]。但在蛋白质结构中,常用的方法是对残基所形成的不同类型相互作用(如疏水、静电、氢键以及盐桥等)和残基特性(如电荷、疏水和极性等)进行综合评判,再通过MD模拟与实验进行验证。然而残基侧链在结构中的位置取向并未被足够重视。
模型蛋白Trp⁃cage 是在模仿肠降血糖素药物艾塞那肽Exenatide 的基础上人工设计提出的[3],该模型蛋白凭借仅由20 个残基组成的典型结构和非常快的折叠速度成为验证蛋白质结构相关问题的理想模型[6⁃7]。目前对该蛋白模拟计算所采用的力场涵盖了现有的常用力场如OPLSAA/L[8]、AMBER 系列[9]、CHARMM 系列[10]以及近年来相关课题组优化开发的各种分子力场等[11]。尽管能够很好地利用这些分子力场研究蛋白质结构,然而经验势函数对原子间非键相互作用的描述精度还不够高,尤其是对相互作用庞大复杂的蛋白质结构,随着原子相互作用数目增大而计算误差迅速积累,会导致模拟计算结果错误。为此吴云东课题组对蛋白质数据库(PDB)中不在二级结构内部的卷曲残基进行统计分析,得到的残基固有局部构象偏好与常用力场OPLSAA/L 给出的残基侧链构象分布(骨架二面角φ、ψ 与侧链扭转角χi)存在明显差异。基于PDB 构建的局部构象偏好卷曲库(Coil library)[12],通过引入特殊局部非键相互作用对OPLSAA/L 力场优化得到新的RSFF1 力场[13]。该力场可得到与PDB 统计相一致的φ,ψ,χi 分布,且MD 模拟得到的结构与PDB结构偏差(RMSD)更小[13⁃14]。
因此,本文利用更为精确描述侧链位置特性的RSFF1力场对野生态模型蛋白及其两个丝氨酸突变体研究其各自折叠过程,并揭示该残基侧链位置特性对其蛋白结构折叠的影响,进一步为蛋白结构改造和优化设计提供理论指导。使用全原子MD 模拟,以模型蛋白Trp⁃cage 为例,首先研究其野生态结构从变性态构象到其天然态构象的折叠转变过程,并将其作为对照。在残基突变点的选择方面并未选取早期研究中[15]已证明对其结构稳定形成具有重要影响的残基如Y3、W6、P12 和P19 等或是形成盐桥的残基D9/R16,而是选择310⁃螺旋结构上的两个相邻的丝氨酸残基(S13和S14),其中残基S13侧链朝向外侧溶剂,而残基S14 侧链朝向蛋白内部疏水核心结构,这为研究残基的位置取向与其折叠之间的关系提供了重要基础。分别对该蛋白结构上残基S13 和S14 进行甘氨酸点突变,构建了S13G 和S14G 突变体,并采用与野生态MD模拟一致的计算参数对其进行折叠模拟,通过对比这两个突变体的折叠模拟过程与野生态之间的差异,研究残基S13和S14分别在结构折叠过程中的贡献差异,进而深入理解残基侧链位置特性对其结构折叠转变的影响。
1 模拟计算体系与方法
1.1 模拟计算体系
模型蛋白Trp⁃cage(TC5b)的晶体结构来自蛋白质数据库(https://www.rcsb.org/),PDB ID 为1L2Y,其氨基酸序列为NLYIQ WLKDG GPSSG RPPPS[16]。将该晶体结构中的第一帧作为其天然态构象,如图1(a)所示,并在之前的工作中[17]通过温控分子动力学(TC⁃MD)模拟获得了该蛋白的变性态结构,如图1(b)所示,该结构呈错误塌缩状。以此变性态结构作为野生态Trp⁃cage 的模拟起始构象,并对变性态结构中的残基S13 和S14 分别进行甘氨酸点突变优化获得突变体S13G 和S14G 的起始构象具体如图1(c)和图1(d)。为更好地模拟蛋白质在细胞正常生理环境条件,将构建好的起始结构分别置于长约43 Å 的正方体盒子中,并向其中填充约2 545 个TIP4p⁃Ew 溶剂水分子[18],为确保体系的电中性,使用2 个Na+和3 个Cl-共替换5 个水分子获得0.05 mol/L 的离子浓度,模拟体系环境pH 设置为7。体系构建完成后,分别进行2 ns 的最陡下降(Steepest decent,SD)能量最小化和5 ns 的位置限制MD 模拟来消除蛋白质结构中的不良接触并优化整个体系。
1.2 分子动力学(MD)模拟参数
野生态Trp⁃cage 及其S13G 和S14G 突变体1.0⁃μs的MD 模拟均使用GROMACS 2016 软件包[19]完成,模拟过程中采用RSFF1 力场和TIP4p⁃Ew 水模型,并使用V⁃rescale 和Berendsen 算法分别耦合温度和压力为300 K 和1.0 atm;LINCS 算法约束所有含有氢原子的共价键;PME(Properties⁃method⁃event)方法计算长程静电相互作用;库仑力和范德华相互作用的截断半径均设置为9.0 Å。每个体系的模拟时长均为1 000 ns,每500 ps保存一次构象。
1.3 实验轨迹数据分析
使用GROMACS 2016 和VMD 1.9.3 软件对所有MD模拟轨迹进行分析,并通过骨架结构的均方根偏差(RMSD_BD);α⁃螺旋的均方根偏差(RMSD_α⁃helix);310⁃螺旋的均方根偏差(RMSD_310⁃helix);骨架结构的回旋半径(Rg);疏水核心的回旋半径(Rgcore);天然接触的形成概率(Q)和Cα原子的均方根波动(RMSF)等参数来描述模型蛋白Trp⁃cage及其突变体的结构动力学特征。均方根偏差和天然接触的计算都是以NMR实验测定得到的天然态结构为基准计算。并通过gmx do_dssp命令调用DSSP程序计算模拟过程中二级结构的形成情况。同时为更清晰地获得结构的折叠转变路径,使用RMSD和Q 两个参数构建了结构的自由能地形图。文中所有结构图像均采用开源的蛋白质图像显示软件Pymol 绘制。参数Q将通过以下公式[20]计算:

其中:i 和j 为至少间隔3 个残基的所有非氢原子对;天然态结构中i和j原子之间的接触距离(r0ij)<4.5 Å将被判定为形成天然接触;β设置为5.0,λ设置为1.4。
2 结果与讨论
2.1 残基S13和S14突变对其二级结构形成的影响

图1 野生态模型蛋白Trp⁃cage的天然态(a)和变性态起始结构(b)及其两个突变体S13G(c)和S14G(d)的变性态起始结构Figure 1 Schematic diagram of the natively folded state(a)and starting denatured state(b)of Trp⁃cage structure and its two starting denatured states of the S13G(c)and S14G(d)mutations

图2 野生态模型蛋白Trp⁃cage及其S13G和S14G突变体的α⁃螺旋(a)和310⁃螺旋(b)的RMSD随时间的变化Figure 2 The time⁃evolution RMSD curves of α⁃helix(a)and 310⁃helix(b)of Trp⁃cage and its S13G and S14G mutations as functions of simulation time
如图2(a)所示,野生态Trp⁃cage 及其S13G 和S14G 突变体的α⁃螺旋RMSD 值在模拟结束时都已达到平衡且分别稳定在3.2 Å、3.4 Å 和3.2 Å。但是α⁃螺旋的折叠稳定性在不同的结构体系中存在一定的差异,如:野生态与突变体S14G 的α⁃螺旋RMSD 曲线在模拟起始后快速上升后下降,然后保持小幅的波动,并分别在500 ns 和400 ns 后达到稳定;而变体S13G 的α⁃螺旋RMSD 曲线虽然在模拟的前200 ns 内一直处于频繁的波动中,但是在200 ns 后保持稳定。这些结果与DSSP 所绘制的二级结构保持一致(图3)。
模型蛋白Trp⁃cage 的另一个二级结构310⁃螺旋的RMSD 随时间的变化如图2(b)所示,野生态结构在模拟过程中一直稳定在2.4 Å 左右,结合其二级结构变化图表明其310⁃螺旋在结构中一直保持稳定。这同时也说明野生态Trp⁃cage 能够在模拟早期快速形成二级结构,这与先前的实验与模拟中发现二级结构在模拟早期快速形成保持一致[21⁃24]。突变体S13G的310⁃螺旋RMSD 值与其α⁃螺旋相似,均在模拟的前200 ns 出现增长,表明其二级结构处于解折叠状态。其310⁃螺旋结构在600 ns 后稳定在2.1 Å,但是图3(c)的DSSP 结果却显示其310⁃螺旋结构并未形成,这与野生态结构的310⁃螺旋RMSD 值为2.4 Å 且DSSP 显示其310⁃螺旋结构形成出现矛盾。通过分析发现,突变体S13G 结构中组成310⁃螺旋的残基(G10⁃S14)中同时存在两个限制螺旋形成的残基(P12 和G13),这两个残基的存在可能导致了组成310⁃螺旋结构残基的RMSD 虽然小于野生态的RMSD 但仍未被判定形成310⁃螺旋结构的现象。突变体S14G 的310⁃螺旋RMSD 曲线在模拟的前600 ns 内一直在2.1~3.9 Å 范围内波动,这意味着组成310⁃螺旋结构的残基构象一直处于频繁转换中。尽管其RMSD 值在600 ns 后达到稳定(3.6 Å),但是突变体S14G 结构中组成310⁃螺旋的残基未能形成,且与天然态构象相差较大,这与图3 的DSSP 结果相一致。通过上述结果发现该蛋白野生态的二级结构在模拟早期就可快速稳定形成,而突变体S13G 和S14G 的α⁃螺旋结构折叠稳定性发生不同程度下降;在310⁃螺旋结构的折叠形成中,残基S13 突变并未产生较高的影响,但是残基S14 突变将导致其结构无法正确形成,这说明残基S14 对于310⁃螺旋结构的形成至关重要。这主要是由于残基S14 的侧链朝向结构内部容易与其他残基发生相互作用,而残基S13 侧链朝向溶剂更容易与溶剂水分子发生作用而出现侧链摆动。相同侧链由于位置朝向和邻近空间特征的不同导致对于螺旋结构折叠稳定性贡献程度产生明显差异,这对于螺旋结构特征的多肽类抑制剂药物如细胞内雌激素受体多肽抑制剂[25]和β⁃淀粉样蛋白等多肽抑制剂[26]结构稳定性的优化提供重要参考。

图3 DSSP计算的野生态模型蛋白Trp⁃cage及其S13G和S14G突变体的二级结构随时间的变化Figure 3 The time⁃evolution secondary structure of Trp⁃cage and its S13G and S14G mutations obtained by DSSP calculation
2.2 残基S13和S14突变对其三级结构形成的影响
如图4 所示,在模拟结束前的350 ns 内野生态及其S13G 和S14G 突变体的Rg值分别约为6.8 Å、6.6 Å和6.5 Å,Rgcore值约为5.4 Å、5.8 Å 和5.9 Å,这表明所有模拟结构在模拟结束时都已完成折叠过程且体系平衡。与二级结构的变化类似,三级结构的折叠塌缩虽然相似,但是在折叠转变过程和最终形成的结构状态仍存在明显差异。其中突变体S13G 的Rg和Rgcore值在模拟的前400 ns 内小范围波动,并在214 ns 时出现一次峰值(Rg=14.1 Å,Rgcore=16.0 Å),此时二级和三级结构完全解折叠,通过对比该时刻前后的构象发现该解折叠事件是突变体S13G 折叠过程中由错误折叠态调整至折叠态结构的关键步骤。突变体S14G 的Rg和Rgcore值在模拟前600 ns 内的变化同其310⁃螺旋的RMSD 变化一致,均出现较大范围的波动。这说明突变体S14G 结构在折叠/解折叠之间不断频繁转换。虽然在模拟结束时突变体S14G 的Rg和Rgcore值与野生态相近,但是结合构象分析发现其三级结构仍处于错误塌缩状态。这表明了残基S14 所发生的氢键相互作用(S14⁃OH→O=C⁃D9 和R16⁃NH→OH⁃S14)的缺失也导致了蛋白整体结构折叠稳定性下降且结构不能正确折叠形成天然态构象。

图4 野生态模型蛋白Trp⁃cage及其S13G和S14G突变体的Rg和 随时间的变化Figure 4 The time⁃evolution Rg and Rg core curves of Trp⁃cage and its S13 and S14 mutations as functions of simulation time
2.3 残基S13和S14突变对其折叠过程的影响
野生态模型蛋白及其突变体S13G 和S14G 结构的骨架RMSD 和Q 随时间的变化如图5所示,野生态Trp⁃cage 的骨架RMSD 并未像其二级结构和三级结构在模拟早期达到稳定,而是在500 ns之后达到稳定,并且在模拟最后的100 ns 再次下降,结合构象分析发现此时主要为残基S14⁃R16 构象的细微调整。模拟过程中骨架RMSD 最小为0.67 Å(图6),这表明野生态Trp⁃cage结构最终折叠至天然态构象,同时也说明通过PDB 卷曲库优化后获得的RSFF1 力场能够很好地描述Trp⁃cage 的折叠形成,这与蒋帆等[11,13,20]的研究结果一致。但是野生态Trp⁃cage 的Q 值在模拟结束时仅为0.7 左右,这与天然态构象的1.0 仍有一定差距。通过结构分析发现,野生态Trp⁃cage 虽然在骨架上与天然态之间的差异非常小,但是在侧链上仍然存在一定的差异,特别是未参与疏水相互作用的残基侧链,如残基Q5和K8 等,它们的侧链朝向溶剂且在溶剂中不断摆动,这是Q 值较低的主要原因。突变体S13G 的骨架RMSD曲线变化同野生态类似,均在500 ns时稳定在1.9 Å左右,但是其在模拟后期并未能如野生态进一步折叠,且其RMSD 曲线也在214 ns 时出现峰值(10.18 Å),即此时结构解折叠,这与其Rg和Rgcore曲线一致。突变体S14G 的骨架RMSD 曲线同样与其二级结构和三级结构参数保持一致,在模拟的前600 ns 内处于剧烈波动中,之后稳定在3.8 Å 左右,表明在模拟结束时,突变体S14G 结构仍处于错误的折叠状态。且通过结构对比发现其310⁃螺旋和C⁃端残基与野生态构象存在较大差异。如图5(c)的RMSF 所示,突变体S14G 在整个折叠过程中结构转换幅度较大,明显高于野生态和突变体S13G。

图5 野生态模型蛋白Trp⁃cage及其S13G和S14G突变体的RMSD、Q和RMSF随时间的变化Figure 5 The time⁃evolution RMSD,Q and RMSF curves of Trp⁃cage and its S13 and S14 mutations as functions of simulation time
2.4 野生态Trp⁃cage 及其S13G 和S14G 突变体的折叠路径
通过结构序参数比较发现,通过均方根偏差(RMSD)和天然接触(Q)构建的自由能地形图能够准确描述其折叠形成转变路径[27-28]。如图7 所示,野生态的折叠由变性态(D)开始折叠至具有正确构象的第一个中间态(I1),尽管此时RMSD 有所下降,但Q 并未增长,此时二级结构基本形成,但是三级结构并未正确塌缩。直至折叠形成第二个中间态(I2),其疏水核心正确塌缩折叠,并且I2的构象与天然态非常相似,这种与天然态相似的中间态在之前的研究中也有所发现[22,24,29]。之后结构进一步调整至近天然态(N)构象。野生态Trp⁃cage 的二级结构率先形成,疏水核心再形成的折叠过程与先前研究中发现的扩散⁃碰撞模型相一致[21⁃24]。
突变体S13G 同样由变性态D 开始,与野生态不同之处在于其结构折叠过程中存在一个过渡态(Ts),这在其结构参数上均有所体现。该过渡态结构发生解折叠和水合作用,其疏水核的解折叠导致了Rgcore和Rg的增大,促进了残基侧链与溶剂水分子的相互作用,并引起了疏水残基的旋转和正确再折叠,经过Ts后,RMSD和Rg值分别急剧下降至2.5 Å 和6.9 Å,形成其中间态(I1),随后该结构又进一步调整至近天然态(N’)构象。尽管突变体S14G 的α⁃螺旋在结构折叠过程中较为稳定,但其310⁃螺旋和C⁃端结构始终未正确形成,这在各结构态的代表性构象中均有所体现,并且最终的错误折叠构象与天然态构象相差甚远,这表明残基S14 所参与形成的氢键作用对其在折叠过程中关键天然接触的形成发挥重要作用,尤其是310⁃螺旋结构的准确折叠,而310⁃螺旋结构的消失对于疏水核心结构残基L7和P12 的准确塌缩也产生一定影响,这也再次表明蛋白质二级结构与三级结构折叠形成的协同性[30]。在对Trp⁃cage(TC5b)的稳定性模拟计算中同样发现具有相同侧链的S13 和S14 由于其侧链所处位置的不同导致其对结构稳定性的贡献程度存在差异[15],同样也发现残基S14对模型蛋白的折叠影响明显高于残基S13,这主要是因为:侧链朝向结构内部的残基S14 与D9、R16 的侧链形成了较强的内部氢键(S14⁃OH→O=C⁃D9和R16⁃NH→OH⁃S14)和天然接触(S14⁃D9,S14⁃G10,S14⁃G11,S14⁃P12,S14⁃R16);而侧链朝向溶剂的残基S13 仅仅通过骨架的羰基氧与G10 的氨基氮原子和溶剂水分子发生氢键作用,且受到溶剂水分子的运动而波动,相比残基S14 对其结构稳定性和折叠转变影响要小。除了模拟计算之外,Andersen 课题组[3]通过圆二色谱(CD)和核磁共振实验(NMR)研究发现:该模型蛋白(TC10b)突变体S13A 增加了其结构折叠稳定性(折叠率为91%,野生态TC10b 的折叠率为90%),这说明处于蛋白结构表面的极性基团如丝氨酸S13 侧链上的羟基外置不利于结构的稳定;而模型蛋白(TC9b)突变体S14allo⁃T 的结构稳定性没有明显降低(折叠率为82%),S14T 的稳定性略有降低(折叠率为76%),说明同样侧链内置丝氨酸变为苏氨酸后,羟基朝向位置的调整对其结构折叠稳定性可产生一定影响,L⁃别⁃型苏氨酸略高于L⁃型苏氨酸。在TC10b 突变体S14A 结构中,S14 侧链的羟基被丙氨酸的甲基取代后导致该内置羟基所形成的氢键网络和天然接触全部消失,彻底破坏了该模型蛋白结构的稳定性,折叠率仅为44%。实验和计算结果都表明残基侧链结构化学特性的改变会彻底破坏其参与形成残基间相互作用网络,侧链位置朝向的不同也会引发其发生残基相互作用的改变。在基于蛋白⁃蛋白相互作用的药物设计中,除了研究残基侧链本身的电荷、疏水、极性等化学特性外,还要充分考虑到残基侧链所处邻近的空间特征和自身朝向位置特性,这为艾塞那肽药物(模型蛋白Trp⁃cage 的前体Exendine⁃4)的结构稳定性优化和多肽成药性的提高提供重要理论参考。
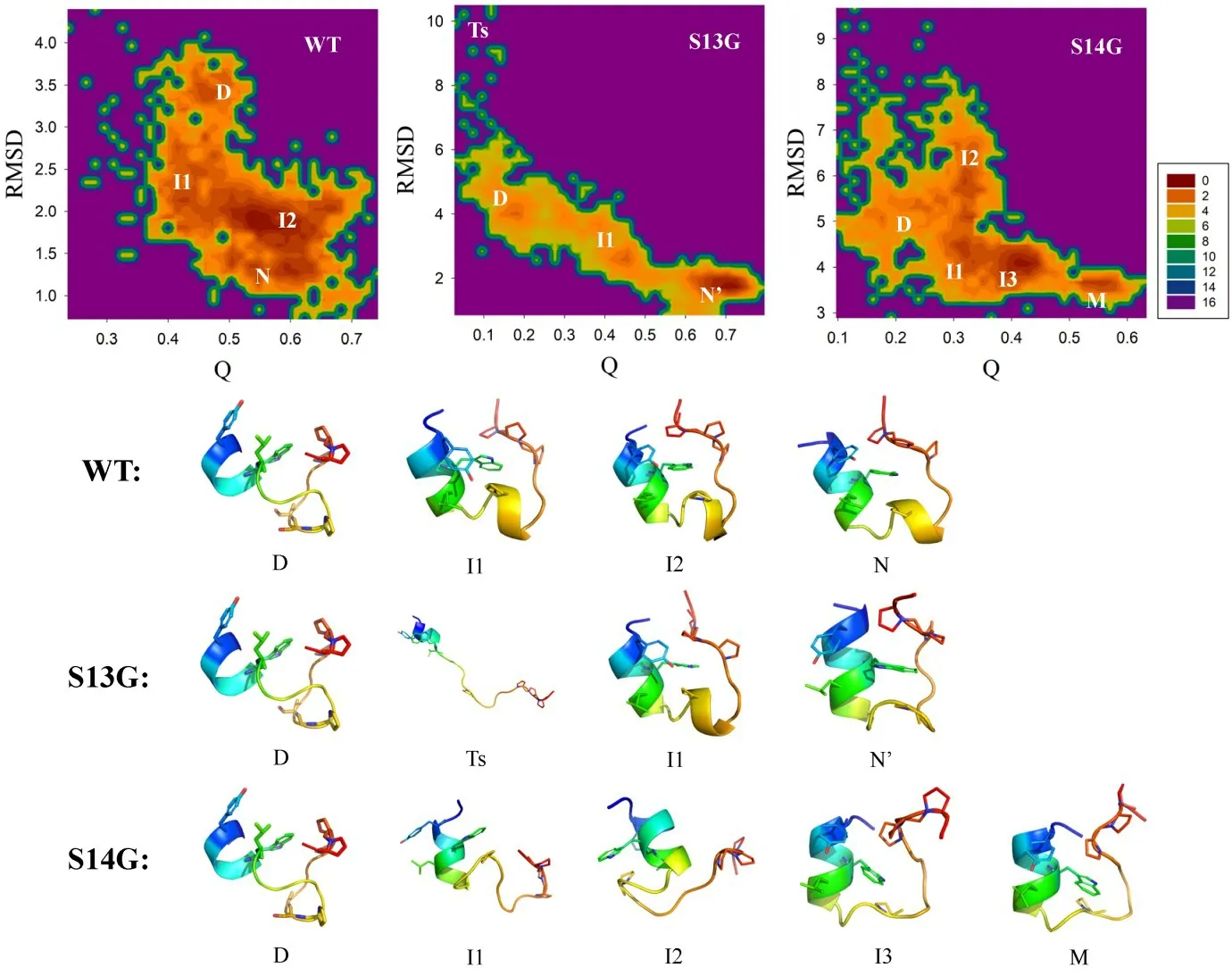
图7 野生态模型蛋白Trp⁃cage及其S13G和S14G突变体的自由能地形图和各结构态的代表性构象Figure 7 The free energy landscapes of Trp⁃cage and its S13 and S14 mutations and representative conformations corresponding to different states
3 结论
利用更为精确描述侧链位置特性的RSFF1力场使用全原子MD 模拟研究野生态模型蛋白Trp⁃cage 及其S13G 和S14G 突变体的折叠过程。结果表明,野生态Trp⁃cage 能够在模拟开始后快速形成二级和三级结构,并在结构折叠过程中形成了与天然态相似的中间态结构和近天然态构象,其结构折叠路径为D ⇌I1 ⇌I2 ⇌N,其折叠机制符合扩散⁃碰撞折叠模型。突变体S13G 的折叠路径为D ⇌Ts ⇌I1 ⇌N’,虽然这与野生态的折叠路径存在一定差异,但其N’结构RMSD比N高,这说明残基S13 突变仅对其结构折叠产生一定影响。与侧链朝向相反的残基S14 则不同,其突变体S14G 的折叠路径为D ⇌I1 ⇌I2 ⇌I3 ⇌M,由于其侧链朝向结构疏水核内部,并与D9 和R16 形成氢键相互作用。在突变体S14 中侧链的羟基被氢原子所替代,导致稳定螺旋二级结构的这些氢键受到破坏,因此该突变体的N端α⁃螺旋局部结构解旋松散、310⁃螺旋结构消失,二级结构的破坏也引发三级疏水核心结构的错误塌缩。
致谢:本模拟计算实验的完成是以北京大学吴云东院士课题组提供RSFF1 力场的参数文件为基础的,在此感谢吴云东院士课题组对实验的大力支持和论文撰写方面的指导。
