类茶氨酸合成酶基因家族在番茄中的鉴定及表达研究
2021-04-19毛鹏王丽鸳白培贤韦康阮丽张亚真成浩
毛鹏,王丽鸳,白培贤,韦康,阮丽,张亚真,成浩
类茶氨酸合成酶基因家族在番茄中的鉴定及表达研究
毛鹏1,2,王丽鸳1*,白培贤1,韦康1,阮丽1,张亚真1,成浩1*
1. 中国农业科学院茶叶研究所/国家茶树改良中心/农业部茶树生物学与资源利用重点实验室,浙江 杭州 310008; 2. 中国农业科学院研究生院,北京 100081
茶树体内的茶氨酸合成酶(Theanine synthetase synthetase,TS)和部分谷氨酰胺合成酶(Glutamine synthetase,GS)家族成员都具有催化乙胺和谷氨酸合成茶氨酸的活性。我们将植物中这类与茶树TS序列高度相似,且具有茶氨酸合成活性的酶,统称为类茶氨酸合成酶(Analogue theanine synthase,ATS)。为了探究ATS在非茶植物中的分布,以及乙胺在茶氨酸合成中的作用,本研究以Mico-Tom番茄为试验材料,对水培番茄幼苗进行了外源添加乙胺处理。结果发现,仅在处理组叶片中检测到茶氨酸,并且处理组叶片中的谷氨酰胺(Glutamine,Gln)含量也极显著增加,而丙氨酸含量与对照相比无显著变化。利用生物信息学方法分别从茶树和番茄基因组中鉴定出12个和5个家族成员,它们均含有谷氨酰胺合成酶催化功能的结构域Gln-synt_C;qRT-PCR检测发现,处理组叶片中有2个(和)表达量极显著提高。上述结果表明,ATS广泛存在于植物中,而乙胺是合成茶氨酸的关键限制因子,能够提高基因的表达,触发茶氨酸的合成。
番茄;茶氨酸;乙胺;类茶氨酸合成酶;基因家族
茶树()起源于中国西南地区,在全球60多个国家和地区有着高达700万公顷的种植面积,并与咖啡树、可可树一起列为世界三大非酒精饮料经济植物[1-2]。茶氨酸是茶树特征性非蛋白质游离氨基酸,在提升茶叶品质,增强茶汤鲜爽味,以及改善人体健康等方面发挥了重要作用[3-6]。在目前的文献报道中,含有茶氨酸的植物主要是茶树,其嫩芽和叶片中茶氨酸的含量达到干重的1%~4%,约占游离氨基酸总量的60%~70%[2]。此外,在山茶属的个别成员,以及茶梅、玉米和一些细菌中也发现有少量的茶氨酸[7-9]。
茶氨酸主要是在茶树根部由谷氨酸和乙胺在茶氨酸合成酶(Theanine synthetase,TS)的催化作用下合成[10]。TS在茶苗匀浆中被首次发现[11],但以往的研究未能对其进行有效的分离和纯化。目前已从茶树cDNA文库中克隆得到TS候选基因(DD410896)、(DD410895)和(TEA015198.1)等;其中,主要在根、芽和叶中表达,主要在芽中表达,而(TEA015198.1)则是在根部特异性表达[9,12-13]。属于Type Ⅰ GS,并与腐臭假单胞菌中的(BAE44186)基因同源,编码的FluG蛋白和都被证实具有合成茶氨酸的能力[9,14]。Deng等[13]对TS与谷氨酰胺合成酶(Glutamine synthetase,GS)进行核酸序列分析,发现与(AB117934)相似性达99%,与(AB115183)相似性达97%。茶氨酸的生物合成方式与谷氨酰胺合成的酶促反应类似[15],且上述反应的产物在结构上也很相似,都属于酰胺类似物(图1)。目前通过试验已证实茶树谷氨酰胺合成酶基因(MG778703)、(MG778704)、(MG778705)(MG778706)以及拟南芥谷氨酰胺合成酶基因均具有合成茶氨酸的功能[8,16]。此外,利用工程菌的方法也证实了微生物中的GS[17]、-谷氨酰甲胺合成酶(-glutamylmethylamide synthetase,GMAS)[18]、-谷氨酰基转肽酶(-glutamyltrans-peptidase,GGT)[19-20]和-谷氨酰半胱氨酸合成酶(-glutamylcsteine synthetase,-GCS)[21]均具有合成茶氨酸的能力。

图1 茶氨酸与谷氨酰胺的生物合成途径
基于上述研究背景,我们推测能够催化谷氨酸和乙胺合成茶氨酸的酶是一类同工酶。因此将这类与茶树TS基因序列高度相似,且具有茶氨酸合成能力的基因,统称为类茶氨酸合成酶(Analogue theanine synthase,ATS)基因。本研究选取了生长周期短、植株矮小、自身不含有乙胺和茶氨酸的Mico-Tom番茄作为试验材料[8,22],对水培番茄幼苗进行了外源添加乙胺的试验处理,以检测幼苗叶中是否存在ATS;随后,利用生物信息学方法,在茶树和番茄基因组中进行家族成员的鉴定,并对番茄中的几个潜在的关键进行表达分析,以此探究ATS在非茶植物中的存在情况,以及乙胺在茶氨酸合成中的作用。
1 材料与方法
1.1 材料与处理
从中国农业科学院茶叶研究所嵊州基地选取一年生龙井43扦插苗,清水冲洗干净,取根和叶放入冷冻干燥机(TF-FD-1,上海田枫实业有限公司)进行冷冻干燥。
将野生型Micro-Tom番茄种子(购自武汉伯远生物科技有限公司)播种在湿润的海绵上,2周后将萌发的幼苗移入装有1/2 Hoagland营养液的45 cm×30 cm×15 cm水槽,光照培养(白天25℃,夜晚18℃)。一周后选择长势一致的番茄幼苗,将其随机分为对照组和处理组。对照组和处理组均用Hoagland营养液培养,其中处理组中添加10 mmol·L-1盐酸乙胺。每5 d更换1次营养液,并且每天用增氧气泵(ACO-005,森森集团股份有限公司)向营养液中注入空气10 h。水培25 d后,分别采摘处理组和对照组中番茄成熟叶放入液氮中速冻,取一部分叶片放入冷冻干燥机中进行干燥备用,另一部分置于–80℃冰箱保存。
1.2 氨基酸和乙胺的提取及测定
用0.1 mol·L-1盐酸分别溶解茶氨酸、谷氨酸、丙氨酸、谷氨酰胺和乙胺标准品,配制成2.5 μmol·L-1的溶液。取茶氨酸溶液400 μL、谷氨酸溶液100 μL、丙氨酸溶液100 μL、谷氨酰胺溶液100 μL、乙胺溶液100 μL、超纯水200 μL于1.5 mL离心管中制成一个1 mL的混合标样。用分析天平称取经冷冻干燥后的茶树和番茄样品并研磨成粉末,其中茶树组织加入5 mL沸水,番茄组织加入3 mL沸水,在100℃的水浴中浸提30 min,期间晃动2~3次,4 000 r·min-1离心10 min后,上清液经0.22 µm过滤器过滤待用。
使用AccQ·Tag Ultra衍生试剂盒(Waters,MA,USA)进行氨基酸及乙胺的柱前衍生反应。将标准品溶液或样品溶液(混标溶液和茶树提取液为10 μL,番茄提取液为20 μL)与70 μL AccQ·Tag Ultra硼酸盐缓冲液混匀,再加入20 μL AccQ·Tag试剂并充分混匀,混合液转移至微量进样瓶中,置于55℃下反应10 min后,用于超高效液相色谱仪UPLC(Agilent,California,USA)检测。流动相组成:100% AccQ·Tag Ultra洗脱液A作为溶剂A,10% AccQ·Tag Ultra洗脱液B作为溶剂B,超纯水作为溶剂C,100% AccQ·Tag Ultra洗脱液B作为溶剂D。洗脱梯度为:0~0.29 min(10% A,90% C),0.29~5.49 min(9.9% A,90.1% C),5.49~7.10 min(9.0% A,80% B,11% C),7.10~7.69 min(8.0% A,15.6% B,57.9% C,18.5% D),7.69~7.99 min(7.8% A,70.9% C,21.3% D),7.99~8.68 min(4% A,36.3% C,59.7% D),8.68~10.20 min(10% A,90% C);设置二极管阵列检测器(Photo-diode array,PDA)的波长为260 nm[23]。
1.3 ATS家族成员鉴定及理化性质分析
从Tea Plant Information Archive(TPIA)(http://tpia.teaplant.org)[24]和Phytozome12(https://phytozome.jgi.doe.gov/pz/portal.html)中分别下载茶树及番茄的基因组、CDS序列和蛋白质序列文件。从NCBI中下载(DD410896)、(DD410895)、(MG778703.1)、(MG778704.1)、(MG778705.1)(MG778706)和(TEA015198.1)核酸序列并作为筛选茶树家族成员的标准基因,利用TBtool[25]中的BLASTP工具与NCBI中的UniProtKB/Swiss-Prot(swissprot)数据库,基于E值<10-5及注释信息去除中的冗余部分。随后将从茶树基因组中确定的家族成员作为筛选番茄家族成员的标准基因,按照相同方法在番茄基因组数据库中进行筛选鉴定。最后利用ExPASy(http://web. expasy. org/protparam)分析上述家族成员的理化性质。
1.4 系统进化关系及蛋白结构域分析
将鉴定得到的基因核酸序列转化成蛋白序列,采用MEGA 6.0软件中的邻接法Neighbor-Joining(参数:Poisson mode,Complete deletion,Bootstrap=1 000)进行多序列比对并构建进化树,随后利用Web CD-Search Tool(https://www.ncbi.nlm.nih.gov /Structure/bwrpsb/bwrpsb.cgi)工具(参数:Search against database=Pfam-17919 PSSMs,其余参数为默认值)对蛋白结构域进行分析,最后利用Evolview(https://www.evolgenius. info/evolview)对数据可视化处理。
1.5 ATS家族成员组织表达模式分析
从TPIA茶树数据库网站中下载茶树品种舒茶早基因在不同组织中表达量数据包(Eight_Tissues_TPM.txt.gz),并从中提取茶树家族成员的表达量数据;从TomExpress(http://tomexpress.toulouse.inra.fr)[26]中下载番茄家族成员的表达量数据,利用TBtool和Excel 2016软件对数据进行可视化处理。
1.6 ATS的定量PCR测定
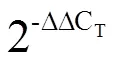
1.7 数据统计分析
显著性检验采用SAS9.4 M3(SAS Institute Inc,USA)中检验(*<0.05,**<0.01)和邓肯多重比较,文本和图表中的数据表示为平均值±标准差(n≥3)。
2 结果与分析
2.1 处理组番茄叶片中乙胺、茶氨酸、丙氨酸及谷氨酰胺含量分析
番茄幼苗水培25 d后,以鲜重计,仅在处理组番茄叶片中检测到(0.095 5±0.008 0)mg·g-1的乙胺和(0.017 0±0.003 5)mg·g-1的茶氨酸(图2-C)。谷氨酸在处理组叶片中的含量极显著降低(<0.01),仅为对照组的30%,变化趋势与茶树根和叶中类似(图3-A),表明番茄体内应该存在ATS,可以将吸收的乙胺转化成茶氨酸,且合成方式与茶树相似。丙氨酸是茶树合成乙胺的前体,它在茶树根部的含量显著低于叶片(图3-A);但在对照组和处理组番茄叶片中,丙氨酸含量差异不显著(图3-B),表明番茄体内可能不存在类似于茶树中的乙胺形成途径,使得乙胺可能成为茶氨酸合成的关键限制因子。此外,与对照组相比,处理组番茄叶片中的谷氨酰胺含量提高了12.66倍,表明外源乙胺也增加了谷氨酰胺的合成量(图4)。

表1 qRT-PCR 荧光定量引物序列

注:A为超高效液相色谱下乙胺、茶氨酸、谷氨酸、丙氨酸和谷氨酰胺标准品的出峰时间图;B、C分别为对照组和处理组番茄叶片中乙胺和茶氨酸的出峰时间图

注:A为龙井43一年生扦插苗根和叶片干重下的谷氨酸、丙氨酸、乙胺含量对比;B为处理组和对照组番茄叶片鲜重下的谷氨酸、丙氨酸、乙胺含量对比

图4 乙胺处理下番茄叶中茶氨酸和谷氨酰胺的含量(鲜重)
2.2 ATS家族成员的鉴定及理化性质分析
在茶树中共鉴定得到12个家族成员基因,其中的7个基因在文献中已有注释和命名,其余的5个,根据它们序列与茶树和相似性依次命名为、、、、。在番茄基因组中共筛选到5个,其中将与高度相似的Solyc10g083830.2.1命名为,其余4个番茄的序列经NCBI中的BLASTn检索,分别注释为、、、(表2)。
采用ExPaSy软件对鉴定的家族成员进行理化性质分析,茶树与番茄的基因CDS编码序列为597~2 562 bp,编码198~853个氨基酸残基,分子量为21.51~94.97 kDa,等电点pI为5.43~7.69,不稳定指数为36.34~46.05,脂肪指数为73.53~96.06,亲水性系数为–0.470~–0.040。其中茶树与番茄GS基因的理化性质较为一致,而与之间的理化性质有所不同。除了CsTSI-3为碱性蛋白外,其余均为酸性蛋白。CsTSI、SlTSI不稳定指数小于40,脂肪指数大于GS成员,表明CsTSI、SlTSI蛋白整体上要比GS稳定。
2.3 系统进化树的构建与蛋白结构域分析
为了进一步明确ATS的同源关系,将筛选得到的茶树与番茄ATS氨基酸序列进行多序列比对,并构建了茶树和番茄ATS的系统进化树(图5)。结果表明,ATS家族被分为3个亚族,Ⅰ亚族中包含11个茶树与番茄的GS1成员,Ⅱ中是CsGS2和SlGS2,Ⅲ中是4个TSI成员,因此将Ⅰ、Ⅱ和Ⅲ分别注解为GS1、GS2和TSI簇。随后利用Web CD-Search Tool对不同亚族的蛋白序列进行结构域分析,共得到6个功能结构域,即谷氨酰胺合成酶催化域Gln-synt_C(PF00120),谷氨酰胺合成酶N端结构域Gln-synt_N(PF03951)和Gln-synt_N_2(PF16952),氨基水解酶结构域Amidohydro_2(PF04909),Cornichon(PF03311)和具有DNase活性的蛋白结构域TatD_DNase(PF01026)。其中谷氨酰胺合成酶催化域Gln-synt_C是ATS家族成员所共有,也是谷氨酰胺合成酶行使功能的关键domain位点,表明ATS可能属于谷氨酰胺合成酶家族。与Ⅰ、Ⅱ簇中的GS1、GS2相比,Ⅲ簇中的TSI还含有Gln-synt_N_2、Amidohydro_2、Cornichon及TatD_DNase等结构域(图5)。

表2 茶树及番茄ATS基因家族理化性质

图5 茶树及番茄ATS的系统进化树及保守结构域
2.4 ATS家族成员组织表达模式分析
由于从茶树基因表达数据中只获取了、、、和等6个茶树家族成员的表达量数据,因此对这部分的成员进行了表达分析。结果发现,所有组织中的和表达量均较低,其余4个基因在组织间存在表达差异。在根中表达量最高,成熟叶次之,在其他组织都有不同程度的表达;在花中表达最高,根和顶芽次之;在嫩叶、成熟叶和老叶中的表达相对较高,只在根中特异性表达(图6)。
在TomExpress中未获得番茄家族成员的表达量数据,其余4个基因和在不同样本中的表达量存在较大差异。在不同生长期的所有组织中,的表达量均最高,的表达量最低接近于0;而和在根、叶、花和籽粒中有一定的表达量(图7)。
2.5 番茄ATS在乙胺处理下的基因表达分析
为了进一步研究番茄在茶氨酸合成中的作用,在外源乙胺处理25 d后,对表达量较高的和进行了qRT-PCR分析。结果显示,对照组中的表达量最高,与的表达量相近;而在处理组中,的表达量最高,次之,与对照组相比分别提高了19.5倍和4.1倍,最低且和对照组基本相同(图8)。这表明和对外源乙胺的诱导敏感,可能在番茄叶部的茶氨酸合成中发挥了作用。
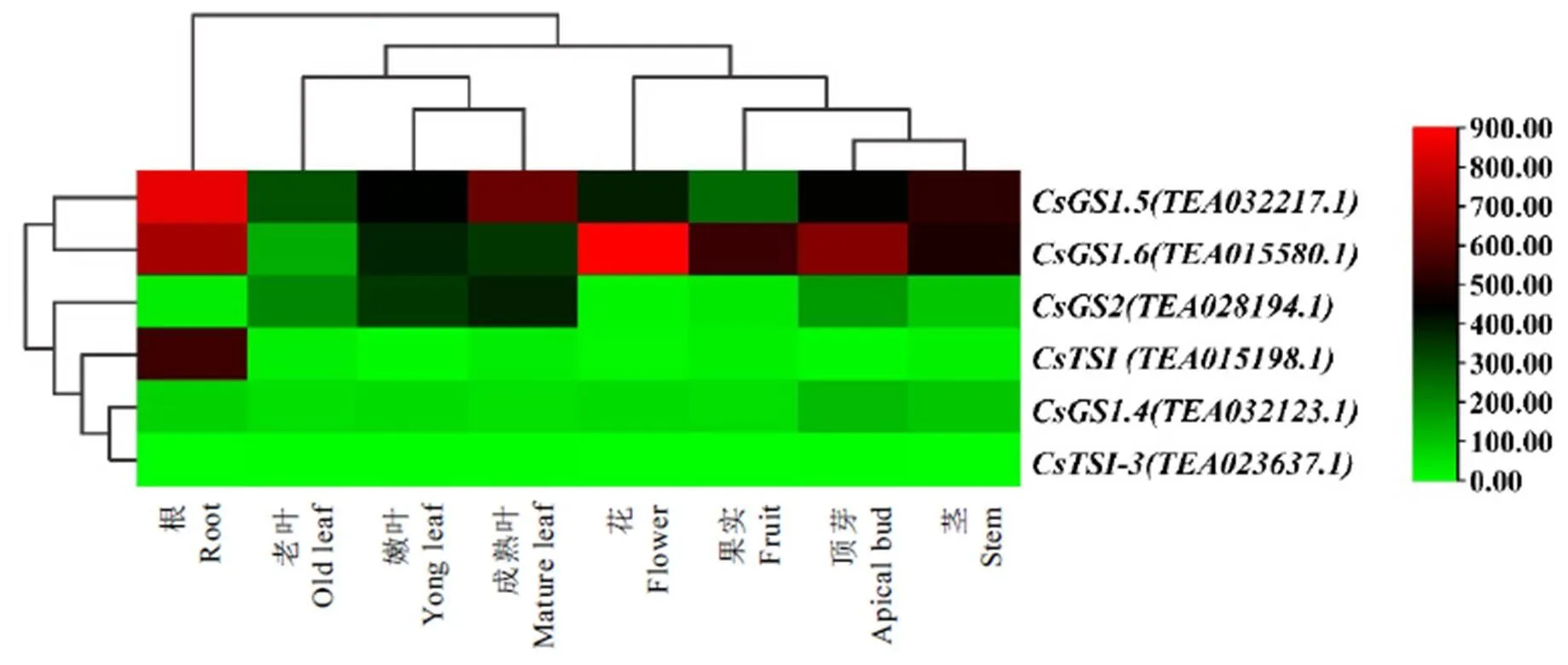
图6 茶树ATS基因家族在茶树组织中的表达

图7 番茄ATS基因家族在番茄不同生长期组织中的表达

图8 乙胺处理下番茄叶中ATS的表达水平
3 讨论
3.1 ATS广泛存在于植物的谷氨酰胺合成酶家族中
本研究通过对水培番茄幼苗进行外源乙胺处理。发现仅在处理组叶片中检测到茶氨酸的存在,并且在茶氨酸积累的同时,谷氨酰胺含量也显著增加;蛋白结构分析表明,从茶树和番茄基因组中鉴定到的均含有谷氨酰胺合成酶的关键催化位点Gln-synt_C。这表明番茄体内存在ATS,且应该属于谷氨酰胺合成酶家族。
Sasaoka等[27]将乙胺添加到豌豆种子的丙酮粉提取物中,发现有茶氨酸的生成,推测这个过程可能是TS的非特异性反应。Okada等[12]通过原核表达技术证实具有合成茶氨酸的能力,Deng等[13]和陈琪等[28]对上述两个基因进行生物信息学分析,发现它们与GS基因序列高度相似。Cheng等[8]试验证实,拟南芥的谷氨酰胺合成酶基因和能够催化乙胺和谷氨酸生成茶氨酸。Fu等[16]研究表明,茶树谷氨酰胺合成酶基因、和分别在烟草中过表达能够提高茶氨酸的合成量(≤0.05)。目前的文献表明,在用乙胺培养过的豌豆、拟南芥、玉米和烟草等植物的组织中都能检测到茶氨酸[8-9,16,27]。因此,推断植物中应该广泛存在ATS,且该酶属于植物的谷氨酰胺合成酶家族。
3.2 乙胺是合成茶氨酸的关键限制因子
已有研究表明,与茶氨酸生物合成过程相关的酶有谷氨酸脱氢酶(Glutamate dehydrogenase,GDH)谷氨酸合成酶(Glutamate synthase,GOGAOT)GGT、丙氨酸脱羧酶(Alanine decarboxylase,AlaDC)和TS等[29-30]。此外,Dong等[31]证实有6个茶树氨基酸通透酶CsAAP具有转运茶氨酸能力,其中CsAAP1可能主要介导了茶氨酸从根部到茎部的转运。AAP属于氨基酸/生长素渗透酶家族AAAPs并广泛存在植物中[32-33]。Zhang等[34]发现转录因子TF家族中的MYB和bHLH可能调控了茶氨酸的形成,而MYB和bHLH在植物叶中普遍存在,其中在拟南芥中有超过100个成员[35]。尽管上述转运蛋白和TF对茶氨酸合成有一定的作用,但它们是植物中比较常见的调控、转运模块,更多是对次级代谢产物的二次分配。
本研究结果表明,受到外源乙胺诱导后,番茄体内的表达量相比于对照组有很大程度的提高,导致了茶氨酸的合成与积累。表明乙胺作为茶氨酸的前体物质,可以诱导表达,触发茶氨酸的合成。Wei等[9]曾用乙胺水溶液水培茶苗12 d后,发现根中的表达升高到最高水平,同时茶氨酸积累量也上升到峰值,认为乙胺具有激活茶氨酸代谢途径的作用。但本研究中发现,与对照组相比,番茄叶中的丙氨酸含量并没有显著变化,这说明番茄只能利用外源的乙胺来合成茶氨酸,自身并不能利用丙氨酸合成乙胺。乙胺在植物中的分布与茶氨酸相似,主要存在于茶树中,且在已发现含有茶氨酸的物种中都有乙胺的存在[7-8]。而催化丙氨酸脱羧生成乙胺的关键酶是AlaDC,目前仅在茶树中有报道[36]。之前文献表明,只有经过乙胺处理过的豌豆、拟南芥、玉米和烟草才能在体内合成出茶氨酸[8-9,16-27]。综上,我们认为乙胺是制约植物合成茶氨酸的关键限制因子,且对茶氨酸的合成存在底物诱导效应。
[1] Chen Z M, Chen L. Delicious and healthy tea: an overview [M]//Chen L, Apostolides Z, Chen Z M. Global Tea Breeding. Berlin: Springer, 2012.
[2] Zhao J, Li P, Xia T, et al. Exploring plant metabolic genomics: chemical diversity, metabolic complexity in the biosynthesis and transport of specialized metabolites with the tea plant as a model [J]. Critical Reviews in Biotechnology, 2020, 40(5): 667-688.
[3] 毛雅琳, 汪芳, 尹军峰, 等. 不同茶树品种碾茶的品质分析[J]. 茶叶科学, 2020, 40(6): 782-794. Mao Y L, Wang F, Yin J F, et al. Quality analysis of tencha made from different tea cultivars [J]. Journal of Tea Science, 2020, 40(6): 782-794.
[4] Yamaguchi S, Ninomiya K. Umami and food palatability [J]. The Journal of Nutrition, 2000, 130(4): 921-926.
[5] Yan Z M, Zhong Y Z, Duan Y H, et al. Antioxidant mechanism of tea polyphenols and its impact on health benefits [J]. Animal Nutrition, 2020, 6(2): 115-123.
[6] Yu Z, Yang Z. Understanding different regulatory mechanisms of proteinaceous and non-proteinaceous amino acid formation in tea () provides new insights into the safe and effective alteration of tea flavor and function [J]. Critical Reviews in Food Science and Nutrition, 2020, 60(5): 844-858.
[7] Deng W W, Ogita S, Ashihara H. Distribution and biosynthesis of theanine in Theaceae plants [J]. Plant Physiology and Biochemistry, 2010, 48(1): 70-72.
[8] Cheng S H, Fu X M, Wang X Q, et al. Studies on the biochemical formation pathway of the amino acid-theanine in tea () and other plants [J]. Journal of Agricultural and Food Chemistry, 2017, 65(33): 7210-7216.
[9] Wei C L, Yang H, Wang S B, et al. Draft genome sequence ofvar.provides insights into the evolution of the tea genome and tea quality [J]. PNAS, 2018, 115(18): 4151-4158.
[10] Sasaoka K, Kito M, Onishi Y. Some properties of the theanine synthesizing enzyme in tea seedlings [J]. Agricultural and Biological Chemistry, 1965, 29(11): 984-988.
[11] Sasaoka K, Kito M, Inagaki H. Studies on the biosynthesis of theanine in tea seedlings [J]. Agricultural and Biological Chemistry, 1963, 27(6): 467-468.
[12] Okada Y, Koseki M, Chu M. Protein and cDNA sequences of two theanine synthetases from: JP2006254780 [P/OL]. 2006-09-28.
[13] Deng W W, Ogita S, Ashihara H. Biosynthesis of theanine (-ethylamino--glutamic acid) in seedlings of[J]. Phytochemistry Letters, 2008, 1(2): 115-119.
[14] Yamamoto S, Wakayama M, Tachiki T. Cloning and expression ofY-30 gene encoding glutamine synthetase: an enzyme available for theanine production by coupled fermentation with energy transfer [J]. Bioscience, Biotechnology and Biochemistry, 2006, 70(2): 500-507.
[15] Kei S, Makoto K. Synthesis of theanine by tea seedling homogenate [J]. Agricultural and Biological Chemistry, 1964, 28(5): 313-317.
[16] Fu X M, Liao Y Y, Cheng S H, et al. Nonaqueous fractionation and overexpression of fluorescent-tagged enzymes reveals the subcellular sites of-theanine biosynthesis in tea [J]. Plant Biotechnology Journal, 2020, 19(1): 98-108.
[17] 朱文娴, 黎星辉, 王丽鸳, 等. 利用GS基因构建茶氨酸生物合成工程菌的研究[J]. 茶叶科学, 2008, 28(4): 242-248. Zhu W X, Li X H, Wang L Y, et al. Construction ofrecombinant engineered strain for theanine biosynthesis with GS gene embedded [J]. Journal of Tea Science, 2008, 28(4): 242-248.
[18] Kimura T, Sugahara I, Hanai K, et al. Purification and characterization of-glutamylmethylamide synthetase fromsp. AA-30 [J]. Bioscience Biotechnology and Biochemistry, 1992, 56(5): 708-711.
[19] Chen X Y, Su L Q, Wu D, et al. Application of recombinant Bacillus subtilis γ-glutamyltranspeptidase to the production of-theanine [J]. Process Biochemistry, 2014, 49(9): 1429-1439.
[20] 王丽鸳, 王贤波, 成浩, 等. 基因工程菌生物合成茶氨酸条件研究[J]. 茶叶科学, 2007, 27(2): 111-116. Wang L Y, Wang X B, Cheng H, et al. Research on conditions of theanine biosynthesis by genetically engineered[J]. Journal of Tea Science, 2007, 27(2): 111-116.
[21] Miyake K, Kakita S. A novel catalytic ability of-glutamylcysteine synthetase ofand its application in theanine production [J]. Bioscience Biotechnology and Biochemistry, 2009, 73(12): 2677-2683.
[22] Martí E, Gisbert C, Bishop G J, et al. Genetic and physiological characterization of tomato cv. Micro-Tom [J]. Journal of Experimental Botany, 2006, 57(9): 2037-2047.
[23] Zhang Y, Wang L, Wei K, et al. Differential regulatory mechanisms of secondary metabolites revealed at different leaf positions in two related tea cultivars [J]. Scientia Horticulturae, 2020, 272: 109579. doi: 10.1016/j.scienta.2020.109579.
[24] Xia E H, Li F D, Tong W, et al. Tea plant information archive: a comprehensive genomics and bioinformatics platform for tea plant [J]. Plant Biotechnology Journal, 2019, 17(10): 1938-1953.
[25] Chen C, Chen H, Zhang Y, et al. TBtools: an integrative toolkit developed for interactive analyses of big biological data [J]. Molecular Plant, 2020, 13(8): 1194-1202.
[26] Zouine M, Maza E, Djari A, et al. TomExpress, a unified tomato RNA-Seq platform for visualization of expression data, clustering and correlation networks [J]. The Plant Journal, 2017, 92(4): 727-735.
[27] Sasaoka K, Kito M, Onishi Y. Synthesis of theanine by pea seed acetone powder extract [J]. Agricultural and Biological Chemistry, 1964, 28(5): 318-324.
[28] 陈琪, 江雪梅, 孟祥宇, 等. 茶树茶氨酸合成酶基因的酶活性验证与蛋白三维结构分析[J]. 广西植物, 2015, 35(3): 384-392, 377. Chen Q, Jiang X M, Meng X Y, et al. Enzymatic activity verification and protein three-dimensional structural analysis of theanine synthetase gene in[J]. Guihaia, 2015, 35(3): 384-392, 377.
[29] Li F, Dong C X, Yang T Y, et al. Seasonal theanine accumulation and related gene expression in the roots and leaf buds of tea plants (L.) [J]. Frontiers in Plant Science, 2019, 10: 1397. doi: 10.3389/fpls.2019.01397.
[30] Crocomo O J, Fowden L. Amino acid decarboxylases of higher plants: the formation of ethylamine [J]. Phytochemistry, 1970, 9(3): 537-540.
[31] Dong C X, Li F, Yang T Y, et al. Theanine transporters identified in tea plants (L.) [J]. The Plant Journal, 2020, 101(1): 57-70.
[32] Fischer W N, Kwart M, Hummel S, et al. Substrate specificity and expression profile of amino acid transporters (AAPs) in[J]. Journal of Biological Chemistry, 1995, 270(27): 16315-16320.
[33] Yao X H, Nie J, Bai R X, et al. Amino acid transporters in plants: identification and function [J]. Plants, 2020, 9(8): 972. doi: 10.3390/plants9080972.
[34] Zhang S H, Cheng Y, He X B, et al. Identification oftranscription factors regulating theanine biosynthesis in tea plant using omics-based gene coexpression analysis [J]. Journal of agricultural and food chemistry, 2020, 68(3): 918-926.
[35] Millard P S, Konrad W, Kragelund B B, et al. Specificity of MYB interactions relies on motifs in ordered and disordered contexts [J]. Nucleic Acids Research, 2019, 47(18): 9592-9608.
[36] Bai P X, Wei K, Wang L Y, et al. Identification of a novel gene encoding the specialized alanine decarboxylase in tea () plants [J]. Molecules, 2019, 24(3): 540. doi: 10.3390/molecules24030540.
Identification and Analysis of Analogue Theanine Synthase Gene Family in Tomato
MAO Peng1,2, WANG Liyuan1*, BAI Peixian1, WEI Kang1, RUAN Li1, ZHNAG Yazhen1, CHENG Hao1*
1. Tea Research Institute, Chinese Academy of Agricultural Sciences, National Center for Tea Improvement, Key Laboratory of Tea Biology and Resource Utilization, Ministry of Agriculture, Hangzhou 310008, China; 2. Graduate School of Chinese Academy of Agricultural Sciences, Beijing 100081, China
Theanine synthetase (TS) and some members of the glutamine synthetase (GS) family in tea plants show catalytic activity for the synthesis of theanine from ethylamine and glutamic acid. The enzymes in plants that show catalytic activity for the synthesis of theanine and are highly similar to TS sequence of tea plants are named as the analogue theanine synthase (ATS). To explore the existence of ATS in non-tea plants and effect of ethylamine on theanine synthesis, ‘Mico-Tom’ tomato was used as materials. As tomato seedlings was treated hydroponically with ethylamine, theanine was successfully detected in the leaves of the treated group. The content of glutamine (Gln) in the tomato leaves was also significantly increased, while the content of alanine did not change compared with the control group. Then, 12 and 5 members of thefamily were identified respectively in tea and tomato through bioinformatics analysis and all of them contained the Gln-synt_C domain of glutamine synthase. Further qRT-PCR results show that the expressions of 2(and) in the leaves were significantly increased under the treatment of ethylamine. In conclusion, the ATS may exist widely in plants, and ethylamine would be a key limiting factor in the synthesis of theanine as an important precursor. Meanwhile, ethylamine can greatly accelerate the production of theanine by promoting the transcripts of.
tomato, theanine, ethylamine, analogue theanine synthase, gene family
S571;Q52
A
1000-369X(2021)02-173-11
2021-01-06
2021-01-22
现代农业产业技术体系(CARS-19)、浙江省农业新品种选育重大专项(2016C02053)
毛鹏,男,硕士研究生,主要从事茶树资源与育种研究。*通信作者:wangly@tricaas.com;chenghao@tricaas.com
(责任编辑:黄晨)
