基于TCGA 数据分析CHRDL1 基因在乳腺癌中的表达特征及生物学功能
2021-03-05刘新兰
郭 婷, 刘新兰
(1.宁夏医科大学临床医学院,银川 750004; 2.宁夏医科大学总医院,银川 750004)
乳腺癌已成为全球最常见的女性恶性肿瘤,且呈不断增长趋势[1]。2015 年女性发病首位为乳腺癌,年发病人数约为30.4 万,女性癌症死因乳腺癌排第五位[2]。因此,寻找能提高乳腺癌早期诊断的肿瘤标志物至关重要。目前,单基因腱蛋白样蛋白1(chordin like 1,CHRDL1)在乳腺癌中研究较少,通过癌症基因组图谱(The Cancer Genome Atlas,TCGA)数据库筛选乳腺癌Top100 差异表达基因(differentially expressed genes,DEGs)包括CHRDL1(Log FC 为-5.26,P=1.87×10-153,FDR=3.07×10-151)。此外,通过对Gene Expression Omnibus(GEO)数据集GES42568、GSE8977 及GSEA50428联合分析后进行Hub 基因筛选,含CHRDL1。CHRDL1 是一种分泌性蛋白,是骨形态发生蛋白(BMP)的拮抗剂。BMP 作为BMP 受体Ⅱ(BMPRII)的激活剂,介导细胞外到细胞内的信号传递,参与肿瘤的发生和转移。Pei 等[3]研究证实CHRDL1 在胃癌组织中的表达下调,且预后较差,CHRDL1 是一种抑癌基因。孙丽等[4]证实BMP抑制剂CHRDL1 可抑制小鼠乳腺增生。Mithani等[5]综合遗传学分析CHRDL1 等基因在恶性黑色素瘤呈低甲基化,抑制恶性黑色素瘤增殖。本研究主要以单基因CHRDL1 为研究对象,通过TCGA数据挖掘,利用生物信息学方法分析CHRDL1 在乳腺癌中的表达、临床相关性及生存预后分析,使用基因富集分析(Gene Set Enrichment Analysis,GSEA)CHRDL1 可能参与的信号通路,揭示其参与乳腺癌发生发展的可能分子机制。
1 材料与方法
1.1 人乳腺癌基因芯片表达数据及临床数据的收集和处理
人乳腺癌患者的RNA-Seq 数据集和相应的临床资料来自TCGA(https://gdc.nci.nih.gov)[6]。在创建数据集的过程中,从TCGA 数据门户网站下载总共1075 例非配对乳腺癌组织样本和114 例正常样本及配对样本(乳腺癌组织样本111例、正常组织样本111 例)的原始RNA 测序读数和相应的临床信息,所有样本的RNA 表达水平均已在R 统计环境(V.3.6.0)中使用BioConductor软件包进行处理和归一化。根据样本ID 匹配CHRDL1 mRNA 表达数据矩阵和临床信息文件,CHRDL1 在乳腺癌甲基化水平,Beta 值表示DNA甲基化的水平,范围从0(未甲基化)到1(完全甲基化);甲基化过高(Beta 值:0.7~0.5)或甲基化过低(Beta 值:0.3~0.25);PMID:29027401、23291739)。
1.2 CHRDL1 在人乳腺癌中的表达差异及其与临床病理特征的关系
采用R 3.6.0 软件对TCGA 下载的数据进行处理,使用limma 包对芯片数据作差异分析,计算CHRDL1 在乳腺癌组织(114 例)和乳腺正常组织(1097 例)、乳腺癌组织和与其相匹配的癌旁正常组织(111 例)的差异表达,使用beeswarm包作图。使用http://ualcan.path.uab.edu[7]网络平台与秩和检验分析CHRDL1 在乳腺癌中的表达及其与TNM 分期、分子分型的关系。
1.3 乳腺癌组织CHRDL1 表达水平与乳腺癌预后的关系
使用Kaplan-Meier Plotter 数据库(https://kmplot.com/analysis)[8]对基因芯片中209763_at 探针数据进行生存和预后分析。按CHRDL1 在乳腺癌组织芯片中的表达均值进行分组,高于均值为高表达组,低于均值为低表达组。分别分析CHRDL1差异表达与乳腺癌患者总生存期(OS)和无远处转移生存(RFS)的关系。
1.4 基因集富集分析
使用KEGG 的基因集[Gene Sets Debates:C2.CP.KEGG.V6.2.Symbols.gmt(Curated)]进行GSEA富集分析[9]评估CHRDL1 可能参与的信号通路。根据CHRDL1 表达中位数将基因表达数据分为高、低两组,每次分析进行1000 次基因集排列。在整个过程中,CHRDL1 的表达水平被认为是一个表型,根据标准化富集分数(NES)、标准化显著性水平(NOM p-val)和矫正多重假设检验(FDR q-val)对每种表型的富集途径进行分类,NES 绝对值≥1.0,NOM p-val≤0.05,FDR q-val≤0.25确认为有意义的基因集合。
1.5 PPI 网络构建和信号通路关键基因筛选
通过在线分析网站String(https://string-db.org/)[10]得到已筛选有意义信号通路中差异表达基因(DEGs)的蛋白互作网络,以TSV 格式导出,将所得源文件导入Cytoscape 进行可视化分析,用插件cytoHubba 进行Hub 基因分析,筛选出潜在的关键基因。
1.6 CHRDL1 与筛选出的信号通路关键基因的相关性分析
通过在线分析网站CANCERTOOL[11](http://web.bioinformatics.cicbiogune.es/CANCERTOOL)分析CHRDL1 与GSEA 富集分析中筛选出来的关键基因CAV1 和FYN 的相关性。绘制的值对应于指定数据集中每个患者的两个基因(X 轴和Y轴)的log2归一化基因表达值。黑线表示线性回归,灰色区域表示置信区间的界限,r 和P 分别表示Pearson 相关系数和统计显著性。
1.7 统计学方法
所有统计分析和绘图均使用R(v.3.6.0)软件。用Wilcoxon 秩和检验和Wilcoxon 符号秩和检验分析CHRDL1 在非配对样本和配对样本中的表达。采用秩和检验分析CHRDL1 表达与临床病理特征的关系,使用Kaplan-Meier 法作生存分析,P≤0.05 为差异有统计学意义。两基因间相关性分析使用Pearson 相关性分析,P≤0.05 并且|r|≥0.3 为差异有统计学意义。
2 结果
2.1 CHRDL1 在乳腺癌和乳腺正常组织中的差异表达及生存分析
基于TCGA 数据分析结果显示CHRDL1 在90%以上肿瘤中呈现低表达状态,仅在肉瘤中表达增高。在非配对乳腺癌组织和正常组织及配对的乳腺癌组织与其癌旁正常组织中的差异表达分析显示CHRDL1 在乳腺癌中的表达低于正常组织及癌旁组织(P=1.0×10-12、P=3.556×10-13)。通过Kaplan-Meier Plotter 数据库分析CHRDL1的表达与乳腺癌患者预后的关系,结果显示CHRDL1的高表达为乳腺癌不良预后因素,在OS 和RFS 分析中,CHRDL1 高表达者的OS 和RFS 均低于CHRDL1 低表达者(P=9.2×10-5、P=2.4×10-8)。见图1。
2.2 CHRDL1 在乳腺癌中的表达与临床病理特征的关系
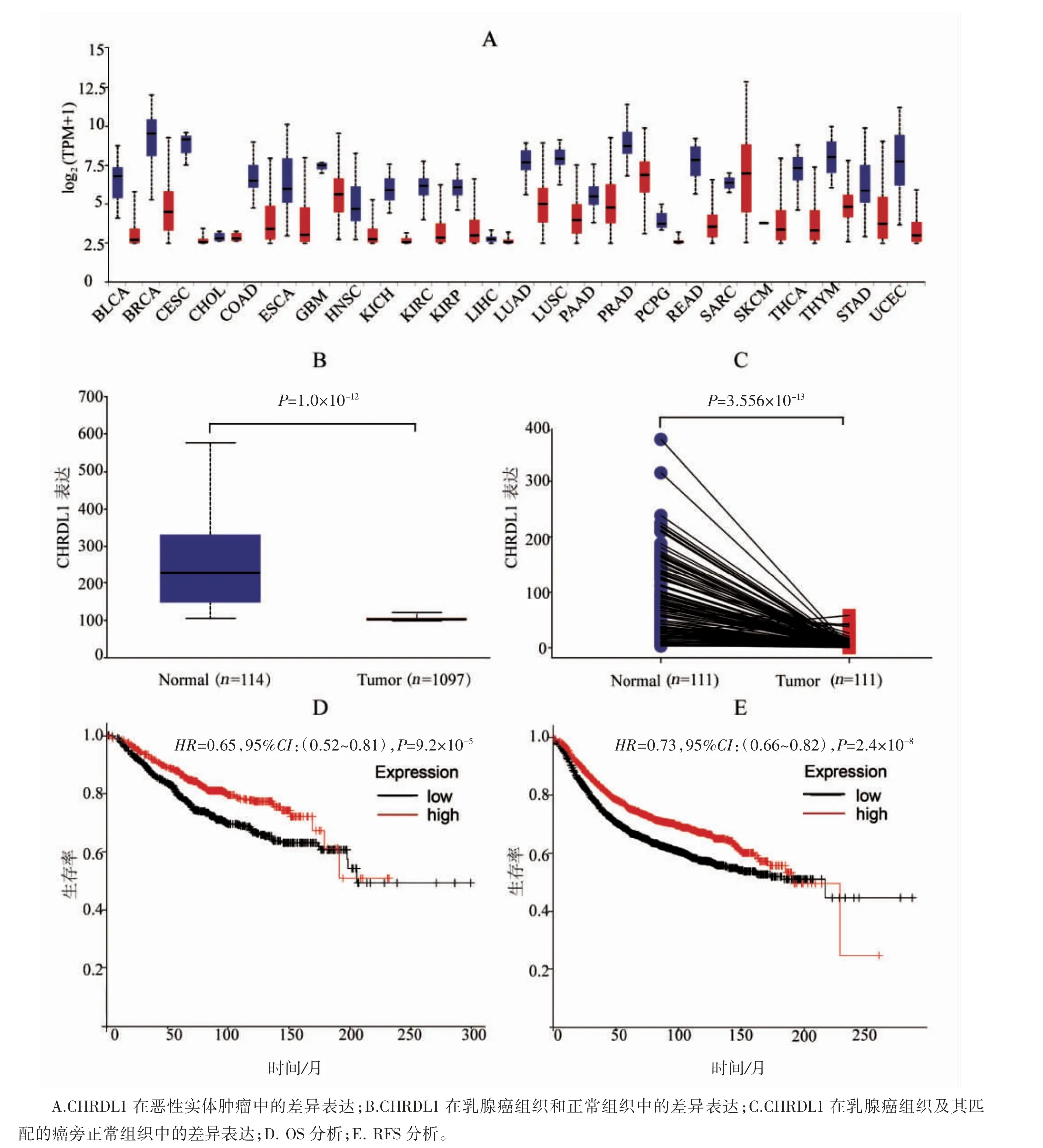
图1 CHRDL1 在乳腺癌中表达及生存分析
在乳腺癌组织大小即T 分期中,T3 期中CHRDL1 的表达均高于其他组(P 均<0.05),说明CHRDL1 在肿块>5 cm 时表达程度较高;N 分期中,不同淋巴结转移分级的CHRDL1 表达差异无统计学意义(P=0.165);M 分期中,无远处转移组CHRDL1 表达高于存在远处转移组(P=0.049);乳腺癌四种不同分子分型组的CHRDL1表达差异有统计学意义(P=1.48×10-12),HER2 过表达组、三阴性乳腺癌(TNBC)组、Luminal 组均低于正常组(P 均<0.05),CHRDL1 在HER2 过表达组中表达最低,在三阴性乳腺癌组中表达最高(P 均<0.05)。见图2。
2.3 乳腺癌组织中CHRDL1 启动子甲基化水平分析
乳腺癌组织CHRDL1 启动子与正常组织的甲基化均为过低,Beta 值处于0.3~0.25,但乳腺癌组织中CHRDL1 启动子甲基化值高于正常组织(P=1.0×10-12)。不同的分子分型分析结果显示,Luminal 组CHRDL1 启动子区甲基化值高于正常组(P=1.62×10-12),Luminal 组和HER2 过表达组CHRDL1 启动子区甲基化值均高于TNBC 组(P=2.53×10-7、P=0.034)。见图3。

图2 乳腺癌CHRDL1 的表达与临床特征的关系
2.4 CHRDL1 调控信号通路GSEA 富集分析
GSEA 富集分析结果显示共有27 个信号通路和生物过程在CHRDL1 和乳腺癌之间具有不同程度的富集,筛选条件为同时满足NOM p-val<0.05 且FDR q-val<0.05。与肿瘤相关的信号通路和生物过程有Jak- STAT 信号通路、MAPK 信号通路、细胞因子通路、凋亡、氧化磷酸化、糖代谢以及脂代谢相关的信号通路等。见表1、图4。
2.5 CHRDL1 在MAPK 信号通路中蛋白互作网络分析
GSEA 富集分析发现CHRDL1 在MAPK 信号通路中发挥重要作用。通过Cytoscape 软件构建CHRDL1 在乳腺癌侵袭及转移相关的信号通路MAPK 通路中关键基因的蛋白互作网络,其中CAV1、FYN 与CHRDL1 具有直接互作关系。为进一步对筛选出的Hub 基因与CHRDL1 作相关性分析,发现其与CHRDL1 呈正相关性,相关系数分别为0.39 和0.39,P 值分别为2.2×10-16和1.45×10-12。见图5。
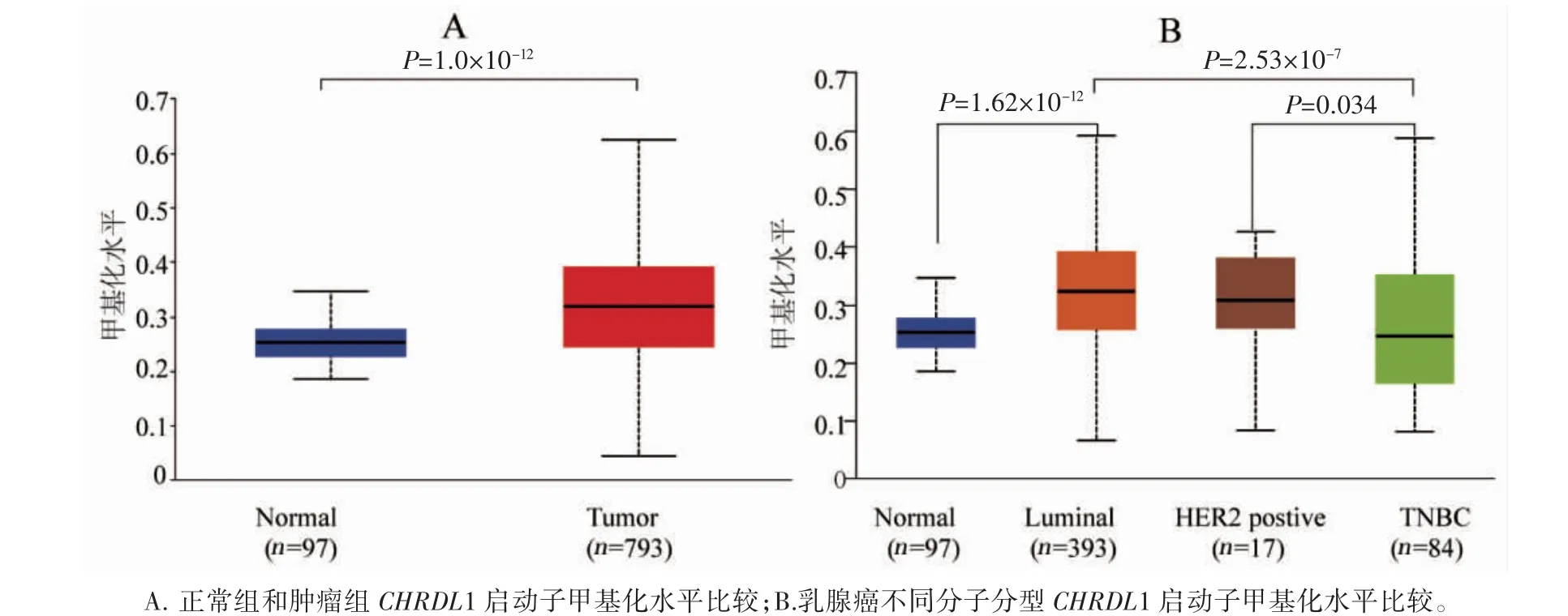
图3 乳腺癌易感基因CHRDL1 启动子区甲基化水平

表1 GSEA KEGG 富集分析
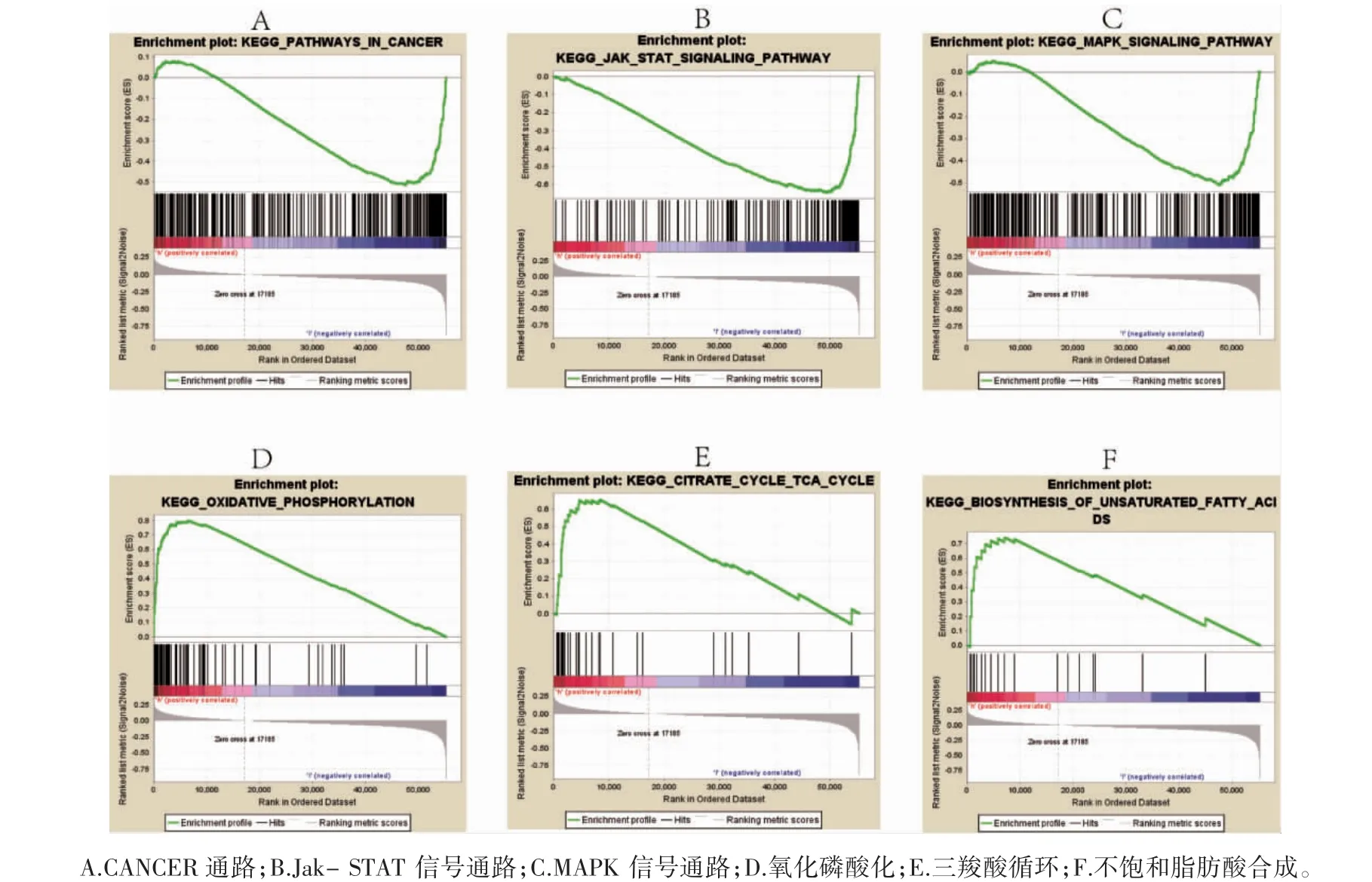
图4 GSEA 信号通路富集分析
3 讨论
CHRDL1 是一种骨形态发生蛋白BMP 抑制剂,参与调节BMP 信号通路活性,CHRDL1 蛋白可以竞争性结合BMP,并通过分泌到细胞外基质来拮抗其功能[12]。BMPs 首先被确定为诱导异位骨形成,并在发育期间形态发生中起重要作用,它们是多功能生长因子,属于转化生长因子-β(TGFβ)的超家族[13]。Cyr-Depauw 等[14]证实,在降低的ShcA 信号传导条件下,刺激TGF 后,CHRDL1 表达在大量乳腺癌细胞中上调。体外实验[14]证实CHRDL1 作为BMP4 诱导迁移和侵袭的抑制剂,CHRDL1 表达是乳腺癌患者的有利预后因素。另一项研究[3]揭示了CHRDL1 在胃癌组织中表达下调,且与低存活率相关。通过临床和病理数据观察到CHRDL1 的低表达与转移之间密切相关,CHRDL1 的表达下调通过BMP 受体II 促进肿瘤细胞的增殖和迁移。此外,CHRDL1 是抑制肿瘤增殖和转移的抑癌基因[3]。以上研究表明,CHRDL1的表达缺失可能是肿瘤起始的分子病因。本文主要依赖于TCGA 数据库获得的高通量测序数据,利用生物信息学工具证实CHRDL1 作为乳腺癌的抑癌因子在乳腺癌发生发展中的调控作用。

图5 MAPK 信号通路中蛋白互作网络及共表达基因筛选
本研究通过挖掘TCGA 数据明确CHRDL1在乳腺癌中的表达、临床相关性及预后价值。首先利用Ualcan 网络平台分析CHRDL1 在大多数恶性实体肿瘤中呈现低表达,如膀胱癌、乳腺癌、结肠癌、食道癌、头颈鳞状细胞癌、肺癌、胰腺癌、前列腺癌、胸腺癌、胃癌、脑胶质瘤、肾癌、肝癌、皮肤黑色素瘤和甲状腺癌;仅在肉瘤呈高表达。本文结果显示,CHRDL1 在乳腺癌组织的表达低于正常乳腺组织,在配对分析中得到了一致的结果。CHRDL1 在不同临床分期中均呈低表达,T分期中结果显示在T3 组中CHRDL1 表达最高,说明CHRDL1 在5 cm 以上乳腺癌肿块组织中表达具有一定诊断意义,且存在远处转移的患者CHRDL1 表达低于未发生转移者,证实CHRDL1为抑癌因子。而CHRDL1 启动子甲基化不论是在正常组织还是癌组织均呈低甲基化,与正常组织相比,乳腺癌组织中CHRDL1 启动子甲基化值高于正常组织;在不同分子分型组间,CHRDL1 启动子区甲基化值在Luminal 组较高,Luminal 组和HER2 过表达组CHRDL1 启动子区甲基化值均高于三阴性乳腺癌组。Kaplan-Meier Plotter 数据库包含54675 个基因的10461 个癌症样本的生存数据,其中包括3955 个乳腺癌样本,可以进行乳腺癌有关基因的临床预后相关数据的分析[15]。通过Kaplan-Meier Plotter 数据库进行生存分析显示,CHRDL1 高表达乳腺癌患者的OS 和RFS 均低于CHRDL1 低表达者,以上生物信息学数据分析结果显示CHRDL1 有望成为乳腺癌抑癌基因,且其在乳腺癌中的高表达与患者OS 和RFS 较低相关。
为进一步研究CHRDL1 在乳腺癌中的功能及调控机制,基于TCGA 数据利用GSEA 软件进行KEGG 基因集富集分析。结果显示,在乳腺癌中共有27 个信号通路和功能在CHRDL1 的高表达表型中有富集,与肿瘤相关的信号通路和生物过程:Jak- STAT 信号通路、MAPK 信号通路、细胞因子通路、凋亡、氧化磷酸化、糖代谢以及脂代谢相关的信号通路等。MAPK 信号通路是众多细胞因子信号通路的共同途径,广泛参与了细胞增殖、分化、凋亡及炎症过程[16]。MAPK 通路的激活与逆转肿瘤化疗耐药及增敏化疗有关,目前成为癌症治疗的热点[16-17]。通过String 在线网站针对筛选出的MAPK 信号通路与CHRDL1 相关的基因进行蛋白互作PPI 网络构建,使用Cytoscape软件再次筛选出与CHRDL1 具有直接互作关系的关键基因CAV1 和FYN。通过CANCERTOOL 在线网站,对CHRDL1/CAV1 及CHRDL1/FYN 的相关性进行了分析,分析结果显示CHRDL1 与CAV1 及FYN 呈正相关。CAV1 是一种完整的膜蛋白,在乳腺癌中,CAV1 的突变增加了乳腺癌的患病风险[18-19]。CAV1 的缺失可能导致肿瘤的增殖、进展和血管生成[20]。CAV1 调控多种癌症相关过程,如细胞转化、肿瘤生长、细胞死亡和存活、血管生成、细胞迁移、侵袭、凋亡和转移[21]。在乳腺癌中,CAV1 基因促进了PI3K/AKT、EGFR-MAPK和MAPK 信号转导[22]。Elias 等[23]的研究表明FYN在三苯氧胺耐药中起重要作用,其在激素受体阳性乳腺癌细胞的表达可能是乳腺癌内分泌治疗效果评估的一个新的重要生物标志物。
综上所述,通过挖掘TCGA 数据库,应用生物信息学技术推测CHRDL1 可能作为潜在的诊断乳腺癌和预测其预后的肿瘤标记物,其内在的调控机制可能是通过调控CAV1 和(或)FYN 介导MAPK 信号通路影响乳腺癌细胞的增殖、侵袭和转移,尚需要进行一系列的实验来验证本文的预测结果,为乳腺癌的早期诊断和疗效评估提供新的靶点。
