酸性稻田土n-damo细菌的多样性和丰度
2021-01-14常佳丽张元勤马旭光
常佳丽,张元勤,江 滔,马旭光
(乐山师范学院 化学与资源环境学院,四川 乐山614000)
0 引言
亚硝酸盐依赖型甲烷厌氧氧化(nitrite-dependent anaerobic methane oxidation, n-damo)是由一类特殊功能菌介导完成、在厌氧条件下耦合甲烷氧化与亚硝酸盐还原的酶促反应过程[1-2]。Ettwig等通过厌氧富集培养和宏基因组学技术手段,在河流底泥中检测到n-damo功能菌的存在,并命名为CandidatusMethylomirabilis oxyfera (M.oxyfera)[3-5]。该类菌在厌氧条件下,通过新型胞内自产氧途径氧化甲烷,并获取生长代谢的能量[5]。甲烷单加氧酶是甲烷氧化代谢途径的关键酶,编码甲烷单加氧酶ɑ亚基的pmoA基因可作为鉴定n-damo细菌的遗传分子标记[6]。
在污水厌氧生物处理领域,存在厌氧段温室气体甲烷无序排放、出水总氮不达标等问题,限制了厌氧技术在水处理领域的大规模推广应用。N-damo过程可利用原位产生的甲烷进行脱氮,无需外源添加其他碳源,同时实现温室气体的减排和出水总氮的达标,是兼具环境友好型和经济节约型的新型脱氮工艺,具有潜在应用前景[7-8]。
近年来,越来越多研究证实n-damo功能菌在自然界中分布广泛,河流底泥、湿地、河口沉积物、淹水稻田土、剩余污泥等生态系统中均不同程度检测到n-damo细菌的存在[9-15],但鲜有研究报道酸性环境中该类菌的赋存情况。此外,n-damo细菌生长代谢速率低、代时长,富集培养耗时久,限制了n-damo在实际污水处理中的推广应用[16]。因此,从其他生境中寻找更多n-damo菌源进行富集培养驯化是未来该领域发展的重要方向之一,也是突破应用瓶颈的关键所在。
基于此,本研究重点关注酸性厌氧生境典型代表——发育自酸性红壤的淹水稻田土,借助微生物分子生态学检测手段,解析其中n-damo细菌的多样性和丰度,以期为后续的富集培养研究提供菌源。
1 材料与方法
1.1 土样的采集及理化性质的测定
本研究选取四川眉山(经纬度:E 103°14′20″;N 45°51′58.87″)稻田土作为研究对象,采用五点随机采样法采集三块不同点位的稻田土壤样品(分别命名为MS-I,MS-II和MS-III)。采样时间为2019年6月,正值水稻种植晚期,稻田为淹水状态,自水土交界面向下延伸20 cm采集表层土样。采集的土样经充分混匀后,无菌条件下,取0.5 g置于2 mL bead-beating离心管中,低温运输至实验室,-80℃保存,用于后续分子生物学检测。剩余土样风干后,剔除植物残体、石砾,研磨过筛,常温保存,用于土壤理化性质的测定。土壤有机质和全氮含量采用国家标准检测方法(GB9848-44和HJ-717-2014)进行测定,土壤分别用去离子水和1 mol/L KCl浸提后,采用酸度计(雷磁,PHS-25)测定pH值。
1.2 土样核酸DNA的提取
采用土壤核酸提取试剂盒(MP FastDNASpin Kit for Soil),参照试剂盒方法说明书,提取土壤样品的核酸DNA。采用1%琼脂糖凝胶电泳,检测土样DNA的提取情况,采用超微量分光光度计(Biofuture,MD2000D)测定核酸浓度和纯度。
1.3 基于n-damo细菌16S rRNA基因和pmoA基因的聚合酶链式反应和克隆测序
以1.2提取的DNA为模板,采用巢式聚合酶链式反应(nested-polymerase chain reaction, nested-PCR)扩增样品中n-damo细菌的16S rRNA基因和pmoA基因,PCR扩增引物参见表1;采用琼脂糖凝胶DNA回收试剂盒(天根生化)纯化目标基因的扩增产物;利用DNA T4连接酶将纯化后的目标基因连接至pMD19-T质粒;将重组质粒转化至大肠杆菌JM109的感受态细胞中,利用蓝白斑筛选,得到含有目标基因的阳性克隆子;分别随机挑选30个含有n-damo细菌16S rRNA基因和pmoA基因的阳性克隆子,送样至上海生工测序平台进行一代Sanger法测序。

续表
1.4 系统发育树的构建
将测序结果上传至NCBI BLAST(https://blast.ncbi.nlm.nih.gov/Blast.cgi),获取相应参比序列,利用MEGA 5.0软件,经Neighbor-joining计算方法比对建树后,明确待测菌株的遗传学分类。
1.5 基于n-damo细菌16S rRNA基因和pmoA基因的定量PCR
以1.2提取的DNA为模板,采用定量PCR技术(quantitative polymerase chain reaction, qPCR)检测样品中n-damo细菌的丰度,qPCR检测试剂为Power SYBR Green PCR Master Mix(Promega),检测仪器为实时荧光定量PCR仪(ABI 7500),qPCR引物详见表1。
2 结果与讨论
2.1 眉山稻田土理化性质分析
本研究共采集3份土样,去离子水浸提pH值均小于6.0,1 mol/L KCl浸提pH值均小于5.5,有机质和总氮含量分别在20~30 g/kg和1.0~2.0 g/kg范围内波动(表2)。根据2015年洪雅县土肥站的土壤养分调研报告[17],本研究所涉及土样均属于微酸性中等养分水平土壤。已知n-damo细菌在中性环境分布广泛[9-15],但鲜有研究报道酸性环境中该类群的多样性及丰度,故解析酸性土样中n-damo菌的赋存情况,对开辟新的菌源意义重大。此外,寻找特殊生境条件下的n-damo细菌,有助于富集培养具有独特生理代谢功能的微生物,例如嗜酸性的n-damo细菌,用于低pH污水的深度脱氮处理。

表2 土样的基本理化性质
2.2 N-damo细菌的定性检测
根据文献可知,巢式PCR第二段扩增反应的目标基因长度分别为460 bp(16S rRNA基因)和389 bp(pmoA基因)[4,6]。本研究成功扩增得到n-damo细菌特异性的16S rRNA基因和pmoA基因,如图1所示,泳道1、3和5为核酸DNA Marker(100 bp ladder,自下而上依次为100 bp ~ 600 bp),2和4分别对应n-damo细菌特异性pmoA基因和16S rRNA基因片段,扩增条带清晰、亮度高、无非特异性扩增条带、扩增长度与文献报道相吻合,可进行后续克隆测序分析。

图1 n-damo细菌16S rRNA和pmoA基因巢式PCR第二段扩增产物电泳图谱
2.3 酸性稻田土中n-damo细菌的多样性
本研究分别对n-damo细菌特异性的16S rRNA基因和pmoA基因进行系统发育分类鉴定,以便更客观准确地反映n-damo细菌的多样性赋存情况。基于n-damo 16S rRNA和pmoA基因序列相似度的聚类分析结果得出,样品中的n-damo细菌分别聚为4和3个OTUs(Operational taxonomic units,可操作分类单元),如图2和图3中的红色圆圈所示。
系统发育分析表明,本研究检测到的n-damo细菌位于CandidatusMethylomirabilis oxyfera系统发育分支,绝大多数酸性稻田土中的n-damo细菌与其它中性生境中检测到的n-damo细菌亲缘关系较近。例如,图2的OTU1与太湖底泥环境样品克隆子TH4-27相似度达98%以上[12],其余OTUs与西溪湿地、红树林湿地沉积物、黄河河口底泥、中性稻田土壤等环境样品中的n-damo细菌均具有较高的相似度(图2和图3)[9-11]。此外,研究还发现,基于n-damo 16S rRNA和pmoA基因的系统发育分析结果均指示,酸性稻田土中存在未知OTU(图2的OTU4和图3的OTU3),与现有数据库中的序列均不匹配,推测本研究土样中可能含有独特的n-damo细菌。后续研究工作可增加样本量,采集更多酸性环境土壤样本,以验证本研究的检测结果。

图2 基于n-damo细菌16S rRNA基因的系统发育树
基于n-damopmoA基因的系统发育树树根为好氧甲烷氧化菌Methylomonas,从图3可知,本研究扩增所得pmoA基因序列全部归属于CandidatusMethylomirabilis oxyfera系统发育分支,未见好氧甲烷氧化菌,证实了cmo182-cmo586这对引物的专一性,利用该引物对能够靶向鉴定环境样品中n-damo厌氧甲烷氧化菌的赋存情况[6]。
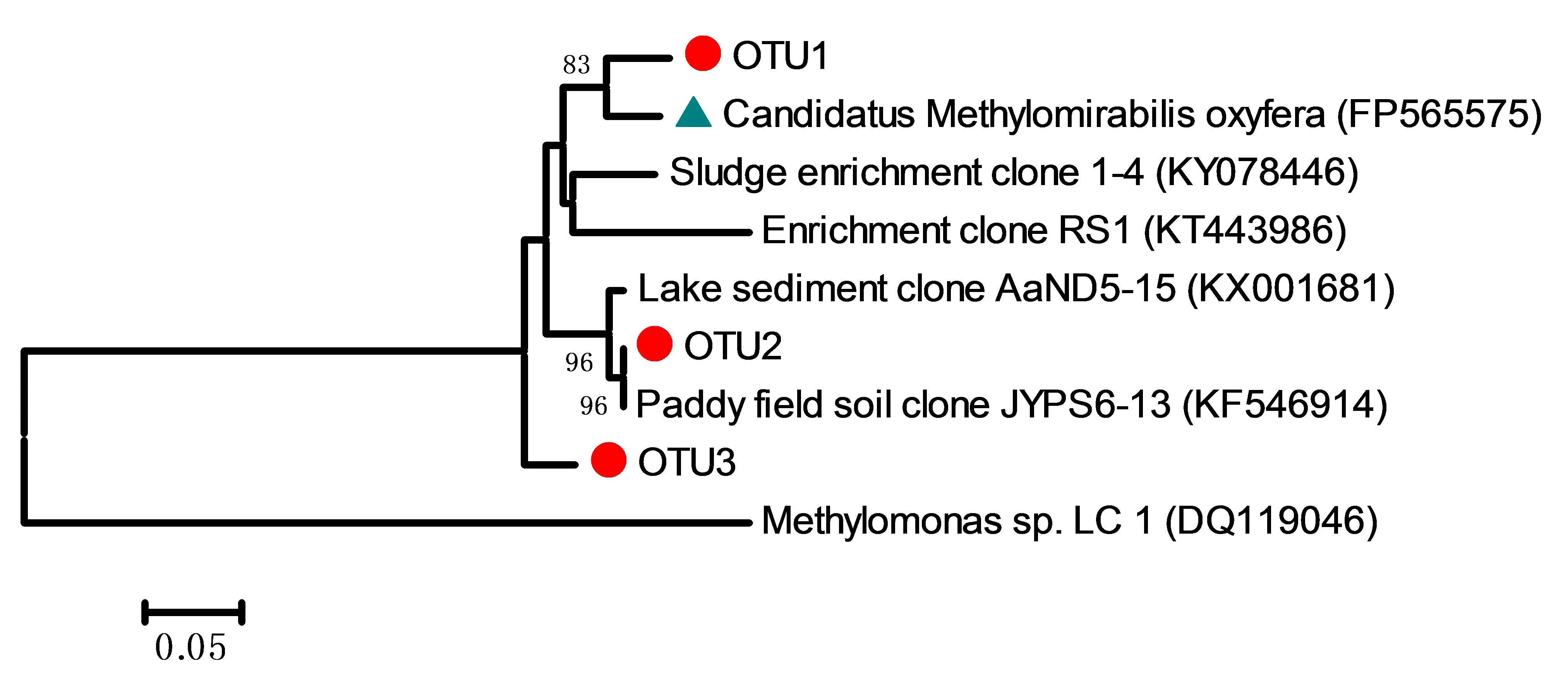
图3 基于n-damo细菌pmoA基因的系统发育树
2.4 酸性稻田土中n-damo细菌的丰度
本研究采用针对n-damo细菌16S rRNA基因和pmoA基因的定量PCR,检测酸性稻田土中n-damo细菌的丰度。研究共检测了三块样田,MS-I、MS-II和MS-III样田中n-damo 16S rRNA基因拷贝浓度分别可达1.25 × 108、2.31 × 108和1.34 × 108拷贝每克干重,pmoA基因拷贝浓度分别为8.66 × 106、2.54 × 107和1.55 × 107拷贝每克干重。其中,pmoA基因普遍比16S rRNA基因丰度低一个数量级,可能的原因在于n-damo细菌单个细胞中含有多个16S rRNA基因拷贝,但通常pmoA基因为单拷贝。对比其他研究发现,不同生境中的n-damo细菌16S rRNA基因丰度值波动较大,其中, 三峡江水水位波动带最低,仅为4.70 ×102copies /g, 长江河口沉积物最高,可达9.77×107copies / g[18]。本研究16S rRNA基因拷贝浓度与长江河口沉积物生境的丰度相当,甚至更高,暗示酸性稻田土可能是n-damo功能菌的适宜生境。
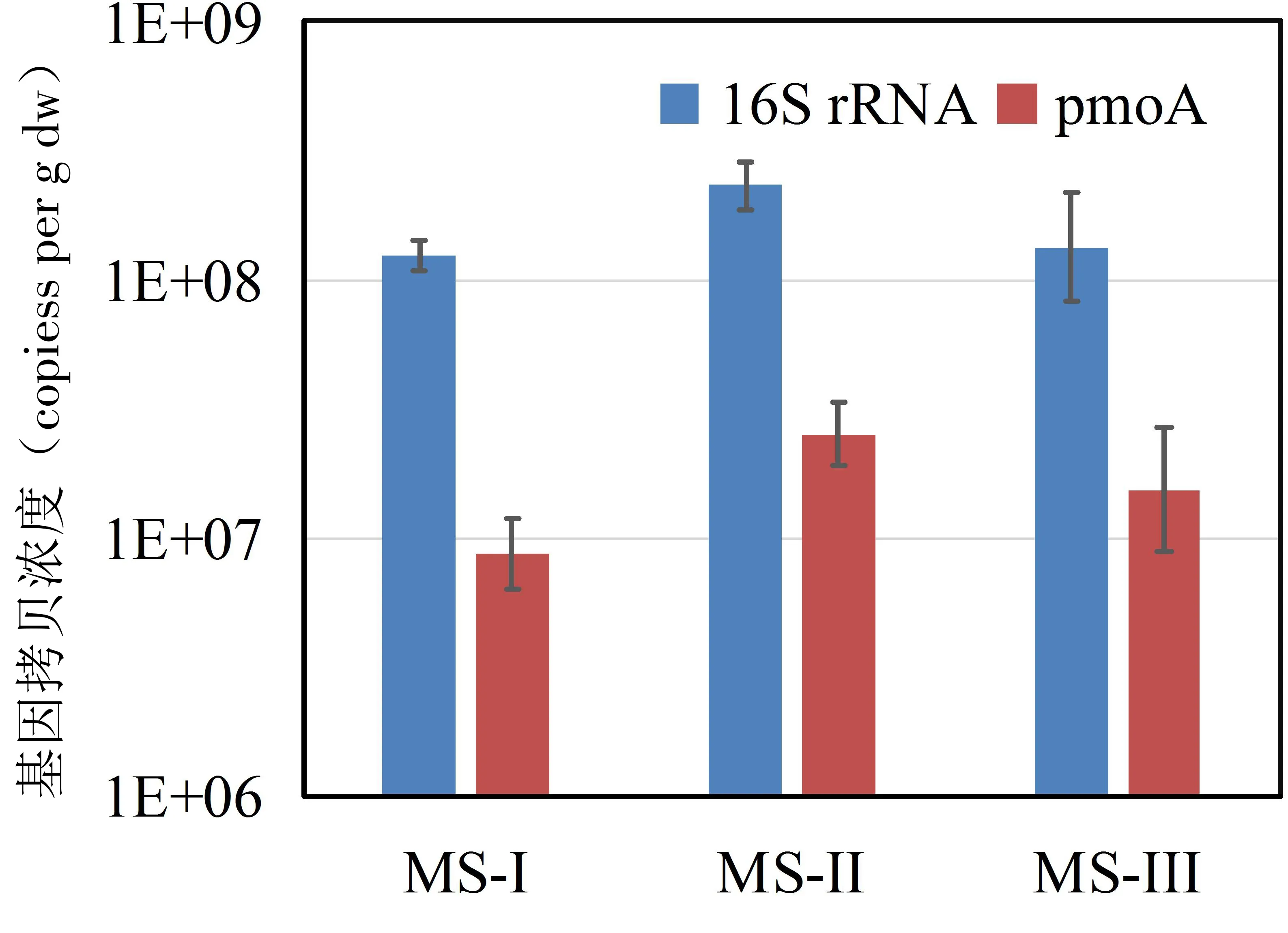
图4 基于n-damo细菌16S rRNA和pmoA基因的定量分析
3 结论与展望
本研究利用分子克隆和定量PCR技术对眉山稻田土壤中亚硝酸盐依赖型甲烷厌氧氧化细菌进行定性定量检测,得出以下结论:
a)采集自眉山洪雅县的稻田土样为弱酸性中等养分水平土壤;
b)酸性稻田土中存在n-damo细菌,且含有潜在的嗜酸性n-damo功能菌;
c)酸性稻田土n-damo细菌丰度较高,与其它中性环境样品n-damo细菌丰度相当。
结合本研究结果和该领域最新进展,对后续研究工作做出下列展望:
a)可针对嗜酸性n-damo功能菌的富集培养开展系列试验;
b)除酸性稻田土,还可采集茶园土、酸性尾矿、酸性泥炭沼泽地等其它低pH生境样品,扩大对酸性环境中n-damo细菌分布多样性的认知;
c)针对多种酸性生境中n-damo细菌群落的分布模式、结构特征以及生理生态影响机制展开研究,筛选富集得到最优功能菌;
d)采用先进的组学技术深度挖掘n-damo细菌的生理代谢功能,为实现其在实际废水深度脱氮中的应用奠定基础。
