融合加权随机森林的自动3D椎骨CT图像主动轮廓分割方法
2021-01-08刘侠,甘权,李冰,刘晓,王波
刘 侠,甘 权,李 冰,刘 晓,王 波
融合加权随机森林的自动3D椎骨CT图像主动轮廓分割方法
刘 侠,甘 权,李 冰,刘 晓,王 波*
哈尔滨理工大学自动化学院,黑龙江 哈尔滨 150080
为了解决CT图像主动轮廓分割方法对初始轮廓的敏感和分割不准确的问题,本文提出一种融合加权随机森林的自动3D椎骨CT主动轮廓分割方法WRF-AC。该方法提出加权随机森林算法和包含边缘能量的主动轮廓能量函数。首先,通过提取椎骨CT的3D Haar-like特征值训练加权随机森林获得的椎骨中心作为分割的初始轮廓,然后,求解包含边缘能量的主动轮廓能量函数最小值完成椎骨CT图像的分割。实验结果表明,本方法在相同数据集上能够更加准确、快速地分割脊柱CT图像提取椎骨部分。
3D分割;CT图像;加权随机森林;主动轮廓

1 引 言
医学图像分割已广泛应用于医学影像诊断技术,并成为临床治疗不可缺少的手段之一[1]。现代医学中使用计算机处理分析脊柱CT图像已经成为一个重要的研究方向,并有十分重要的临床实际应用价值。由于椎体结构复杂,差异较小,人们很难精确地提取椎体感兴趣区域[2]。
在早期的工作中,自适应阈值处理、区域生长和边界调整等无监督图像处理方法应用于图像分割[3],但是准确度较低。在之后的工作中,相关研究人员将改进的水平集框架和分水岭分割方法[4-6]用于椎骨CT图像分割,这些方法虽然对CT图像分割达到了一定的分割精度,但需要手动辅助不具有普适性。随着机器学习等算法的发展,一些学者将随机森林算法用于图像定位分割[7-9],这些方法不能准确分割兴趣区域,有较大误差。现今学者们提出许多基于轮廓模型和形状模型的分割方法[10-12]用于椎骨CT图像的分割。后续研究人员将模糊理论融入主动轮廓模型中并提出基于模糊能量的主动轮廓模型[13-15],此模型对噪声和初始轮廓不敏感可以实现快速收敛,但不能正确分割灰度不均匀图像。在之前的研究中我们尝试直接手动设置初始轮廓,构造交互式半自动分割方案,但是由于人体脊柱的椎骨数量较多且形状相似,手动设置初始轮廓点需要有一定的医学基础,并需要消耗大量的时间。因此,提出一种三维空间内的全自动椎骨CT图像分割方法十分必要。由于上述方法大多数针对二维区域进行图像分割,不能充分利用CT图像的三维空间信息,并且大多数需要根据期望区域对原始模型进行手动修改,这样做不仅耗费人力且耗时巨大。
针对上述问题,本文提出加权随机森林算法解决初始轮廓选取的问题;为了能够更好地突出图像的边缘信息,本文提出包含边缘能量的主动轮廓能量函数。通过提取椎骨CT的3D Haar-like特征值训练加权随机森林获得的椎骨中心并作为分割的初始轮廓位置,求解包含边缘能量的主动轮廓能量函数最小值完成椎骨CT图像的三维分割。实验结果与对比表明,本文方法不仅能够快速准确地分割健康椎骨CT图像,并且能够高效地分割患病椎骨CT图像。
2 理论基础
3D Haar-like[16]特征能在完整描述输入数据的同时提供良好的泛化能力,可充分利用椎骨CT图像的三维特征信息。选取合适的分割初始轮廓其本质是椎骨中心点的选取问题,利用提取的图像特征,随机回归森林能在三维区域内定位椎骨中心候选点,并结合随机分类森林去除假椎骨中心候选点,准确地找到椎骨中心。将3D主动轮廓分割的初始轮廓置于椎骨中心,解决了手动选取初始轮廓的问题和初始轮廓选取不佳导致分割效果不佳的问题。通过定义求解联合边缘能量的主动轮廓能量函数最小值完成分割,解决了主动轮廓分割不能准确分割椎骨边缘的问题。本文的流程图如图1所示。
2.1 特征提取
在提取图像3D Haar-like特征时,为加快计算用式(1)计算积分图,用式(2)计算引用位置强度的和差值sum。



图1 本文的流程图
本文除了在原始CT图像中提取特征之外,在横断面、矢状面、冠状面3个方向上提取梯度特征,这样每个体素就得到了4个特征,分别对体素单元使用[2,2,2]、[4,4,4]和[8,8,8]盒过滤器[17],盒过滤器(box filter)的响应作为CT图像中单个体素的3D Haar-like特征值,最终获得一个48维的特征来表示每个体素。
2.2 随机森林
随机决策森林(Random decision forests)是根据决策树算法,由Breiman[18]提出的利用多棵决策树对样本进行训练并预测的一种分类器。根据应用目的的不同,随机森林分为分类森林和回归森林,分类森林与回归森林在决策树每个节点的判别原理上是基本相似的,只是叶子节点的输出原理不同[19]。
本文中利用随机回归森林得到椎骨中心候选点,通过Mean-shift算法对候选点进行聚类操作,减少候选点个数,利用随机分类森林去除非椎骨区域候选点,最终建立3D距离图谱,选取距离最小的点作为椎骨中心点。
2.3 主动轮廓分割
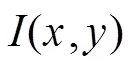
定义为
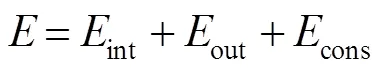

通过求解式(4)的最小值,使轮廓曲线收敛在图像的最大梯度点,而图像的最大梯度一般在目标的边缘处取得,也即检测出了目标的边缘。
3 分割算法
3.1 问题描述
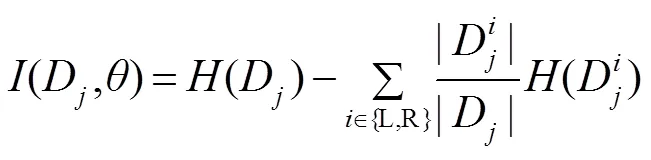
3.2 算法改进
3.2.1 加权随机森林
随机森林算法中样本特征相对集中,不同特征对结果的影响会有很大差异,导致有些决策树对结果有很大影响,而有些决策树则不会产生显著的影响结果。不同决策树选择不同训练样本特征,导致不同决策树的性能不同。为了提高决策树的性能,有必要改进决策树在随机森林中所占权重。为此,本文对传统随机森林决策树的权重进行改进权衡。加权投票时,如果权重合理,可以提高随机森林的分类回归效果,但如果权重不适合则会导致数据过拟合,降低决策效果。因此随机森林中决策树的性能越好,其权重应越高。本文由后验概率结果表示的决策树可靠性,分别提出改进的加权随机森林算法。
本文提出随机分类森林权重公式为
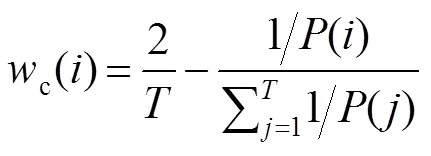
同理,随机回归森林权重公式为

本文提出的加权参数均满足归一化:
会计的职业道德建设是一项长期的、艰苦的实践活动,任重而道远,要靠我们每个会计人员的不懈努力。有道是:“人无信不立,国无信不强”,只有进一步加强会计职业道德建设,全面提升会计从业人员的道德素质,牢固树立良好的职业道德,让“诚实守信”这一道德规范深深植根于每个会计人员的心中,我国的会计职业道德水平才会跃上一个新的台阶。
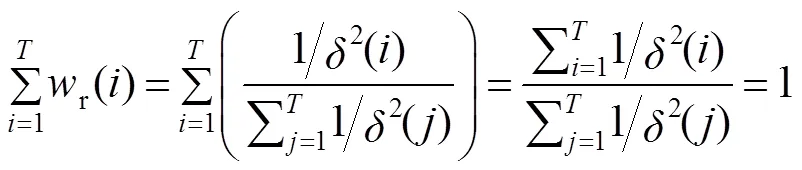

3.2.2 3D边缘能量的主动轮廓分割


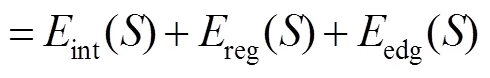
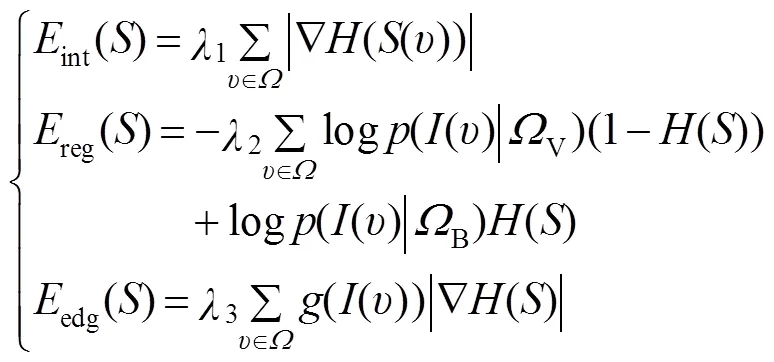



由于本文方法第一次进行样本训练需要一定的时间,在之后的测试集实际应用中不需要再次训练模型,为了减少训练时间,本文在以下几个方面做出改进:
1) 为加快运算,将权重小于0.005的树权重赋值为0,剩余权重按比例重新赋值,减少决策树数量;
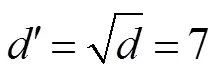
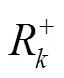
4) 为了能更准确找出中心点位置,选择回归后的距离图谱中值最小的150个点进行Mean-shift聚类得到更准确的椎骨中心点的候选点。
综上所述,本文提出的融合加权随机森林的自动3D椎骨CT图像主动轮廓分割方法具体实现步骤如下。
步骤1:导入训练集后,提取图像特征向量,构建随机森林并初始化参数;
步骤2:训练随机森林,根据式(6)、式(7)进行加权运算,将权重小于0.005的树权重重新赋值为0,其余权重按比例重新赋值;
步骤3:重复步骤2,直至到达预定迭代次数,输出椎骨中心点位置;
步骤4:根据式(10)、式(11)定义联合边缘能量的主动轮廓分割能量函数,并初始化参数;
步骤5:以随机森林输出的中心点为初始轮廓中心构造初始轮廓球;
步骤6:使用梯度下降法根据式(12)更新距离符号函数S ;
4 实验结果及分析
4.1 实验环境与数据集
本文分割方法在公开的CT数据集上进行评估,本文数据来源于Forsberg[24]与Hammernik[25]实验所用数据集,CSI(Collaborative Platform for Research on Spine Imaging and Image Analysis)挑战提供发布于Spine Web网站(http://spineweb.digitalimaginggroup.ca /spineweb/index.php?n=Main.Datasets)上,该数据共20组CT图像(包括健康椎骨和患病椎骨),总计10991张切片图像,数据集覆盖整个胸椎和腰椎,没有静脉对比。本文椎骨分割算法实验环境为Intel(R) Core(TM) 3.4 GHz CPU,32 GB RAM,4 GHz GPU,Caffe,Matlab R2016b。我们用ITK和VTK工具包结合Python语言编程实现分割结果可视化。为使图像强度大致在相同的范围之内,通过调整窗宽/窗位对图像进行归一化处理。
4.2 分割结果
本文从分割常用指标DC(Dice coeffcient,用DC表示)、ASD(absolute surface distance,用ASD表示)、正确分类率(CCR,简写为CCR)和Jaccard[26]对本文提出的方法进行评估和对比,其定义如下:
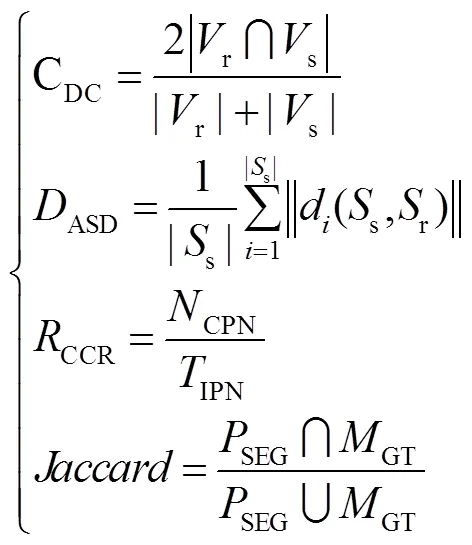

图3 椎骨中心及初始轮廓
4.3 随机森林中心点定位
4.3.1 初始轮廓选取
本文使用均值漂移聚类来找到具有最高回归概率的150个点中的中心候选点,通过随机分类森林去除非椎骨点,将候选点中概率最大的点确定为椎骨中心点,建立一个三维距离图,图2(a)给出了第4节腰椎的回归点及其中心点;图2(b)显示了回归森林得到的回归点,蓝色点为Mean-shift聚类中心,经过随机分类森林去除非椎骨区域的点为椎骨中心候选点,黄色点为随机森林最终选定的椎骨中心点,红色点为专家标记中心点。
为了避免分割时陷入局部能量极小值,将初始轮廓球置于随机森林选取的中心点位置,设定曲线由内向外演化,如图3所示。图3(a)为健康椎骨的中心点(黄色圆点)及初始轮廓球(白色圆圈),图3(b)对应为患病椎骨的中心点及初始轮廓球。
4.3.2 初始轮廓和边缘能量
如图4所示,从左到右、从上到下依次为CT原始图像(a)、无边缘能量主动轮廓分割的任意初始轮廓(b)、边缘能量主动轮廓分割的任意初始轮廓(c)、无边缘能量主动轮廓分割(weighted random forest,WRF)初始轮廓(d)、WRF-EAC(weighted random forest-active contour)分割(e)和通过后操作平滑边界最终分割的结果(f)。初始轮廓分割不能将椎骨区域和目标外部区域正确地分割。随机森林位置的主动轮廓分割产生分割缺陷和过分割。与前三种方法相比,该方法可以提供椎骨的准确分割。

图4 控制变量实验分割结果
分割效果的定量表征结果如表1所示。不同方法对于多个椎骨的分割结果如图5所示,第一行是任意初始轮廓和非边缘能量项方法的分割结果。该方法不能完成CT图像的器官分割。第二是任意初始轮廓和边缘能量项方法的分割结果。该方法可以找到准确的器官轮廓并完成分割,并且一些非器官部分也被分割是不够的。第三行是权重RF初始轮廓和非边缘能量项方法的分割结果。但是没有足够的细分和过度细分。第四行是本文的分割结果,该方法可以准确地分割目标区域并正确分割目标区域,分割结果最佳。
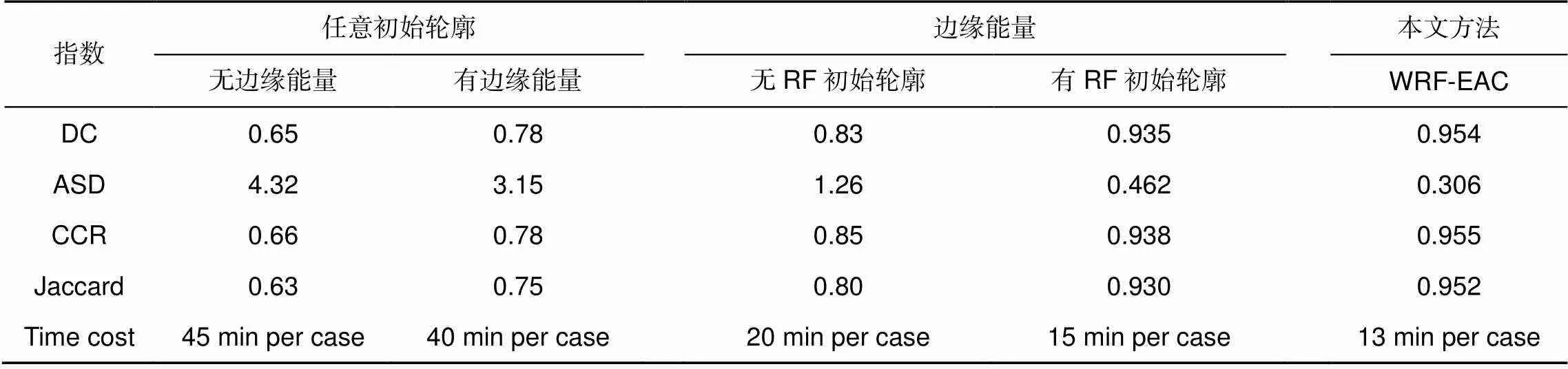
表1 定量实验分割结果
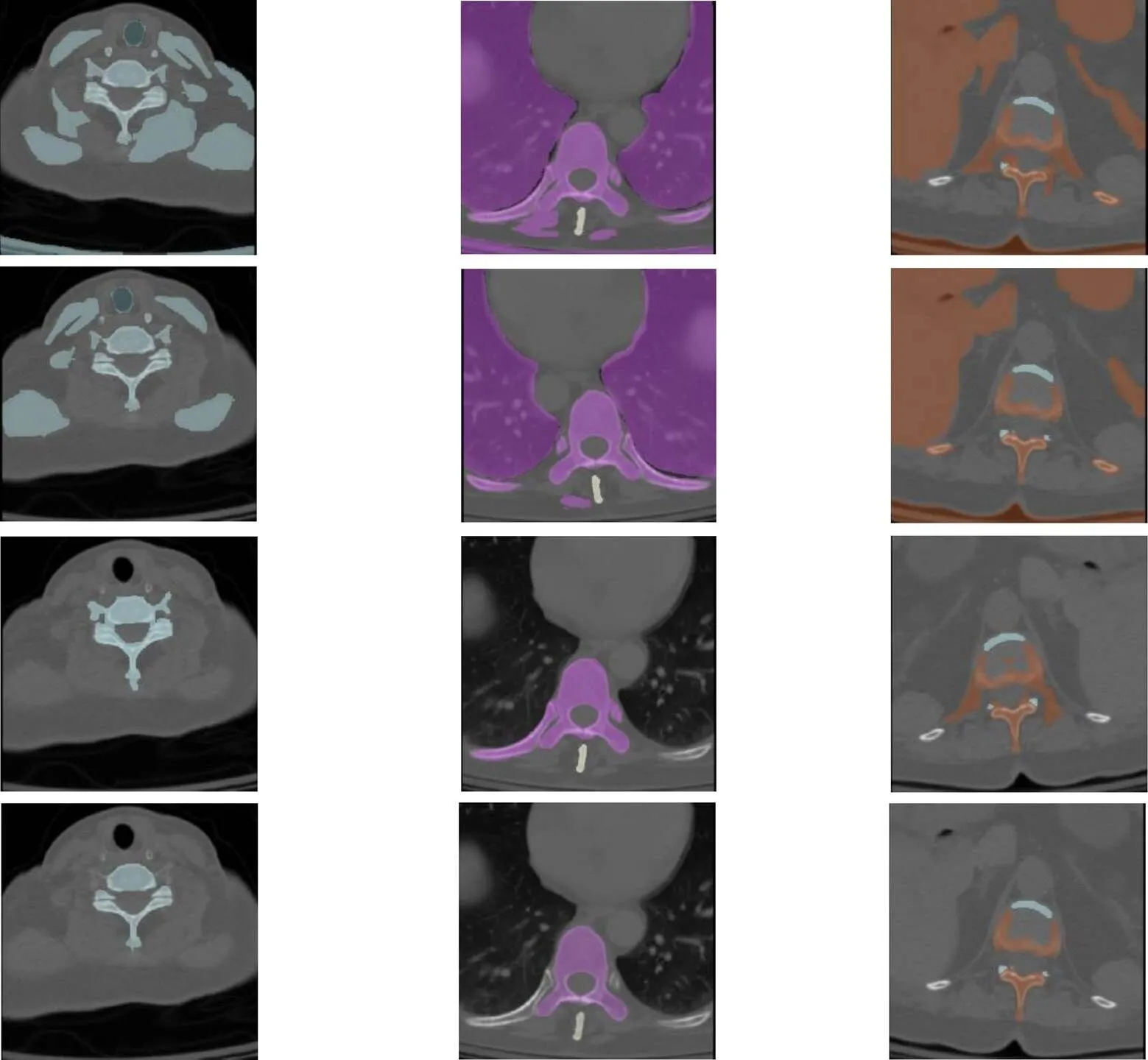
图5 分析实验分割结果
4.4 本文分割方法结果比较
为了便于比较,将脊柱分成三个区段(T1~T6,T7~T12和L1~L5)并计算分割的DC和ASD系数。本文的脊柱CT分割方法对健康和患病的椎骨具有良好的分割效果,具体系数对比结果如图6所示,分割效果的各评价指标如表2所示。由于健康椎骨的形状和形状差异较小,患病椎体的差异显著,健康椎骨分割的结果明显优于患病椎骨的分割结果。胸椎的尺寸和骨密度小于腰椎的尺寸和骨密度,肋骨关节和周围结构的成像更复杂。这些原因导致腰椎分割的结果优于胸椎分割。图7为10例健康和10例非健康椎体的CT数据分割结果的DC系数和ASD系数。无论DC系数或ASD系数如何,健康的椎骨分割结果明显优于患病的椎骨分割结果。DC系数越大,分割结果的ASD系数越低,反之亦然。
如图8所示,第一行是健康椎体CT的横断面分割结果(a);第二行是患病椎体患病部位的横断面分割结果(b);第三行是整个椎骨表面的水平分割结果(c);第四行是对应于患者椎骨的分割结果(d)。将椎骨分割结果(红色部分)放在专家手动分割数据(白色部分)和原始CT数据上,第五行是椎骨的部分矢状切面(e),第六行是整个椎骨冠状面的分割结果(f)。

图6 DC和ASD统计结果

图7 20例DC系数和ASD系数

表2 分割结果各评价指标
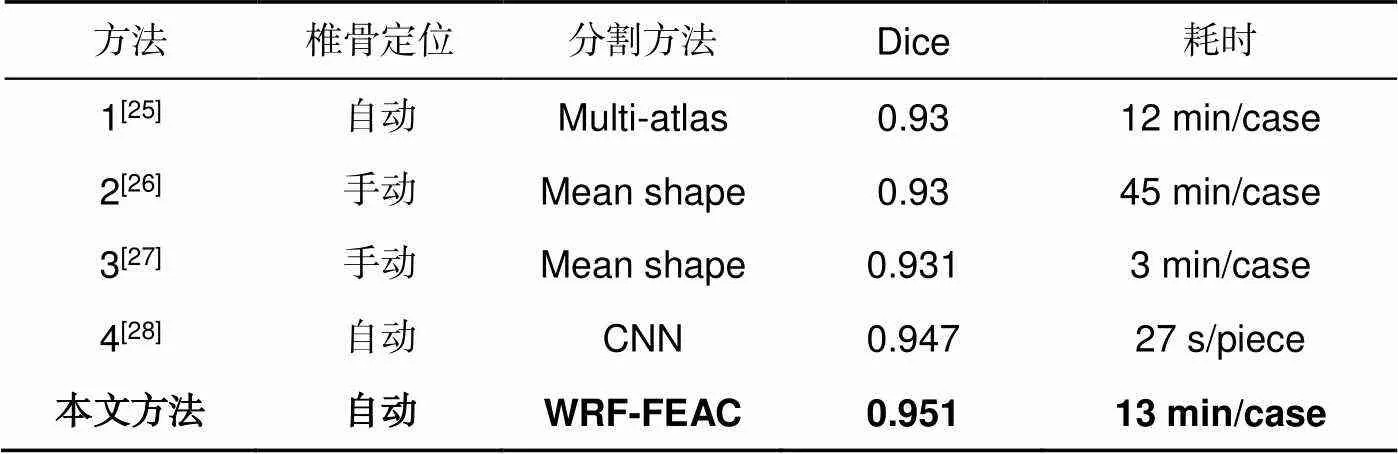
表3 分割结果的DC系数比较

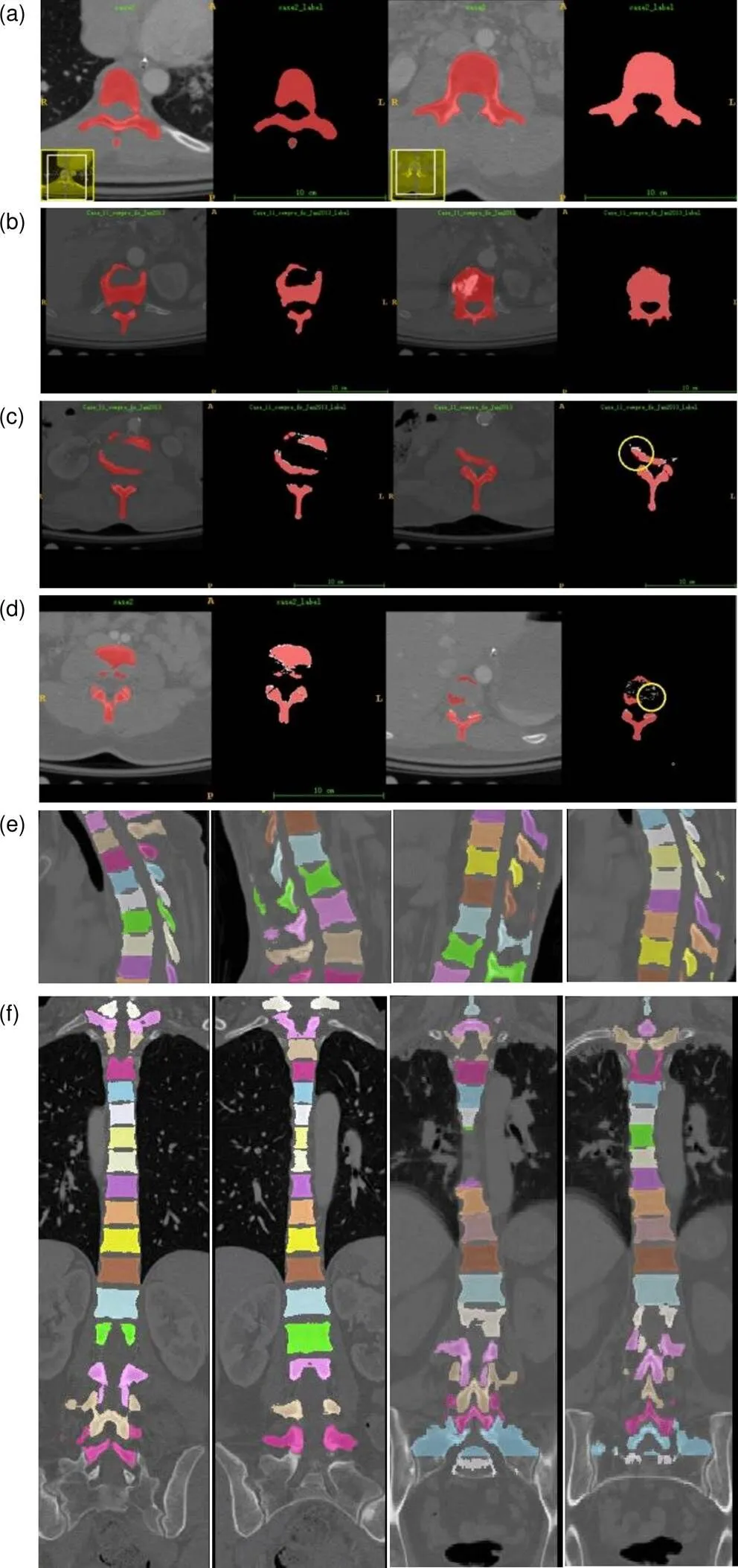
图8 方法分割结果
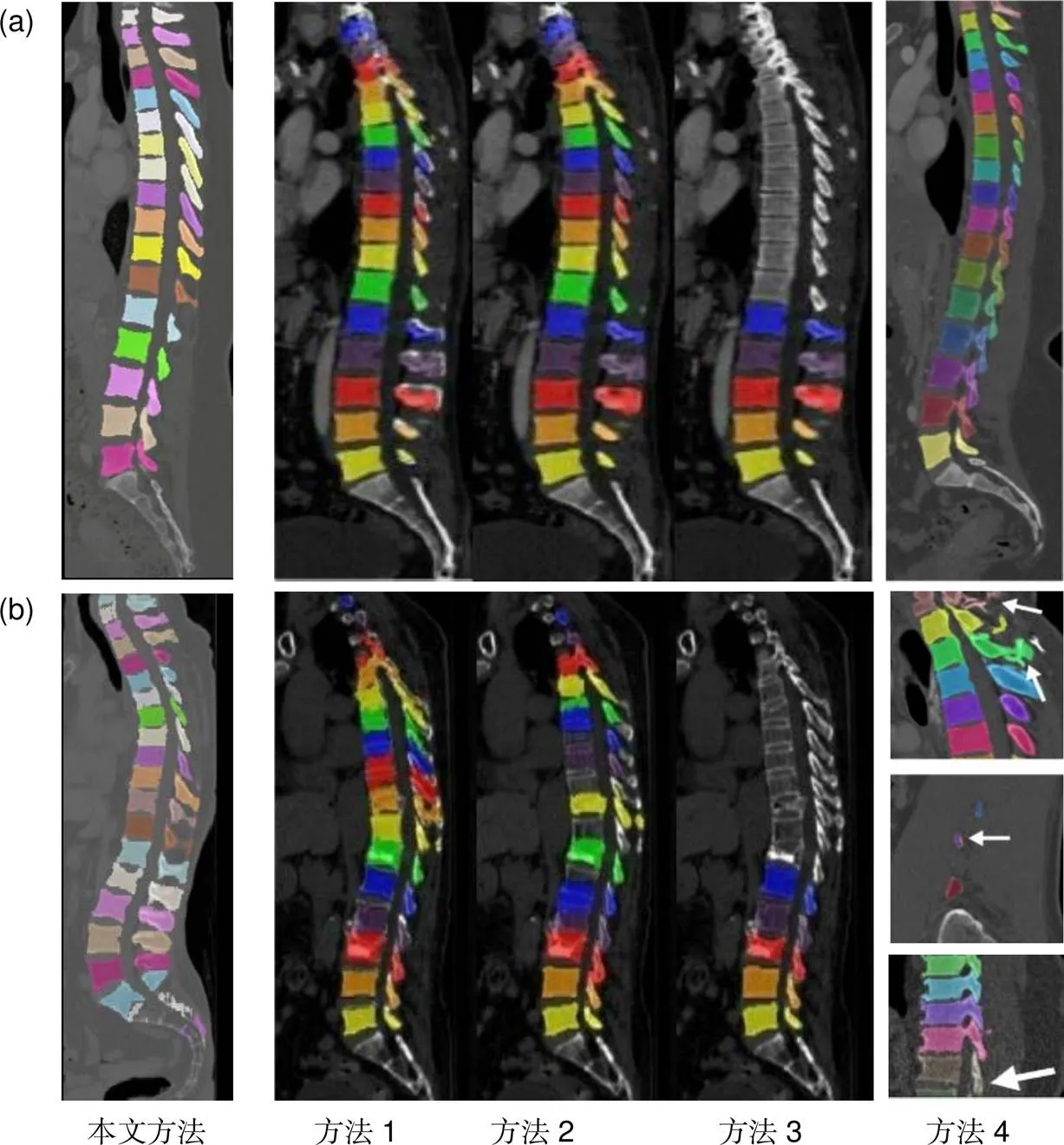
图9 本文方法与其他方法之间分割效果的比较
为了能够更直观的观察椎体的分割效果,用VTK对脊柱分割结果进行三维重建如图10所示。图10(a)为健康椎骨分割结果三维重建图,图10(b)为患病椎骨分割结果三维重建图。
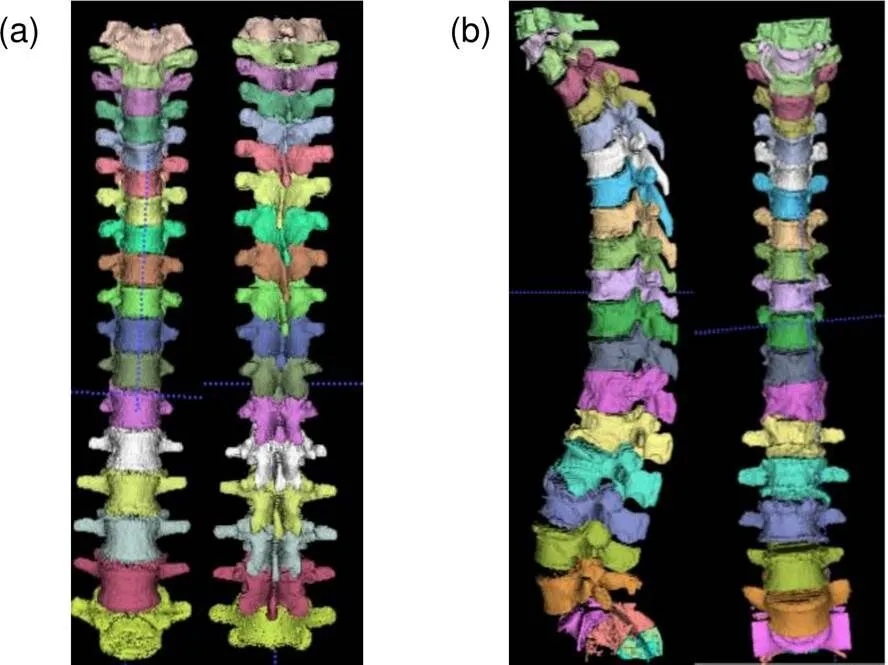
图10 椎骨三维重建可视化效果
5 结 论
实验结果表明,本文提出的融合加权随机森林的3D椎骨CT图像主动轮廓分割方法平均分割准确率能达到92%以上,虽然第一次进行样本训练需要一定的时间,但在之后的测试集实际应用中不需要再次训练模型,本文提出的分割方法能够帮助临床医生快速、高效地进行诊断治疗。本文方法存在一定的优势:自动定位椎骨中心并准确分割椎骨区域;脊柱CT图像易于获取,利用本文提出的分割模型,进行椎骨区域分割,结合后续的三维重建、3D打印工作易于帮助临床应用、治疗。由于椎骨CT数据较难收集,在之后的研究中需要补充更多分割数据进行模型训练,提高分割模型的分割准确率,实现脊柱的多级分割。
[1] Roth H R, Farag A, Lu L,. Deep convolutional networks for pancreas segmentation in CT imaging[J]., 2015, 9413: 94131G.
[2] Glocker B, Feulner J, Criminisi A,. Automatic localization and identification of vertebrae in arbitrary field-of-view CT scans[M]//Ayache N, Delingette H, Golland P,.. Berlin Heidelberg: Springer, 2012: 590–598.
[3] Kang Y, Engelke K, Kalender W A. A new accurate and precise 3-D segmentation method for skeletal structures in volumetric CT data[J]., 2003, 22(5): 586–598.
[4] Lim P H, Bagci U, Bai L. Introducing Willmore flow into level set segmentation of spinal vertebrae[J]., 2013, 60(1): 115–122.
[5] Huang J Y, Jian F Z, Wu H,. An improved level set method for vertebra CT image segmentation[J]., 2013, 12: 48.
[6] Tang L M, Tian X Q, Huang D R,Image segmentation model combined with FCMS and variational level set[J]., 2014, 40(6): 1233–1248.
唐利明, 田学全, 黄大荣, 等. 结合FCMS与变分水平集的图像分割模型[J]. 自动化学报, 2014, 40(6): 1233–1248.
[7] Criminisi A, Shotton J, Robertson D,. Regression forests for efficient anatomy detection and localization in CT studies[M]//Menze B, Langs G, Tu Z W,.. Berlin Heidelberg: Springer, 2010: 106–117.
[8] Criminisi A, Robertson D, Konukoglu E,. Regression forests for efficient anatomy detection and localization in computed tomography scans[J]., 2013, 17(8): 1293–1303.
[9] Cuingnet R, Prevost R, Lesage D,. Automatic detection and segmentation of kidneys in 3D CT images using random forests[M]//Ayache N, Delingette H, Golland P,.Berlin Heidelberg: Springer, 2012: 66–74.
[10] Klinder T, Ostermann J, Ehm M,. Automated model-based vertebra detection, identification, and segmentation in CT images[J]., 2009, 13(3): 471–482.
[11] Huang S H, Chu Y H, Lai S H,. Learning-based vertebra detection and iterative normalized-cut segmentation for spinal MRI[J]., 2009, 28(10): 1595–1605.
[12] Roberts M G, Cootes T F, Pacheco E,. Segmentation of lumbar vertebrae using part-based graphs and active appearance models[M]//Yang G Z, Hawkes D, Rueckert D,.. Berlin Heidelberg: Springer, 2009: 1017–1024.
[13] Chen K, Li B, Tian L F. A segmentation algorithm of pulmonary nodules using active contour model based on fuzzy speed function[J]., 2013, 39(8): 1257–1264.
陈侃, 李彬, 田联房. 基于模糊速度函数的活动轮廓模型的肺结节分割[J]. 自动化学报, 2013, 39(8): 1257–1264.
[14] Thieu Q T, Luong M, Rocchisani J M,. Efficient segmentation with the convex local-global fuzzy Gaussian distribution active contour for medical applications[J]., 2015, 75(1–2): 249–266.
[15] Sun W Y, Dong E Q, Cao Z L,A robust local segmentation method based on fuzzy-energy based active contour[J]., 2017, 43(4): 611–621.
孙文燕, 董恩清, 曹祝楼, 等. 一种基于模糊主动轮廓的鲁棒局部分割方法[J]. 自动化学报, 2017, 43(4): 611–621.
[16] Viola P, Jones M J. Robust real-time face detection[J]., 2004, 57(2): 137–154.
[17] Tu Z W, Zhou X S, Bogoni L,. Probabilistic 3D polyp detection in CT images: the role of sample alignment[C]//, New York, 2006: 1544–1551.
[18] Breiman L. Random forests[J]., 2001, 45: 5–32.
[19] Li X H. Using “random forest” for classification and regression[J]., 2013, 50(4): 1190–1197.
李欣海. 随机森林模型在分类与回归分析中的应用[J]. 应用昆虫学报, 2013, 50(4): 1190–1197.
[20] Kass M, Witkin A, Terzopoulos D. Snake: active contour models[J]., 1988, 1(4): 321–331.
[21] Malladi R, Sethian J A, Vemuri B C. Shape modeling with front propagation: a level set approach[J]., 1995, 17(2): 158–175.
[22] Hajiaghayi M, Groves E M, Jafarkhani H,. A 3-D active contour method for automated segmentation of the left ventricle from magnetic resonance images[J]., 2017, 64(1): 134–144.
[23] Osher S, Fedkiw R, Piechor K. Level set methods and dynamic implicit surfaces[J]., 2004, 57(3): B15.
[24] Forsberg D. Atlas-based segmentation of the thoracic and lumbar vertebrae[M]//Yao J H, Glocker B, Klinder T,.Cham: Springer, 2015: 215–220.
[25] Hammernik K, Ebner T, Stern D,. Vertebrae segmentation in 3D CT Images based on a variational framework[M]// Yao J H, Glocker B, Klinder T,.Cham: Springer, 2015: 227–233.
[26] Dice L R. Measures of the amount of ecologic association between species[J]., 1945, 26(3): 297–302.
[27] Castro-Mateos I, Pozo J M, Lazary A,. 3D vertebra segmentation by feature selection active shape model[M]//Yao J H, Glocker B, Klinder T,.. Cham: Springer, 2015: 241–245.
Automatic 3D vertebrae CT image active contour segmentation method based on weighted random forest
Liu Xia, Gan Quan, Li Bing, Liu Xiao, Wang Bo*
School of Automation, Harbin University of Science and Technology, Harbin, Heilongjiang 150080, China

Random forest center point positioning. (a) 3D distance map; (b) Regression points obtained by regression forest
Overview:Medical image segmentation has been widely used in medical image diagnosis technology and has become one of the indispensable means of clinical treatment. The use of computer processing to analyze spine CT images in modern medicine has become an important research direction, and has very important clinical application values. Due to the complicated structure of the vertebral body and the small difference, it is difficult for people to accurately extract the vertebral body of interest. In previous studies, we tried to manually set the initial contour directly to construct an interactive semi-automatic segmentation scheme. However, due to a large number of vertebrae in the human spine and the similar shape of the vertebrae, the manual setting of initial contour points requires a certain medical foundation and consumes much time. In order to solve the problems of sensitive initial contours and inaccurate segmentation caused by active contour segmentation of CT images, this paper proposes an automatic 3D vertebral CT active contour segmentation method combined weighted random forest called “WRF-AC”. This method proposes a weighted random forest algorithm and an active contour energy function that includes edge energy. First, the weighted random forest is trained by extracting 3D Haar-like feature values of the vertebra CT, and the ‘vertebra center’ obtained is used as the initial contour of the segmentation. Then, the segmentation of the vertebra CT image is completed by solving the active contour energy function minimum containing the edge energy. The experimental results show that this method can segment the spine CT images more accurately and quickly on the same datasets to extract the vertebrae. Experimental results show that the average segmentation accuracy of the active contour segmentation method of 3D vertebra CT image fusion weighted random forest proposed in this paper can reach more than 92%. This method has certain advantages: it can automatically locate the center of the vertebrae and accurately segment the vertebral area; it is easy to obtain CT images of the spine, using the segmentation model proposed in this paper to segment the vertebral area, and combining the subsequent 3D reconstruction and 3D printing can easily help clinical applications and treatment. Due to the difficulty in collecting CT data of vertebrae, it is necessary to add more segmentation data for model training in the subsequent research to improve the segmentation accuracy of the segmentation model and achieve multi-level segmentation of the spine.
Citation: Liu X, Gan Q, Li B,. Automatic 3D vertebrae CT image active contour segmentation method based on weighted random forest[J]., 2020,47(12): 200002
* E-mail: hust_wb@126.com
Automatic 3D vertebrae CT image active contour segmentation method based on weighted random forest
Liu Xia, Gan Quan, Li Bing, Liu Xiao, Wang Bo*
School of Automation, Harbin University of Science and Technology, Harbin, Heilongjiang 150080, China
In order to solve the problems of sensitive initial contours and inaccurate segmentation caused by active contour segmentation of CT images, this paper proposes an automatic 3D vertebral CT active contour segmentation method combined weighted random forest called “WRF-AC”. This method proposes a weighted random forest algorithm and an active contour energy function that includes edge energy. First, the weighted random forest is trained by extracting 3D Haar-like feature values of the vertebra CT, and the 'vertebra center' obtained is used as the initial contour of the segmentation. Then, the segmentation of the vertebra CT image is completed by solving the active contour energy function minimum containing the edge energy. The experimental results show that this method can segment the spine CT images more accurately and quickly on the same datasets to extract the vertebrae.
3D segmentation; CT images; weighted random forest; active contour
National Natural Science Foundation of China (61172167) and Natural Science Foundation of Heilongjiang Province (QC2017076)
10.12086/oee.2020.200002
TP391.4
A
刘侠,甘权,李冰,等. 融合加权随机森林的自动3D椎骨CT图像主动轮廓分割方法[J]. 光电工程,2020,47(12): 200002
: Liu X, Gan Q, Li B,Automatic 3D vertebrae CT image active contour segmentation method based on weighted random forest[J]., 2020, 47(12): 200002
2020-01-02;
2020-04-16基金项目:国家自然科学基金资助项目(61172167);黑龙江省自然科学基金资助项目(QC2017076)
刘侠(1975-),男,博士,教授,硕士生导师,主要从事模式识别,图像处理的研究。E-mail:liuxia@hrbust.edu.cn
王波(1982-),男,博士,副教授,硕士生导师,主要从事模式识别,机器学习,医学影像分析与处理,自然语言处理等的研究。E-mail:hust_wb@126.com
