Curcin和curcin C与腺嘌呤互作位点的比较分析
2020-12-04邓聿杉
吴 朋, 邓聿杉, 陈 放, 徐 莺
(四川大学生命科学学院 生物资源与生态环境教育部重点实验室,成都 610065)
1 引 言
核糖体失活蛋白(Ribosome inactivating proteins,RIPs)能通过其rRNA N-糖苷酶活性去除核糖体28S rRNA上特异位置腺嘌呤的糖苷键从而抑制蛋白质合成[1].因其具有抗肿瘤、抗病毒和抗真菌等多种生物学活性而受到关注[2].
Curcin和curcin C均是从大戟科麻疯树属植物麻疯树(Jatrophacurcas)中分离得到的核糖体失活蛋白.1976年,Stripe首次从麻疯树胚乳中分离出curcin[3],Barbieri等认为其属于I型RIPs[4].2002年,林娟[5]等人确定其分子量为28.2 kDa,具有rRNA N-糖苷酶活性.2017年,Zhang等人[6]从麻疯树完全伸展开的子叶中分离纯化出一种新型I型核糖体失活蛋白curcin C.研究表明,curcin和curcin C的氨基酸序列同源性达到81.88%,但抗肿瘤活性存在较大的效率差异(例如后者对骨肉瘤细胞的细胞毒性可达前者的10倍)[6-7],这表明两者的催化效率或作用机制可能存在差异.我们注意到两种核糖体失活蛋白的蛋白体外翻译抑制的IC50分别为0.19和0.05nM,暗示它们的rRNA N-糖苷酶活性可能同样也存在差异[6-7].由于造成酶活性差异的主要原因是蛋白的底物结合和催化中心,RIPs在完成其rRNA N-糖苷酶活性时,首先需要与底物分子特异位点的腺嘌呤结合.两种蛋白是否因为在结合腺嘌呤上存在差异,因而进一步导致活性的不同目前还不清楚.为了回答这一问题,本文利用生物信息学方法,首先预测出curcin、curcin C蛋白的三维结构模型,并对模型质量进行优化和评估,之后采用分子对接方法探究curcin,curcin C与腺嘌呤相互作用位点的比较分析,为阐明其活性差异的潜在机制提供一种的线索.
2 材料与方法
2.1 材 料
麻疯树核糖体失活蛋白curcin (GenBank登记号:AAL58089.1) 以及curcin C (GenBank登记号:ABZ04128.1) 的氨基酸序列由NCBI蛋白数据库 (http://www.ncbi.nlm. nih.gov) 检索获得.
2.2 方 法
2.2.1 序列检索与三维结构预测 利用在线预测软件SWISSMODEL[8-12](https://swissmodel.expasy.org/)预测curcin、curcin C的三维模型.
2.2.2 模型优化 采用SYBYL软件,选用AMBER7 FF99力场和AMBER电荷对curcin和curcin C模型进行能量最小化处理.
2.2.3 模型评估 为了进一步确定模型的有效性,我们采用PROCHECK[13]、VERIFY 3D[14-15]、ERRAT[16]对模型进行评估.
2.2.4 分子对接 分子对接实验使用AutoDock 4.2[17],以鉴定curcin、curcin C与腺嘌呤可能的相互作用残基.对蛋白质进行预处理:加氢、计算电荷、分配原子AD4类型;对腺嘌呤小分子做加氢以及计算电荷处理.设置GridBox大小在X、Y、Z轴分别为60、64、50 Å,间距为0.375 Å[18].蛋白质为刚性结构,小分子配体为柔性结构.选择遗传算法(Genetic Alogorithm),寻找蛋白质受体上配体合适的结合位置.
3 结果与讨论
3.1 Curcin和curcin C的三维建模和初步评估
通过序列和结构比对,寻找一个与curcin、curcin C同源的由实验测定的蛋白质结构,本次建模选择的模型PDB id为1IFS,为蓖麻ricinA链和游离腺嘌呤形成的复合体[19].其中ricin A链与curcin、curcin C的氨基酸序列相似度分别为36.48%、34.69%,均大于30%,满足了SWISSMODEL三维结构模型预测的需求.在此基础上,预测出curcin、curcin C的三维结构示意图(图1A、B).为了判断所预测结构的质量,采取QMEAN软件对上述预测结果进行检验(图1C、D).分值分别是-2.97、-2.87,表明curcin、curcin C模型质量合格.其中curcin模型具有10个α-螺旋,10个β-折叠;curcin C模型具有12个α-螺旋,8个β-折叠.导致α-螺旋差异的两处位置分别是curcin的Ala56-Ala57对应curcin C中为 Thr56-Thr57、Pro134对应为Ser134.造成β-折叠差异的两处位置分别是curcin的Ser78对应curcin C的Leu78、Glu81对应为Lys81.
3.2 Curcin和curcin C模型的评估
蛋白质三维结构的稳定性主要取决于原子的最小能量排列,通过能量最小化的优化可以使预测的蛋白质结构更接近自然状态[18].将上述预测出的模型经SYBYL软件能量最小化优化后,curcin、curcin C模型总能量分别从-3 033.609 kcals/mol和-3 072.181 kcals/mol降低至-5 073.993 kcals/mol和-5 316.474 kcals/mol.为了进一步验证优化前后curcin、curcin C模型的准确性,采取PROCHECK、VERIFY 3D、ERRAT三个软件对模型质量进行评估.结果如下:应用PROCHECK的评估表明,优化前curcin、curcin C的允许区域和额外允许区域百分比之和分别为98.1%(图2A)和97.8%(图2B),优化后分别为97.7%(图2C)和97.8%(图2D).可以发现优化对于模型的分值影响不大,所有的分值均大于90%.对curcin C来说,没有发生变化;对于curcin来说,分值甚至减小.VERIFY 3D评估表明,优化使得两个模型的分值都得到提高.前curcin、curcin C分别有88.21%(图2E)、80.97%(图2F)的残基拥有大于0.2的3D/1D值,优化后分别为94.31%(图2G)、86.23%(图2H).ERRAT的评估结果类似于VERIFY 3D,优化前curcin、curcin C的得分分别是96.581(图2I)、97.046(图2J),优化后分别为98.312(图2K)、90.377(图2L),即优化使得模型的质量提高,不过值得指出的是优化前后的分值均大于85.综上分析结果提示优化前后的curcin和curcin C模型均具有较好的质量,都可用于下一步分析.
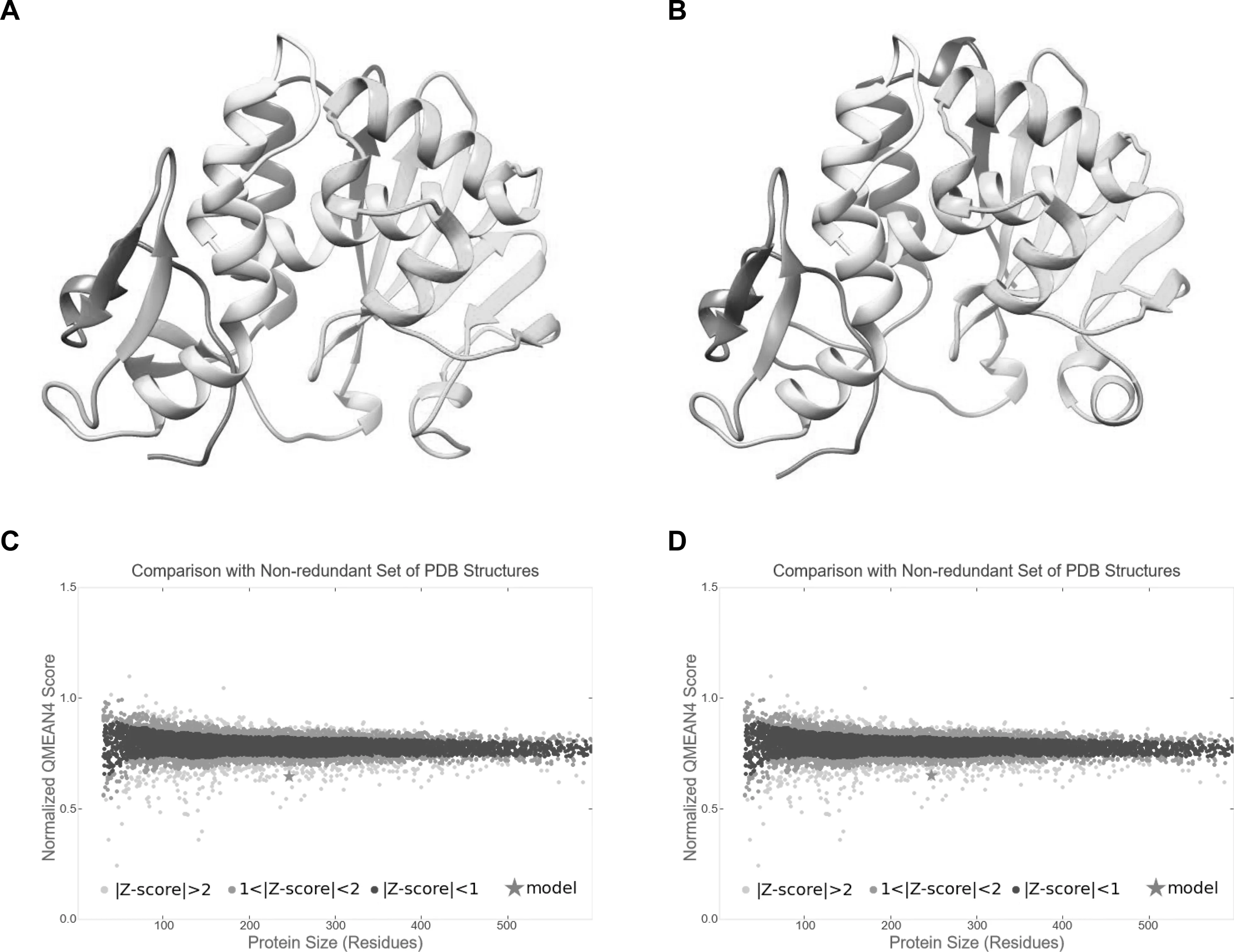
图1 基于SWISS MODEL预测的curcin和curcin C三维结构示意图

图2 Curcin、curcin C模型的质量评估
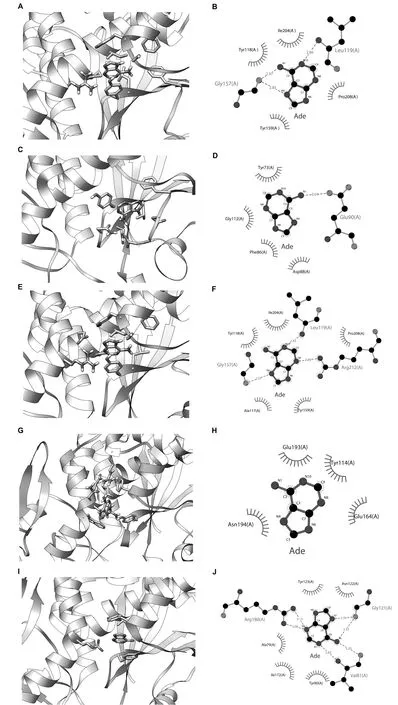
图3 Curcin和curcin C与腺嘌呤相互作用示意图
3.3 与腺嘌呤分子的对接研究
为了预测和比较curcin和curcin C与腺嘌呤的相互作用位点,采用AutoDock 4.2软件将腺嘌呤分别与优化前后的curcin和curcin C模型进行对接分析,选取结合能最低的对接结果并使用UCSF Chimera v.1.12.1[20]和LigPlot+[21]软件进行可视化(图3).可以看见优化前后的curcin模型氨基酸残基与腺嘌呤相互作用的结合能分别为-7.52kJ/Mol和-5.32kJ/Mol.优化前的curcin模型与腺嘌呤分子共形成3个氢键,其中Leu119与腺嘌呤的N10形成氢键,Gly157分别与腺嘌呤的N1、N4形成氢键,Tyr118、Tyr159、Ile204、Pro208则与腺嘌呤形成疏水相互作用(图3A,3B).优化后的curcin模型则是通过Glu90与腺嘌呤的N1形成一个氢键, Try73、Phe86、Asp88、Gly112与腺嘌呤具有疏水相互作用(图3C,3D).优化前后的curcin C模型氨基酸残基与腺嘌呤相互作用的结合能分别为-8.2kJ/Mol和-5.29kJ/Mol.优化前curcin C与腺嘌呤也形成了3个氢键,分别为Leu119与腺嘌呤的N10、Gly157与N4、Arg212与N8之间,Ala117、Tyr118、Tyr159、Ile204、Pro208共5个氨基酸残基则参与了与腺嘌呤的疏水相互作用(图3E,3F).优化后的curcin C模型的氨基酸残基与腺嘌呤未形成氢键,仅Tyr114、Glu164、Glu193以及Asn194共4个氨基酸残基参与了与腺嘌呤的疏水相互作用(图3G,3H).
在上述四个对接组合中,可以看出,结合能最低的组合是优化前的curcin C模型与腺嘌呤组合,该组合中分别有3个氨基酸残基参与了氢键的形成,5个氨基酸残基参与了与腺嘌呤的疏水互作,从数量上也是最多的.并且,我们还注意到,参与腺嘌呤互作的氨基酸残基及其位置与ricin A非常类似,后者也是有3个氨基酸残基(Val81、Gly121、Arg180)参与了氢键形成,5个氨基酸残基(Ala79、Tyr80、Asn122、Tyr123、Ile172)参与了疏水互作(图3I,3J).唯一的区别是Pro208替代了Ser158(ricin A中为Asn122)参与疏水作用.同样地,我们发现优化前的curcin模型与腺嘌呤组合的结合能也低于另外两个优化后模型组合,参与的氨基酸数量虽然相对较少,但也非常类似于ricin A.前期的研究显示,在Ricin A链中Tyr80、Tyr123、Glu177、Arg180、Trp211是rRNA N-糖苷酶活性的关键活性位点,而且这些位点在RIPs中高度保守.因此,优化前的模型结构虽然分值相对较低,但可能更接近真实状态.造成这一现象的原因则可能是在能量最小化优化时,没有考虑到该模型是在有腺嘌呤参与时形成的.当然这也意味着优化前的模型并非两个核糖体失活蛋白游离时的状态.至于curcin和curcin C与腺嘌呤的相互作用存在差异,则提示它们的rRNA N-糖苷酶活性可能存在差异,至于这种差异是导致它们的抗肿瘤活性差异的原因需要进一步的实验验证.
