基于生物信息学分析PKM2在肺腺癌中的表达及临床意义*
2020-12-03张轶贾方王秋月崔文静王菊勇
张轶,贾方,王秋月,崔文静,王菊勇
200032 上海,上海中医药大学附属龙华医院 肿瘤研究所
肺癌作为严重威胁人类健康的主要公共卫生问题之一,2018年全球新发病例约209万例,死亡约180万例,在我国肺癌的发病与死亡人数均居恶性肿瘤首位[1-2]。随着分子诊断技术的发展,早期发现、诊断并接受治疗已成为降低肺癌死亡率、延长患者生存期的关键措施。肺癌中最常见的病理类型为肺腺癌(lung adenocarcinoma,LUAD),因其早期临床症状不明显,明确诊断时已属中晚期,5年生存率不足17%[3-4]。因而从分子水平探寻影响LUAD发生发展及预后生存的新靶点具有重要意义。
恶性肿瘤代谢重编程的主要表现为有氧糖酵解,这种即使在氧气充足的条件下仍以糖酵解为首要产能途径的代谢方式在肿瘤细胞的快速生长中起着至关重要的作用。M2型丙酮酸激酶(pyruvate kinase M2,PKM2)是一种在糖酵解后期过程中起主要作用的限速酶,可催化磷酸烯醇式丙酮酸生成丙酮酸并释放能量[5],在调节细胞代谢活动和肿瘤生长中起着关键作用[6]。临床研究表明,多种类型癌症的不良预后与PKM2过表达相关,PKM2被认为是肺、胃肠道、卵巢、皮肤、肾和胆管等恶性肿瘤的潜在诊断标志物[7-8]。有研究指出,敲除PKM2可抑制人类非小细胞肺癌的发生并促进凋亡[9],但目前尚无针对其在LUAD中的临床预后和生物学过程的系统分析。
本研究采用生物信息学的方法,联合基因表达数据库(Gene Expression Omnibus,GEO)、癌症基因组图谱(The Cancer Genome Atlas,TCGA)和人类蛋白质表达图谱(Human Protein Atlas,HPA)等癌症公共数据库,分析PKM2在LUAD中的表达情况及其潜在的临床预后意义,为进一步探究LUAD发生的分子机制和治疗靶点提供新思路。
1 资料与方法
1.1 Oncomine和TCGA数据库提取基因数据
Oncomine数据库(https://www.oncomine.org /resource /login.html)是一个基于GEO和TCGA数据库的生物信息学分析平台[10]。本研究首先在Oncomine数据库中检索PKM2相关数据集,限定条件为:Gene:PKM2;Cancer Type: Lung Adenocarcinoma;Analysis Type: CancervsNormal Analysis; Data Type: all;Gene Summary:P<0.05,FOLD CHANGE>2,GENE RANK=Top 10%;Order By: Over-expression。提取数据集中关于PKM2基因的表达信息,对各基因芯片进行荟萃分析,选择箱式图展示结果。随后使用基于TCGA数据库的UALCAN平台(http://ualcan.path.uab.edu/analysis.html)对PKM2在LUAD中的表达结果进行验证。
1.2 HPA数据库分析PKM2蛋白表达
HPA数据库(https://www.proteinatlas.org/)利用免疫检测技术提供了多种人类蛋白质的组织和细胞分布信息,本研究选取PKM2抗体(CAB019421)对正常肺组织和LUAD组织进行的免疫组化实验结果进行分析。
1.3 Kaplan-Meier Plotter在线生存分析
在Kaplan-Meier Plotter数据库(http://kmplot.com/analysis/)的肺癌数据集中评估PKM2表达对LUAD患者总体生存时间(overall survival ,OS)的影响,设置筛选条件如下:Gene symbol:PKM2(Affymeteix ID:201251_at);Survival: OS;Histology: Adenocarcinoma。
1.4 LinkedOmics临床病理特征及KEGG分析
LinkedOmics分析平台(http://www.linkefomics.org)包含TCGA数据库内32种癌症的多组数据。进入LinkFinder模块设置目标肿瘤为“LUAD”,目标基因为“PKM2”。选择Clinical数据集,获取LUAD患者临床信息;选择RNAseq的数据集,获取PKM2共表达基因,筛选P<0.001且Statistic>0.4的正相关表达基因导入Metascape工具进行基因KEGG通路富集分析。
1.5 String-DB蛋白互作(protein-protein interaction,PPI)调控网络构建
String-DB网站(https://string-db.org/)是专门研究已知蛋白和预测蛋白之间相互作用的在线分析平台。本研究利用该网站预测了与PKM2关系最为密切的10个相互作用蛋白并进行可视化PPI网络构建。
1.6 统计学分析
正常肺组织与LUAD中PKM2基因表达差异,使用配对t检验进行配对样本的组间比较,使用Wilcoxon秩和检验进行非配对样品的组间比较;PKM2基因表达与LUAD临床病理相关性采用非参数检验;Pearson相关性检验筛选PKM2正相关表达基因;PKM2表达与LUAD患者OS的关系采用Kaplan-Meier模型分析。以双侧检验P<0.05被认为差异具有统计学意义。
2 结 果
2.1 PKM2在实体肿瘤和LUAD中的表达情况
Oncomine数据库共检索出410个关于PKM2基因在16种恶性肿瘤中表达差异的研究,与正常组织相比,肿瘤组织中PKM2高表达数据集53个,低表达数据集5个。在肺癌中,PKM2表达上调的有4个数据集,表达下调的有0个数据集(表1)。对该数据库内LUAD芯片数据进一步统计发现,自2001年开始共有9项研究、964个样本涉及PKM2在LUAD与正常组织中的表达,荟萃分析差异有统计学意义(P<0.05)的8项研究,结果显示PKM2在LUAD中所有差异表达基因中的中位数排名为373,差异有统计学意义(P<0.001),提示该基因在LUAD组织中显著高表达(图1)。

表1 Oncomine数据库中PKM2在不同肿瘤中的表达情况Table 1. Expression of PKM2 Gene in Different Tumors in the Oncomine Database

图1 PKM2基因在Oncomine数据库中肺腺癌中的表达Figure 1. Expression of PKM2 in LUAD in the Studies Identified in the Oncomine DatabasePKM2: Pyruvate kinase M2; LUAD: Lung adenocarcinoma.
2.2 PKM2基因在各LUAD芯片中的差异表达结果
获取Oncomine数据库内不同LUAD研究芯片中PKM2基因的表达数据,选取基因差异性表达排序前10%的芯片作箱式图比较分析。结果显示在Selamat、Hou、Beer、Landi、Selamat、Okayama、Su等7个数据集研究中,PKM2在LUAD中的表达量显著高于正常组织,差异均具有统计学意义(P<0.05)(图2)。 利用UALCAN平台进一步验证TCGA数据库中59例正常肺组织与515例LUAD组织中PKM2的差异表达情况,与正常组织相比,PKM2在LUAD组织中表达升高了1.95倍(图3)。

图2 Oncomine数据库中PKM2在不同肺腺癌研究芯片中的表达(*P<0.05)Figure 2. Expression of PKM2 mRNA in LUAD Tissues in Different Studies Identified in the Oncomine Database (P<0.05)
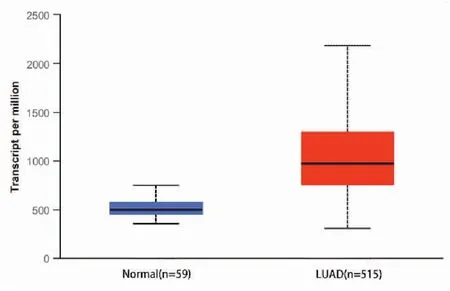
图3 PKM2在TCGA数据集肺腺癌患者中的表达情况Figure 3. Expression of PKM2 in LUAD Patients (TCGA)
2.3 PKM2蛋白在LUAD组织和正常组织之间的表达差异
通过HPA数据库中的免疫组化结果对PKM2在LUAD中的蛋白表达水平进行分析,以验证上述趋势,结果显示LUAD组织中PKM2蛋白免疫反应阳性显著强于正常肺组织(图4)。数据分析结果皆显示PKM2的表达增加有助于LUAD的发生,表明其在LUAD中扮演促癌基因的作用。

图4 PKM2蛋白在正常肺组织与肺腺癌组织表达的免疫组化检测结果Figure 4. Immunohistochemical Examination for Expression of PKM2 Protein in Normal Lung and LUAD Tissues
2.4 PKM2与LUAD患者预后的关系
为明确PKM2表达水平是否对LUAD患者预后存在影响,本研究获取Kaplan-Meier Plotter肺癌数据库内探针号为201251_at的芯片数据,对PKM2高、低表达组各360例的配对样本进行生存分析,结果表明PKM2高表达组的OS劣于低表达组,差异存在统计学意义(P<0.001,HR=2.56, 95%CI:1.99~3.28),提示LUAD患者预后不良与PKM2表达水平有关(图5)。

图5 PKM2表达与肺腺癌患者总体生存时间的关系Figure 5. Relationship between the Expression of PKM2 and the Overall Survival of LUAD Patients
2.5 PKM2表达与LUAD临床病理特征的关系
利用LinkedOmics数据库的LinkFinder功能检索PKM2基因,发现PKM2的表达量与LUAD患者的病理分级(图6A,P=0.011,FDR=0.033,n=507)呈正相关,与T分期(图6B,P=0.006,FDR=0.024,n=512)和N分期(图6C,P=0.004,FDR=0.023,n=501)呈显著正相关,而与M分期(图6D,P=0.363,FDR=0.544,n=371)无关。

图6 PKM2基因的表达与肺腺癌临床病理特征的关系Figure 6. Correlation between PKM2 Expression and Pathologic Characteristics in LUADA. Correlation between PKM2 expression and pathological grade; B. Correlation between PKM2 expression and T stage; C. Correlation between PKM2 expression and N stage; D. Correlation between PKM2 expression and M stage.
2.6 LUAD中PKM2潜在分子调控机制的预测
为进一步预测PKM2在LUAD恶性进程中可能参与的信号通路,本研究通过LinkedOmics数据库获得与PKM2表达正相关基因8 638个(图7),进一步筛选出78个基因导入Metascape数据分析平台进行KEGG分析,结果显示,LUAD中PKM2正相关表达基因与糖酵解、癌症中央碳代谢、HIF-1信号通路、磷酸戊糖途径、内分泌及其他因子调节的钙重吸收、细胞凋亡、IF-17信号通路、甲状腺激素信号通路和HTLV-1感染等有关(图8)。

图7 PKM2在TCGA肺腺癌数据库中的表达相关性及正相关基因热图Figure 7. PKM2 Association Result Identified in TCGA Database and the Heat Map of Positively Correlated Genes

图8 PKM2正相关基因的KEGG通路富集分析Figure 8. Enrichment Analysis of PKM2 Positively Correlated Genes by KEGG Pathway
2.7 PKM2蛋白相互作用网络分析
利用String网站绘制人源PKM2可能存在的PPI网络图(图9),结果显示,与PKM2相互作用排名前10的蛋白分别有烯醇化酶1(enolase 1,ENO1)、烯醇化酶3(enolase 3,ENO3)、葡萄糖-6-磷酸异构酶(glucose-6-phosphateisomerase,GPI)、烯醇化酶4(enolase 4,ENO4)、烯醇化酶2(enolase 2,ENO2)、果糖二磷酸醛缩酶A(aldolase A,ALDOA)、丙酮酸羧化酶(pyruvate carboxylase,PC)、苹果酸酶1(malic enzyme1,ME1)、乳酸脱氢酶A(lactate dehydrogenase A,LDHA)、甘油醛-3-磷酸脱氢酶(glyceraldehyde-3-phosphate dehydrogenase,GAPDH)。
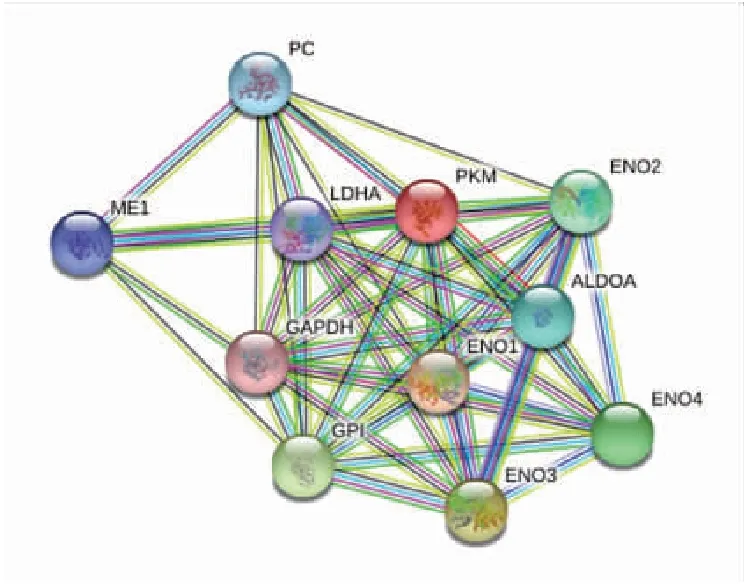
图9 PKM2的蛋白互作网络图Figure 9. Protein-Protein Interaction Network of PKM2
3 讨 论
丙酮酸激酶(pyruvate kinase,PK)是一种参与葡萄糖分解代谢的重要调节蛋白,哺乳动物体内PK有4种不同的亚型,以满足不同组织特定的能量需求[11]。PKL和PKR主要分别在肝肾组织和红细胞中表达,PKM1大量存在于肌肉和脑中,而PKM2作为PKM1的剪接变异体已经在胚胎细胞、成人干细胞和各种肿瘤细胞中被发现[12]。PKM2与癌症的代谢重编程和炎症反应直接相关,肺癌、胃癌、肝癌、胆管癌、乳腺癌和前列腺癌等肿瘤细胞中仅表达PKM2,且高表达水平的PKM2对肿瘤生长和预后有着不良的影响[13]。Suzuki等[14]报道了沉默PKM2基因可抑制LUAD H1299细胞的生长和迁移,shRNA-PKM2的H1299细胞异种移植小鼠模型中移植瘤显著缩小。因此,PKM2作为糖酵解过程的最终限速酶对肺癌的发生和发展至关重要。
本研究首先在Oncomine数据库中挖掘出涉及PKM2表达的数据集8个,其中7个数据集均显示LUAD组织中PKM2mRNA水平较正常肺组织升高;再对TCGA肺癌数据库中大样本规范数据统计分析,发现PKM2基因在LUAD组织中的表达约是正常肺组织的1.95倍,与Oncomine数据库分析结果相符。随后进一步分析了HPA数据库中LUAD与正常肺组织采用PKM2免疫组织化学抗体的染色强度,结果显示PKM2蛋白在正常肺组织中呈低表达或未检出,而在LUAD组织中呈现明显中等强度染色,与基因表达结果相一致。临床病理特征分析表明,该基因与LUAD的病理分级、T分期和N分期呈正相关,与M分期无关。
尽管医疗技术的进步使LUAD的诊疗手段有所提高,靶向药物和免疫疗法在临床也已广泛应用,但由于基因突变位点存在差异性,使得靶向药物疗效因人而异,患者5年生存率依旧较低[15]。An等[16]对73例LUAD病理标本研究表明,与PKM2表达低的患者相比,PKM2高表达患者的总存活率和无病存活率均降低。本研究通过分析Kaplan-Meier Plotter肺癌数据库中PKM2高、低表达各360例的LUAD患者OS发现,PKM2高表达组患者具有更高的病死率和更差的预后。提示PKM2可作为潜在有价值的生物标志物监测LUAD患者病情进展。
为了进一步探究PKM2的调控网络及信号通路,本研究利用LinkedOmics数据库对其共表达基因行Pearson相关性分析,得到与PKM2正相关的基因8 638个,随后将正相关基因进行KEGG通路富集分析发现,PKM2参与细胞凋亡、癌症中央碳代谢、HIF-1和IF-17等多种信号通路。有研究指出PKM2能显著增加HIF-1α的转录活性,从而增加其靶基因包括GLUT1、LDHA、ENO1和PDK1的表达,促进肺癌细胞的代谢重编程,分泌的PKM2通过激活整合素Beta1/FAK信号通路促进肺癌转移[16-18]。此外,PKM2参与调控肿瘤细胞的代谢方式向有氧糖酵解为主的改变,导致乳酸生成增加,酸性肿瘤微环境有助于肿瘤免疫逃避的发生[19],而且PKM2与人LUAD细胞程序性死亡配体1的表达水平呈正相关[20]。因此,PKM2可能通过其调控网络影响正相关表达基因的功能从而发挥癌基因的作用。
综上所述,本研究通过多种生物信息学数据库分析发现,PKM2在LUAD表达上调并影响预后,可能在LUAD中发挥生物预测作用,为进一步探索PKM2的生物学作用和分子机制提供了理论依据。
作者声明:本文全部作者对于研究和撰写的论文出现的不端行为承担相应责任;并承诺论文中涉及的原始图片、数据资料等已按照有关规定保存,可接受核查。
学术不端:本文在初审、返修及出版前均通过中国知网(CNKI)科技期刊学术不端文献检测系统的学术不端检测。
同行评议:经同行专家双盲外审,达到刊发要求。
利益冲突:所有作者均声明不存在利益冲突。
文章版权:本文出版前已与全体作者签署了论文授权书等协议。
