长江、淮河水系安徽区段黄颡鱼的群体遗传结构研究
2020-11-20胡玉婷段国庆周华兴潘庭双
胡玉婷,段国庆,凌 俊,周华兴,潘庭双,江 河
( 安徽省农业科学院 水产研究所,水产增养殖安徽省重点实验室,安徽 合肥 230031 )
黄颡鱼(Pelteobagrusfulvidraco),属鲇形目,鲿科,广泛分布于除西部高原和新疆外的中国各大水系,特别是长江中下游的湖泊、溪流等水域中[1-2]。黄颡鱼是鲿科鱼类中最常见的物种,也是我国重要的经济鱼类和主要养殖对象。近年来,由于黄颡鱼、杂交黄颡鱼(P.fulvidraco♀×P.vachelli♂)的人工养殖日益发展,以及各地黄颡鱼的相互交易,导致黄颡鱼种质混杂难以避免;考虑到栖息地破坏、水质污染、过度捕捞等因素,野生黄颡鱼资源日益减少。安徽所属长江、淮河水系是黄颡鱼的重要栖息地之一,目前未有对其群体遗传状况研究,而黄颡鱼资源的保护及进一步开发必须建立在了解其种质遗传现状的基础上。因此,开展黄颡鱼群体遗传结构研究很有必要。
鱼类线粒体细胞色素b(Cyt b)基因,除具有动物线粒体的分子小、结构简单、进化中性、检测方便、母系遗传、无重组等特征外,还兼有进化速度适中,易为保守引物扩增等特性,广泛用于鱼类的群体遗传学和系统发育学研究[3-8],在黄颡鱼种群遗传变异和系统演化关系方面也已进行了一些研究[9-11]。如库喜英等[9-10]分析了中国6大水系(长江、珠江、辽河、闽江、富春江和韩江)、长江中下游5个湖泊(鄱阳湖、巢湖、滆湖、洪泽湖、太湖)的黄颡鱼群体遗传变异,结果表明,黄颡鱼群体遗传多样性表现中等,但群体间遗传分化极小、缺乏明显的地理结构;而张鹤千等[11]研究了珠江水系5个江段黄颡鱼的遗传结构,结果显示,不同江段间存在显著的遗传分化。但上述研究均不涉及淮河水系以及安徽长江水系,笔者采用线粒体Cyt b部分序列为分子标记,探讨安徽长江、淮河两大水系野生黄颡鱼群体的遗传多样性和遗传结构,为黄颡鱼的育种和资源保护提供理论基础,并对比较不同水系间黄颡鱼的遗传变异,维护黄颡鱼种质资源的生物多样性具有重要意义。
1 材料与方法
1.1 样品采集
黄颡鱼样品于2018—2019年采集于安徽长江和淮河水系8个地点(表1、图1)。取新鲜样品鱼肌肉于无水乙醇中固定,于冰箱4 ℃密封保存。

表1 黄颡鱼样本采集信息Tab.1 Information on samples of yellow catfish P. fulvidraco
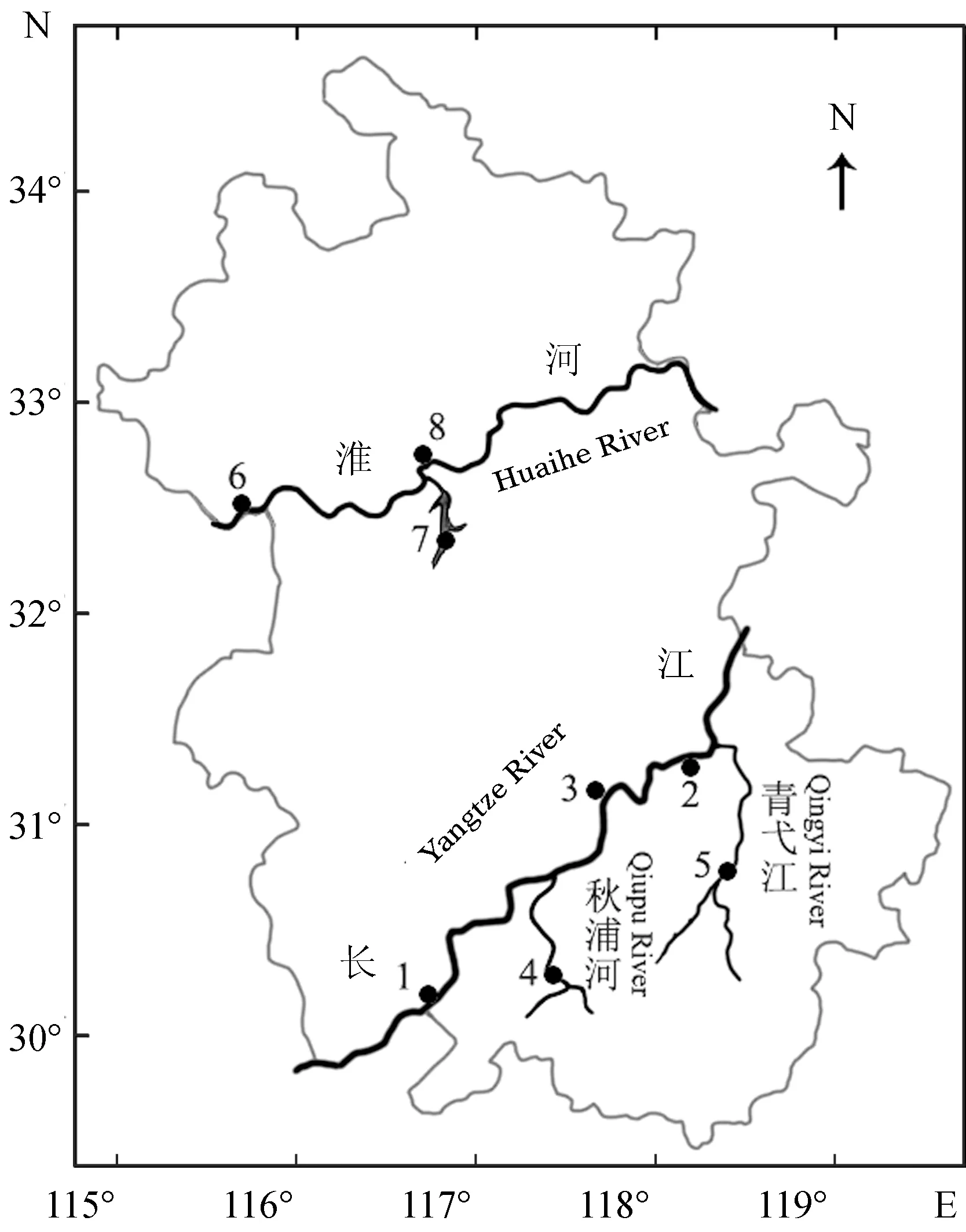
图1 黄颡鱼采样示意Fig.1 Sampling localities of yellow catfish P. fulvidraco in the study
1.2 基因组DNA提取、PCR扩增和测序
采用动物基因组提取试剂盒提取黄颡鱼基因组DNA。PCR扩增与测序引物均为通用引物L14724和H15915[12],反应体系和反应条件参照文献[10]中的Cyt b序列的扩增方法。经1%的琼脂糖凝胶电泳检测的目标PCR产物委托生物公司双向测序。
1.3 序列比对分析
采用软件Seaview[13]和Bioedit[14]拼接校正所得序列,并通过美国国立生物技术信息中心的Blast比对分析,验证这些序列均为目的基因。
多态位点数目、单倍型数目、单倍型多样性与核苷酸多样性由软件DNAsp 5.0[15]计算。MEGA 4.0[16]计算序列碱基组成、变异率、转换与颠换比值、群体内及群体间遗传距离;基于群体间遗传距离构建群体间系统进化树,采用1000次自展分析进行重复检验。基于Network 4.600[17]中中间连接法绘制黄颡鱼单倍型进化网络图。
由Arlequin 3.5[18]计算群体分化指数(FST)和进行分子变异分析,分别用于评估群体间遗传差异和检验群体间遗传变异来源,都通过1000次重抽样来检验统计学显著性。设定两种分子变异分析检验黄颡鱼的群体遗传结构:将所有群体划分为一个组群和将所有群体根据水系划分为两个组群以验证是否存在显著遗传分化。群体间基因流Nm=(1/FST-1)/2[19]。
2 结 果
2.1 序列多态性
212尾黄颡鱼Cyt b同源序列长度均为1122 bp(Cyt b基因第10至1131位),碱基T、C、A、G的含量分别为26.3%、32.1%、27.7%和13.9%,存在明显的A/T碱基偏向(A/T含量54.0%大于G/C含量46.0%)和较强的反G偏倚,G含量明显低于其他碱基,尤其在密码子第二(13.4%)和第三位(2.5%),而第一位的碱基组成相近(T:25.1%,C:26.8%,A:22.5%,G:25.7%)。这些都与脊椎动物线粒体DNA的特征一致[20-22]。
所有序列共有变异位点48个(多态率4.28%),其中简约信息位点20个、单突变位点28个。变异主要发生于密码子第3位(32个),然后为第1位(14个)、第2位(2个);变异多为同义突变的现象符合鱼类线粒体中氨基酸变化速率较慢的特征[20]。序列变异中转换明显大于颠换(比率7.795)、转换偏向的核苷酸替换特征符合线粒体的一般规律[23]。
2.2 单倍型分析
212条Cyt b序列片段共检出49种单倍型,单倍型间的最大变异率仅0.89%,单倍型在各地理群体间的分布见表2。多数单倍型(32个)为独有单倍型,仅包含1个样品,共有单倍型17个,但包含绝大多数样品(84.9%)。单倍型间简约进化网络图结果见图2。黄颡鱼单倍型在不同地理群体间并非随机分布的,具有一定的地理遗传结构。

表2 黄颡鱼单倍型在各群体的分布Tab.2 Distributions of haplotype in 8 populations of yellow catfish P. fulvidraco

(续表2)

图2 黄颡鱼49种单倍型的简约进化网络Fig.2 Statistical parsimony network of 49 haplotypes of yellow catfish P. fulvidraco圆大小与单倍型频率大小相一致,单倍型间斜体数字为碱基置换数,无斜体数字则表示碱基置换数为1,三角形为丢失的单倍型(节点).Circle sizes are proportional to the frequencies of the haplotype; the italic numbers on the line indicate the number of substitutions between two haplotypes, and the italic number without numbers on the line connecting haplotypes indicates single substitution between two haplotypes; the triangle represents the missing haplotype (node).
2.3 群体遗传多样性分析
黄颡鱼8个地理群体的遗传多样性参数见表3。单倍型多样性较高(0.930),除石台外(0.554),其余群体为0.816~0.949;尤其泾县群体最高(0.949)。而所有群体核苷酸多样性均偏低(0.00105~0.00221),其中泾县群体最高、石台群体最低,与单倍型多样性一致。
2.4 群体遗传距离和分化
群体遗传距离结果(表4)显示,群体间最大遗传距离是石台与瓦埠湖群体间(0.00382),最小遗传距离为望江与龙窝湖群体间(0.001778)。群体内遗传距离为0.00105~0.00222;大于部分群体间遗传距离,如泾县群体内遗传距离(0.00222)大于其与3个群体(望江、龙窝湖、无为)间的遗传距离,但小于其与其他4个群体间的遗传距离。由于群体内的遗传距离反映了该群体的遗传多样性,而不同群体间的遗传距离反映了其遗传组成的分化程度。这显示部分群体内存在较高的遗传多样性,但这种遗传多样性在其他一些群体中由于建群个体遗传多样性较低或遗传漂变等原因尚未积累太大的区别。这种现象反应了不同群体间复杂的遗传进化史,也显示了不同群体间亲缘关系的差异,体现了该区域总体遗传分化、部分群体遗传相似的复杂地理结构。群体间系统进化树(图3)显示,8个地理群体可分为两大进化枝,长江群体、淮河水系群体各聚为一枝。

表3 黄颡鱼群体遗传多样性参数Tab.3 Parameters of genetic diversity in 8 populations of yellow catfish P. fulvidraco

表4 黄颡鱼群体内(对角线)和群体间遗传距离(对角线下)Tab.4 Pairwise genetic distances among population (below diagonal) and genetic distance within population (diagonal)of yellow catfish P. fulvidraco

图3 基于线粒体Cyt b序列片段的黄颡鱼群体间系统进化树Fig.3 Phylogenetic tree of 8 populations of yellow catfish P. fulvidraco based on Cyt b gene fragments
黄颡鱼群体间的遗传分化和基因流分析结果见表5。群体分化指数为0.0012~0.5975、基因流为0.337~416.167,总的群体分化指数和基因流分别为0.2590、1.4305,这表明黄颡鱼地理群体间有明显遗传分化。
分子变异分析结果(表6)显示,第一种分析中地理群体内的遗传变异占比74.1%,群体间变异为25.9%(P<0.001),显示黄颡鱼地理群体间已产生一定程度的遗传分化;第二种分析中来自不同水系即长江群体与淮河群体之间的遗传变异为23.58%,来自同一水系内不同地理群体间的遗传变异仅为10.29%,而来自地理群体内的遗传变异为66.13%,显示不同水系的黄颡鱼地理群体间具有较高程度的地理差异,存在明显的地理结构。综合研究结果表明,安徽黄颡鱼不同地理群体间的遗传分化主要是由水系间的群体遗传差异产生的。

表5 基于Cyt b序列的黄颡鱼种群成对群体分化指数(对角线下)和基因流(对角线上)Tab.5 F-Statistics (below diagonal) and gene flow (Nm) (above diagonal) from haplotype frequencies of yellow catfishP. fulvidraco based on Cyt b sequences

表6 基于线粒体Cyt b序列的黄颡鱼所有样品的分子变异分析Tab.6 AMOVA of all samples of yellow catfish P. fulvidraco based on Cyt b gene
3 讨 论
3.1 黄颡鱼群体遗传多样性
单倍型多样性和核苷酸多样性是衡量群体遗传多样性的重要参数,其数值大小表示生物适应环境变化能力的强弱。研究结果显示,长江、淮河水系安徽区段黄颡鱼群体单倍型多样性(平均0.930,0.554~0.949)较高而核苷酸多样性(平均0.00236,0.00105~0.00221)较低,其遗传多样性处于中等水平。这低于长江中下游5个湖泊(巢湖、滆湖、洪泽湖、鄱阳湖和太湖)黄颡鱼的单倍型多样性指数(0.945)和核苷酸多样性指数(0.00419)[10];高于珠江流域黄颡鱼的单倍型多样性(0.849)而显著低于其核苷酸多样性(0.04817)[11],而与库喜英等[9]分析的中国6个水系黄颡鱼的遗传多样性结果(单倍型多样性0.857,核苷酸多样性0.0023)相比,本研究单倍型多样性指数略高而核苷酸多样性指数相近。上述研究结果均是基于Cyt b序列分析,一致显示我国野生黄颡鱼群体单倍型多样性尚处于较高水平,但不同地区核苷酸多样性水平相差较大。
同属鱼类相比较,丁言伟[24]通过线粒体ND4基因探讨黄颡鱼属鱼类的种群遗传结构,瓦氏黄颡鱼(P.vachelli)的种群遗传多样性最高,光泽黄颡鱼(P.nitidus)和长须黄颡鱼(P.eupogon)的种群遗传多样性次之,黄颡鱼的种群遗传多样性最小;而且这种遗传多样性差异主要体现在核苷酸多样性水平上。与区域内其他鱼类相比较,远低于安徽长江和淮河水系黄鳝(Monopterusalbus)的平均核苷酸多样性(0.01904、0.01882)[25-26]、也低于淮河野生鲇鱼(Silurusasotus)和淮河源区(Hemiculterleucisculus)的核苷酸多样性(0.0038、0.0046)[27-28];与长江水系大眼鳜(Sinipercakneri)的核苷酸多样性0.00237相近[29],而与上述鱼类在单倍型多样度上差异不大。上述研究表明,黄颡鱼核苷酸多样性低是一种普遍现象,而本研究的黄颡鱼核苷酸多样性几乎最低。由于单倍型多样性是基于核苷酸多样性在群体中的分布,而核苷酸多样性比单倍型多样性更易受到遗传漂变等的影响、且一旦降低更难恢复。近年来,黄颡鱼及杂交黄颡鱼的人工养殖日益发展,以及各地黄颡鱼的相互交易,导致黄颡鱼种质混杂难以避免;又由于栖息地破坏、水质污染、过度捕捞等因素,野生黄颡鱼资源日益减少。考虑到本地黄颡鱼遗传多样性较低的现状,相较于其他区域,长江、淮河安徽区段的黄颡鱼遗传多样性形势较为严峻,在黄颡鱼种质资源保护上要加以关注。两水系间相比较,长江干流黄颡鱼群体遗传多样性略高于淮河群体,长江支流群体的遗传多样性水平差别较大,这与当地的自然环境条件及人为影响不同有关。表明长江干流是安徽本地选育黄颡鱼良种的合适产地,应当加大野生群体的资源保护。
3.2 黄颡鱼群体遗传分化
群体分化指数常用来表示群体间的遗传分化程度,其值越大则群体间的分化程度越高。当群体分化指数为0~0.05、0.05~0.15、0.15~0.25和0.25~1.00时,分别表示无分化、中度分化、遗传分化较大和遗传分化极大或高度分化[30]。本研究中黄颡鱼地理群体间的总群体分化指数为0.2590,属高度分化;两两群体间群体分化指数为0.0012~0.5975,表明各群体间呈现不同程度的分化,部分群体间遗传分化显著。基因交流程度是影响群体结构的重要因素。当基因流<1时,遗传漂变是影响群体遗传结构的主导因素;基因流≥4时,基因流则成为主导因素;1<基因流<4时,基因流和遗传漂变均是影响群体遗传结构的重要因素[31]。本研究中各地理群体间基因流为0.337~416.167,总基因流为1.4305,表明基因流和遗传漂变在黄颡鱼群体遗传结构中均发挥着重要作用。
不同群体间比较可发现,望江、龙窝湖、无为和泾县4个地理群体间群体分化指数较低且P>0.05,表明这些群体间遗传差异不显著。这是由于除泾县外,3个群体样本均采自长江干流或近岸水域(龙窝湖),而泾县处于长江下游支流青弋江干流上,该段以下河道宽敞、与长江干流畅通无阻,因而群体间基因交流频繁(基因流>4)、遗传分化不显著。另外,长江下游支流秋浦河上的石台群体位于皖南山区,该地山高沟深、地势落差大、河道狭窄多变,地理阻隔较大,因而具有较独立的遗传特点。黄颡鱼淮河群体中,瓦埠湖为淮河沿岸湖泊、与凤台距离较近,因而两群体间无显著遗传差异;阜南和凤台群体样本虽然均采自干流,但阜南黄颡鱼群体采集王家坝上游,大坝的阻隔导致群体间基因交流困难,因此群体间遗传分化显著。
库喜英等[9]分析了长江、珠江、辽河、闽江、富春江和韩江6个水系70尾黄颡鱼的遗传变异,结果显示,黄颡鱼群体缺乏明显的地理结构;但又认为,27个单倍型中的26个均是不同水系的特有单倍型,因此认为黄颡鱼缺乏显著的地理结构的状况已发生改变。而分子变异分析也显示,将样本按照6个水系划分为6个组时,得到的组间遗传差异最大;这表明水系的隔离可能限制了群体之间的基因交流,导致不同水系的地理群体产生了不同的变异模式。本研究中长江、淮河水系黄颡鱼群体间的显著遗传分化结果即证明了上述可能性。钟立强等[10]对长江中下游5个湖泊黄颡鱼种群间的遗传分化结果也显示,虽然这些群体间不存在显著的系统地理格局,但群体间的相对遗传距离能反映群体间的亲缘关系;而且一些来自不同湖泊的黄颡鱼群体间存在较大程度的遗传分化,如洪泽湖与鄱阳湖两群体间遗传分化系数为0.21846。另外,珠江水系5个野生黄颡鱼地理群体间的群体分化指数为0.0260~0.4143,也显示了同一水系间亦存在显著的遗传分化[11]。这与本研究中同一水系不同地理群体间存在明显遗传分化的结果相一致。
以上结果均说明,黄颡鱼不同水系间甚至同一水系不同地理群体间存在遗传分化的现象较为普遍,库喜英等[9]“黄颡鱼群体缺乏明显的地理结构”的结果可能是由一些水系所用样本过少引起的,如富春江水系仅有1个采样点2个样本,辽河仅有2个采样点共5个样本,闽江仅有3个采样点共6个样本。
分子变异分析结果也表明,长江、淮河水系安徽区段黄颡鱼地理群体间存在着显著的遗传分化,这些遗传分化主要是由不同水系间的群体遗传差异(变异百分比23.58%)产生的。单倍型进化网络图和群体间系统进化树表明,黄颡鱼8个地理群体分为2个大的进化枝,长江、淮河水系群体各为1枝。因此,认为水系间的地理阻隔可能是导致这些黄颡鱼群体遗传分化的主要原因。
4 结 论
长江、淮河水系安徽区段黄颡鱼群体具有中高度的单倍型多样性、低的核苷酸多样性;群体间存在显著的遗传分化,这种遗传分化主要来源于长江、淮河两水系的群体间,表明长江、淮河水系安徽区段的黄颡鱼具有明显的地理遗传结构,分析认为不同水系间的地理阻隔可能是导致黄颡鱼群体遗传分化的主要原因。研究结果为黄颡鱼的遗传管理和资源保护提供重要的基础信息。
