基于TCGA数据库数据NME基因家族生物学功能、在肝癌诊断和预后预测中的应用分析
2020-10-14姚清媚宋伟王鹏飞刘玲珑周素芳
姚清媚,宋伟,王鹏飞,刘玲珑,周素芳
广西医科大学基础医学院,南宁 530021
肝癌是我国常见的恶性肿瘤,具有高度转移性,中位生存期为6~9个月,晚期预后极差[1]。目前报道的生物学指标有基因LARP4B、长链非编码RNA-OGDHL、膜蛋白-POSTN、调节分子 Treg 和IL-10等,但敏感性差,因此有必要寻找新的肝癌预后评估指标。人类NME是编码核苷二磷酸激酶的基因家族[2],由10个基因组成,参与细胞增殖、分化、发育、凋亡、纤毛功能、转移等多种生理病理过程[3]。目前研究最多的NME基因家族成员是NME1基因和NME2基因,均与肿瘤转移相关。2019年9~11月, 本研究采用生物信息学方法分析NME基因家族的生物学功能和信号通路,从数据库中获取肝癌组织和癌旁组织NME基因家族的表达水平、并进行比较,利用ROC分析NME 1~7对肝癌患者预后的诊断效能,同时探讨NME 1~7与肝癌预后的关系,以期寻找良好的肝癌预后指标。
1 材料与方法
1.1 数据获取 从加州大学圣克鲁斯分校平台(https://xenabrowser.net/datapages/)中的TCGA公共数据库中下载NME基因家族在肝癌组织和癌旁组织中的表达水平及肝癌患者相关临床信息包括生存时间、死亡状态、年龄、性别、危险因素、TNM分期、肿瘤浸润情况、是否治疗新发肿瘤等。最终得到365例肝癌患者临床信息和NME基因表达信息完整的数据。
1.2 NME基因家族的生物功能分析 利用生物信息注释数据库DAVID(the Database for Annotation, Visualization and Integrated Discovery,http://david.abcc.nciferf.gov/)在线数据库对NME基因进行基因本体功能(GO)分析和KEGG代谢富集分析,设置P<0.05为差异有统计学意义。
1.3 肝癌组织和癌旁组织NME基因的表达观察 TCGA数据库中获取的NME基因表达数据经以2为底的对数转换,比较其在肝癌和癌旁正常组织中的表达水平。
1.4 NME基因家族对肝癌的诊断效能评价 应用GraphPad Prism v.7.0绘制7个NME基因的ROC评价其对肝癌患者预后的诊断效能,以P<0.05为差异有统计学意义。
1.5 NME基因家族对肝癌预后的预测分析 单因素生存分析(Kaplan-Meier法)和Log-rank检验分析肝癌患者生存时间与临床病理资料的关系,寻找影响肝癌患者生存时间的临床因素。再以NME基因表达值的中位数分为高表达组和低表达组,预测7个NME基因与肝癌患者生存期的相关性采用单因素生存分析(Kaplan-Meier法)和Log-rank检验。对影响肝癌患者预后的因素采用COX回归分析,得到校正后的P值、风险比(HR)和95%置信区间(CI)。
2 结果
2.1 NME基因家族的生物学功能及信号通路 NME基因家族生物学功能主要是具有核苷二磷酸激酶活性、参与细胞凋亡、发育等生物学过程,包括分子功能(MF)、生物过程(BP)和细胞成分(CC)。NME基因家族与新陈代谢、抗生素合成、嘌呤和嘧啶代谢途径相关。
2.2 肝癌组织及癌旁组织中NME基因家族的表达比较 肝癌组织中NME1、NME2、NME3、NME6、NME7的水平分别为3.476±0.006、3.686±0.005、3.259±0.006、2.965±0.005、2.895±0.007,癌旁正常组织中分别为3.334±0.009、3.593±0.007、3.172±0.010、2.846±0.007、2.807±0.014,肝癌组织中各指标水平均高于癌旁正常组织(t分别为8.927、7.044、5.267、8.370、4.349,P均<0.000 1),而NME5在肝癌组织中水平低于癌旁组织(0.952 6±0.006 比 1.606±0.087,t=4.306,P<0.000 1),NME4在两种组织中的水平差异无统计学意义(3.458±0.006 比 3.477±0.012,t=1.403,P=0.1613)。
2.3 NME基因家族对肝癌的诊断效能 ROC结果显示,除NME4的ROC标准化诊断值P=0.200 8>0.05 无统计学意义外,其他6个NME1、NME2、NME3、NME5、NME6、NME7对肝癌患者的预后有较好的诊断价值,曲线下面积(AUC)分别为0.887 2、0.826 2、0.745 9、0.745 7、0.871 5、0.725 5,诊断预测效能良好。
2.4 NME家族基因表达与肝癌患者预后相关性 回顾性分析365例肝癌患者的临床资料,不符合分析条件的归为删失结果(见表1)。单因素生存分析显示,吸烟等危险因素、TMN分期、肿瘤浸润情况和是否治疗新发肿瘤影响肝癌患者的生存时间,P值分别为0.006、<0.001、<0.001、0.010。这四个影响因素作为COX回归生存分析的校正因子。单因素生存分析结果显示,NME6和NME7与肝癌患者的预后相关,P分别为0.02、0.017,均<0.05,加校正因子进行多因素COX生存分析后,发现NME5、NME6和NME7与肝癌患者预后相关,校正后P分别为0.025、0.001、0.042,均<0.05,且NME5、NME6和NME7低表达肝癌患者的生存时间比NME5、NME6和NME7高表达者长,详见表2。
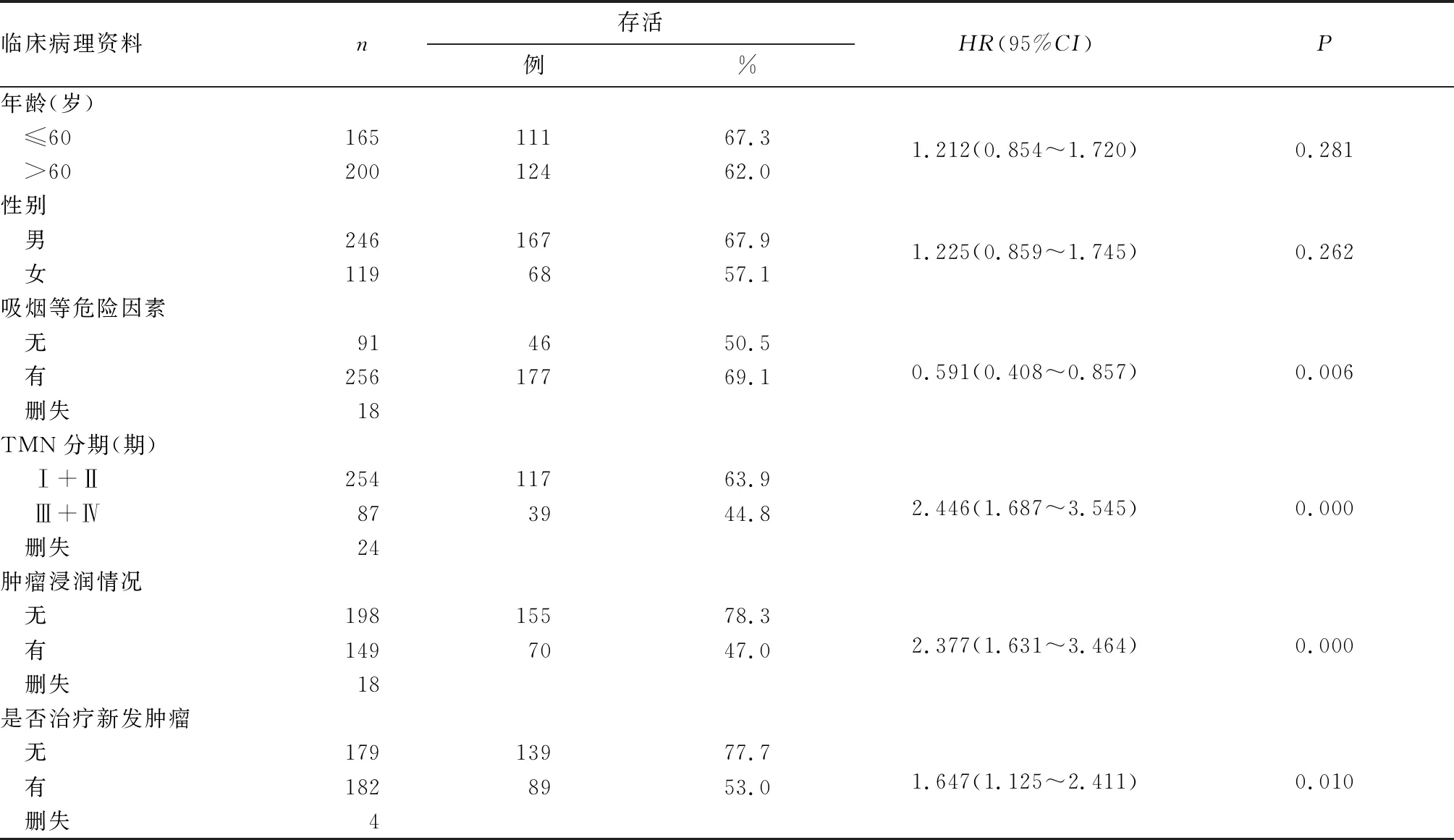
表1 临床病理资料与肝癌患者生存率的关系
3 讨论
目前,一些研究机构在公共数据库中上传各种肿瘤组织和正常组织的人类全基因组表达芯片数据,研究者可下载芯片数据进行相应基因的表达分析[4]。NME基因家族曾被称为nm23基因家族,它们是1988年Steeg等在美国国立癌症研究所从鼠的黑色素瘤细胞株中提取的与恶性肿瘤转移过程相关的基因[5]。NME1参与内皮细胞的血管生成和集体迁移等生物过程[6],而NME2对胃癌细胞的凋亡具有抑制作用[7]。还有研究[8~12]表明,NME基因家族在软组织肉瘤、结直肠癌、乳腺癌、非小细胞肺癌和白血病中存在表达失调且影响肿瘤的发展和预后情况。提示NME基因家族参与各种细胞生物学过程,且在多种肿瘤组织中存在表达失调,影响肿瘤的发展。
对NME基因进行GO富集分析,结果表明NME基因主要参与的生物学过程有核苷二磷酸激酶活性、细胞凋亡和发育,KEGG通路富集分析结果主要涉及新陈代谢、抗生素合成、嘌呤和嘧啶代谢途径。提示NME基因在肝癌细胞中主要通过影响肝癌细胞的磷酸化、凋亡和物质代谢等对肝癌的进展产生一定的影响。

表2 肝癌不同NME家族水平患者预后的生存分析结果
本研究中发现除NME4在肝癌组织中不存在表达失调的情况外,其他的几个基因家族均存在表达失调的情况,其中NME6和NME7在肝癌组织的表达高于癌旁组织,与文献中NME6在结直肠癌中的表达高于癌旁组织[13]和NME7在转移癌中的明显过表达[14]相符。而NME5在肝癌组织的表达量低于癌旁组织,与其在乳腺癌[15]和尿路上皮细胞癌和原发性尿路上皮细胞癌[16]中表达情况一致。
ROC诊断分析中,当AUC>0.5时,AUC值越接近1,表明诊断标志物的诊断效能越好。本研究结果显示,除NME4外,其余的NME基因的AUC在0.7~0.9之间,表明它们诊断预测肝癌的效能良好。在生存分析中,NME6和NME7在肝癌组织中高表达,而单因素生存分析和COX回归分析表明NME6和NME7表达量低的肝癌患者预后更好,生存时间延长,提示NME6和NME7可能为癌基因。而COX回归分析显示低表达的NME5也可作为肝癌预后的生物标志物,且其低表达的肝癌患者预后较好。研究[17]报道,NME5过表达诱导胰腺癌细胞耐药,相反,下调能逆转耐药,原理可能是NME5可减弱吉西他滨等药物诱导的凋亡和细胞周期阻滞作用,使敏感性下降,引起癌细胞耐药。
综上所述,NME基因家族主要富集在具有核苷二磷酸激酶活性、参与细胞凋亡、发育等生物学过程,主要涉及新陈代谢、抗生素合成、嘌呤和嘧啶代谢途径等相关通路。NME4在肝癌组织和癌旁组织无明显差异,NME5在肝癌组织中低表达,其余的5个NME基因在肝癌组织中高表达。ROC表明除NME4外,其他6个NME基因预测肝癌效果良好,生存分析显示,NME5、NME6、NME7的低表达与肝癌患者的预后良好相关。本研究首次报道了NME5、NME6、NME7下调与肝癌患者的良好预后有关,这3个基因有可能成为肝癌患者预测预后的生物标志物,但本研究只是对NME基因家族的生物学功能及其在肝癌患者预后中的作用进行初步探讨,将NME基因家族作为靶向治疗仍需进一步研究。 另外,本研究样本量小,还需要扩大样本以验证目前的发现。
