2019新型冠状病毒S蛋白的结构和功能分析
2020-09-05谭玉靓
谭玉靓, 唐 标
(湖南中医药大学 医学院,湖南 长沙 410208)
新型冠状病毒肺炎(Corona Virus Disease 2019, COVID-19)自爆发以来,以数月时间在全世界引起了大流行[1]。COVID-19的传播性极强,目前尚无针对该病毒的特效药,对人类具有严重的健康威胁,WHO已将其认定为国际公共卫生紧急事件[2]。 COVID-19是由严重急性呼吸系统综合症冠状病毒2(Severe acute respiratory syndrome coronavirus 2, SARS-CoV-2)引起, WHO将其命名为2019新型冠状病毒(2019 novel Coronavirus, 2019-nCoV)[3]。2019-nCoV借助其表面的纤突(Spike, S) 糖蛋白,与宿主细胞膜发生融合并实现传播,S蛋白是疫苗、治疗性抗体以及临床诊断的潜在靶点。S蛋白是一种三聚体糖蛋白,其功能发挥依赖两个基本结构域:受体结合域S1亚基和融合域S2亚基[4-5]。S1亚基是S蛋白与宿主细胞膜相互作用的“门户”,即功能发挥区;S2亚基在S1亚基的激活下,能加强S1与宿主细胞的融合,进而促进2019-nCoV在宿主细胞的增殖,即功能辅助区。此外,研究发现,S1亚基对S2亚基起激活的作用,而激活的S2亚基能保护S1亚基躲避宿主的免疫攻击,二者在功能上呈现协同作用[6-8]。因此,研究S蛋白,预测其理化性质,如相对分子质量、疏水性和等电点等,有利于蛋白质的分离与鉴定;预测其进化、结构、功能和抗原表位特征,有利于了解其致病性,为疫苗和抗病毒药物的研制提供思路。本研究通过生物信息学,对 2019-nCoV S蛋白的理化特征、结构、功能和抗原表位进行预测分析,旨在为该病毒的生物学特性研究提供思路,为COVID-19疫苗和抗病毒药物的研发提供参考。
1 材料与方法
1.1 材料
1.1.1 2019-nCoV S蛋白的氨基酸序列 从美国国立生物技术信息中心(National Center for Biotechnology Information, NCBI)获取2019-nCoV S蛋白的氨基酸序列。NCBI由美国国立医学图书馆成立,提供多种基因组、染色体和蛋白质的序列信息,涉及细菌、病毒和真菌等多种种属,供全球科研人员共享。NCBI网址:https://www.ncbi.nlm.nih.gov/
1.1.2 主要数据库 利用ExPASy分析S蛋白的氨基酸结构、原子结构和理化性质等;利用TMpred预测S蛋白的跨膜螺旋结构;利用SignaIP v5.0预测S蛋白的信号肽;利用 NetPhos3.1预测S蛋白的磷酸化位点;利用Pfam预测S蛋白的结构域;利用PSIPRED预测S蛋白的二级结构;利用SWISS-MODEL预测S蛋白的三级结构;利用IEDB预测S蛋白的T细胞表位和B细胞表位;利用BLAST分析2019-nCoV S蛋白与其他物种的相似性;利用MEGA分析2019-nCoV与其他物种的进化关系。
1.2 方法
1.2.1 获取2019-nCoV S蛋白的氨基酸序列 使用NCBI数据库,以2019-nCoV S蛋白为检索条件,筛选适合本研究的S蛋白全长氨基酸序列。下载S蛋白的Fasta文件信息,以便后续操作。
1.2.3 预测2019-nCoV S蛋白的结构、功能与抗原表位 利用TMpred预测S蛋白的跨膜螺旋结构;利用TMHMM预测S蛋白的跨膜区;利用 NetPhos3.1预测S蛋白的磷酸化位点;利用Pfam预测S蛋白的结构域;利用PSIPRED预测S蛋白的二级机构;利用SWISS-MODEL构建S蛋白的三级结构;利用免疫表位数据库IEDB预测S蛋白的T细胞和B细胞表位。
1.2.4 分析2019-nCoV S蛋白与其他物种的相似性 运用BLAST软件分析2019-nCoV S蛋白与其他物种的相似性。为进一步了解2019-nCoV与BLAST输出的物种的进化关系,利用MEGA分析获得这些物种的进化关系图。
2 结果与分析
2.1 理化性质
2.1.1 氨基酸的组成 研究所选用的2019-nCoV S蛋白序列编号为BBW89517。通过ExPASY的ProtParam软件分析S蛋白的氨基酸结构。结果显示,S蛋白由1 273个氨基酸组成,详细信息如表1所示。其中,亮氨酸Leu(L) 108个,占比8.5%;丝氨酸Ser (S)99个,占比7.8%;缬氨酸Val(V)和苏氨酸Thr (T)均为97个,占比7.6%。脂溶性氨基酸Ala、Val、Ile和Leu的总数达360个,占S蛋白总序列的28.3%。带负电荷的氨基酸残基 (天冬氨酸Asp + 谷氨酸Glu) 共有110个,带正电荷的氨基酸残基 (精氨酸Arg + 赖氨酸Lys) 共有103个,等电点(isoelectric point, PI)6.24。
2.1.2 疏水性分析 ProtParam软件分析结果显示,S蛋白的脂溶指数(aliphatic index,AI)为84.67,亲水性的总平均值 (Grand average of hydropathicity, GRAVY)为-0.079(GRAVY范围介于2和-2之间,负值越大表明亲水性越好)。AI为蛋白质脂肪侧链占蛋白质的相对含量;GRAVY为蛋白质中所有氨基酸亲水值的总和与氨基酸数量的比值。进一步利用ProtScale的Kyte﹠Doolittle预测S蛋白的疏水性,见图1。横轴为氨基酸位置,纵轴为疏水评分,正值越大,疏水性越强。在1 273个氨基酸组成的序列中,疏水性和亲水性氨基酸的分布较均匀,且数目相差不大。第679位Asn亲水性最强,评分为-2.589;第7位Leu疏水性最强,评分为3.222。以上结果表明,S蛋白亲水性低,脂溶性高。
2.1.3 原子结构分析 由ExPASY的ProtParam软件分析S蛋白的原子结构。结果显示,S蛋白共由19 710个原子构成,相对分子质量为141 178.47,化学分子式为C6336H9770N1656O1894S54。
2.1.4 其他理化性质 ProtParam分析结果显示,S蛋白的不稳定系数(instability index, II)为30.01,可认为是稳定蛋白(当II>40时,认为该蛋白为不稳定蛋白)。消光系数在280 nm的水中测量得到,单位为L/(mol·cm)。假设S蛋白的所有Cys残基都来自胱氨酸,并形成二硫键,则其消光系数为148 960 L/(mol·cm);假设所有二硫键打开,其在水溶液中(A280 nm) 的摩尔消光系数则为 146 460 L/(mol·cm)。预测结果显示,S蛋白氨基端可能为甲硫氨酸 (Met)。此时,其在哺乳动物体外网织红细胞中的半衰期为30 h,在酵母体内的半衰期大于20 h,在大肠埃希菌体内大于10 h。
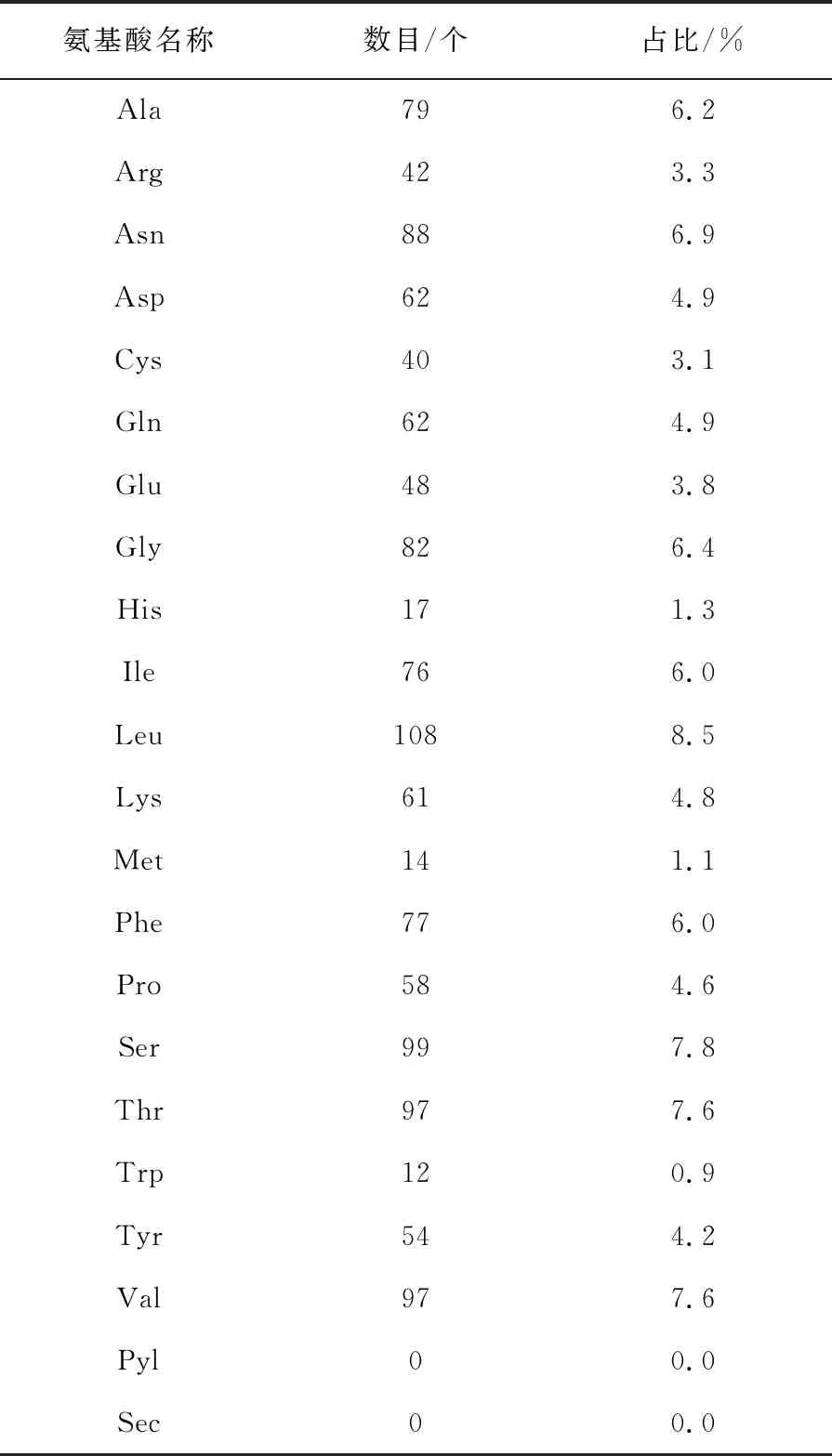
表1 S蛋白的氨基酸组成信息

图1 S蛋白的疏水性分析Fig.1 Hydrophobicity analysis results of S protein
2.2 功能相关序列和结构分析
2.2.1 跨膜螺旋分析 利用TMpred预测S蛋白的跨膜螺旋结构。结果显示,从里往外(inside-outside, i-o)的可能螺旋共有12个,详细信息如表2所示。其中,在110~131和941~963氨基酸序列处的螺旋具有重要意义。从外往里(outside-inside, o-i)的可能螺旋有11个,如表3所示,1 279~1 298这个螺旋方向具有重要意义。进一步分析得到S蛋白的跨螺旋分析图,见图2。结果显示,整条S蛋白氨基酸序列中,有4个螺旋被认为是有意义的(当分值高于500时,认为该螺旋有意义),即58~79、110~131、942~963以及1 279~1 298,其中1 279~1 298序列的螺旋程度最高。
首先,要注重材料采购管理的作用,特别是招标管理文件内容,应确保供应商是一般纳税人,且对外合同签订名称要统一,确保专用发票的购买方信息和企业始终是相同的。
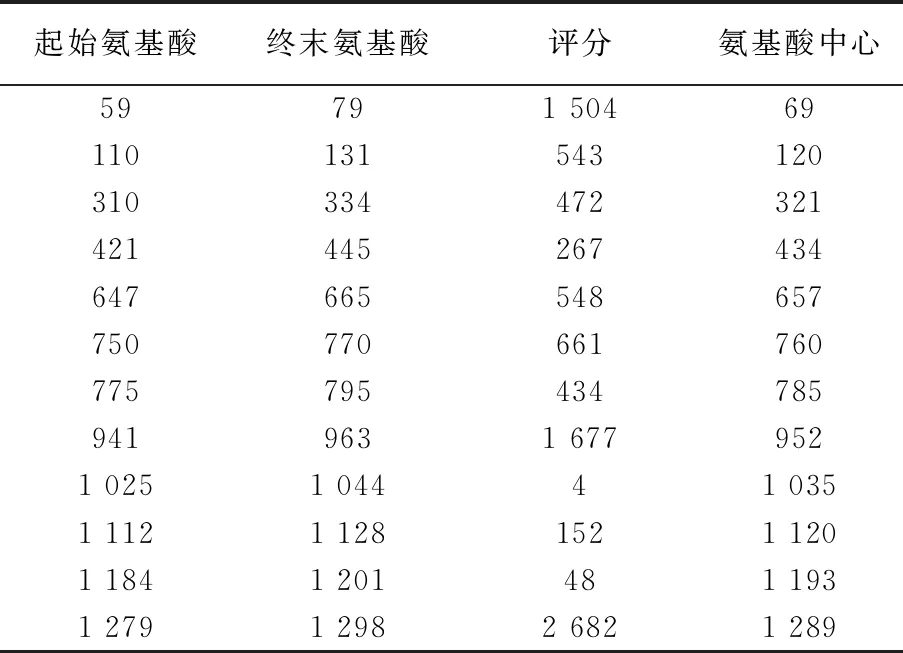
表2 从里往外的跨膜螺旋信息

表3 从外往里的跨膜螺旋信息
2.2.2 跨膜区预测 利用TMHMM预测S蛋白的跨膜区,得到一个跨细胞膜的区域。跨膜区的原子吸收光谱(atomic absorption spectrum, AAS)为23.973 03,第一个60原子吸收光谱为0.015 58,N-in的总概率为0.000 77。
2.2.3 信号肽预测 利用SignaIP v5.0预测S蛋白的信号肽,信号肽值(Signal Peptide, Sec/SPI)为0.968 9,高于cutoff值0.5,提示S蛋白为分泌型蛋白。
2.2.4 磷酸化位点预测 磷酸化位点是药物作用的重要区域,利用 NetPhos3.1预测S蛋白的磷酸化位点,设定0.8为有效阈值,得到了38个磷酸化位点。包括24个丝氨酸位点(S),11个苏氨酸位点(T),3个酪氨酸位点(Y),见表4。
2.3 S蛋白的结构
2.3.1 结构域预测 利用Pfam预测S蛋白的结构域,得到2个具有意义的保守结构域,如图3所示。结果显示,S蛋白由纤突蛋白受体结合域和S2糖蛋白组成。其中,S2糖蛋白,即S2亚基,属于Corona S2家族,CL0595族,E值为1.4e-266,Bit值为885.9;纤突蛋白结合域,即S1亚基,其HMM长度为212,E值为6.6e-75,Bit值为251.5(此处命名遵循冠状病毒命名原则:受体结合亚基为S1,融合蛋白为S2)。
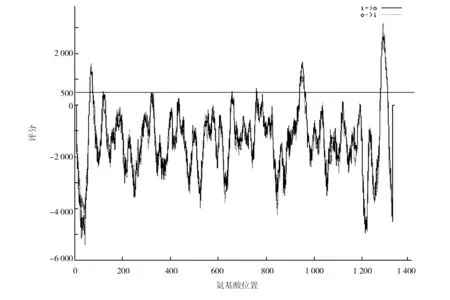
图2 S蛋白的跨膜螺旋拓扑图Fig.2 The topology diagram of S protein transmembrane spiral

图3 S蛋白功能结构域Fig.3 The analysis results of functional domain of S protein
2.3.2 二级结构特征 利用PSIPRED预测S蛋白的二级结构。图4显示S蛋白的线性区、螺旋区、膜相互作用区和无序区。结果表明,S蛋白以无规则卷曲和螺旋结构为主。S蛋白发挥功能的氨基酸位置如图5所示,每种颜色所代表的意义:亲水性/极性(红色)、小非极性(黄色)、疏水性/非极性(绿色)、半胱氨酸和芳香烃(蓝色),可以看出S蛋白的疏水氨基酸总数显著多于亲水氨基酸,疏水性强。
2.3.3 三级结构预测 利用SWISS-MODEL预测S蛋白的三级结构,得到22个模型,以下仅展示覆盖度最高的模型,如图6所示。该模型包含两个重要的结构:纤突糖蛋白(Spike glycoprotein, S)和血管紧张素转化酶2 (angiotensin converting enzyme 2, ACE2)复合体,ACE2复合体由ACE2受体和“向上”结构域RBD组成。
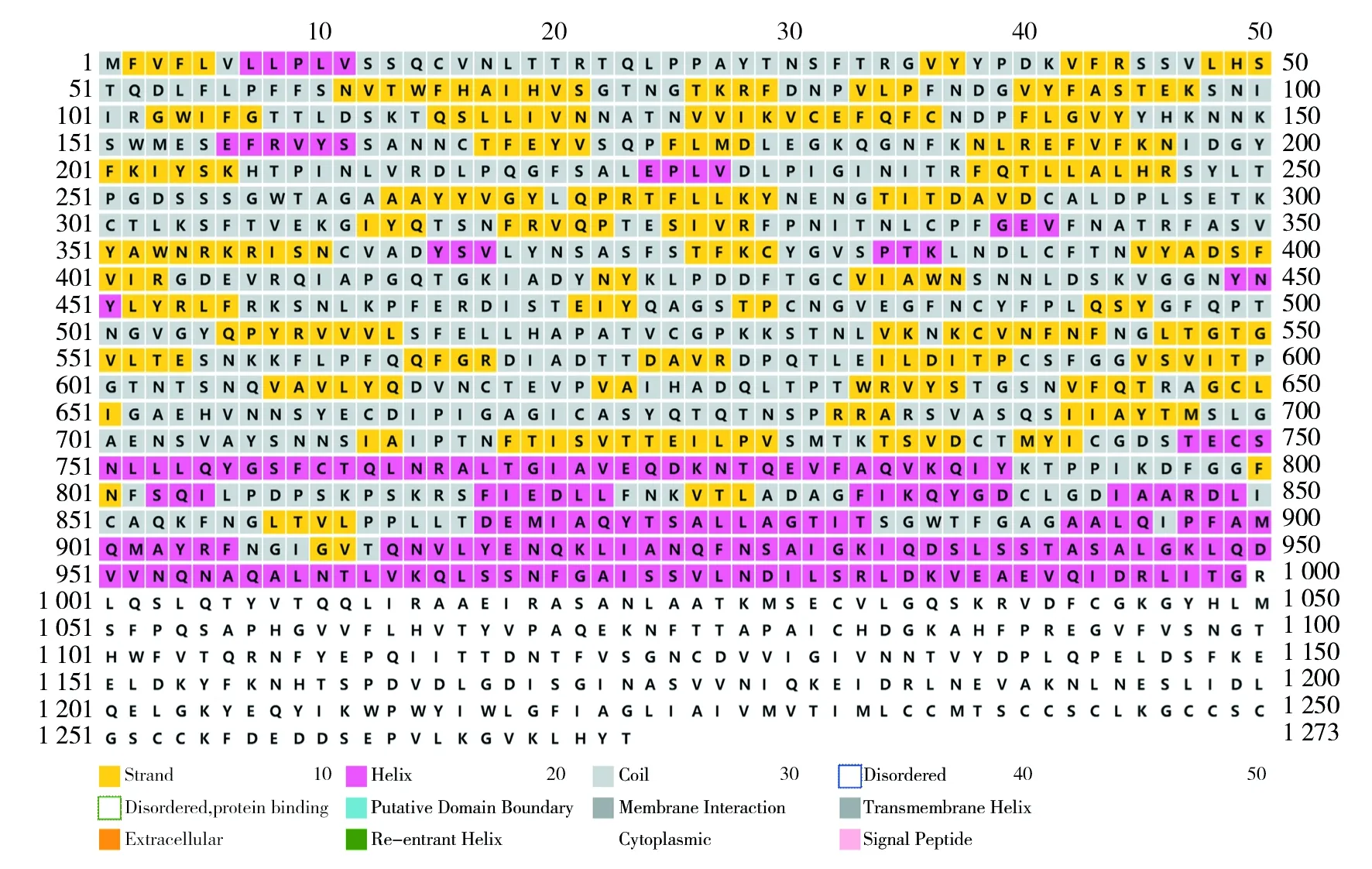
图4 S蛋白二级结构预测模型Fig.4 The prediction of secondary structure of S protein
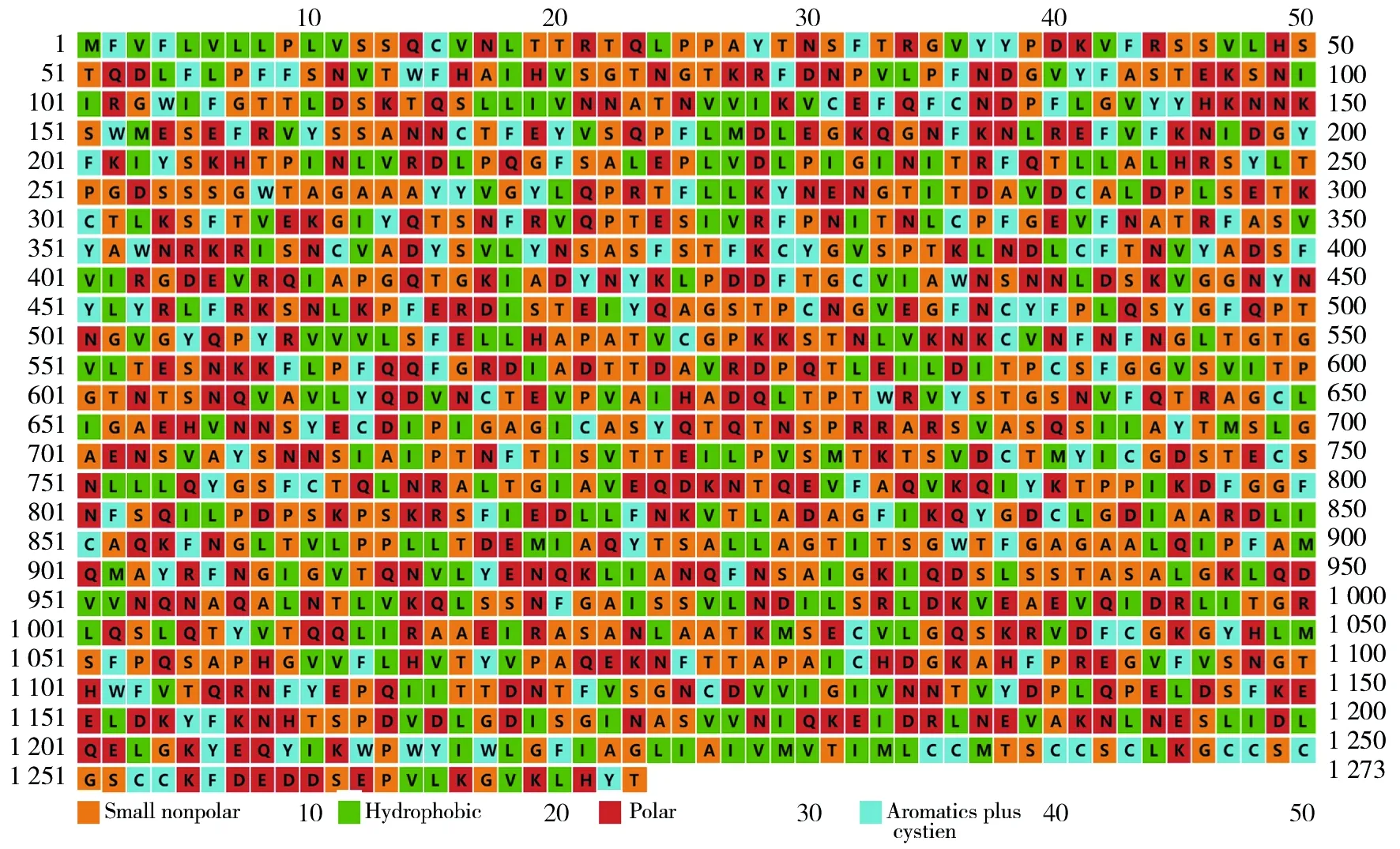
图5 S蛋白二级极性结构预测模型Fig.5 The prediction of secondary structure in polar property of S protein

图6 S蛋白预测三级结构的拓扑结构图Fig.6 The prediction of tertiary structure of S protein
2.4 抗原表位
2.4.1 T细胞表位预测 利用IEDB预测S蛋白的T细胞表位。结果显示,在1 203~1 209位氨基酸的抗原性最强,有4个阳性抗原表位,3个阴性抗原表位,应答频率最高达到0.85,最低0.5。其次为1 167~1 175、411~419、978~981和541~544位氨基酸。
2.4.2 B细胞表位预测 利用IEDB预测S蛋白的B细胞表位。结果显示,在1 202~1 210位氨基酸的抗原性最强,有4个阳性抗原表位,应答频率最高达到1,最低0.85,其次为978~985、1 167~1 175和411~420位氨基酸。其他区域的氨基酸B细胞表位抗原性较弱。
2.5 2019-nCoV S蛋白与其他物种的比较与进化分析
利用BLAST分析2019-nCoV S蛋白与其他物种的相似性,见表5。结果显示,2019-nCoV S蛋白与蝙蝠冠状病毒RaTG13的纤突糖蛋白相似性高达97.41%,与蝙蝠SARS样冠状病毒的纤突蛋白相似性达到80.32%,与SARS样冠状病毒WIV16的纤突蛋白相似性达到77.07%,与重组冠状病毒的纤突糖蛋白相似性达到77.38%。为进一步探究其间的进化关系,利用MEGA绘制进化树(图7)。结果显示,2019-nCoV、蝙蝠冠状病毒RaTG13、蝙蝠SARS样冠状病毒、SARS样冠状病毒WIV16和重组冠状病毒源自同一个祖先。

图7 2019-nCoV S蛋白与其他物种的进化关系分析Fig.7 Evolutionary analysis of S protein between 2019-nCoV and other species
3 讨 论
生物信息学利用高通量测序比对技术,分析蛋白质的基本理化性质、功能和结构等,能从多种角度进行蛋白质研究,为疾病诊断和疫苗研制提供方向[9]。本研究通过生物信息学,得到了2019-nCoV S蛋白的理化特征、结构、功能、进化和抗原表位的预测结果。
理化性质的预测结果显示,S蛋白在哺乳动物体外网织红细胞中的半衰期高达30 h,等电点为6.24,且不稳定系数预测S蛋白为稳定蛋白。冠状病毒体外膜融合实验显示,当病毒处于弱酸性环境时,更易转化为不稳定状态,与宿主细胞膜融合[10]。S蛋白的极性分析结果表明,2019-nCoV S蛋白脂溶性氨基酸数目显著高于水溶性氨基酸,亮氨酸(脂溶性)数目最多,高达108个,为高脂溶性蛋白。冠状病毒是一种膜包膜病毒,其融合肽S2亚基通常是脂溶性的,以利于病毒与宿主细胞膜磷脂双分子层融合,进而实现入侵和传播机制[11]。以上结果表明,2019-nCoV S蛋白具备入侵宿主和稳定繁殖的条件。
进化分析显示,2019-nCoV S蛋白和蝙蝠冠状病毒的纤突蛋白源自同一祖先,其氨基酸序列具有高度同源性。海外有学者发现,在中国境内,几乎所有哺乳动物体内的冠状病毒都起源于蝙蝠体内的祖先病毒;甚至在世界范围,蝙蝠和冠状病毒的起源都存在着密切的联系,且蝙蝠能通过长距离飞行,实现传播机制[12-13]。说明COVID-19的流行与蝙蝠之间可能具有一定的联系,包括起源和传播。
经过进化分析,2019-nCoV S蛋白与SARS样冠状病毒和重组冠状病毒具有高度同源性。提示2019-nCoV与18年前的SARS-CoV之间具有联系,或许2019-nCoV是一种SARS-CoV突变病毒,抑或是在祖先病毒进化过程中相关基因突变的产物[12]。S蛋白的结构域预测结果进一步证实了这一想法。结果显示,2019-nCoV与SARS-CoV的S蛋白高度保守,皆由两个重要的结构域组成:纤突蛋白受体结合域S1亚基和糖蛋白S2亚基。国外有学者利用3D精细化造图技术重建了2019-nCoV的整体结构,其与SARS-CoV S蛋白高度相似,二者在超过959个Cα原子上的均方根偏差(root-mean-square deviation, RMSD)仅为3.8埃(Å);Wrapp等[4,8]利用冷冻电镜技术解析了2019-nCoV S蛋白的超微结构,得到了S1亚基和S2亚基。以上结果表明,2019-nCoV与SARS-CoV具有同源性,结构域高度保守。
相关研究表明,2019-nCoV与SARS-CoV的传播方式相似,皆可表现为跨物种传播和人传人[2,14]。但是,相比2002~2003年的SARS,此次COVID-19人传人的速度更快。国内有学者发现,2019-nCoV S蛋白与宿主细胞ACE2之间的亲和力是SARS的10~20倍[4], 表明ACE2在COVID-19的传播过程中发挥重要作用。
本研究对S蛋白的三级结构进行预测,发现ACE2复合体对S蛋白的三级结构具有重要意义;而ACE2复合体由ACE2受体结合域和“向上”结构域RBD组成。这表明ACE2受体结合域和“向上”结构域RBD对ACE2复合体功能的发挥具有重要作用,可能是2019-nCoV入侵宿主,实现增殖和传播的重要机制。 “向上”构象RBD是宿主受体可达的结构域,呈不稳定的活跃状态,当2019-nCoV侵入人体时,仅当其表面的RBD处于“向上”状态,ACE2受体结合域才能与人体细胞膜表面的ACE2分子发生相互作用,进而入侵宿主细胞,实现增殖和传播,而当RBD处于“向下”构象时,S蛋白则进入“静止”的稳定状态[4,8,15]。ACE2受体结合域是2019-nCoV S蛋白与ACE2受体结合的关键结构,其与ACE2的亲和力可能是决定病毒传播速度的关键因素。更为重要的是,二者的相互作用可激活宿主免疫系统,启动 “炎症风暴”,而阻断炎症风暴,能有效干预病情进展,提示阻断ACE2受体结合域与ACE2结合是干预2019-nCoV的重要靶点[16]。
抗原表位是抗原引起机体免疫反应的重要媒介,寻找抗原表位能为疫苗研制提供思路[17-18]。本研究预测S蛋白具有多个线性抗原表位,且T细胞表位的数目多于B细胞表位。提示S蛋白具有强烈的免疫原性,或许能引起较强的T细胞免疫反应,与2019-nCoV造成的器官损伤机制相关。已有研究表明,T细胞表位在SARS-CoV S蛋白呈偏态分布。此外, 中东呼吸综合征冠状病毒(Middle East respiratory syndrome coronavirus, MERS-CoV)进入人体后,能引起强烈的T细胞免疫,而T细胞免疫相关细胞因子水平在死亡患者体内低于幸存患者[17]。在所有的抗原表位中,1 202~1 210位氨基酸区域间的抗原性最强,阳性抗原表位数目较多,所引起的宿主免疫应答较强。表明该段氨基酸序列免疫原性和反应原性强,或许能作为疫苗研制的候选序列。磷酸化位点是药物作用的重要靶点[19],本研究预测到S蛋白具有多个有效的磷酸化位点,或许能作为抗病毒药物作用的潜在区域。以上结果表明,S蛋白具有较强的免疫原性和反应原性,且有多个可供抗病毒药物作用的磷酸化位点,提示S蛋白或许能用于疫苗研制和抗病毒研究,为COVID-19提供防治方向。
本研究对2019-nCoV表面的糖蛋白S进行生物信息学分析,预测基本理化性质、结构特征、功能特征、进化特征和抗原表位特征,这些对S蛋白的深入研究具有重要意义,或许能为COVID-19抗病毒药物和疫苗的研制提供思路,但仍需进一步临床和实验验证。