利用四交RIL群体定位小麦籽粒脱水速率QTL
2020-08-03朱冬梅胡文静别同德陆成彬赵仁慧高德荣
朱冬梅,胡文静,别同德,陆成彬,赵仁慧,高德荣,2
(1.江苏里下河地区农业科学研究所/农业农村部长江中下游小麦生物学与遗传育种重点实验室,江苏扬州 225007;2.扬州大学/江苏省粮食作物现代产业技术协同创新中心,江苏扬州 225009)
小麦蜡熟末期,籽粒的营养物质积累量最高,达到生理成熟,此后的脱水特性至关重要,关系到收获时间、烘干晾晒成本和安全储存。近年来,长江中下游麦区水稻直播面积扩大,导致小麦晚播现象严重[1],收获期经常遇连续阴雨,籽粒水分过高易发生霉变,影响及时入库。朱冬梅等[2]研究认为,不同小麦品种生理成熟期后的脱水速率存在差异;何贤芳等[3]认为,品种对蜡熟至收获期小麦籽粒脱水速率有极显著影响。因此,开展小麦籽粒生理成熟后脱水速率的遗传机制研究,挖掘快速脱水的基因资源及相应的QTL,对培育籽粒脱水快的品种尤为迫切和重要。已报道的关于谷物籽粒脱水速率遗传特性的研究多集中在玉米上,研究结果表明,玉米籽粒自然脱水速率是可稳定遗传的数量性状,受多基因控制,广义遗传力较高,以加性效应遗传为主[4-6]。学者们相继利用各种遗传群体定位到一些与玉米籽粒脱水速率相关的QTL[7-9]。迄今未见关于小麦籽粒脱水速率相关QTL定位的报道。
与传统的双亲本定位群体RIL相比,多亲本RIL群体定位 QTL具有以下优势:(1)多亲本RIL群体中包含来源于不同亲本的多个等位基因,可以发现不同来源的等位基因对某个性状的影响[10-11];(2)可选用育种中性状优异的材料作为亲本,经过多次重组创造大量的遗传变异,群体中出现的优良株系可用做育种中间材料或品系[12]。Huang等[13]构建了小麦中第一套4亲本RIL群体;随后8亲本和16亲本的小麦MAGIC群体也相继构建完成[14]。水稻中,Bandillo等[15]选用育种中产量、抗性、米质等性状优异的材料作为亲本构建了4套MAGIC群体。大麦、玉米等其他重要农作物中也都完成MAGIC群体的构建[16-17],学者们相继利用这些多亲本RIL群体进行了多个农艺性状的QTL定位,获得了许多性状优异的育种群体,为分子标记辅助育种提供了材料和技术支撑。
扬麦16、扬麦20和扬麦22系江苏里下河地区农业科学研究所培育的小麦品种,扬麦16是近年来长江中下游推广面积最大的小麦品种,生理成熟后籽粒脱水快[2]。镇麦168是镇江丘陵地区农业科学研究所培育的品种,籽粒脱水亦较快。本研究以镇麦168、扬麦20、扬麦16和扬麦22为亲本创建的四亲本RIL群体为材料,利用小麦15K SNP芯片构建连锁图谱,定位小麦籽粒脱水速率的QTL,为小麦籽粒脱水性状的研究和选育脱水快的品种提供理论支撑。
1 材料和方法
1.1 试验群体
将4 个小麦亲本镇麦168(Zhengmai 168,ZM168)和扬麦20(Yangmai 20,YM20)、扬麦16(Yangmai 16,YM16)和扬麦22(Yangmai 22,YM22)(表1)两两成对杂交(ZM168×YM20、YM16×YM22),产生 2对双亲本杂交种,将 2对双亲本杂交种 F1再成对杂交产生四亲本杂交种(ZM168/YM20//YM16/YM22),四亲本杂交种通过单粒传法加代,自交 6 代,建成四交RIL群体,获得 158个稳定株系的ZM168/YM20//YM16/YM22群体。

表1 用于构建四交RIL群体的品种来源及其主要特征Table 1 Origin and agronomic characteristics of the four varieties for developing the four-way wheat population
1.2 田间设计和脱水速率测定
试验群体及亲本于2018年在江苏里下河地区农业科学研究所湾头试验基地种植,适期播种,采用4重复随机区组设计,每系种植3行,行长1.3 m,行距23 cm。肥料运筹为基施复合肥(N、P和K含量均为15%)800 kg·hm-1,壮蘖肥(尿素)50 kg·hm-1,拔节肥(尿素)200 kg·hm-1。开花期用多酮和吡虫啉防治赤霉病、白粉病、蚜虫等。其他管理与大田生产一致。在小麦开花期,每系选择开花时期、穗型大小一致且无病虫害的单穗50个挂牌标记,生理成熟期及其7 d后分别取样,每系取10个标记的穗子,快速剥取籽粒,称鲜重计数,在105℃下烘30 min杀青,80℃烘至恒重,计算籽粒含水率,籽粒含水率=(籽粒鲜重-籽粒干重)/籽粒鲜重×100%。前期试验结果表明,所有系的籽粒水分含量从生理成熟期降至13%时的最短时间间隔是7 d。
脱水速率(%)=(生理成熟期籽粒含水率- 7 d后籽粒含水率)/7。
1.3 SNP基因型和群体结构分析
采用中国农业科学院作物科学研究所与中玉金公司合作开发的小麦15K SNP芯片对试验群体及亲本进行基因型分析,按以下 3 个步骤过滤 SNP 基因型数据,一是将所有杂合基因型作为缺失数据,删除缺失率10%以上标记数据的株系; 二是剔除所有稀有等位基因频率低于3%的标记; 三是删除标记间相关性很高(在0.95 以上)的关联标记。使用 TASSEL V5.2.3 软件评估群体结构,由主成分分析(principal components analysis,PCA)中的 PC1 和 PC2 来揭示。
1.4 数据分析和 QTL 定位
使用 SAS V9.2(SAS Institute Inc. Cary NC,USA) PROCGLM对试验群体和亲本的表型进行统计分析。利用GAPL软件(http://www.isbreeding.net)[18-19]对群体进行连锁作图和QTL定位;以LOD=2.5为阈值确定与小麦脱水速率显著相关的QTL。QTL的命名方法为“Q”加性状缩写,加单位名称缩写,加QTL所在的染色体,同一染色体上多个QTL用.1、.2、.3……来表示。为了与前人结果比较,将连锁标记或者基因的序列与中国春在Ensembl Plants数据库(http://plants.ensembl.org/)中的参考基因组序列进行比对,获得标记或者基因的物理位置。
2 结果与分析
2.1 亲本及群体的脱水速率表现
由表2可以看出,扬16和镇麦168的平均脱水速率显著高于扬麦20和扬麦22,群体脱水速率的最小值和最大值之间差异明显,与亲本的表型值相比存在超亲分离。统计分析表明,脱水速率的遗传力是0.85,群体的偏度和峰度绝对值均小于1,分布呈连续的正态分布。
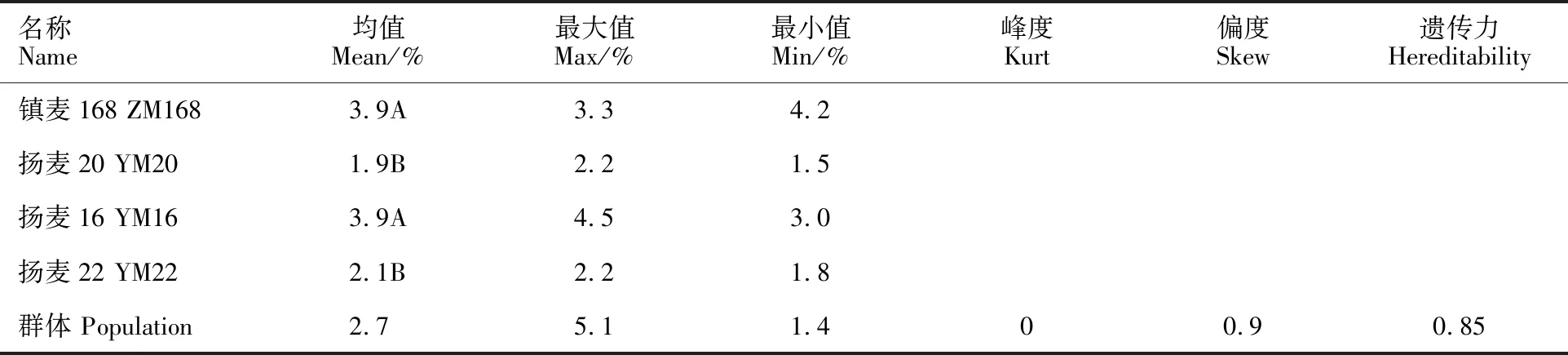
表2 亲本和群体的脱水速率表型值统计分析Table 2 Statistic analysis of dehydration rate for the parents and population
2.2 SNP分析和群体结构
过滤15K SNP基因型数据,最终在群体中得到2 761个高质量 SNP 位点用于群体结构分析,主成分分析结果表明,群体的 PC1 和 PC2分别是5.9%和5.8%,没有表现出明显的群体结构(图1)。
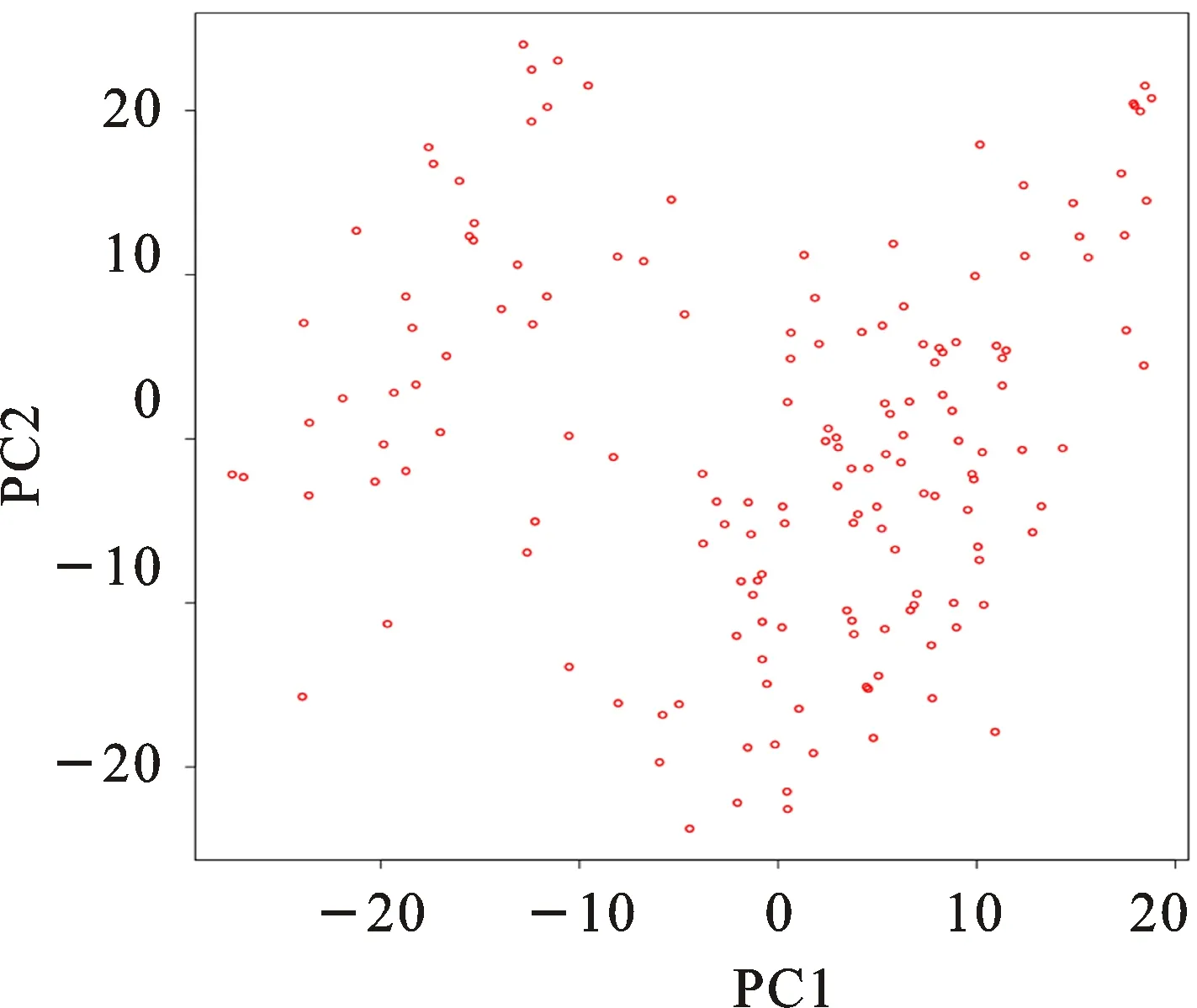
图1 群体的主成分分析结果
2.3 连锁作图
利用过滤后得到的2 761个高质量SNP位点,经过GAPL(http://www.isbreeding.net)的SNP功能剔除无效标记和BIN功能去冗余分析后,得到672个SNP标记构建连锁图谱。图谱覆盖小麦21条染色体,长度为13 167.1 cM,标记间平均距离是19.6 cM。分布于小麦A、B和D染色体组的标记数分别为273、241和158个,连锁长度分别为5 361.8、4 744.0和3 061.3cM。
2.4 脱水速率 QTL 定位
共检测到12个与脱水速率显著相关的QTL(图2,表3),将连锁标记序列与中国春的EnsemblPlants数据库(http://plants.ensembl.org/)参考基因组序列进行比对,发现12个QTL分布在1AL(2)、2AL、3BS、3BL、4AS、5BL、6DL、7AL、7BS、7BL和7DS上,其中,脱水速率增效基因QDR-yaas-6DL、QDR-yaas-1AL.1和QDR-yaas-3BL仅来自扬麦16(加性效应为正),分别位于6D染色体的445.8~450.7 Mb、1A染色体的519.3~521.3 Mb和3B染色体的493.1~493.6 Mb 处,LOD值分别是2.9、3.9和3.1,可解释表型变异率的8.1%、7.6%和2.8%。脱水速率增效基因QDR-yaas-1AL.2、QDR-yaas-2AL、QDR-yaas-4AS和QDR-yaas-7DS仅来自镇麦168(加性效应为正),分别位于1A 染色体的 563.3~ 589.7 Mb、2A染色体的697.4~698.2 Mb、4A染色体的 23.2~26.0 Mb和7D染色体的 72.5~ 75.2 Mb,LOD值分别是2.5、3.0、2.6和2.7,可解释表型变异率的3.6%~4.2%。脱水速率增效基因QDR-yaas-5BL仅来自扬麦20(加性效应为正),位于5B染色体的682.7~ 685.3 Mb,LOD值是2.7,可解释表型变异率 5.4%。脱水速率增效基因QDR-yaas-3BS和QDR-yaas-7BS同时来自扬麦20和扬麦16(加性效应为正),分别位于3B染色体的15.2~15.7 Mb、7B染色体的124.1~135.0 Mb,LOD值分别是3.4和3.8,可解释表型变异的3.2%和 3.8%。QDR-yaas-7AL和QDR-yaas-7BL脱水速率增效基因同时来自镇麦168和扬麦16(加性效应为正),分别位于7A染色体的593.5~611.5 Mb、7B染色体的692.9~693.2 Mb,LOD值分别是4.3和3.3,可解释的表型变异的4.8%和5.9%。
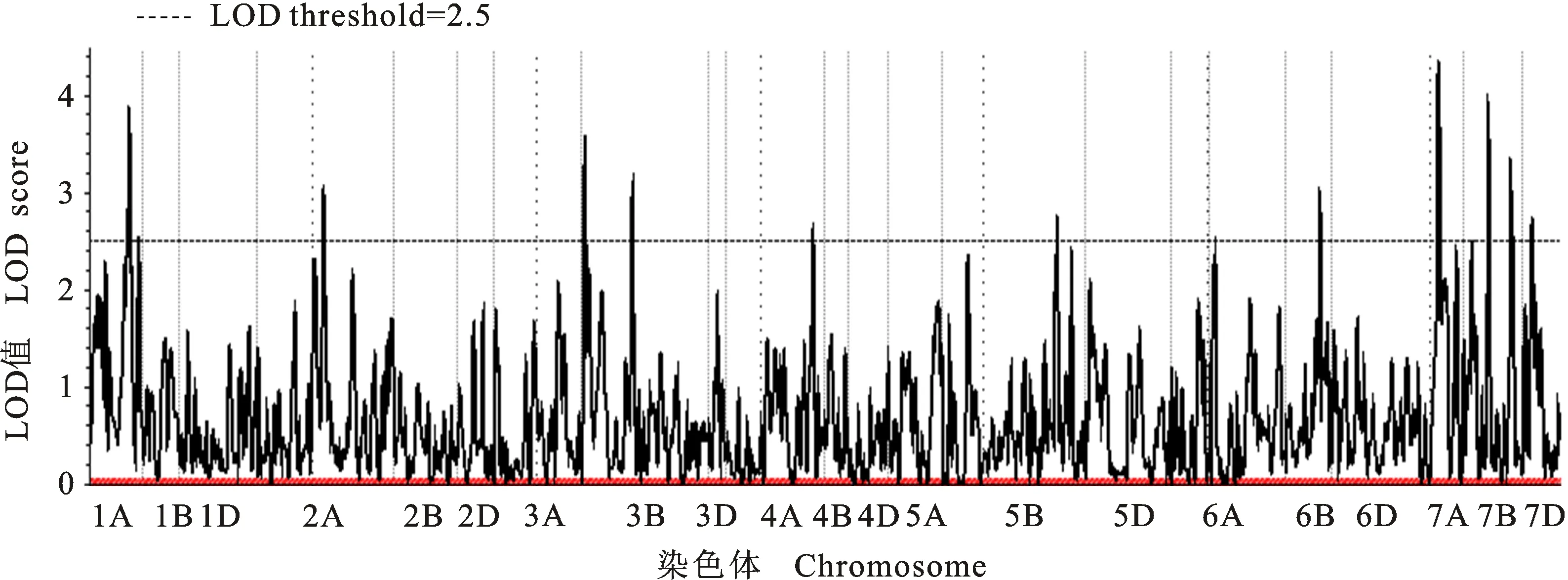
虚线表示LOD阈值=2.5。
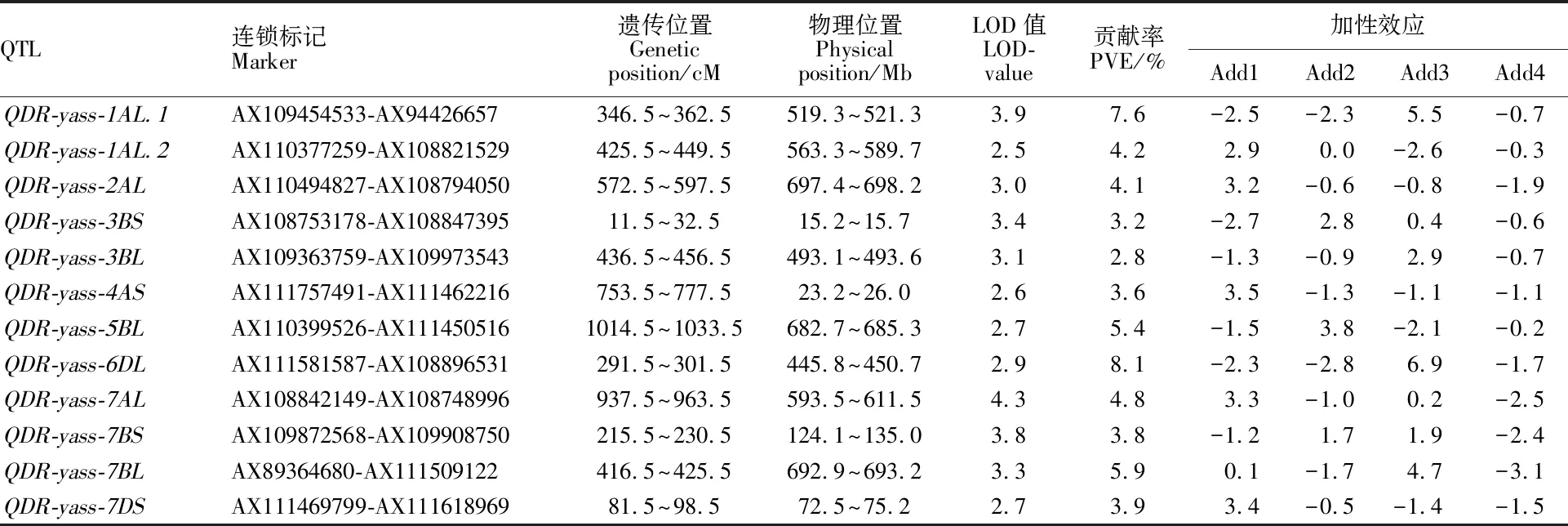
表3 群体中检测到的脱水速率相关QTLTable 3 Quantitative trait loci(QTLs) for dehydration rate in population
3 讨 论
3.1 同类QTL的研究比较
前人研究玉米籽粒脱水遗传特性,发现籽粒脱水速率与一些其他性状相关。如Capelle 等[20]测定了玉米29个性状共找到了 78个QTL,其中与玉米籽粒脱水相关的QTL位点43个,控制ABA含量有关的QTL位点20个,有10个位点在控制ABA含量与控制水分的QTL间具有共线性。向 葵[21]整理了已发表的玉米籽粒含水量 QTL和抗穗粒腐病性QTL的文献,利用元分析方法找到了 14 个“一致性”QTL 位点。国内外关于小麦籽粒脱水速率相关QTL的定位研究未见报道,本研究首次利用小麦四交RIL群体定位出12个与小麦籽粒脱水速率相关的QTL,我们后续将对该群体与脱水速率显著相关农艺性状(如灌浆速率、千粒重)和抗病QTL进行深一步研究,以期明确小麦籽粒生理成熟后与脱水相关的一些性状的分子遗传机理。
3.2 QTL的遗传分析
扬麦16和镇麦168均表现脱水速率快。本研究定位结果显示,扬麦16中对脱水速率增效的QTL有7个,镇麦168中对脱水速率增效的QTL有6个,其中来自扬麦16的QDR-yass-6DL对脱水速率的贡献率最大,为8.1%。扬麦16的组合是扬麦158优系(91F138)/扬90-30,镇麦168的组合是苏麦6号/扬97G59(扬97G59是扬麦158的抗白粉病回交系),已有研究表明,扬麦158成熟后期籽粒脱水较快[2],因此推测扬麦158对镇麦168与扬麦16的脱水性状有一定贡献。扬麦20脱水速率慢,QTL定位结果表明其对脱水速率增效的QTL有3个,扬麦20的组合是扬麦10号/扬麦9号,扬麦10号是以扬麦158为轮回亲本育成的抗白粉品种,推测扬麦158为扬麦20贡献了较少的对脱水速率增效的基因。说明小麦籽粒脱水速率属于多基因控制的复杂数量性状,在遗传背景的影响下,脱水速率慢的材料中也会存在增效基因。
3.3 QTL的有效性分析
由于本研究所用芯片通量不高,造成在群体中呈现多态的标记数量有限,所定位的置信区间较大,因而无法有效实现脱水速率相关QTL的剖析,使得多亲本群体在基因定位上的优越性受到一定程度的制约。已有研究表明,玉米籽粒脱水速率是数量性状,不仅受遗传因素影响和后期环境因素影响,还受2者互作效应的影响,因此在不同产区测出的结果不尽相同[22]。本研究选择在扬州点进行1年小麦四交RIL群体籽粒生理成熟后脱水速率测定及相关QTL定位,可以作为一个初级定位结果,其有效性和稳定性还有待进一步试验。为此本研究小组在2019年已经对该四交RIL群体增加了相对环境较稳定易控制的温室评价,以验证初定位的结果,并且正在利用小麦参考基因组序列,针对目标区域开发分子标记,增加图谱密度,对目标QTL区域进一步精密定位和验证。
4 结 论
本研究在四亲本RIL群体中共检测到与小麦籽粒脱水速率相关的QTL 12个,多数脱水速率快的QTL来源于扬麦16和镇麦168,其中来源于扬麦16的QDR-yaas-6DL、QDR-yaas-1AL.1效应较大;发现小麦籽粒脱水速率性状存在超亲分离现象。本研究为小麦生理成熟后籽粒脱水性状的深入研究奠定基础。
致谢:感谢中国农业科学院作物科学研究所数量遗传课题组张鲁燕研究员提供GAPL软件并在进行四亲本RIL群体定位QTL中给予指导。
