浮游动物DNA宏条形码标志基因比较研究
2020-07-02高旭杨江华张效伟
高旭,杨江华,*,张效伟,2
1. 污染控制与资源化研究国家重点实验室,南京大学环境学院,南京 210023 2. 江苏省环境保护化学品安全与健康风险研究重点实验室,南京 210023
作为一种新型的生物监测方法,DNA宏条形码(DNA metabarcoding)利用环境DNA序列差异快速、准确和高效地识别物种,适用于从微生物、浮游植物到鱼类等各个生物群落,在未来生态环境监测中有巨大的应用潜力[1-6]。DNA宏条形码技术不仅能实现物种有无监测,还能通过DNA丰度在一定程度上反映生物量的大小,打破现有以生物个体为主的研究模式,可用于研究环境胁迫对生物群落的影响,甄别环境关键污染因子和推导建立地域化环境基准[7-9]。目前浮游动物DNA宏条形码技术仍在发展阶段,尚缺乏统一的操作方法,需要对其(采样方法、引物选择和数据分析等)进行标准化和调整,然后才能用于常规流域生态监测。其中,如何选择合适的PCR扩增引物是DNA宏条形码生物监测的关键。
DNA宏条形码主要基于特定标志基因序列差异辨别物种,不同标志基因的物种覆盖度(coverage)和物种敏感性(sensitivity)存在巨大差异。高敏感性、低覆盖度的标志基因会导致大量物种的漏检,而高覆盖度、低敏感性的标志基因则不能有效地区分近源物种。因此,选择合适的标志基因是DNA宏条形码生物监测的基本保障。传统核糖体18SrDNA基因具有较高的物种覆盖度,能扩增出包括原生动物、真核藻类、真菌和后生动物[10]在内的大部分真核生物类群,但由于基因序列过于保守,物种敏感性较差,难以识别到种水平[11]。细胞色素C氧化酶Ι(cytochrome c oxidase Ι,COI)是最常用的动物标志基因,具有大量条形码序列积累[12-15]。COI是一个编码蛋白质的基因,存在“第三位密码子变异(third codon wobble)”,使传统COI引物在部分物种的鉴定中存在缺陷(taxonomic bias)[16-17]。线粒体12S和16S基因同样可用于DNA宏条形码研究[18-19],但是这2对引物多是为整个真核生物类群设计的,对浮游动物特异性未知。截至目前,大部分浮游动物研究仍基于18S[10,15,20-22]或28SrDNA基因[1]。Zhan等[23]比较了COI、16S和18S等标志基因在浮游动物条形码研究中的差异,发现传统COI引物难以提供稳定的PCR产物,建议选择18S和16S基因进行研究。虽然,Leray和Knowlton[24]在研究鱼类肠道浮游动物时设计了一个更适合高通量测序的COI引物,但其并未就该引物对浮游动物的覆盖度和敏感性进行深入探讨。
除各种通用引物外,学者们也针对不同生物类群设计了各种引物,譬如针对底栖动物、鱼、浮游植物、昆虫[25-27]和甲壳动物[1,28]的引物,也有很多引物的初衷是想覆盖整个真核生物类群[29-30]。对于浮游动物群落来说,究竟何种引物更为合适目前尚未明确。本研究利用Ion Torrent PGM测序平台比较了3对常见的宏条形码引物(COI、16S和18SV9)对浮游动物群落表征的差异,探究引物对淡水浮游动物宏条形码监测的影响,从物种检出、物种多样性和群落结构解析等方面分析最适合浮游动物宏条形码监测的引物,为初步建立标准化的浮游动物宏条形码监测提供依据。
1 材料与方法 (Materials and methods)
1.1 样品采集
浮游动物采样点位如表1所示,分别在2013年4月、8月和11月采集样品。每个点位用13号浮游生物网浓缩20 L环境水样(半定量采样),浓缩液现场用冰盒储存,在实验室用0.45 μm的滤膜(Millipore, USA)进一步过滤去除水分,过滤后的滤膜装入5 mL离心管中-20 ℃储存直至DNA提取。
1.2 浮游动物DNA提取
用E.Z.N.A. water DNA kit (Omega, USA)试剂盒提取滤膜DNA。简要操作为:将滤膜剪碎后(3 mm大小)装进5 mL无菌离心管中,加入0.2 mg的直径为2 mm的玻璃珠(试剂盒提供),加入1 mL裂解液,用MoBio Vortex-Genie2 (MoBio Laboratories Inc., CA, USA)在最高转速下均质5 min后按照试剂盒说明书完成后续操作。
表1 采样点位经纬度信息Table 1 Basic information of sampling sites
1.3 PCR扩增
PCR扩增体系为50 μL,包含5 μL的10×PCR High Fidelity PCR buffer、2 μL 50 mmol·L-1的MgSO4、1 μL的10 mmol·L-1dNTP mix、0.2 μL的Platinum Taq DNA polymerase (Invitrogen, USA)、2 μL的模板DNA和1 μL的引物(表2)。PCR程序为:预变性95 ℃、30 s,35个扩增循环中95 ℃变性10 s、退火30 s(COI为48 ℃,16S为55 ℃,18S为65 ℃),72 ℃延伸30 s,72 ℃延伸5 min。PCR产物通过2%琼脂糖凝胶电泳检测后用MinElute gel extraction kit试剂盒(Qiagen, CA, USA)进行纯化,纯化后用Qubit dsDNA HS assay kits (Invitrogen, USA)定量。
1.4 Ion Torrent PGM高通量测序
为了保证样本获得大致等量的序列数(Reads),所有的PCR产物均被稀释到10 ng·μL-1的浓度,然后将所有样点样品等体积混合在一起。混库后取100 ng建库,用Agencourt AMPure XP kit (Beckman Coulter, Germany)纯化测序文库,去除短片段(<100 bp),用Ion PGM测序仪(Life Technologies, USA)进行测序。
1.5 数据分析1.5.1 质量控制
测序结束后用Fastx toolkits将FASTQ文件转为FASTA和QUAL文件[31]。在QIIME(Quantitative Insights into Microbial Ecology v1.8.0)[32]中去除低质量(Q<20)的序列、错配序列和长度<150 bp的序列。利用UCHIME[33]识别并去除PCR嵌和子,以上操作均在Bio-Linux 8系统[34]下完成。利用R “Biostrings”包[35]进行序列比对,去除离群序列。
1.5.2 生物信息学分析
利用UPARSE[36]进行操作分类单元(operational taxonomic unit, OTU)聚类。序列注释在Statistical Assignment Package软件中以默认参数进行[37],其中,序列相似性>95%且后验概率>50%的OTU被注释到物种水平。

表2 引物序列Table 2 Primer sequences
2 结果与分析(Results and analysis)
2.1 不同引物的OTU多样性
随着测序深度的增加,COI、16S和18SV9检出的OTU数量亦不断增加。当测序深度达到2 000 000左右时,16S引物检出的OTU数量趋于饱和,平均每10 000条序列新增1.9个OTU;测序深度达到30 000 000时,COI引物检出的OTU数量趋于饱和,平均每10 000条序列新增1.8个OTU;18SV9引物在测序深度达到16 000 000时检出的OTU才逐渐趋于平衡,增加率为平均每10 000条序列1.2个OTU(图1)。
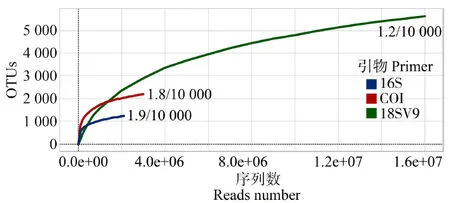
图1 不同测序深度下检出的操作分类单元(OTU)数量Fig. 1 The number of operational taxonomic unit (OTU) in different sequencing depth
18SV9引物共检出超过16 000个OTU,其中有6 891个OTU(占总序列的8.6%)属于未知序列,最后能够被注释的OTU有9 450个(占总序列的91.4%)。仅有1 461个18SV9的OTU隶属于浮游动物群落,其中,轮虫438个(占总序列的17.3%),枝角类159个(占总序列的3.78%),桡足类864个(占总序列的44.75%)(图2)。
COI引物共检出OTU 2 259个,其中有1 669个OTU能够被注释(占总序列的93.4%),这其中浮游动物OTU有1 211个,包括轮虫729个(占总序列的27.4%),枝角类235个(占总序列的14.7%)和桡足类246个(占总序列的30.63%)(图2)。
16S引物共检出1 277个OTU,其中有390个OTU(占总序列的6.62%)无法确定物种来源,能被注释的OTU有887个(占总序列的93.4%),这其中有782个OTU来自浮游动物类群,包括轮虫548个(占总序列的59.6%),枝角类235个(占总序列的26.11%)和桡足类46个(占总序列的3.1%)(图2)。
尽管18SV9引物检出的总OTU数量远大于16S和COI,但其中有大量OTU来自于藻类和原生动物,其检出的浮游动物OTU数量与16S和COI引物接近。
2.2 检出浮游动物种类差异
COI和16S引物检出的浮游动物物种种类较接近,有64种浮游动物能够同时被COI和16S引物检出,有28个物种仅能被COI引物检出,另有10个物种仅能被16S检出。18SV9引物获得的大部分序列都无法注释到物种水平,因此,在物种检出上其与COI和16S差别较大,仅5个物种能够同时被18SV9、COI和16S检出(图3)。

图2 不同引物检测到的OTU数和序列Reads组成注:(a)不能注释OTU比例;(b)不能注释Reads的比例;(c)OTU组成;(d)Reads组成。Fig. 2 The composition of OTU and Reads in zooplankton metabarcoding with different primersNote: (a) proportion of “unknown” OTU; (b) proportion of “unknown” Reads; (c) composition of OTU; (d) composition of Reads.

图3 不同引物OTU注释出浮游动物种数注:(a)浮游动物OTU数量和被注释的等级;(b)检出物种数。Fig. 3 The number of zooplankton species detected by different metabarcoding primersNote: (a) the number of zooplankton and the level of taxonomic assignment in each primer; (b) number of species detected by different primers.
COI和16S引物对轮虫和枝角类检测结果的一致性要好于对桡足类检测结果的一致性。大部分轮虫和枝角类都能同时被这2对引物检出,且物种OTU数量也较为接近(图4)。虽然,2对引物检出的桡足的种类差别不大,但每个物种对应的OTU数却相差明显。例如16S引物发现指状许水蚤(Schmackeriaforbesi)仅6个OTU,但是COI引物却发现其OTU数量超过30个。类似的情况还出现在汤匙华哲水蚤(Sinocalanusdorrii)和中剑水蚤(Cyclopsp.)中。18SV9引物中除萼花臂尾轮虫(Brachionuscalyciflorus)的检出与COI和16S存在较高的一致性外,其他物种的检出均与另外2对引物存在较大差异。18SV9中有大量OTU被注释为鄂足纲(Maxillopoda),并不能往下细分。
2.3 引物对浮游动物多样性的影响
COI桡足类多样性与18SV9桡足类多样性存在明显的正相关(R2=0.4,P<0.05),而16S桡足类多样性与COI及18SV9桡足类多样性之间没有明显相关性。这部分说明,18SV9引物和COI引物能更稳定地表征桡足类多样性(图5(a)、图5(b)和图5(c))。
对枝角类而言,16S引物检出的枝角类多样性与COI(R2=0.35,P<0.05)和18SV9存在明显相关性(R2=0.43,P<0.05),但是COI和18SV9之间却没有明显相关;这表明在物种多样性的角度上,虽然16S、18SV9和COI引物都能表征枝角类多样性,但16S监测的结果相对更加稳健(图5(g)、图5(h)和图5(i))。
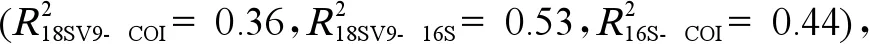
2.4 浮游动物的季节差异
COI、18SV9和16S都能很好地表征浮游动物群落组成的季节差异。其中,COI和16S能够明确区分3个采样季节对浮游动物群落的影响。18SV9显示8月份的浮游动物组成明显与12月份和4月份不同,但4月份和12月份间的差别并不明显(图6)。宏条形码监测除了能反映季节差异,还反映了不同水体类型中浮游动物组成的差异,尤其是河流的浮游动物组成与太湖的浮游动物组成存在明显差异,河流中轮虫比例更高,而太湖中枝角类和桡足类更为丰富。
3 讨论(Disscussion)
3对引物中,18SV9引物具有物种覆盖度高、测序成本低的特点,更适合种以上水平的物种多样性监测。虽然,18S引物具有很高的物种覆盖度,但其对浮游动物的扩增特异性较差[11]。尽管本研究的采样方法已经提前对浮游动物进行了富集,18SV9测序中仍有很大一部分OTU来自藻类、真菌和原生生物,说明18SV9引物更加适合原生生物和藻类多样性研究[38]。此外,18S的V9可变区长度仅130 bp左右,长度过短可能是物种识别敏感性较差的主要原因。有研究表明,即便是产物长度更长的18SV4高变区对物种的辨识度也不如同等长度的COI和16S[39]。18SV9测序结果中大量的OTU仅被注释到纲,导致物种水平多样性评估效果并不理想。另一方面,尽管大量18SV9 OTU无法注释到物种,但是基于OTU依然可以有效地反映季节和水体类型对浮游动物群落的影响[40]。因此,在不注重物种水平的多样性时,18SV9引物亦可用于宏条形码研究,毕竟其更为通用,能覆盖更多的物种类群,更全面反映整体生物多样性,且18SV9产物长度短、易于扩增,测序成本更低。
线粒体16S引物具有更高的浮游动物扩增特异性,更适合枝角类和轮虫多样性调查。16S测序产生的OTU大部分都来自浮游动物,说明其对浮游动物具有更好的特异性,使测序结果更加“高效”和“纯净”,数据浪费少,而另外2对引物测序都有相当比例的序列不属于浮游动物类群。16S引物具有更长的扩增产物(340 bp),能够很好地在物种水平上区分浮游动物。大部分轮虫和枝角类都能同时被16S和COI所检出。在16S发现的782个浮游动物OTU中,来自轮虫的有548个(占总序列的59.6%),枝角类235个(占总序列的26.11%),而桡足类仅有46个(占总序列的3.1%),对桡足类的检出效果稍差,会低估水体中桡足类多样性。
COI引物的扩增产物长度适中,能更加全面、均衡地反映整个浮游动物群落的完整性,为浮游动物宏条形码研究的理想引物之一。COI作为最常用于动物物种鉴定的条形码基因[41],为了能区别近缘物种,传统COI引物的扩增产物的长度为648 bp[29],超出了当前高通量测序的读长(目前主流的测序平台也只能稳定地测到550 bp左右),无法用于宏条形码研究。本研究中使用的COI引物的扩增产物长度为313 bp,在满足高通量测序读长要求的同时还兼顾了物种辨识敏感性。COI引物测序中共检出1 669个浮游动物OTU,其中,轮虫有729个(占总序列的27.4%),枝角类235个(占总序列的14.7%),桡足类246个(占总序列的30.63%),序列组成上在3个浮游动物群落中比较均衡。COI引物所发现的物种数量在3对引物中也是最多的,更为适合浮游动物群落组成的研究。理论上,引物的偏好性是不可避免的,本研究中讨论的3种标志基因也都不可能完全覆盖浮游动物群落的所有物种,需要根据不同的研究目的选择合适的引物。在进行探索性多样性调查及本底调查时,可采用多对引物的组合,以尽量检出更多的物种,丰富物种编目。而进行生态健康评估或者生态质量评价时应选择某种固定引物(如COI引物)进行调查,以减少多引物产生的不确定性。


图5 不同引物检出浮游动物多样性间的相关性Fig. 5 The correlation of zooplankton diversities detected by different primers
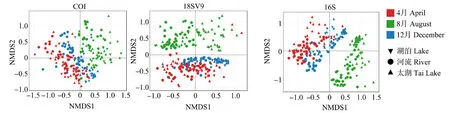
图6 浮游动物群落的季节差异注:基于OTU数据的非度量多维尺度法(NMDS)分析。Fig. 6 The difference of zooplankton community among different seasonNote: Non-metric multidimensional scaling (NMDS) analysis was based on the OTU data.
