拉氏鱥mtDNA COI区段的遗传多样性分析
2020-04-27郭芮名倩张兴华苗建新李崇文
白 云, 郭芮名倩, 王 妍, 胡 莹,张兴华, 苗建新, 李崇文, 董 仕
(1.天津师范大学 生命科学学院,天津 300387;2.天津师范大学 天津市动植物抗性重点实验室,天津 300387;3.天津市天祥水产有限责任公司,天津 301500)
拉氏鱥(Phoxinus lagowskii),又称洛氏鱥,俗称“柳根鱼”、“柳根子”[1],属于鲤形目(Cypriniformes)鲤科(Cyprinidae)雅罗鱼亚科(Leuciscinae)鱥属(Phoxinus),主要分布在黑龙江、图们江、鸭绿江、黄河中上游等水域,是所有鱥属中生长最快、个体最大的一种.拉氏鱥体质量一般为20~40 g, 人工养殖个体最大可达到500 g[2].拉氏鱥体型较长,呈纺锤型,体背部灰黄色并带有小的黑色斑点,具有味道鲜美、肉质好、蛋白质和不饱和脂肪酸含量高等优点[1],因此价格较高.近年来,随着环境变化和过度捕捞,拉氏鱥的数量逐渐减少,急需人工养殖来满足市场需要[3].由于拉氏鱥特殊的繁殖习性, 人工催产自然产卵的难题始终未能解决,因此难以实现苗种的批量生产,养殖苗种无法得到保障.现阶段我国拉氏鱥的人工养殖开始起步,在东北三省的人工养殖有零星报道[2].
DNA 条形码技术是 Hebert 等[4-5]于 2003 年提出的“测定线粒体DNA COI 区段碱基序列, 进而进行物种鉴定”的一种方法.线粒体DNA 又称mtDNA,为闭合环状结构,是细胞核外具备自主DNA 复制、转录以及翻译能力的双链DNA 分子[6].COI 区段全称为线粒体细胞色素C 氧化酶I 亚基(cytochrome C oxidase subunit Ⅰ,COI),具有进化速率快、包含遗传进化的信息量大以及各类群间序列差异较明显等优点,因此在DNA条形码技术中,此区段具有较高的物种鉴定的可靠性[7-8].利用DNA COI 区段进行鱼类遗传多样性的研究已有一些报道.如张大莉等[9]对大口黑鲈北方亚种和佛罗里达亚种的COI 区段序列分析表明, 后者mtDNA COI 区段的遗传多样性高于前者,该序列可作为鉴别2 个亚种的DNA 条形码.沙航等[10]报道了长江中上游6 个群体鲢COI 的遗传变异主要来自群体内的个体间,群体间具有明显的遗传分化.谢佳燕等[11]对蒙古鲌5 个群体的COI 基因序列的分析结果表明,不同种群间的遗传多样性存在一定差异但尚未形成明显的谱系地理结构.
本研究利用DNA 条形码技术,检测分析了40 尾拉氏鱥mtDNA COI 区段的碱基排列顺序及遗传多样性,并进行了不同大小拉氏鱥遗传差异的比较,其结果可为拉氏鱥的物种鉴定和育种提供资料.
1 材料与方法
1.1 实验材料
2018 年2 月28 日,从天津市天祥水产有限责任公司采集2017 年春季同批次人工繁殖、 同池养殖的拉氏鱥865 尾, 测量体长和体质量.拉氏鱥的个体大小差异明显,体质量范围为1.33~96.99 g.按照体质量从中选取大、中、小不同个体40 尾,进行组间遗传差异的比较和拉氏鱥mtDNA COI 区段遗传多样性分析.3 种体型拉氏鱥的体长和体质量如表1 所示.

表1 拉氏鱥个体的数量、体长和体质量Tab.1 Length and body mass of Phoxinus lagowskii
1.2 总DNA的提取
剪取大小约1.0 cm×1.0 cm 的鳍条,采用苯酚-氯仿抽提法提取总DNA,保存在4 ℃下备用[12].
1.3 mtDNA COI区段的PCR扩增
依据GenBank 上的KC967217 设计了用于扩增拉氏鱥mtDNA COI 区段的一对引物,LSGCO1 序列为5′-TAT TCT GCG TTT CTG GAT TT-3′,LSGCO2 序列为5′-TCT TCT ATT ACG GGT GAT GC-3′,结合位点分别位于tRNACys和COⅡ2 个编码区上,PCR 反应体系参照王淞等[13]的方法.获得的PCR 产物经电泳检测后,委托苏州金唯智生物科技有限公司进行测序.
1.4 数据处理
使用Bioedit 软件将序列转为Fasta 格式, 经Seqverter 软件进行整合,用Clustalx1.83 软件将序列的突变位点与峰图比对后获得排列清晰的序列, 再用MEGA7.0 软件得出碱基差异和碱基频率,基于Kimura2-parameter 计算组内和组间遗传距,并构建NJ 系统树.使用Dnasp 软件计算单倍型多样性指数(Hd)、核苷酸多样性指数(Pi)以及平均核苷酸差异性(k).
2 结果与分析
2.1 865尾拉氏鱥的体质量分布
本研究采集的865 尾拉氏鱥的体质量分布如图1所示.由图1 可以看出, 中等偏大体型的个体数量最多,其次是小体型,体质量较大的个体数量最少.
2.2 mtDNA COI区段的PCR产物
对选取的40 尾不同体型的拉氏鱥进行PCR 扩增,均获得了约1.85 kb 大小的DNA 片段,部分个体的PCR 产物电泳结果如图2 所示.对PCR 扩增产物测序后,与测序公司提供的测序峰图进行比对,获得碱基排列顺序清晰的DNA 片段长度为1 551~1 552 bp,经与 GenBank 中的 KC967217(物种名:Rhynchocypris cf lagowskii)比对,序列全部在 COI 区段.
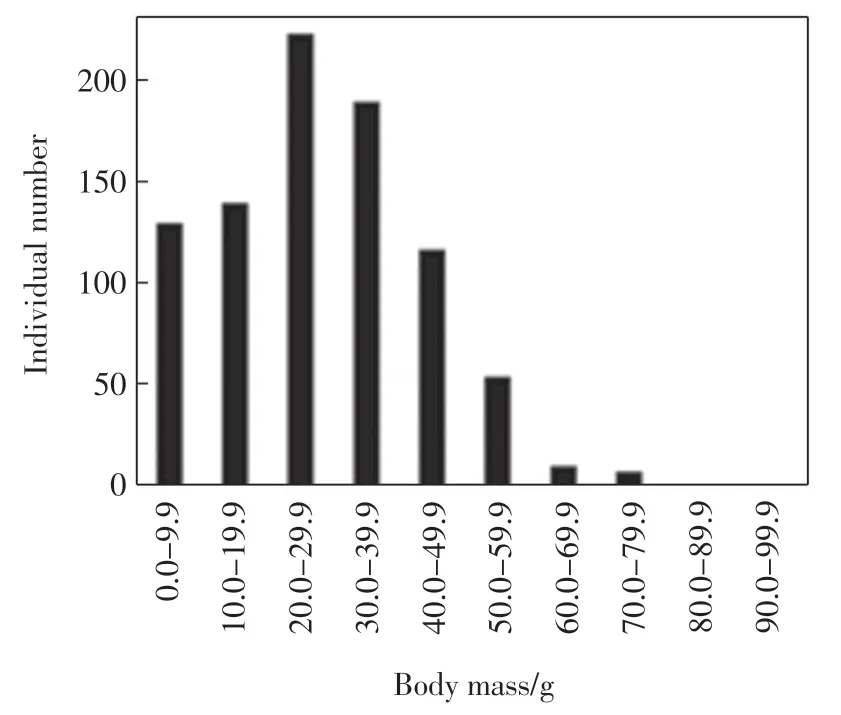
图1 拉氏鱥体质量的分布Fig.1 Distribution of body mass of Phoxinus lagowskii

图2 拉氏鱥mtDNA COI 区段的PCR 产物电泳检测结果Fig.2 Electrophorescs results of the PCR product of mtDNA COI region in Phoxinus lagowskii
2.3 拉氏鱥的碱基差异
40 尾拉氏鱥共有128 个位点存在碱基差异,有23 种单倍型(haplotype).计算单倍型间 Kimura 2-parameter 遗传距,通过与 KC967217、KF734881(物种名:Rhynchocypris lagowskii)、AP009147(物种名:Rhynchocypris lagowskii)比较,23 种单倍型可以分为 2 种类型:类型1 为单倍型Ⅰ~ XX;类型2 为单倍型XXI~XXⅢ.类型1 中的37 尾拉氏鱥有34 个位点存在碱基差异,如表2 所示;类型2 中的3 尾拉氏鱥有8 个位点存在碱基差异, 如表3 所示.类型1 各单倍型中碱基T 和A 的含量小于类型2 中的含量, 碱基C 和G的含量大于类型2 中的含量,如表4 所示.2 种类型各单倍型中不同大小拉氏鱥的个体数如表5 所示.
2.4 拉氏鱥的遗传距和系统树
2.4.1 单倍型间的Kimura2-parameter 遗传距
类型1 拉氏鱥(单倍型Ⅰ~XX)中,20 种单倍型间以及与KC967217、KF734881 间的Kimura 2-parameter遗传距范围在0.000 00~0.010 42 之间.类型2 拉氏鱥(单倍型XXI~XXIII)中,3 种单倍型间以及与AP009147间的遗传距范围在0.001 29~0.003 23 之间.类型1 与类型2 间的遗传距范围在0.073 75~0.080 48 之间.由此认为,类型1 与KC967217 和KF734881 相近,类型2 与 AP009147 相近, 如表 6 所示.2 种类型拉氏鱥间存在明显的遗传差异.
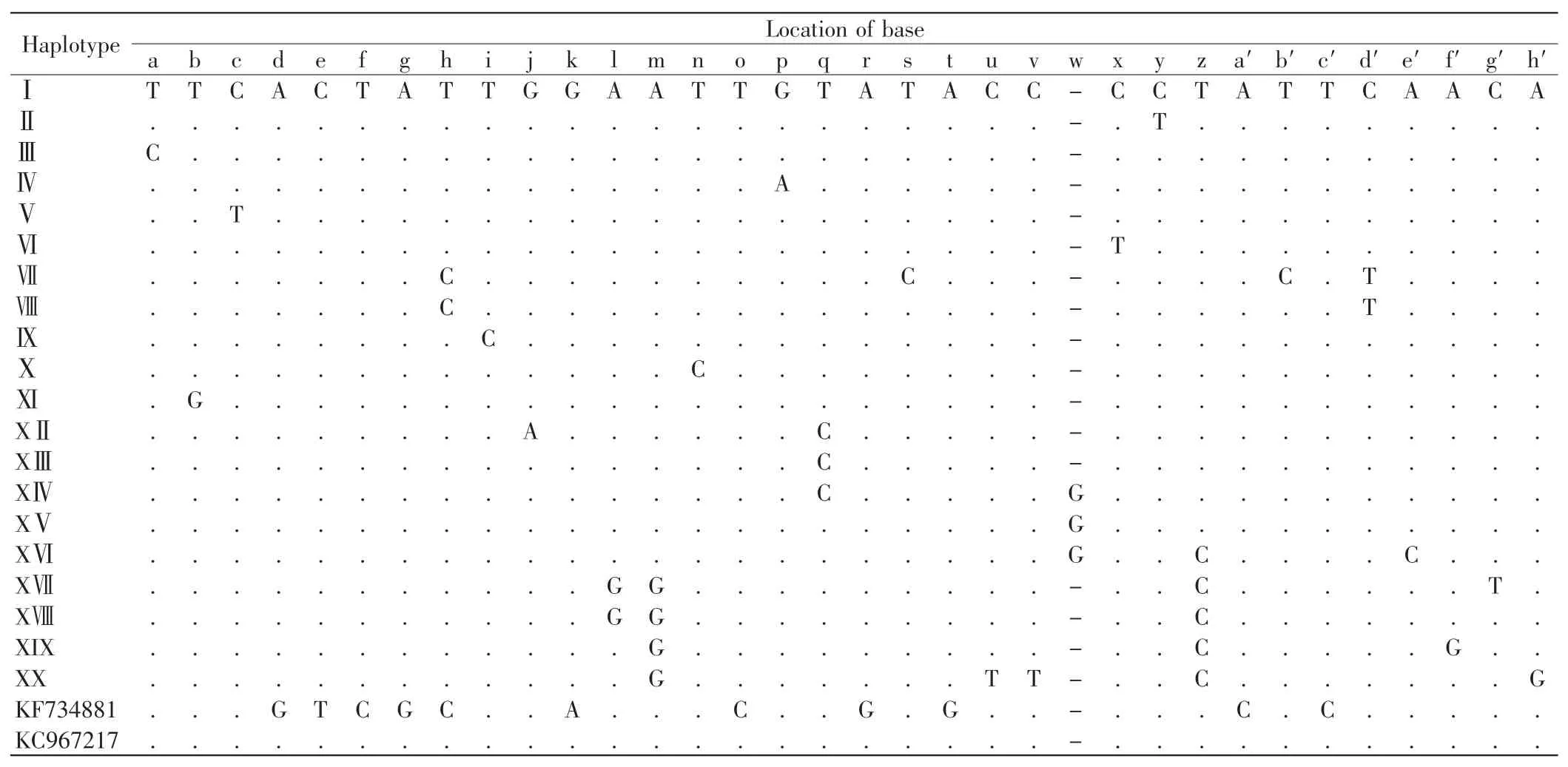
表2 拉氏鱥类型1 中单倍型的碱基差异Tab.2 Nucleotide diversity of the haplotypes in type 1 of Phoxinus lagowskii

表3 拉氏鱥类型2 中单倍型的碱基差异Tab.3 Nucleotide diversity of the haplotypes in type 2 of Phoxinus lagowskii

表4 拉氏鱥单倍型的碱基组成Tab.4 Nucleotide composition of the haplotypes of Phoxinus lagowskii

表5 各单倍型中拉氏鱥个体数Tab.5 Individual number of different haplotypes in Phoxinus lagowskii
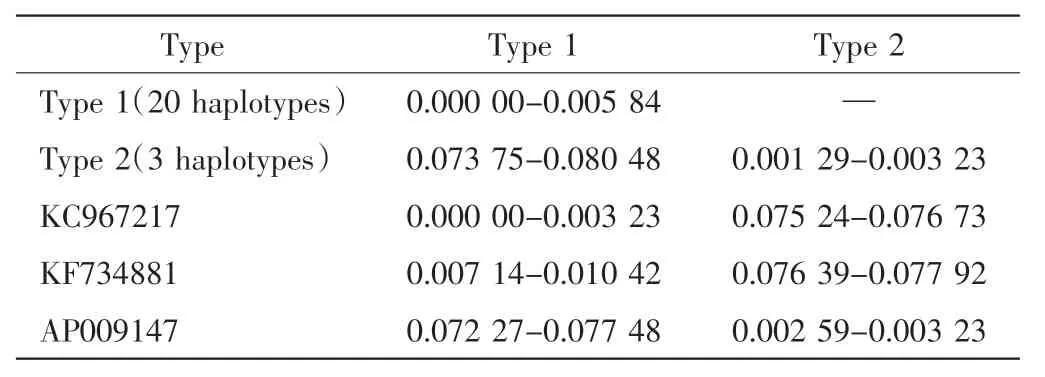
表6 拉氏鱥单倍型间Kimura 2-parameter 遗传距范围Tab.6 Range of Kimura 2-parameter genetic distance among different haplotypes in COI region of Phoxinus lagowskii
2.4.2 类型1 和类型2 的组内与组间遗传距
2 种类型拉氏鱥中组内和组间的遗传距如表7 所示.类型1 中37 尾拉氏鱥组内Kimura 2-parameter 遗传距平均值为0.001 71,与KC967217 和KF734881 间的平均值分别为0.000 93 和0.007 86,与类型2 组间的平均值为0.07596,与AP009147 的平均值为0.073 97.类型2 中3 尾拉氏鱥组内遗传距平均值为0.002 59,与 AP009147 的平均值为 0.002 80, 与 KC967217 和KF734881 间的平均值分别为0.075 74 和0.076 44.

表7 拉氏鱥COI 区段的Kimura 2-parameter 遗传距Tab.7 Kimura 2-parameter genetic distance in COI region of Phoxinus lagowskii
2.4.3 大、中、小拉氏鱥的组内和组间遗传距
类型1 中的大、中、小个体拉氏鱥组内、组间的Kimura 2-parameter 遗传距如表8 所示.遗传距范围为0.001 23~0.001 97,大、中、小3 组拉氏鱥之间的差异不明显.
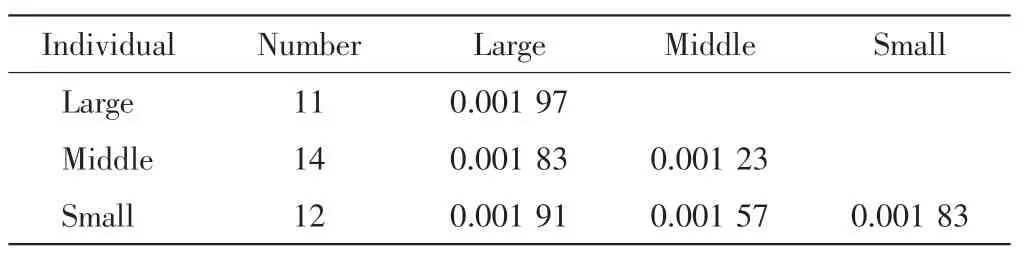
表8 不同体质量拉氏鱥COI 区段的Kimura 2-parameter遗传距离Tab.8 Kimura 2-parameter genetic distance in COI region of different body mass of Phoxinus lagowskii
2.4.4 拉氏鱥的系统树
40 尾拉氏鱥的系统树如图3 所示.类型1 中的37 尾拉氏鱥与 KC967217 和 KF734881 为一支; 类型2 中的3 尾与AP009147 为一支.拉氏鱥的聚类与大、中、小个体间未见显著关联.

图3 不同体质量拉氏鱥mtDNA COI 区段的NJ 系统树Fig.3 NJ phylogenetic tree in COI region of different body mass of Phoxinus lagowskii
2.5 拉氏鱥的遗传多样性指数
类型1 拉氏鱥的单倍型多样性指数(Hd)、核苷酸多样性指数(Pi)及平均核苷酸差异性(k)分别为0.862、0.001 71 和 2.646, 类型 2 拉氏鱥的 Hd、Pi和 k 分别为1.000、0.002 58 和4.000.类型2 拉氏鱥的遗传多样性略高于类型1 的多样性.
3 讨论与结论
对于拉氏鱥所在属及种的中文名、拉丁名,迄今仍没有统一定论.成庆泰等[14]使用的属名、种名为鱥属、洛氏鱥Phoxinus lagowskii;川那部浩哉等[15]命名为Phoxinus lagowski;许旺[16]使用的属名、种名为大吻鱥属、拉氏大吻鱥Rhynchocypris lagowskii,且存在拉氏大吻鱥的相似种Rhynchocypris cf lagowskii.mtDNA COI 区段作为一种DNA 遗传标记常用于遗传多样性检测、系统分类以及物种鉴定等研究[4-8].Hebert 等[4-5]对11 个门类2 238 种动物间的遗传差异进行比较,认为种内遗传距大多数在1%以下,很少超过2%,3%的遗传距可作为种类鉴别的标准.本研究选取了同批次相同条件下培养的40 尾体质量大小不一的拉氏鱥,对它们的线粒体DNA COI 区段进行PCR 扩增, 得到了23 种单倍型.计算 Kimura 2-parameter 遗传距,40 尾拉氏鱥可以明显地分为类型1(37 尾)和类型2(3 尾).2个类型组内单倍型间的遗传距均小于1%,而2 种类型间以及2 种类型的各单倍型间的遗传距均在7%以上,远远大于3%.如以3%的Kimura 2-parameter 遗传距为基准,可认为本研究中的类型1 与类型2 的母本应来自于2 个不同物种.类型1 与KF734881(物种名:Rhynchocypris lagowskii)和 KC967217(物种名:Rhynchocypris cf lagowskii)相近,类型 2 与AP009147(物种名:Rhynchocypris lagowskii)相近.本文认为KF734881 与AP009147 应为不同物种,由于实验中的拉氏鱥为人工繁殖后代,因此2 种类型母本的具体种名有待进一步研究.
在拉氏鱥养殖过程中发现,同时间人工繁殖、同池养殖的拉氏鱥个体间的体长、体质量差异明显.根据体质量将拉氏鱥分为大、中、小3 组,进行组内和组间遗传差异检测分析.大个体拉氏鱥的平均体长是小个体拉氏鱥的2.36 倍,体质量为17.47 倍.类型1 中3 组拉氏鱥的组内、组间遗传距范围为0.001 23~0.001 97,遗传差异不明显.本研究中测量的865 尾拉氏鱥的体长和体质量呈正态分布,养殖过程出现差异明显的个体可能归因于个体间的摄食能力差异.类型1 拉氏鱥的单倍型多样性指数(Hd)、核苷酸多样性指数(Pi)及平均核苷酸差异性(k)分别为 0.862、0.001 71 和 2.646,类型 2 拉氏鱥的 Hd、Pi和 k 分别为 1.000、0.002 58 和4.000.类型2 拉氏鱥的遗传多样性高于类型1.张大莉等[9]报道了大口黑鲈北方亚种COI 区段的Hd、Pi和k 均为 0,佛罗里达亚种的 Hd、Pi、k 分别为 0.784、0.0034 2 和4.573.沙航等[10]报道了长江中上游6 个群体鲢 COI 的 Hd为 0.047 6~0.094 5、Pi为 0.001 96~0.009 82.本研究中2 种类型拉氏鱥的Hd均高于大口黑鲈的2个亚种,Pi和k 指数高于北方亚种, 低于佛罗里达亚种; 拉氏鱥的Hd远高于6 个群体鲢,Pi指数则低于6个鲢群体.
