不同野生群体与家系养殖翘嘴鳜遗传多样性分析
2020-04-23成为为夏儒龙王青云曾可为危起伟邓国乔程颖红
成为为,夏儒龙,王青云,曾可为,宋 文,危起伟,邓国乔,程颖红
(1.农业部淡水生物多样性保护重点实验室,中国水产科学研究院长江水产研究所,武汉 430223;2.武汉市农业科学院,武汉 430207)
翘嘴鳜(Sinipercachuatsi)隶属鲈形目鳜亚科鳜属,是我国鳜属的7个种中个体最大、生长速度最快的种类,其肉质细嫩,无肌间剌,且富含大量的蛋白质和不饱和脂肪酸,具有很高的营养价值,素有“淡水石斑鱼”的美称,是我国特有的名优淡水经济鱼类,为我国重要的名优鱼类养殖对象。历史上长江中下游浅水湖泊均与干流相连通,近年来却因大量的水电建筑(堤、坝和闸等)的兴建而相继阻隔,导致生境异质性下降[1]。近年来由于过度捕捞,河湖面积大减及工业毒污水的严重污染,鳜类资源受到了严重威胁,翘嘴鳜野生种质资源锐减、种质退化、生长和抗逆性能下降等问题变得越来越严重[2-5],同时翘嘴鳜人工养殖规模不断扩大,养殖翘嘴鳜的种质退化明显,抗病力下降,严重制约了翘嘴鳜养殖业的健康可持续发展。
赵金良等[6]对长江水系洞庭湖、鄱阳湖、秋浦河和太湖4个鳜群体共42尾的mtDNA控制区核苷酸序列进行了测定,获得了长度785 bp的同源序列,利用控制区核苷酸序列构建的NJ分子树中,各群体内的个体均未单独成群,而是互有交叉。由于遗传分化低,初步认为洞庭湖、鄱阳湖、秋浦河和太湖鳜群体可能同属一个种群-长江种群。吴旭等[7]2010年对长江中下游不同地理种群鳜遗传结构进行了研究,以长江、通江湖泊(洞庭湖、鄱阳湖)、陆封型湖泊(牛山湖、涨度湖、汤逊湖、肖四海湖)不同水体鳜为研究材料,利用微卫星遗传标记对其种群遗传结构进行分析,结果表明:由期望杂合度(He)和多态信息含量指数(PIC)检测的遗传多样性由大到小的顺序为:长江、通江湖泊群体>无放流陆封型湖泊群体>放流的陆封型湖泊群体,并且发现一些稀有等位基因位点在陆封型湖泊鳜群体中消失;根据遗传距离构建的UPGMA聚类图,鳜群体被分成三支,长江、通江湖泊聚类为一支,无放流陆封型湖泊与放流陆封型湖泊亦分为两支,研究结果表明,江湖阻隔是造成定居性鱼类鳜种群间遗传分化的重要原因之一。吴旭等[8]对肖四海湖野生和人工放流鳜群体遗传结构分析,结果表明野生群体遗传多样性明显高于放流增殖鳜群体,野生鳜群体表现出更丰富的遗传多样性;遗传变异主要存在于群体间,而不是群体内部,这充分反映近交及瓶颈效应会引起养殖群体遗传结构的改变,从而导致群体间的遗传分化。以上研究结果均表明鳜长江野生群体对比通江湖泊和养殖群体均存在较高的遗传多样性,江湖阻隔导致的基因交流的减少是造成鳜种群间遗传分化的重要原因之一。
随着建坝与调水等水利工程的建设以及围垦与人工引种等人们生活活动等,长江流域天然渔业资源量连续呈现出衰退的特征,濒危和灭绝的种类也逐渐增加[9]。另外,人为的江湖阻断也给长江流域淡水鱼类的繁衍产生了极大的影响。这些都为长江流域淡水鱼类生物多样性的维持与保护提出了极大的挑战。长江水系和支流湖泊以及家系养殖翘嘴鳜是否进一步出现了明显的遗传分化和某些稀有等位基因的缺失?基于以上问题,本项目以分子生物学结合生态学的方式,利用分子标记(SSR)研究我国长江水系、通江湖泊、人工养殖的翘嘴鳜群体遗传结构,系统分析群体遗传多样性现状,在此基础上划分管理单元,为鳜类鱼类资源开发与管理提供指导性建议,对合理开发和有效的物种保护策略的制定具有重要实际意义。
1 材料与方法
1.1 样本采集
采集长江石首江段野生翘嘴鳜(YZ)63尾,梁子湖岛野生(LZH)翘嘴鳜63尾,洞庭湖沅江江段(DTH)野生翘嘴鳜10尾,无锡董氏水产有限公司养殖场翘嘴鳜(WX)12尾,武汉鳜鱼健康养殖小区武汉市佳恒水产有限公司(WH)200尾,共计348尾鳍条样本,无水乙醇保存备用。
1.2 PCR扩增及毛细管电泳检测
DNA提取采用磁珠法提取基因组DNA(试剂盒天根DP341),经1%琼脂糖凝胶电泳检测后稀释到约100 ng/μL用于PCR扩增。测定鳜性腺转录组数据,筛选7对高度多态微卫星标记(未发表数据)合成荧光引物(FAM和HEX),在各自特定的退火温度下进行PCR扩增,PCR扩增体系和反应程序如下,PCR反应体系为10 μL,2×Taq PCR Master Mix(含染料)5 μL(包含Taq DNA Polymerase,Mg2+,dNTPS,指示染料和反应Buffer),上下游引物各0.5 μL(10 μmol/L),DNA 100 ng,ddH2O 3 μL。扩增程序为:95 ℃预变性5 min;95 ℃变性30 s,62 ℃→52 ℃退火30 s,72 ℃延伸30 s,10个循环;95 ℃变性30 s,52 ℃退火30 s,72 ℃延伸30 s,25个循环;最后72 ℃末端延伸20 min,4 ℃保存。PCR产物经自动测序仪ABI 3730 xl(Rox-500 standard)微卫星分型并用软件GENEMAPPER V.4.0[10]读取等位基因大小。
1.3 分析方法
采用PopGene32[11]进行数据统计分析,计算群体的等位基因数(Na)、有效等位基因数(Ne)、观测杂合度(Ho)、期望杂合度(He)、遗传相似系数(I)、群体间遗传距离(Ds)。用Cervus[12]软件计算多态信息含量(PIC)。MEGA7.0软件被用来依据遗传距离进行群体UPGMA聚类分析。采用Arlequin V3.5.1.3[13]软件中的AMOVA方法分析群体的遗传变异,计算遗传分化系数Fst来评价群体间的遗传差异,通过1000次重复抽样来检验群体间FST显著性,使用Nm=(1-Fst)/4Fst计算群体间的基因流。用Structure 2.3.4软件[14-15]进行群体遗传结构分析,参数“Length of Burnin Period”设置为200 000,“Number of MCMC Repsafter Burnin”设置为1 200 000,k为1至8,每个k值运行10次。经网址http://taylor0.biology.ucla.edu/struct_harvest在线计算获得最佳k值,并利用CLUMPP_Windows.1.1.2[16]和distruct1.1[17]作遗传结构图。
2 结果与分析
2.1 遗传多样性
7个微卫星位点在5个群体348尾个体检测中均表现为多态性(表1)。7个多态性微卫星位点的等位基因数范围为18~36,平均值为29.14;有效等位基因数范围为6.660~10.629,平均值为8.909;观测杂合度范围为0.784~0.928,平均值为0.853;期望杂合度范围为0.851~0.907,平均值为0.886;平均多态信息量范围为0.841~0.899,平均值为0.875;Fst范围为0.028~0.084,平均值为0.051;基因流范围为2.731~8.788,平均值为5.421(位点特征值详见表1)。
7对微卫星引物在5个群体中共检测到526个等位基因,其中平均等位基因数:长江野生群体>梁子湖群体>武汉养殖群体>洞庭湖群体>无锡养殖群体;平均有效等位基因数:长江野生群体>梁子湖群体>洞庭湖群体>武汉养殖群体>无锡养殖群体;平均观测杂合度:梁子湖群体>无锡养殖群体>长江野生群体>洞庭湖群体>武汉养殖群体;平均期望杂合度:长江野生群体>梁子湖群体>洞庭湖群体>无锡养殖群体>武汉养殖群体;平均多态信息量:长江野生群体>梁子湖群体>洞庭湖群体>武汉养殖群体>无锡养殖群体(详细值见表2)。

表1 7个多态微卫星特征值Tab.1 Characteristic values of 7 polymorphic microsatellites

表2 5个群体在微卫星位点的遗传信息Tab.2 Genetic diversity of microsatellite loci in five populations
2.2 遗传分化
通过PopGene 32得到5个群体间的Nei氏遗传距离(Ds)和遗传相似系数I(表3)。5个群体的遗传距离为0.277~0.813,遗传相似系数为 0.444~0.758;长江野生群体和无锡养殖群体间的遗传距离最大(0.813),遗传相似系数最小(0.444);无锡养殖群体和武汉养殖群体间的遗传距离最小(0.277),遗传相似系数最大(0.758)。

表3 5个群体的Nei氏遗传相似系数(上三角)和遗传距离(下三角)Tab.3 Nei′s genetic similarity coefficient(upper triangle) and genetic distance(lower triangle)in 5 populations
根据Nei氏遗传距离采用MEGA7.0软件进行群体UPGMA聚类分析,结果显示梁子湖群体和洞庭湖群体聚为一支,无锡养殖群体和武汉养殖群体聚为一支,继而这四个群体共聚为一支,最后和长江野生群体聚为一大支(详见图1)。
利用Arlequin软件计算每两个群体之间的遗传分化值,结果显示:梁子湖群体和洞庭湖群体之间的遗传分化系数最小,为0.013(P=0.865>0.05),长江野生群体和武汉养殖群体之间的遗传分化系数最大,为0.077(P=0.000<0.05)(表4)。
AMOVA分析表明:在总遗传变异中,群体间的遗传变异占5.95%,群体内遗传变异占 94.05%(表5)。说明5个群体中大部分的遗传变异来自于群体内。

图1 基于Nei氏遗传距离构建5个地理群体的UPGMA聚类图Fig.1 UPGMA clustering map of 5 populations based on Nei′s genetic distance
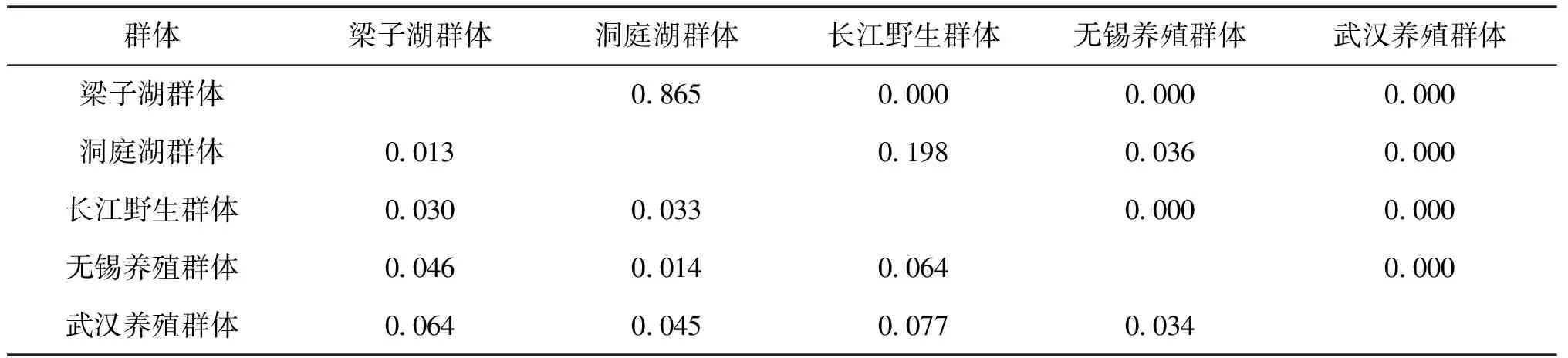
表4 5个群体的遗传分化指数(对角线下)和P值(对角线上)Tab.4 Genetic differentiation index(below diagonal)and P value(above diagonal)in 5 populations

表5 5个群体的 AMOVA分析Tab.5 AMOVA analysis of 5 populations
2.3 种群结构
运用structure2.3.4软件对5个群体进行遗传结构分析。在Deltak值与k值的关系变动图中结果显示在k=2时,Δk值最大(图2),说明这5个群体可分为2个亚群,分别为为亚群I和亚群II。其中亚群I大部分分布于梁子湖群体、洞庭湖群体和长江野生群体,亚群II大部分布于剩余的武汉养殖群体和无锡养殖群体中。

图2 5个群体在k=2的遗传结构图Fig.2 Genetic structure of 5 populations in k=2
3 讨论
3.1 种群遗传多样性
多态性信息含量(PIC)是衡量基因多态性的指标[18]。当PIC>0.5时,该位点为高度多态位点;0.25 基因流对于遗传结构的影响是,基因流越顺畅,遗传分化的程度将越低[19-20]。遗传分化指数Fst若在0~0.05间为无分化,0.05~0.15间为中度分化,0.15~0.25间为高度分化[21]。本研究所显示这几大群体呈现一个中度分化的趋势,并不是一个无分化的大种群,梁子湖群体和洞庭湖群体聚为一支,无锡养殖群体和武汉养殖群体聚为一支,继而这四个群体共聚为一支,最后和长江野生群体聚为一大支,5个群体可分为2个亚群,其中亚群I包括梁子湖群体、洞庭湖群体和长江野生群体,亚群II包括武汉养殖群体和无锡养殖群体。这与吴旭等[7,8](2010)所研究的结果基本一致,本文研究结果显示翘嘴鳜不同种群间的基因交流受到了一定的地理阻碍,特别是养殖群体与长江野生群体表现出较高的遗传分化,一方面可能是内陆湖泊和通江湖泊及长江之间,由于长期的江湖阻隔以及生境的不同,导致不同地理群体间逐渐产生形态或者分子的变异导致遗传分化[22];另一方面鳜鱼野生资源逐渐减少,所产生的瓶颈效应导致后代容易受到偶然因素的影响,加快群体间的分化速度。 综上所述,基因交流的减少是造成鳜种群间遗传分化的重要原因之一。建议加强鳜遗传多样性的监测和评估,降低鳜野生遗传资源的稀释和衰退速度。以鳜遗传背景为基础,根据遗传结构制定合理的策略和科学的保护措施。从分子水平上分析种群系统地理格局的形成机制,追溯和揭示群体水平的进化历史,防止人工繁殖的杂交子代对原始种的混杂与污染,在此基础上制定系统的选育、保种方法,建立种资资源库,使鳜类优良的种质资源得到合理的保护、开发及可持续利用。3.2 群体遗传分化
