基于线粒体D-loop的西江流域粗唇鮠群体遗传多样性分析
2020-04-23韩耀全吴伟军李育森王大鹏雷建军何安尤
彭 敏,韩耀全,吴伟军,李育森,施 军,王大鹏,雷建军,何安尤
(广西水产科学研究院,广西壮族自治区遗传育种及健康养殖重点实验室,南宁 530021)
粗唇鮠(Leiocassiscrassilabris)隶属鲇形目(Siluriformes)鲿科(Bagridae)鮠属(Leiocassis),常栖息于江河、湖泊的底层草丛和岩洞内,为底栖肉食性小型经济鱼类,白天极少活动,夜间外出觅食,以底栖生物,小鱼和小虾为食。在中国广泛分布于长江、珠江、闽江等水系。因其肉质细嫩,味道鲜美,无肌间刺,食用价值较高而深受消费者喜爱。近年调查发现西江主要干支流,粗唇鮠的捕获量有明显下降趋势。粗唇鮠资源量的降低,预示着有效繁殖群体变小,这有可能造成性别失衡、遗传漂变、遗传多样性丢失等现象。陈锋等[1]在2013年对珠江龙滩库尾至长洲坝下江段干支流鱼类调查中发现,由于生境的改变,河口型鱼类分布范围变窄、珍稀濒危特有鱼类种群规模变小、流水性鱼类资源量显著下降;徐田振等[2]在2012-2013年在郁江金陵江段开展了漂流性鱼卵的调查监测发现,与历史资料相比,郁江鱼类产卵场功能发生较大变化,部分鱼类产卵场消失,部分鱼类虽然还能自然补充,但资源量较小。表明河流生境的变化及现行的过度捕捞等因素对鱼类群落结构及早期资源的补充会造成很大的影响。
线粒体DNA因具有母系遗传、进化速度快等特点,常被用于研究群体亲缘关系。粗唇鮠线粒体全基因组包括13个蛋白质编码基因、22个tRNA基因、2个rRNA基因和1个非编码区(D-loop)[3],其中D-loop更是鱼类群体遗传学研究的常用材料。如李珊[4]对清水江的斑鳜(Sinipercascherzeri)种群遗传分析发现其遗传变异程度不高,遗传多样性丰富,种群结构稳定;侯新远等[5]对我国5种虾虎鱼类进行系统进化关系分析,描绘其亲源关系的远近;董微微等[6]对长江流域异鳔鳅鮀(Xenophysogobioboulengeri)的遗传分析,推测其种群扩张时间大概为0.017 3 Ma,均采用线粒体DNA的D-loop序列作为研究对象。刘伟等[7]亦曾使用D-loop序列对贵州都柳江段的粗唇鮠群体种群遗传多样性进行了相关研究,然而广度较小。粗唇鮠作为西江流域广西境内江段的重要经济鱼类之一,在广西各江段群体中遗传多样性如何,是否发生遗传漂变迄今未见报道,这对广西境内粗唇鮠的种质资源监测及保护理论的构建是有所欠缺的。本研究通过对西江流域广西境内7条江段的粗唇鮠群体分别进行mtDNA D-loop 序列测定,旨在了解其遗传结构,分析其不同江段群体的遗传分化程度和基因交流情况,为西江水系粗唇鮠自然种群的合理开发利用提供参考,为粗唇鮠种质资源恢复与可持续利用提供科学依据。
1 材料和方法
1.1 实验材料
实验用粗唇鮠样本共227尾,于2014年6月至2016年12月,分别采自西江流域7段支流或干流,其中都柳江(DLJ)广西段(S1)31尾,红水河(HSH)江段(S2)30尾,柳江(LIUJ)段(S3)50尾,西江(XIJ)段(S4)29尾,右江(YOUJ)段(S5)30尾,郁江(YUJ)段(S6)27尾,左江(ZJ)段(S7)30尾(图1)。材料取自新鲜个体背部肌肉,每尾5 g,分别用95%酒精保存备用,前两天每天更换酒精一次。

图1 粗唇鮠采样点Fig.1 Sampling site of L.crassilabris
1.2 DNA提取、PCR扩增与测序
粗唇鮠的总DNA采用醋酸铵法提取[8],采用微量核酸蛋白分析仪测量DNA浓度和纯度,用1%的琼脂糖凝胶电泳检测DNA质量,经检测合格的DNA模板保存于-20 ℃冰箱备用。
粗唇鮠的D-loop序列扩增采用上海生工生物工程技术服务有限公司合成的D-loop通用引物,序列为D-loop-F(5′-ACC CCT GGC TCC CAA AGC-3′)和D-loop-R(5′-ATC TTA GCA TCT TCA GTG-3′)[9]。
PCR反应体系50.0 μL,包括2×Ex Taq Bufffer 25 μL,10 μmol/L的上、下游引物各2 μL,100 ng/μL DNA模板2.0 μL,用ddH2O补至50.0 μL。退火温度为54.0 ℃。经1.5%琼脂糖凝胶电泳检测合格的PCR扩增产物由深圳华大基因公司正向测序。
1.3 数据处理
测序所得序列采用clustalw软件并辅以人工校对调整,碱基含量及变异位点采用MEGA 4.1软件分析,计算Kimura 2-parameter遗传距离,并以长吻鮠(GenBank登录号:GU596454.1)为外类群构建单倍型的NJ系统树。利用DnsSP 5.10软件统计单倍型及计算遗传多样性参数。采用Arlequin软件进行分子方差分析,评估种群的遗传分化程度,采用Tajima’s D和Fu′s Fs中性检验估测有效种群大小的历史。
2 结果与分析
2.1 mtDNA D-loop碱基组成
获得的粗唇鮠D-loop序列长度均为666 bp,碱基T、C、A、G的平均含量为32.9%、23.3%、29.4%、14.4%,其中A+T(62.3%)明显高于C+G(37.7%),表现出较强的碱基偏倚性,与一般脊椎动物线粒体DNA序列特征相一致。
2.2 遗传多样性分析
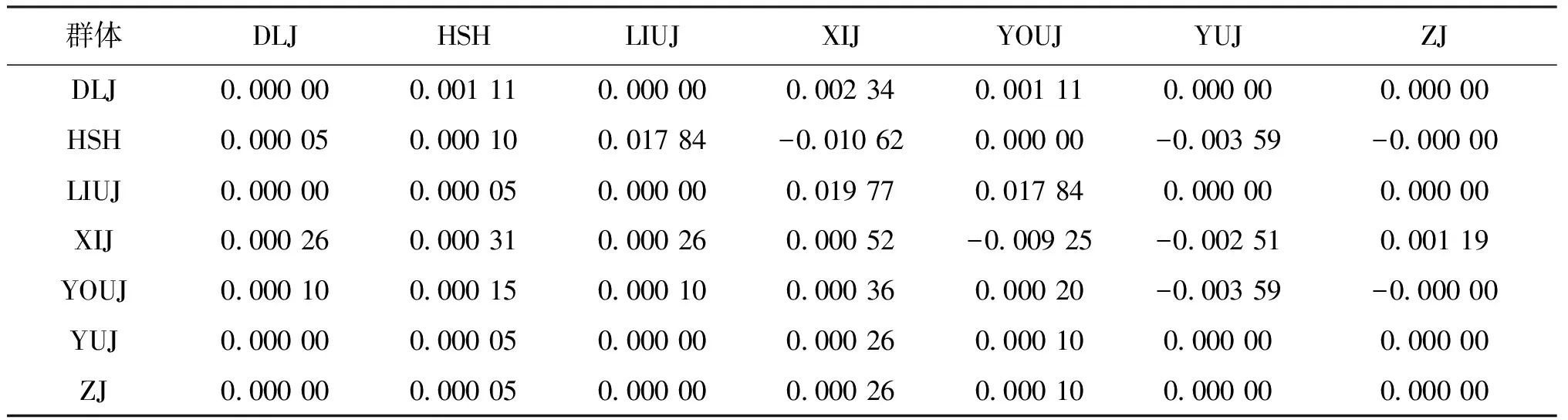
粗唇鮠D-loop序列均未检测到插入或缺失位点,检测到变异位点6个,占总位点数的0.90%,其中简约信息位点2个,单一变异位点4个;DLJ、LIUJ、YUJ、ZJ4个群体未发现变异位点,HSH江段1个,YOUJ段2个,XIJ段5个。转颠换比值R趋于无穷大,说明此基因序列突变未达到饱和状态,受进化噪音的影响可能性较小(表1)。在DAMBE进行DNA序列替换饱和性检验中,Iss=0.004 2,Iss.c=0.774 8,Iss 7个江段的粗唇鮠无论是单倍型多样性指数和核苷酸多样性指数均表现出较低的整体特征。整体单倍型多样性指数为0.052 24,核苷酸多样性指数为0.000 11。其中,单倍型多样性指数与核苷酸多样性指数均为XIJ群体最高,分别为0.199 51、0.000 52;DLJ、LIUJ、YUJ和ZJ群体最低,均为0.000 00。平均核苷酸差异最大为XIJ,为0.344 83,其次为YOUJ,为0.133 33(表1)。 227尾个体共检测出6种单倍型,即hap1~hap6,其中hap1为优势单倍型,在所有群体均有出现,占总个体数的97.36%;hap3仅存在于XIJ与YOUJ群体;有4种单倍型为不同江段群体独享,其中hap2为HSH独享,hap4、hap5为XIJ独享,hap6为YOUJ独享(图2)。 表1 粗唇鮠7个群体遗传多样性指数Tab.1 Genetic diversity index among seven populations of L.crassilabris 图2 粗唇鮠NJ单倍型系统树Fig.2 NJ phylogenetic tree of L.crassilabris 2.3.1 种群遗传距离 MEGA4.1软件分析测序结果表明,粗唇鮠群体内的遗传距离在0.000 00~0.000 52之间。其中XIJ群体内的遗传距离最远为0.000 52;其次为YOUJ,为0.000 20;HSH为0.000 10;而其余4个江段群体内的遗传距离均为0.000 00。各群体间的遗传距离在0.000 00~0.000 36之间,XIJ与其他几个江段群体间的距离均较远,与YOUJ最远为0.000 36,其次与HSH群体,为0.000 31,与YUJ、ZJ、LIUJ、都LIUJ群体间的遗传距离均为0.000 26(表2)。 2.3.2 种群遗传分化系数 7个江段粗唇鮠群体之间的遗传分化系数在-0.010 62~0.019 77之间,XIJ与LIUJ群体的遗传分化系数最大,为0.01977;其次为LIUJ与HSH/YOUJ群体的遗传分化系数均为0.017 84;而HSH与XIJ/YUJ群体之间,XIJ与YOUJ/YUJ群体之间,YOUJ与YUJ群体之间的遗传分化程度小且为负值。 群体间和群体内的分子变异分析结果显示,群体间的遗传分化系数Fst=0.001 30;而且变异几乎均来自群体内部,群体间的变异占0.13%,群体内占99.87% ,整个调查群体基因流Nm=384.1154(表2、表3)。 表2 粗唇鮠群体遗传距离与FstTab.2 Genetic distance and genetic differentiation coefficient Fst of L.crassilabris 注:对角线为群体内遗传距离,对角线上方为遗传分化系数Fst,对角线下方为群体间遗传距离。 表3 粗唇鮠群体间和群体内的分子变异分析(AMOVA)Tab.3 Analysis of interspecific and intraspecific molecular variation of L.crassilabris(AMOVA) 无限突变位点模型的中性检验值Fu’s Fs 检验结果表明,Tajima’s D(-1.857 04,0.000 00,P<0.01)、Fu′s Fs(-9.875 96,0.000 00,P<0.01)均为显著负值,同时核苷酸错配分布结果显示,粗唇鮠种群单倍型和碱基差异呈单峰分布,表明粗唇鮠种群近期经历过种群扩张事件(图3)。 根据公式τ=2ut计算XIJ水系粗唇鮠种群的大约扩张时间,参照鲿科鱼类的线粒体DNA进化速率约为0.18%~0.30%/Ma[10],而粗唇鮠性成熟最早为2龄,通过AMOVA分析表明τ=3.000,因此推测本次调查的粗唇鮠群体基于D-loop基因序列的扩张时间可能是在距今0.75~1.25 Ma前。 图3 粗唇鮠核苷酸错配分布图Fig.3 Mismatch distribution of L.crassilabris 3.1.1 D-loop结构与变异性 7个江段的粗唇鮠D-loop序列长度均为666 bp,表现出较强的碱基偏倚性,与一般脊椎动物线粒体DNA序列特征相一致。碱基转颠换比值R趋于无穷大,说明此基因序列突变未达到饱和状态,受进化噪音的影响可能性较小,Wolstenhome等[11]认为,转颠换的偏倚性可能与序列间的差异存在着一定的相关性,即转颠换的比率与物种从共同祖先进化的时间成反比,由此可以推断XIJ流域的粗唇鮠有效种群是在近期由单一、少数的种群建立的。 3.1.2 遗传多样性指数分析 单倍型多样性指数Hd和核苷酸多样性指数π是评估遗传多样性的两个重要参数[12]。本研究中7个江段群体中,XIJ群体单倍型多样性指数与核苷酸多样性指数均为最高,平均核苷酸差异最大,LIUJ、LIUJ、YUJ和ZJ群体各多样性指数最低,核苷酸差异最小,整体呈现下游高于上游,南方高于北方的现象。这与白晓慧等[13]对天津5个大鳞副泥鳅(Paramisgurnusdabryanus)自然群体的研究结果较为一致。粗唇鮠整体单倍型多样性指数为0.052 24,核苷酸多样性指数为0.000 11,远低于珠江水系的其他鱼类,如底栖肉食性鱼类黄颡鱼(Pelteobagrusfulvidrac)群体(Hd=0.721~0.911,π=0.002 2~0.046 89)[14]、中上层杂食性鱼类赤眼鳟(SpualiobarbusCurriculus)群体(Hd=0.700 0~1.000 0,π=0.011 3~0.013 7)[15]、中下层杂食性鱼类鲮(Cirrhinamolitorella)群体(Hd=0.826,π=0.003 32)[16]和中下层肉食性鱼类鳜鱼(Hd=0.773,π=0.001 95)[17]。程丹汝等[18]在研究铜陵顺安河中不同种类鱼体内磺胺类抗生素累积中发现,在7种不同的鱼类中,7种磺胺类抗生素的浓度水平在粗唇鮠体内含量最高;Li等[19]亦曾报道肉食性、底栖性鱼类在大坝蓄水后汞的生物累积高于其他鱼类。这在一定程度上可推测底栖肉食性鱼类的粗唇鮠相比于其他鱼类可能更容易受环境污染的影响,因此推断环境污染可能是粗唇鮠资源量下降,遗传多样性丢失的其中一个原因。一个物种遗传多样性的高低与其适应能力、生存能力和进化潜力密切相关。遗传多样性为物种的适应能力、生存能力和进化能力提供了潜在的遗传基础和储备。物种遗传多样性越丰富,其适应能力、生存能力和进化潜力越大;遗传多样性的降低可导致其适应能力和生存能力降低、物种退化甚至威胁物种生存[20]。本次调查的粗唇鮠群体单倍型多样性指数与核苷酸多样性指数远低于DLJ易危物种小口白甲鱼(Hd=0.260,π=0.005 75)[21],说明粗唇鮠种群的保护与恢复应受到重视。 单倍型之间的遗传距离是衡量一个物种或群体的mtDNA变异程度的重要指标,遗传距离越大表明群体间亲缘关系越远[22]。本次调查粗唇鮠群体内的遗传距离在0.000 00~0.000 52之间,远远未达到种群划分的界限,表明调查的7个江段粗唇鮠群体具有较近的亲缘关系,无明显的种群分化,可能是处于同一水系的粗唇鮠群体有迁移习性形成基因交流所致。与熊美华等[23]对长江向家坝上、下圆口铜鱼(Coreiusguichenoti)群体的遗传分化调查结果相似,表明大坝并不能阻碍所有鱼类的基因交流。 Wright按照遗传分化指数的大小提出了遗传分化标准(Fst<0.05,无遗传分化;0.05< Fst <0.15,较小遗传分化;0.15 NJ邻接树和单倍型网络图显示,粗唇鮠群体间不存在明显的地理结构和谱系结构,另外,通过AMOVA分析发现7个江段群体的遗传变异系数Fst=0.001 30,群体间变异占0.13%,群体内变异占99.87%,变异几乎全部来自种群内部。可能是XIJ流域粗唇鮠种群最近发生过种群的瓶颈效应或由单一、少数种群所产生的奠基者效应。 Tajima’s D与Fu’s Fs值中性检验常用于推测种群历史发生,如果Tajima’s D与Fu’s Fs值呈负值,且在统计学上达到显著标准,则说明序列中含有比中性进化模型更多的核苷酸位点变化,可能预示着被研究种群曾经经历过一个扩张的历史[26]。中性检测结果表明,Tajima’s D、Fu’s Fs均为显著负值,同时核苷酸错配分布结果呈单峰,故认为粗唇鮠种群最近经历过种群扩张事件,扩张时间推测在距今0.75~1.25 Ma,与采用相同进化速率推算出的鲇形目鲿科的黄颡鱼(0.41~0.69 Ma年前)、光泽黄颡鱼[27](0.51~0.86 Ma年前)扩张时间相近。 研究结果表明,本次调查江段的粗唇鮠群体遗传多样性水平较低,预示着其群体对复杂环境具有更差的适应能力。因此,应通过合理限制捕捞规格,加强禁渔期监管,维持其野生资源的可持续性开发与利用;同时还可以考虑收集XIJ流域上游,如贵州、云南江段的亲鱼进行人工繁育,并合理的增殖放流于广XIJ段以增加其种群多样性,若有必要还需实行捕捞限额制度或减少钓捕、并放生笼捕所获得的小规格鱼种,以促进粗唇鮠种群的恢复与健康发展。

2.3 粗唇鮠群体遗传分化

2.4 群体动态分析

3 讨论
3.1 粗唇鮠的遗传多样性
3.2 粗唇鮠的遗传变异分析
3.3 粗唇鮠的种群动态
3.4 保护建议
