转录组分析鉴定肺腺癌潜在的生物标志物
2020-03-19张伟然林雪峰韩兴鹏孙大强
张伟然 ,林雪峰,李 鑫,张 浩,王 猛,孙 伟,韩兴鹏,孙大强
1. 天津医科大学研究生院,天津 300070;2. 天津市胸科医院胸外科,天津 300222;3. 天津医学高等专科学校护理系,天津300222;4. 天津市南开医院胸外科,天津 300100
肺癌是世界上发病率和死亡率均排第一位的恶性肿瘤。据最新统计数据,2020 年全世界肺癌新发患者228 820 例,死亡病例135 720 例[1]。非小细胞肺癌(nonsmall-cell lung cancer,NSCLC)约占所有肺癌病例的85%[2]。肺腺癌是一种常见的NSCLC,约占NSCLC 的50%[3]。近年来,肺腺癌机制的研究得到了广泛的开展,研究人员发现遗传因素影响了肺腺癌的发生和发展。其中有研究[4]发现,肺腺癌中最常出现的是ALK、ROS1 和RET 重排,另外还有80 多种基因易位。此外,还有很多常见的基因突变影响肺腺癌,包括TP53(46%)、EGFR(20%)、HER2(2%)、KRAS(1%)、ALK(1%)、BRAF(1%)和PIK3CA(1%)等[5]。这些发现对肺腺癌的靶向治疗具有重要意义。例如,克唑替尼对ALK 或ROS1 融合的肿瘤有效,而吉非替尼、厄洛替尼和阿法替尼可用于EGFR 突变的肿瘤患者[6]。但目前临床应用的药物很少,肺腺癌患者的死亡率很高,患者很难获得长期生存[7]。因此,肺腺癌的分子机制亟待进一步探索。
目前,很多研究通过基因芯片数据分析揭示了一些与肺腺癌相关的基因。Tian 等[8]鉴定出9 个lncRNA 和6 个mRNA,它们可能是诊断或预测人类肺腺癌细胞对紫杉醇耐药性的有效标志物。Liu 等[9]通过分析miRNA 基因芯片表达谱,发现miR-136、miR-376a 和miR-31 在小鼠肺癌中均显著过表达,同时miR-31 作为致癌miRNA 在肺癌中靶向抑制特异性的肿瘤抑制因子。Zhu 等[10]报道了hsa_circ_0013958(一种环状RNA)可以作为一种潜在的非侵入性生物标志物用于肺腺癌的早期检测和筛查。另一项微阵列图谱研究[11]表明,hsa_circ_0007385 在NSCLC 肿瘤的形成中起重要作用,为NSCLC 的治疗提供了一个潜在的靶点。本研究综合分析了肺腺癌组织的基因、miRNA和circRNA 表达谱,以探索肺腺癌的潜在生物标志物,并通过癌症基因图谱(the Cancer Genome Atlas,TCGA)对关键基因进行验证。
1 资料与方法
1.1 基因芯片数据
芯 片 数 据GSE104854[12]、GSE102511、GSE85716、GSE83527 和GSE101586[13]从 基因表达数据库(GEO,http://www.ncbi.nlm.nih.gov/geo/) 中 下 载。GSE104854、GSE102511 和GSE85716 是基因表达谱,分别包括3、16和6 个肺腺癌组织及其癌旁组织,测序平台分别是Ion Torrent Proton(Homo sapiens)、Ion Torrent Proton(Homo sapiens)和Agilent-062918 OE Human lncRNA Microarray V4.0 028004。GSE83527 是一个基于Illumina Hiseq 2000(Homo sapiens)平台的miRNA 表达谱,包含36 个肺腺癌样本及其癌旁样本。circRNA 表达谱芯片编号为GSE101586,有10 例样本,包括5 例正常样本及5 例癌症样本,芯片平台为Agilent-069978 Arraystar Human CircRNA microarray V1。
1.2 差异表达分析
对所有的GEO 数据进行处理后,通过Limma V3.34.8函数包(http://www.bioconductor.org/packages/3.6/bioc/html/limma.html)进行差异表达基因(differentially expressed genes,DEGs)、差异表达miRNAs(differentially expressed miRNAs,DEMs) 和 差 异 表 达circRNAs(differentially expressed circRNAs,DECs)分析。GSE104854、GSE102511和GSE85716 的分析参数分别为|Log_FC|>3、|Log_FC|>2、|Log_FC|>2,均P<0.05。进一步筛选出上述3 组表达谱的重叠DEGs。此外,DEMs 和DECs 的纳入标准均为|Log_FC|>1 和P<0.05。采用circBase(http://www.circbase.org/)进一步对DEGs 与DECs 的关系进行分析。
1.3 功能富集分析
用注释、可视化和综合发现数据库(David,https://david.ncifcrf.gov/summary.jsp,6.7 版)、京 都 基 因 和 基因组百科全书(KEGG PATHWAY,http://www.genome.jp/kegg)和Reactome(http://www.reactome.org)对 重 叠DEGs 进行功能富集分析,得到P<0.05 的基因本体(gene ontology,GO)和重叠DEGs 所涉及的重要生物过程和 通路。
1.4 miRNA 靶向基因的筛选
为了探索肺腺癌样本中DEMs 的调控机制,利用miRWall v3.0 数 据 库(http://129.206.7.150/)获 得DEMs的分子靶标和miRNA-基因关系对,进一步筛选出负调控的miRNA-基因关系对。
1.5 综合调控网络构建
利用String V10.5(https://string-db.org)数据库获得重叠DEGs 的高置信(>400 分)的蛋白-蛋白对,基于蛋白-蛋白对和上述负调控的miRNA-基因关系对建立调控网络,并用CytoCope V3.5.1 软件(http://wwwyCytoCAPE.org/Sudio.php)进行可视化。
1.6 关键基因的验证
从TCGA(https://cancer genome.nih.gov/)中下载15例肺腺癌患者的癌组织样本及其癌旁组织进行DEGs 筛选。TCGA 数据筛选参照中国临床肿瘤学会肿瘤标志物专家委员会和中国肿瘤驱动基因协会的专家共识及相关文献,由2 位病理学教授对每例样本进行病理定量和质量控制分析。DEGs 以|Log_FC|>2、P<0.05 为阈值,记为DEGs-TCGA。将DEGs-TCGA 与上述基因表达谱的3 组DEGs 进行比较。此外,采用聚类算法对癌组织样本和癌旁组织进行分离,比较关键基因表达与患者总生存期和肿瘤分期间的相关性。
收集8 例2018 年1 月—10 月在天津市胸科医院就诊的肺腺癌患者的癌组织和癌旁组织,提取总RNA。使用SYBR®Premix Ex Taq ™ Kit 试剂盒(TaKaRa,日本)进 行SFTPC、ITLN2、SLC6A4、ADRA1A 以 及EGFR 的实时荧光定量PCR(qRT-PCR)检测,hsa-mir-141-5p 和hsa-mir-191-3p 采 用All-in-One ™ miRNA qPCR Kit 试 剂盒(GeneCopoeia Inc.,美国)检测。所有引物均由宝日医生物技术(北京)有限公司设计合成,如表1 所示。β-actin 和U6 为内参。收集患者的外周血样本,采用酶联免疫吸附实验(ELISA)检测血清中肿瘤标志物癌胚抗原(CEA)和神经元特异性烯醇化酶(NSE)的表达水平。

表1 SFTPC、ITLN2、SLC6A4、ADRA1A、hsa-mir-141-5p 和hsa-mir-191-3p 的引物序列Tab 1 Primer sequences of SFTPC, ITLN2, SLC6A4, ADRA1A, hsa-mir-141-5p and hsa-mir-191-3p
1.7 统计学分析
2 结果
2.1 DEGs、DEMs 和DECs 筛选
在GSE104854、GSE102511 和GSE85716 中, 与 癌旁组织相比,肺腺癌组织分别检测到412 个(180 个上调和232 个下调)、201 个(71 个上调和130 个下调)和709 个(166 个上调和543 个下调)DEGs,图1 显示了3组DEGs 的Venn 图。此外,3 组DEG 中包含61 个重叠DEGs(表2),其中P 值较小的重叠DEGs 包括SFTPC、SLC6A4、CA4、TMEM100 和ITLn2。这些重叠DEGs 具有相同的调节趋势,即同时上调或下调。
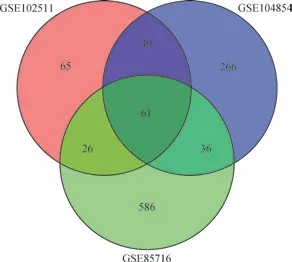
图1 GSE104854、GSE102511 和GSE85716 基 因 表 达 谱 中3 组DEGs 的Venn 图Fig 1 Venn diagram of 3 sets of DEGs in gene expression profiles of GSE104854, GSE102511 and GSE85716
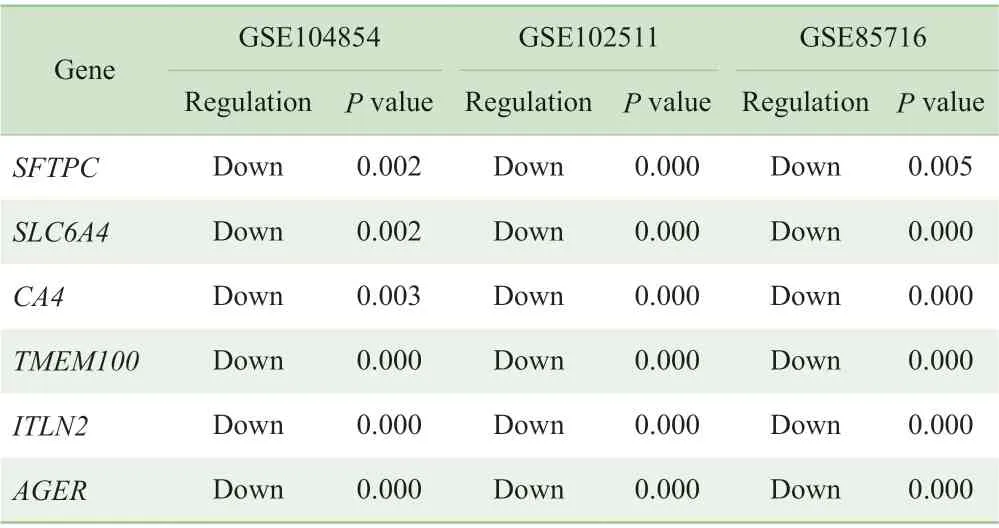
表2 GSE104854、GSE102511 和GSE85716 基因表达谱中的重叠DEGsTab 2 Overlapped DEGs in gene expression profiles of GSE104854, GSE102511 and GSE85716
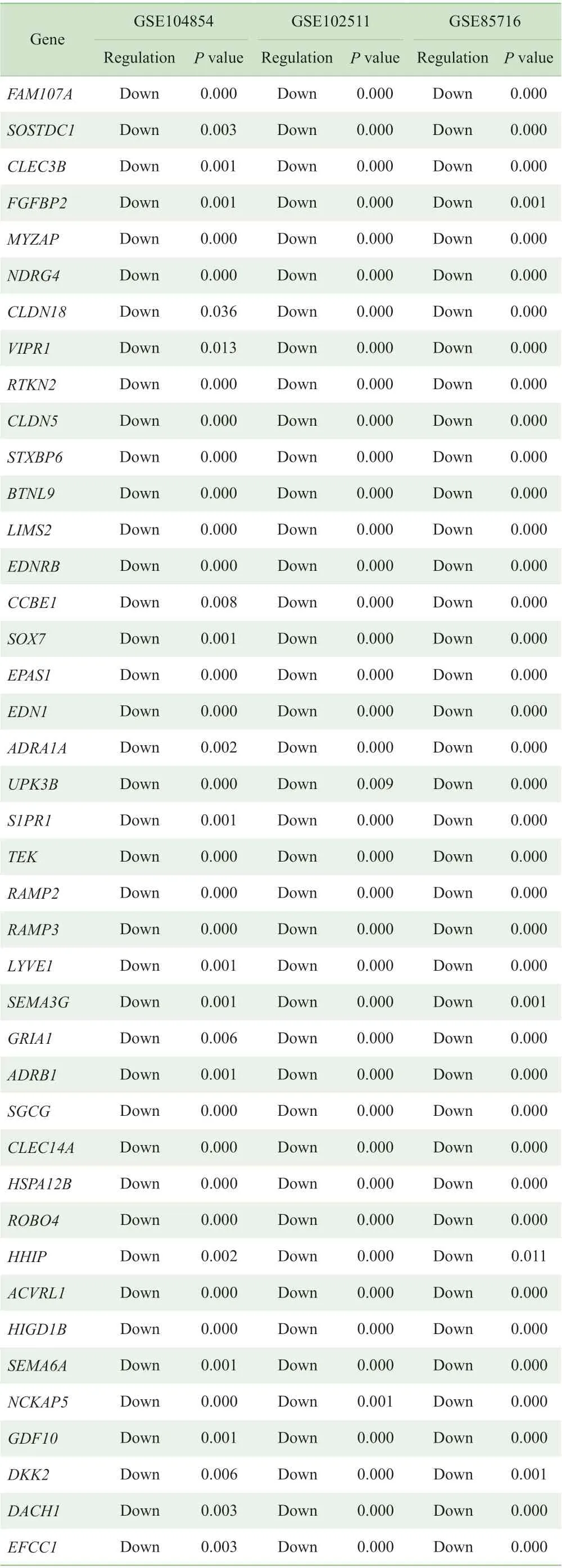
Continued Tab
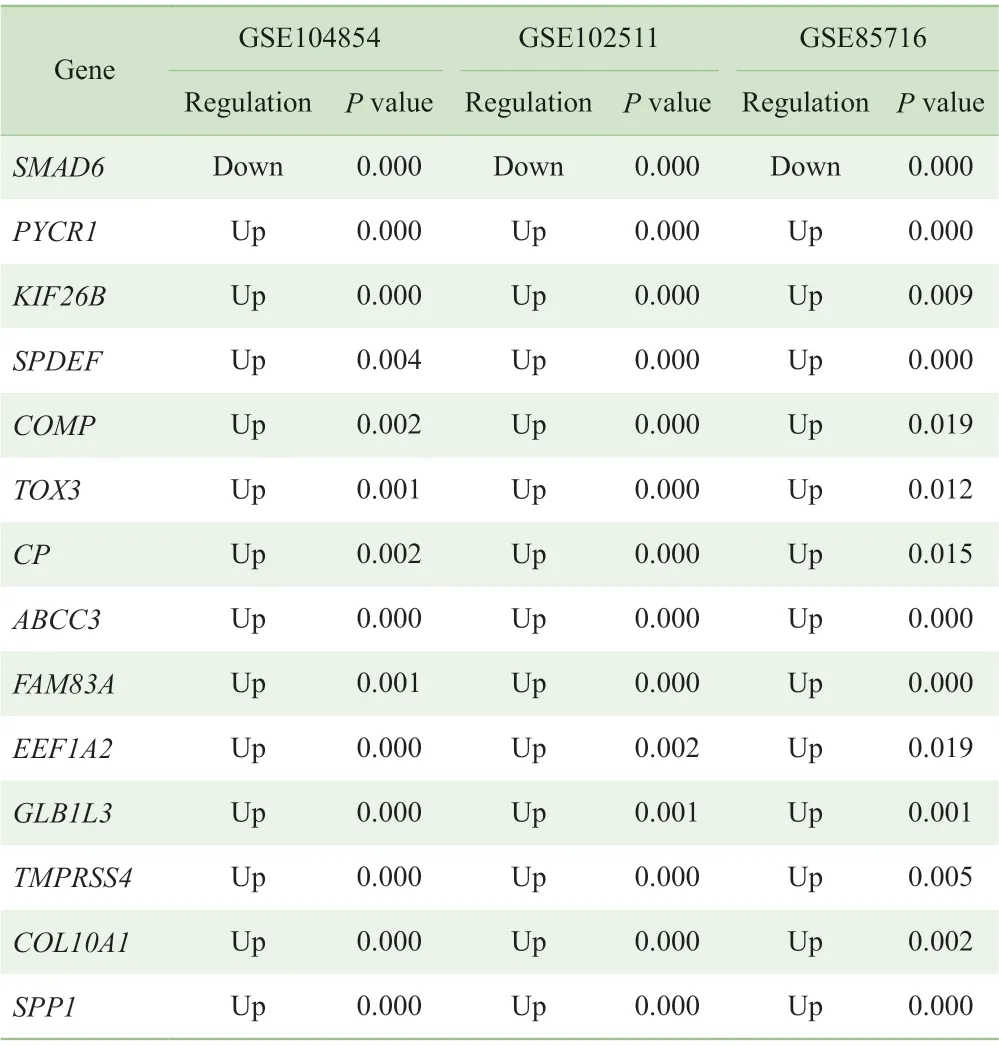
Continued Tab
在GSE83527 中,与癌旁组织相比,肺腺癌组织仅发现24 个DEMs,其中11 个上调,13 个下调(表3)。所有DEMs 在2 组间差异均有统计学意义(P<0.05)。
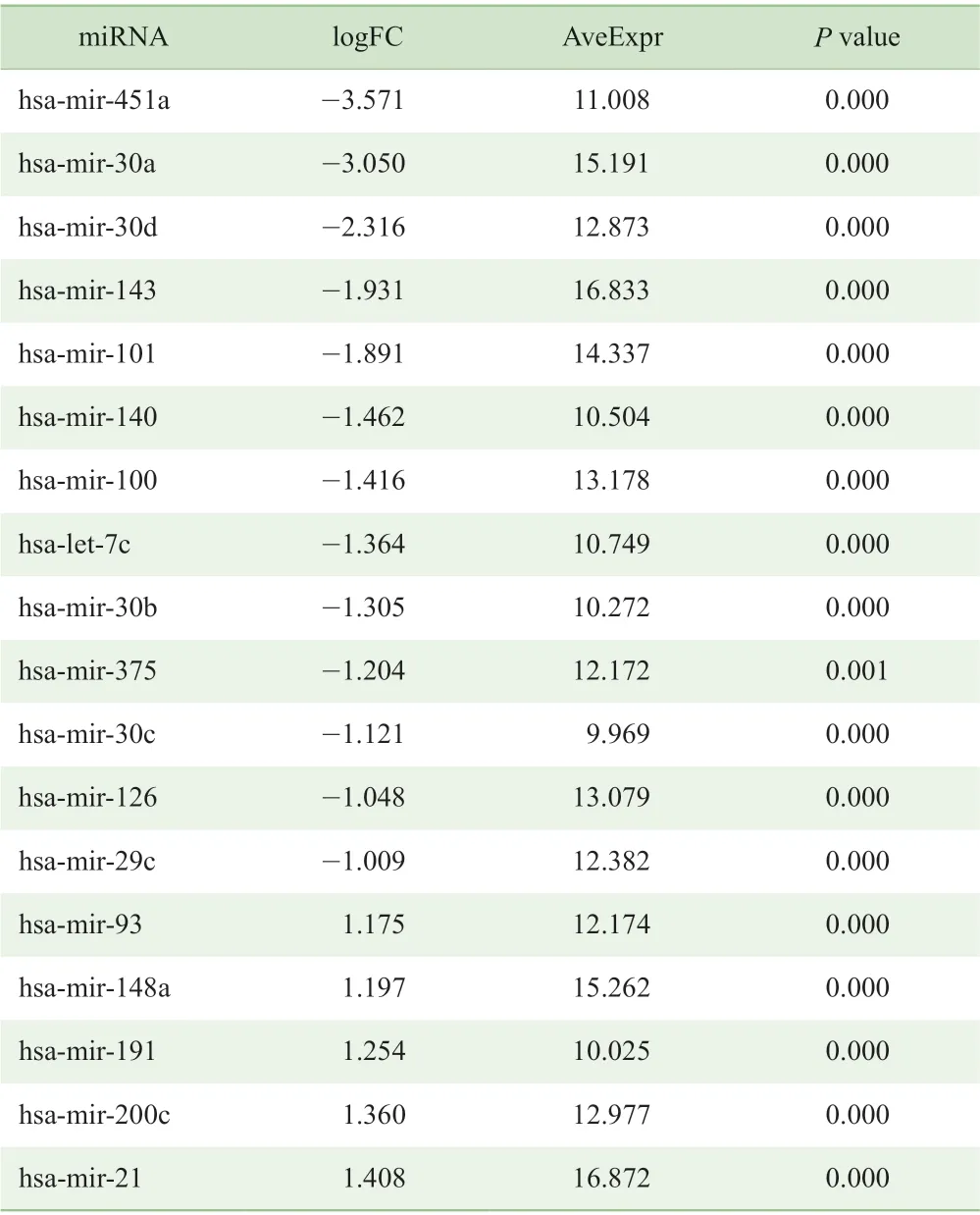
表3 肺腺癌组织相对于癌旁组织的DEMsTab 3 DEMs in lung adenocarcinoma tissues compared with their paracancerous tissues
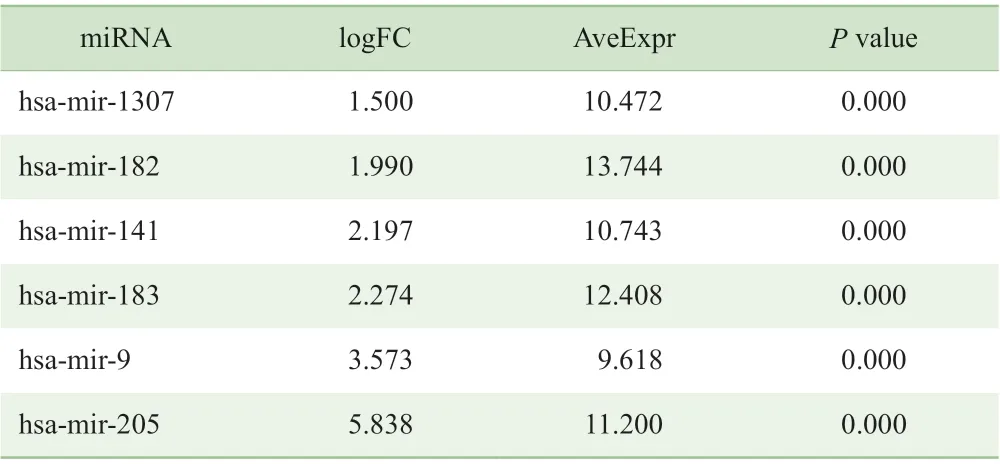
Continued Tab
在GSE101586 中,肺腺癌组织和癌旁组织发现92 个DECs,包括10 个上调DECs 和82 个下调DECs。进一步确定这些DECs 的位置,其中70 个位于编码区的外显子区,18 个位于编码区的内含子区,3 个位于内部转录间隔区,1 个位于非编码区的5'非翻译区(5'UTR)。此外,筛选出与DECs 有关系的基因中,在上述3 组DEGs 中出现过的基因只有LDB2 和PREX1,而重叠DEGs 中没有筛选出与DECs 相关的基因。前30 个差异最显著的DECs 的位置和相应的基因见表4。
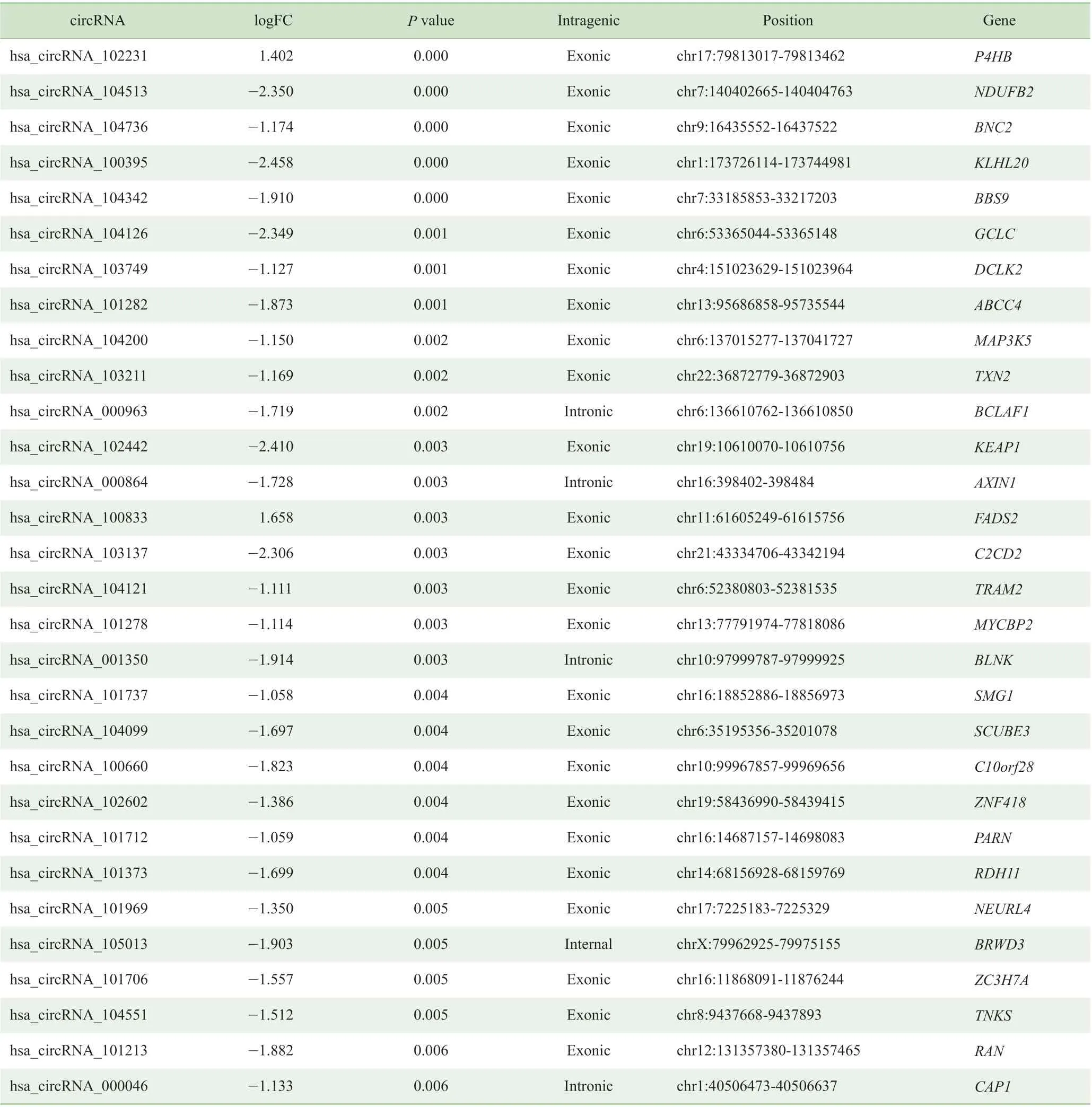
表4 根据P 值排列的前30 个最显著的DECs 及其位置Tab 4 Top 30 most significant DECs according to P value and their position and corresponding genes
2.2 功能通路富集
对DEGs 进行功能和通路富集分析,共得到32 个通路,包括23 个生物过程、6 个细胞组分、1 个分子功能、1 个KEGG 通路和1 个RACTOME 通路(图2)。从结果中可以看出大部分DEGs 显著富集于系统发育、迁移调控、心率调控、血压调控等生物学过程,主要富集于GPCR 及刺激神经组织的受体配体相互作用信号通路。此外,富集到通路中最多的基因分别为S1PR1、EDN1 和ACVRL1,分别富集在24、24 和23 个通路中。
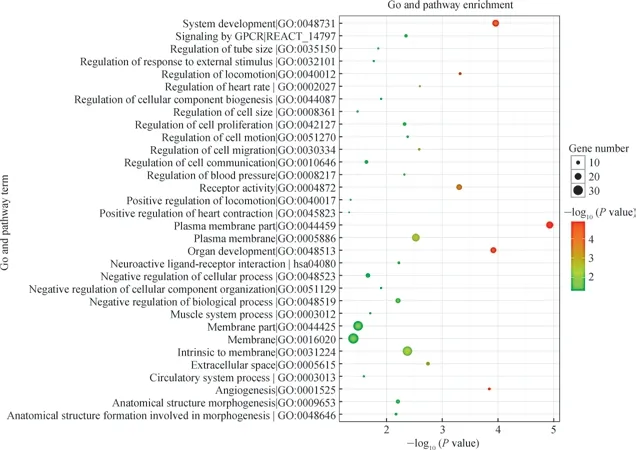
图2 重叠DEGs 富集的GO 和通路Fig 2 Enriched GO and pathway terms of the overlapped DEGs
2.3 DEMs 的靶标预测
根据芯片数据得到了24 个DEMs,并筛选出DEMs的靶基因,仅保留为DEGs 关系对的靶向基因。由此筛选出612 对miRNA-基因关系对,其中303 对为负调控。
2.4 重叠DEGs 与DEMs 的调控网络
经过String 数据库分析,共鉴定出得分在400 分以上的19 对蛋白-蛋白相互作用对,涉及19 个重叠DEGs,EDNRB-EDN1、RAMP2-VIPR1 和RAMP3-VIPR1 是 打 分最高的3 个关系对(分别为997、947 和947 分)。基于19 个蛋白-蛋白对和以上303 个负调控的miRNA-基因对构建了调节网络(图3),其中,hsa-mir-141-5p、hsa-mir-191-3p 和ADRA1A 的节点度最高,被认为是潜在的生物标志物。
2.5 DEGs 的验证
在TCGA 数据库中共筛选出1 175 个DEGs,并与癌旁组织进行比较,其中412 例上调,763 例下调。这些基因中有55 个基因(10 个上调,45 个下调)与上述的重叠DEGs(表2)相同并具有同样的表达趋势。其余6 个未鉴定的基因分别为TOX3、EDN1、SPDEF、FAM83A、FGFBP2 和COMP,说明本研究所鉴定的DEGs 是可靠的。此外,SFTPC、ITLN2 和SLC6A4 的表达量较高,且在2 组DEGs 中均具有显著差异。因此,选择SFTPC、ITLN2 和SLC6A4 作为潜在的标志物。
关键基因SFTPC、ITLN2、SLC6A4、hsa-mir-141-5p、hsa-mir-191-3p 和ADRA1A 的表达水平与患者总生存期和肿瘤分期间的相关性见表5。相关系数用来衡量2 个数据集合的线性关系,相关系数的绝对值越大,相关性越强。
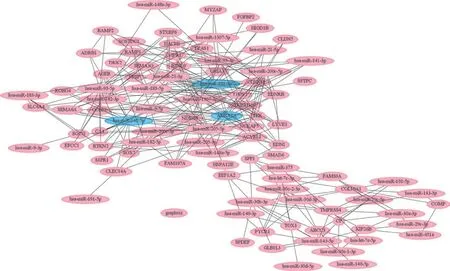
图3 重叠DEGs 和DEMs 的调控网络Fig 3 Regulated network of overlapped DEGs and DEMs
经过统计学分析,上述6 个基因中hsa-mir-191-3p 的表达与患者的肿瘤分期显著相关(P=0.005),其相关系数为正,说明基因的表达水平与肿瘤分期呈正相关;其他基因的表达与患者的总生存期和肿瘤分期间均无相关性(P>0.05)。
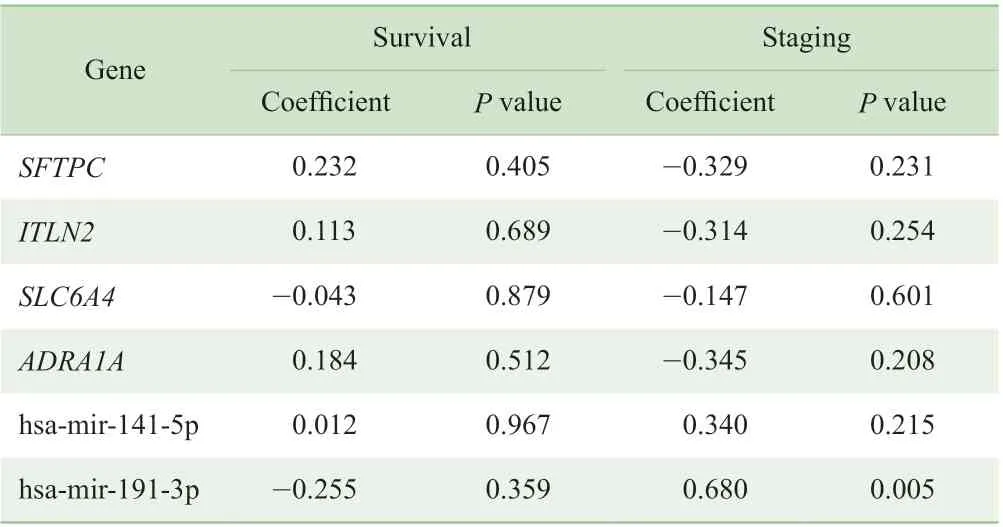
表5 SFTPC、ITLN2、SLC6A4、ADRA1A、hsa-mir-141-5p 和hsa-mir-191-3p 表达与总生存期和肿瘤分期的相关性Tab 5 Correlation of SFTPC, ITLN2, SLC6A4, ADRA1A, hsa-mir-141-5p and hsamir-191-3p expression with overall survival and tumor stage
RT-qPCR 结 果 如 图4 所 示。癌 组 织 中SFTPC 和SLC6A4 的表达水平明显低于癌旁组织,而hsa-mir-141-5p和hsa-mir-191-3p 的表达水平明显升高(P<0.05)。ITLN2和ADRA1A 在癌组织中的表达水平虽然与癌旁组织存在差异,但两者间差异无统计学意义(P>0.05)。EGFR 在癌组织中的表达水平高于癌旁组织,血清中CEA 和NSE 的表达水平分别为5.36 μg/L 和14.69 U/mL。
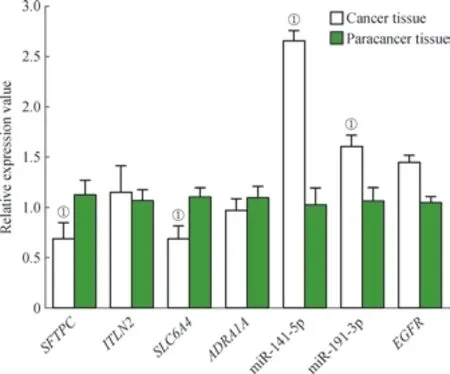
图4 肺腺癌组织和癌旁组织中SFTPC、ITLN2、SLC6A4、ADRA1A、hsamir-141-5p、hsa-mir-191-3p 和EGFR 的相对表达水平Fig 4 Relative expression levels of SFTPC, ITLN2, SLC6A4, ADRA1A, hsa-mir-141-5p, hsa-mir-191-3p and EGFR in lung adenocarcinoma tissues and paracancer tissues
3 讨论
GEO 数据库是由美国国立生物技术信息中心(National Center for Biotechnology Information,NCBI)创建并维护的基因表达数据库,其中收录了世界各国研究机构提交的高通量基因表达数据。GEO 数据库的设计初衷是为了收集整理各种基因表达芯片数据,后来陆续加入了甲基化芯片、lncRNA、miRNA、CNV 芯片等各种芯片,甚至高通量测序数据。在本研究中,从GEO 数据库中下载了3 个基因表达谱、1 个miRNA 表达谱和1 个circRNA表达谱,通过差异表达分析、功能富集分析、miRNA-基因关系对筛选以及综合调控网络构建,最终筛选出hsamiR-141-5p、hsa-miR-191-3p、ADRA1A、SFTPC、ITLN2和SLC6A4 为肺腺癌潜在的标志物。
hsa-mir-141-5p 和hsa-mir-191-3p 在肺癌中的作用尚不清楚,只有少数研究揭示了它们在其他癌症中的潜在作 用。Wang 等[14]确 定 了10 个mRNA(hsa-mir-141-5p、hsa-mir-1271-5p、hsa-mir-574-3p 等),对 人 类 卵 巢 癌 组织和正常组织的敏感度和特异度分别为97%和92%。Plieskatt 等[15]报道了hsa-mir-141-5p 和其他miRNA 结合与viverrini 感染的胆管癌亚型有关。Wu 等[16]发现与非侵袭性垂体腺瘤相比,hsa-mir-191-3p 在侵袭性垂体腺瘤中的表达上调。ADRA1A 在精神分裂症患者中受到广泛关注,它是导致更严重代谢异常的危险因素[17]。在肿瘤中,ADRA1A 是一种抑癌基因;与老年肝癌患者相比,年轻肝癌患者中ADRA1A 含量较少[18]。此外,ADRA1A 在子宫内膜癌患者外周血中高表达,ADRA1A 的表达与国际妇产科协会(FIGO)分期和淋巴结转移有关。因此,血清ADRA1A 的表达可用于评估生存率,并可能参与子宫内膜癌的发病和转移。本研究发现hsa-mir-141-5p、hsa-mir-191-3p 和ADRA1A 可能在肺腺癌中发挥一定的作用,并且调控网络进一步提供了可能的调控对,例如hsa-mir-141-5p 和hsa-mir-191-3p 可能通过靶向ADRA1A 发挥作用。
表面活性蛋白C(surfactant protein C,SFTPC)在肺癌中的研究已经取得了一定的成果。它是肺表面活性物质蛋白之一,与肺细胞的分化有关[19]。Saintigny 等[20]认为SFTPC 在肺腺癌组织中下调,与本研究结果一致。2014年的一项研究[21]表明,SFTPC 的表达与NSCLC 较短的总生存期显著相关。SLC6A4 是5-羟色胺转运体基因,其表达或甲基化与抑郁症的发生、表型及预后密切相关[22-23]。同时,SLC6A4 变异与结直肠癌患者的低生存率相关[24]。此外,SLC6A4 变异是慢性阻塞性肺病合并肺癌的风险因素[25]。然而,并没有发现另一潜在标志物ITLN2 在肺癌中的作用。在本研究中,与正常组织相比,SFTPC、ITLN2和SLC6A4 在肺组织中的表达有显著变化,提示它们可能在肺腺癌中起一定作用,但尚需实验室和临床证实,这也是我们下一步要开展的工作。
我们也试图从circRNA 表达水平揭示肺腺癌的相关机制,并获得了一些DECs。然而,只鉴定了3 组DECs中2 个相应的基因LDB2 和PREX1,而没有发现重叠的DECs。因此,推测circRNAs 的表达可能具时空特异性,可能与肺腺癌无关,具体原因尚需进一步研究。
在本研究中,对于从GEO 数据库中发现的重叠DEGs,通过TCGA 数据库进行了验证。TCGA 数据库是由美国国家癌症研究所和美国人类基因组研究所联合启动的一个项目。作为目前最大的癌症基因信息数据库,TCGA 收录了各种人类癌症的临床数据、基因组变异、mRNA 和miRNA 表达以及甲基化等数据,是肿瘤研究中资源最丰富、数据最权威的数据库[26]。本研究得到的61 个重叠DEGs 中,大多数都在TCGA 数据库中得到了验证,因此被认为是准确和可信的。只有6 个基因,即TOX3、EDN1、SPDEF、FAM83A、FGFBP2 和COMP 未被证实,它们在调控网络中没有起到关键作用。此外,我们 对TCGA 数 据 中SFTPC、ITLN2、SLC6A4、hsa-mir-141-5p、hsa-mir-191-3p 和ADRA1A 的表达水平与患者总生存期和肿瘤分期进行了相关性研究,其中hsa-mir-191-3p与患者的分期间存在显著相关性。这种相关性很可能反映了hsa-mir-191-3p 对肿瘤进展和细胞迁移的影响,有必要进行更多的研究,以进一步阐释hsa-mir-191-3p 作为肿瘤分期标志物的有效性及其在肿瘤细胞行为中的作用。其他基因的表达水平与患者的总生存期和肿瘤分期间均无相关性,但在癌组织和癌旁组织中的表达存在显著差异,说明这些基因在肺腺癌中的差异表达与患者的发病情况相关,与生存和预后无关。因此,这些基因在肺腺癌的临床诊断和治疗中可能具有潜在的应用价值。
在对选定的分子标志物SFTPC、ITLN2、SLC6A4、hsa-mir-141-5p、hsa-mir-191-3p 和ADRA1A 进行验证时,癌组织中SFTPC 和SLC6A4 的表达水平明显高于癌旁组织,hsa-mir-141-5p 和hsa-mir-191-3p 的表达水平明显降低,这些结果与上述生物信息学分析结果一致。而ITLN2和ADRA1A 在癌组织中的表达水平虽然低于癌旁组织,但两者间差异无统计学意义。芯片中的样本来自各个国家和地区,包括白种人、亚洲人和非裔人群等,而本研究收集的病例大部分为本市患者,这种个体差异可能导致不同人群中基因表达水平的差异。因此,后续需要进一步进行临床试验,对所选的标志物进行验证。此外,癌组织中EGFR 的表达水平明显高于癌旁组织,这与以往的研究结果一致[27]。EGFR 是表皮生长因子受体家族成员之一,EGFR 信号通路对细胞的生长、增殖和分化等生理过程发挥重要的作用,EGFR 阳性肺癌患者的生存期明显短于EGFR 阴性患者[28]。CEA 和NSE 是肺癌中常用的肿瘤标志物,在正常人体中的水平分别低于5.0 μg/L 和12.5 U/mL,在临床中经常可以发现肺腺癌患者血清CEA 及NSE水平升高[29]。本研究中检测结果显示CEA 和NSE 在肺腺癌患者中的表达水平分别为5.36 μg/L 和14.69 U/mL,这与以往的研究结果一致。
在本研究中,转录组测序和分析应用于识别重要的miRNA 和与转录组测序相关的基因。ADRA1A、hsa-miR-141-5p、hsa-miR-191-3p、SFTPC、ITLN2 和SLC6A4 可能是肺腺癌的潜在标志物。此外,以上6 个基因组合在临床检测中可能具有更高的准确度、敏感度及特异度,为肺腺癌的早期诊断和靶向治疗提供了理论依据和潜在的研究方向。然而,这些生物标志物的筛选仅仅是通过生物信息学分析,还需要更多的临床试验和更直接的证据进行验证。
参·考·文·献
[1] Siegel RL, Miller KD, Jemal A. Cancer statistics, 2020[J]. CA Cancer J Clin, 2020, 70(1): 7-30.
[2] Pitroda SP, Stack ME, Liu GF, et al. JAK2 inhibitor SAR302503 abrogates PDL1 expression and targets therapy-resistant non-small cell lung cancers[J]. Mol Cancer Ther, 2018, 17(4): 732-739.
[3] Denisenko TV, Budkevich IN, Zhivotovsky B. Cell death-based treatment of lung adenocarcinoma[J]. Cell Death Dis, 2018, 9(2): 117.
[4] Huret JL. Lung: translocations in adenocarcinoma[J]. Atlas Genet Cytogenet Oncol Haematol,2015. DOI: 10.4267/2042/56443.
[5] McGuire S. World cancer report 2014. Geneva, Switzerland: world health organization, international agency for research on cancer, WHO press, 2015[J]. Adv Nutr, 2016, 7(2): 418-419.
[6] Ettinger DS, Wood DE, Aisner DL, et al. Non-small cell lung cancer, version 5.2017, NCCN clinical practice guidelines in oncology[J]. J Natl Compr Canc Netw, 2017, 15(4): 504-535.
[7] Huang WW, Zhu WZ, Mu DL, et al. Perioperative management may improve long-term survival in patients after lung cancer surgery: a retrospective cohort study[J]. Anesth Analg, 2018, 126(5): 1666-1674.
[8] Tian X, Zhang HY,Zhang BJ, et al. Microarray expression profile of long non-coding RNAs in paclitaxel-resistant human lung adenocarcinoma cells[J]. Oncol Rep, 2017, 38(1): 293-300.
[9] Liu X, Sempere LF, Ouyang HX, et al. MicroRNA-31 functions as an oncogenic microRNA in mouse and human lung cancer cells by repressing specific tumor suppressors[J]. J Clin Invest, 2010, 120(4): 1298-1309.
[10] Zhu XL, Wang XY, Wei SZ, et al. Hsa_circ_0013958: a circular RNA and potential novel biomarker for lung adenocarcinoma[J]. FEBS J, 2017, 284(14): 2170-2182.
[11] Jiang MM, Mai ZT, Wan SZ, et al. Microarray profiles reveal that circular RNA hsa_circ_0007385 functions as an oncogene in non-small cell lung cancer tumorigenesis[J]. J Cancer Res Clin Oncol, 2018, 144(4): 667-674.
[12] Ding XX, Zhang S, Li X, et al. Profiling expression of coding genes, long noncoding RNA, and circular RNA in lung adenocarcinoma by ribosomal RNAdepleted RNA sequencing[J]. FEBS Open Bio, 2018, 8(4): 544-555.
[13] Zong L, Sun QC, Zhang HP, et al. Increased expression of circRNA_102231 in lung cancer and its clinical significance[J]. Biomedecine Pharmacother, 2018, 102: 639-644.
[14] Wang L, Zhu MJ, Ren AM, et al. A ten-microRNA signature identified from a genome-wide microRNA expression profiling in human epithelial ovarian cancer[J]. PLoS One, 2014, 9(5): e96472. DOI: 10.1371/journal.pone.0096472.
[15] Plieskatt JL, Rinaldi G, Feng YJ, et al. Distinct miRNA signatures associate with subtypes of cholangiocarcinoma from infection with the tumourigenic liver fluke Opisthorchis viverrini[J]. J Hepatol, 2014, 61(4): 850-858.
[16] Wu SL, Gu Y, Huang YY, et al. Novel biomarkers for non-functioning invasive pituitary adenomas were identified by using analysis of microRNAs expression profile[J]. Biochem Genet, 2017, 55(3): 253-267.
[17] Cheng C, Chiu HJ, Loh El-W, et al. Association of the ADRA1A gene and the severity of metabolic abnormalities in patients with schizophrenia[J]. Prog Neuropsychopharmacol Biol Psychiatry, 2012, 36(1): 205-210.
[18] Wang HW, Hsieh TH, Huang SY, et al. Forfeited hepatogenesis program and increased embryonic stem cell traits in young hepatocellular carcinoma (HCC) comparing to elderly HCC[J]. BMC Genom, 2013, 14(1): 736.
[19] Setoguchi Y, Tamaoki F, Nakai M, et al. The frequency of mutations in the surfactant protein C gene (SFTPC) is much higher in pediatric interstitial lung disease (PILD) than either adult-onset familial or sporadic interstitial pneumonia (IP)[C]. San Diego: American Thoracic Society 2009 International Conference, 2009.
[20] Saintigny P, Massarelli E, Lin S, et al. CXCR2 expression in tumor cells is a poor prognostic factor and promotes invasion and metastasis in lung adenocarcinoma[J]. Cancer Res, 2013, 73(2): 571-582.
[21] Lindskog C, Fagerberg L, Hallström B, et al. The lung-specific proteome defined by integration of transcriptomics and antibody-based profiling[J]. FASEB J, 2014, 28(12): 5184-5196.
[22] Dong C, Wong ML, Licinio J. Sequence variations of ABCB1, SLC6A2, SLC6A3, SLC6A4, CREB1, CRHR1 and NTRK2: association with major depression and antidepressant response in Mexican-Americans[J]. Mol Psychiatry, 2009, 14(12): 1105-1118.
[23] Philibert RA, Sandhu H, Hollenbeck N, et al. The relationship of 5HTT (SLC6A4) methylation and genotype on mRNA expression and liability to major depression and alcohol dependence in subjects from the Iowa Adoption Studies[J]. Am J Med Genet B Neuropsychiatr Genet, 2008, 147B(5): 543-549.
[24] Savas S, Hyde A, Stuckless SN, et al. Serotonin transporter gene (SLC6A4) variations are associated with poor survival in colorectal cancer patients[J]. PLoS One, 2012, 7(7): e38953. DOI: 10.1371/journal.pone.0038953.
[25] de Andrade M, Li Y, Marks RS, et al. Genetic variants associated with the risk of chronic obstructive pulmonary disease with and without lung cancer[J]. Cancer Prev Res (Phila), 2012, 5(3): 365-373.
[26] 谢龙祥, 闫中义, 党艺方, 等. TCGA 数据库: 海量癌症数据的源泉[J]. 河南大学学报(医学版), 2018, 37(3): 223-228.
[27] 赵敏, 田畅, 邸鑫, 等. RBM5、EGFR 和KRAS 在非小细胞肺癌中的表达及其意义[J]. 中国实验诊断学, 2019, 23(2): 303-308.
[28] Arfaoui A, Kriaa L, Znaidi N, et al. Over-expression of EGFR is closely correlated to poor prognosis in Tunisian patients with non-small cell lung adenocarcinoma[J]. J Immunoassay Immunochem, 2014, 35(3): 256-268.
[29] Peng Y, Wang Y, Hao XZ, et al. Utility of multiple increased lung cancer tumor markers in treatment of patients with advanced lung adenocarcinoma[J]. Chin J Lung Cancer, 2017, 20(10): 690-694.
