葡萄糖 -6- 磷酸脱氢酶基因4 种突变及蛋白结构分析
2020-03-19朱之星顾坚磊田国力
朱之星 ,纪 伟,顾坚磊 ,吕 晖 , ,田国力
1. 上海交通大学附属儿童医院,上海市儿童医院生物医学信息研究中心,上海 200040;2. 上海交通大学附属儿童医院儿童精准医学大数据工程技术研究中心,上海 200040;3. 上海交通大学附属儿童医院新生儿筛查中心,上海 200040;4. 上海交通大学生命科学技术学院,上海交通大学-耶鲁大学生物统计与数据科学联合中心,上海 200240
葡萄糖-6-磷酸脱氢酶(glucose-6-phosphate dehydrogenase,G6PD)缺乏症是常见的一种先天性酶缺乏遗传病,由定位于Xq28 上的G6PD 基因(OMIM:305900)突变导致G6PD 酶活性降低或缺乏。该基因编码为一个由515 个氨基酸组成的酶蛋白亚基,其同源二聚体具有酶活性,并能进一步产生酶促活性的四聚体蛋白[1-2]。G6PD 酶活性的降低会引起红细胞不能抵抗氧化损伤而遭受破坏,从而产生急性溶血,部分可引起新生儿期高胆红素脑病,导致智力低下甚至死亡[2]。
G6PD 缺乏症作为我国新生儿疾病筛查主要疾病之一,主要通过检测滤纸干血片血斑样品的酶活性,并结合后续生化或基因检测来明确诊断。随着测序技术的发展,G6PD 基因的突变位点被广泛研究[3-5]。1993 年杜传书团队[5]利用APCR 单链产物以双脱氧链终止法对该基因进行DNA 序列测定,首次在中国人群中发现了6 种G6PD 基因突变位点。如今,G6PD 缺乏症已知突变位点已达近200种[3],其中在中国人群中被报道的有40 多种[4,6-7]。多种基因检测方法不断发展[6,8-10],其中多色探针荧光PCR 熔解曲线法[10]因其检测速度快、简便、低成本等特点而被广泛使用。
然而,基因突变导致G6PD 酶活性降低或缺乏的分子机制尚未明确。有研究[11]认为基因突变导致了G6PD 酶蛋白稳定性的降低,也有研究[12-13]提示突变影响了辅酶NAPD+及底物与酶蛋白结合能力,从而影响其正常的生物学功能。为了更好地了解不同突变从哪些方面影响了酶活性,本研究分析了新生儿筛查G6PD 缺乏症人群的酶活性及基因突变分布情况,从分子、结构及功能的角度将4 种常见突变,即c.95A>G、c.1376G>T、c.1388G>A、c.1024C>T,与野生型G6PD 酶进行比较,并对这些突变蛋白结构及功能进行预测分析,揭示G6PD 突变与酶学结构、功能的关系,以期为遗传咨询提供帮助。
1 对象与方法
1.1 研究对象
纳入上海交通大学附属儿童医院2014—2017 年收集的205 103 例新生儿G6PD 缺乏症筛查记录(为保护患者隐私,在数据进行分析处理前,患者的个人可识别信息均已被剔除)。研究获上海交通大学附属儿童医院伦理委员会批准(2019R075-E01)。
1.2 方法
新生儿出生72 h 后,针刺足跟采血于滤纸上,制成滤纸干血片标本。通过新生儿G6PD 缺乏症筛查及分析并存于医院新生儿筛查中心系统。
1.2.1 G6PD 酶活性检测 采用新生儿G6PD 测定试剂(PerkinElmer,美国)及VICTOR ™D 荧光计(Wallac Oy, 芬兰),遵照实验室建立的标准操作流程检测:在室温下使血斑样品和含有 G6P、NADP+的底物试剂反应30 min,可通过添加硫酸铜减缓反应速度。荧光计设定激发波长为355 nm 和460 nm,进行荧光强度测量(荧光强度与酶活性呈正比),由机载软件处理得到G6PD 酶活性数据。设定酶活性的最低标准,以U/g(每克血红蛋白中)为酶活性单位,活性低于2.0 U/g 判定为初筛阳性。
1.2.2 16 个中国人群常见G6PD 突变位点的PCR 扩增 采用荧光PCR 熔解曲线法,取直径为3 mm 的3 个滤纸干血片,参照全血-824 核酸提取Mini 试剂盒(厦门致善生物科技有限公司,中国)说明书,通过 Lab-aid 824全自动核酸提取仪(厦门致善生物科技有限公司,中国)提取人基因组DNA,-20 ℃保存备用。使用 SLAN-96S荧光定量PCR 仪(厦门致善生物科技有限公司,中国)对靶标基因进行PCR 扩增。
反应的体系均为:DNA 模板2.6 μL,2×PCR 混合体系 14.0 μL,上、下游引物(20 μmol/L)各0.7 μL,用双蒸水补足体积至25.0 μL。反应程序为:50 ℃UnG 酶处理2 min;95 ℃预变性10 min;95 ℃变性15 s,65 ℃~56 ℃退火15 s(每个循环下降 1 ℃),76 ℃延伸20 s 以上过程进行10 个循环;随后95 ℃变性15 s,55 ℃退火15 s,76 ℃延伸20 s 过程进行50 个循环。
采用G6PD 基因突变检测试剂盒,根据靶标与探针杂交产物熔点的差异来检测突变位点,可以定性检测出16种中国人群常见的G6PD 基因突变,分别为:c.95A>G、c.383T>C、c.392G>T、c.487G>A、c.493A>G、c.517T>C、c.519C>T、c.592C>T、c.871G>A、c.1004C>A、c.1024C>T、c.1360C>T、c.1376G>T、c.1381G>A、c.1387C>T、c.1388G>A。
1.3 酶活性及突变情况统计学分析
应用R3.6.3 软件对数据结果进行统计分析,定量数据以M(Q1,Q3)表示,定性数据以n (%)表示。Wilcoxon秩和检验分析酶活性与突变基因类型之间的关系,在显著性水平=0.05 的条件下,对检验假设“H0:μ1=μ2;H1:μ1≠μ2”进行检验,其中μ1、μ2 分别为2 个总体的均值,以秩和W 作为统计量,P<0.05 认为有统计学意义。所有检验均为双侧检验。随后对不同突变的酶活性采用G6PD 缺乏症严重等级分型[14-15]进行描述性统计分析,以比较不同突变对患者造成的影响是否具有差异。
1.4 酶蛋白空间结构预测
G6PD 基因具有13 个外显子,除去1 号外显子不编码蛋白,根据cDNA 翻译的氨基酸序列,包含有515 个氨基酸,此外G6PD 的活性蛋白在人体中以同源二聚体和同源四聚体形式出现[2]。
G6PD 的cDNA 参考序列及氨基酸序列来源于Ensembl基因组数据库(ENST00000393564.6),Psipred[16-17]、SOPMA[18]、JPred4[19]被用来预测蛋白的二级结构。使用SWISS-MODEL[20]预测氨基酸链的空间结构,并参比PDB[21-22]蛋白结构数据库6e08 和6e07.1.A 分别构建蛋白单体及二聚体。
为保证预测可靠性,利用Mupro[23]、SDM[24-25]、CUPSAT[26]、mSCM[27]、DUET[28]、Dynamut[29]6 种基于结构、图形特征、统计势能等蛋白稳定性分析工具同时对突变前后蛋白的稳定性进行预测,以突变后蛋白折叠自由能的变化量ΔΔG作为统计量。若ΔΔG>0[23-29],则表示突变后蛋白结构的稳定性增加,反之表示蛋白结构稳定性降低。蛋白结构及预测图使用PyMOL 完成,LigPlot+[30]用来构建配体与蛋白相关作用预测图。
采用PROVEAN[31]工具预测氨基酸的改变对蛋白功能的影响。为保证更高的有害突变灵敏度,设置阈值为-1.3[31],PROVEAN 得分≤-1.3 的变量被认为是“有害的”,得分> -3 的变量被认为是“中性的”。
2 结果
2.1 G6PD 酶活性检测情况
2014—2017 年新生儿筛查的205 103 例G6PD 活性检测血样,男婴106 654 例(52%),女婴98 449 例(48%),中位采血日龄3 d(3 d,5 d),中位胎龄39+3周 (38+4周,40+1周),中位孕周体质量3 350 g(3 080 g,3 620 g)。通过荧光分析法测定G6PD 酶活性,确诊G6PD 缺乏症230例,其中男211 例(占92%),女19 例(占8%)。
2.2 基因突变检测分析
采用多色探针荧光PCR 溶解曲线法对121 例G6PD酶缺陷样本进行检测分析,检测出8 种点突变,其中7 种单碱基突变,分别是 c.95A>G、c. 1024C>T、c.392C>T、c.1376G>T、c.1388G>A、c.487G>A、c.517T>C,1 种复合点突变为c.95A>G+c.1388G>A。来自全国14 个行政区不同突变的酶活性结果如图1 所示。本研究人群除c.95A>G、c.1376G>T、c.1388G>A 3 种 常 见 突 变,c.1024C>T 也表现出高发生率,4 种突变占检测总人数的91%,且前3 种突变酶活性主要分布在0.2 ~0.6 U/g,c.1024C>T 突变则主要分布在0.4 ~0.8 U/g。
采用Wilcoxon 秩和检验估计4 种主要突变与酶活性之间的关系。表1 显示,c.1376G>T、c.1388G>A、c.95A>G 突变的酶活性两两之间差异均无统计学意义,3种突变与c.1024C>T 突变的酶活性比较,差异均有统计学意义,结果表明部分基因突变位点与酶活性有关 。
对不同突变进行分组,根据 G6PD 缺乏症严重等级分型[14-15]进行统计分析,发现121 例阳性记录仅表现出了Class Ⅲ和Class Ⅳ 2 种等级,且各突变两两之间比较差异均无统计学意义,见表2。
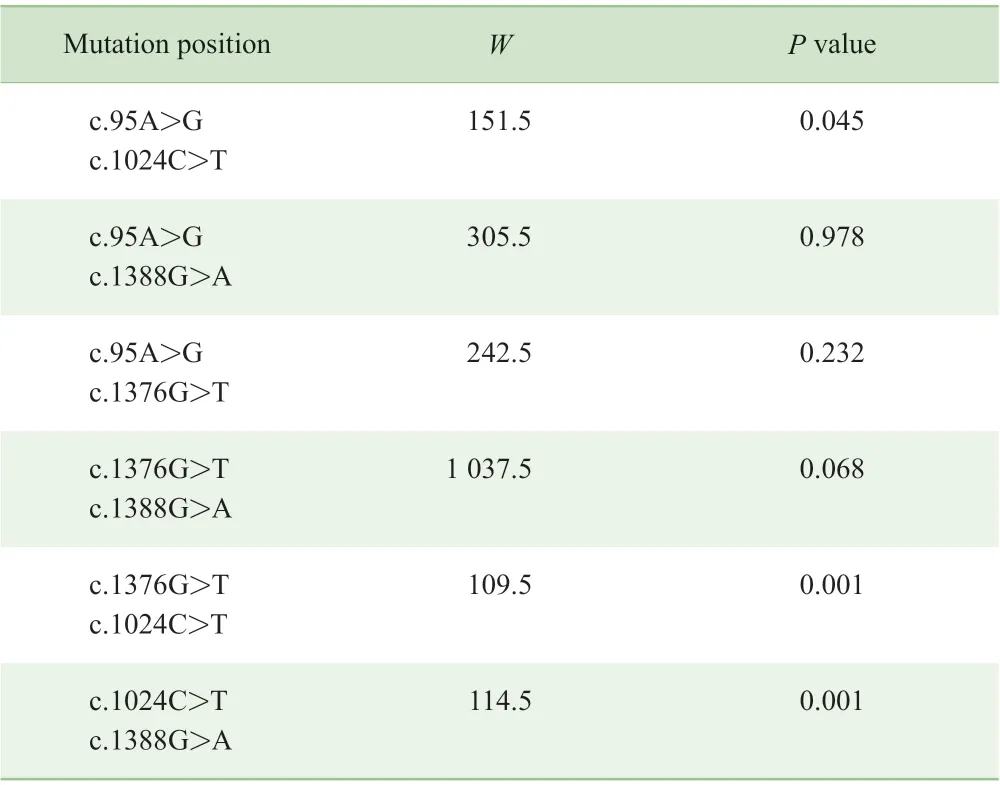
表1 4 种主要突变位点两两间的Wilcoxon 检验结果Tab 1 Wilcoxon test results between four major mutation positions
2.3 G6PD 酶蛋白的空间结构
2.3.1 G6PD 酶蛋白的二级结构 G6PD 野生型蛋白主要包含α-螺旋、β-折叠、β-转角以及无规则卷曲,其中α-螺旋 有228 个氨基酸,β-折叠占66 个氨基酸,β-转角有37个氨基酸,无规则卷曲有184 个氨基酸,4 种二级结构呈现于整个氨基酸链。与野生型蛋白相比,4 种突变蛋白的二级结构均有不同程度的改变,如表3 所示:4 种突变构成α-螺旋的氨基酸均减少,β-折叠的氨基酸均增加,c.95A>G 与c.1388G>A 2 种突变的β-转角氨基酸均减少了2 个,无规卷曲增加了3 个。
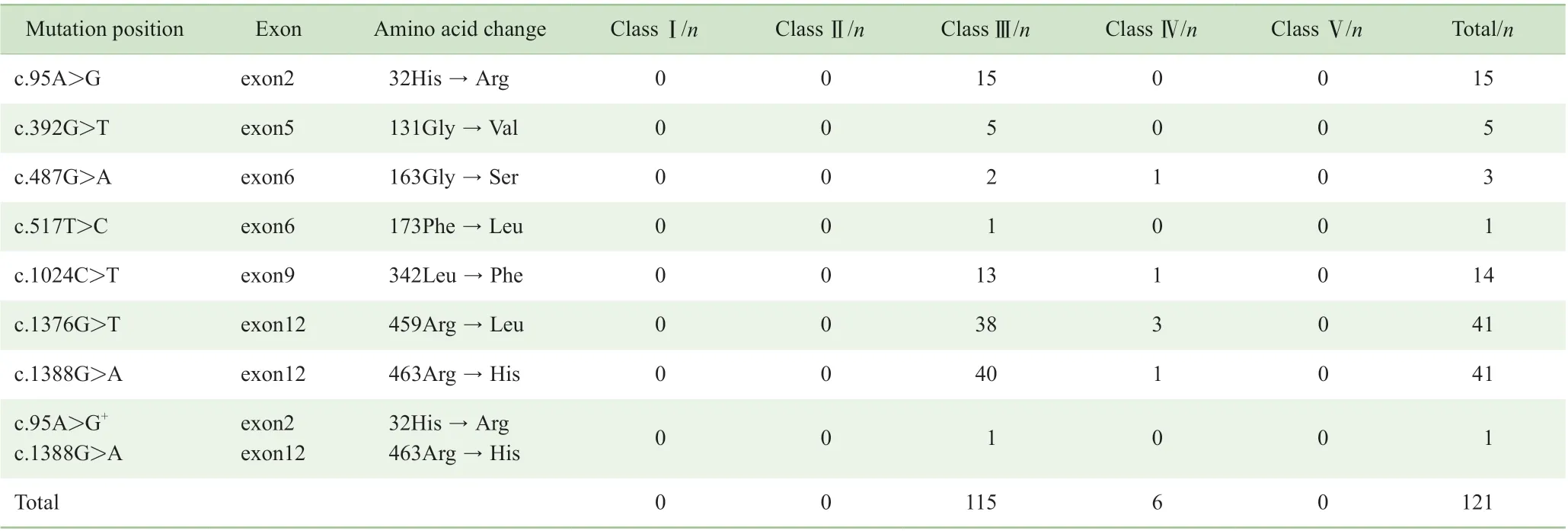
表2 突变位点与酶活性缺乏严重度情况Tab 2 Severity of mutation positions and enzyme activities
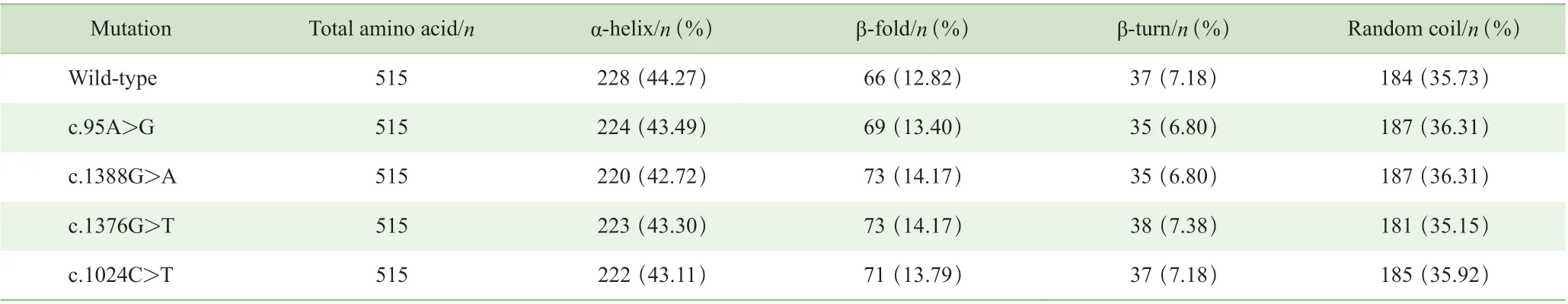
表3 G6PD 野生型及4 种突变蛋白的二级结构预测情况Tab 3 Prediction of secondary structure of G6PD wild type and four mutant proteins
2.3.2 G6PD 蛋白野生型空间构型 G6PD 蛋白单体由多个α-螺旋(图2A 蓝紫)及β-折叠(图2A 粉)构成了2个结构域,即底物结构域和α+β 结构域。前者较小的位于肽链N 端,参与底物的结合;后者主要由9 股β-折叠构成,与亚基的聚合有关;在折叠区还具有NAPD+配体的结合位点。G6PD 活性蛋白由2 个相同的亚基构成(图2B橘黄和蓝紫),具有2 个NADP+配体的结合位点以及多个GOL 位点(图2)。
2.3.3 G6PD 活性蛋白4 种高频突变的空间构型 图3 中显示了4 种错义突变氨基酸情况,以及突变位点与其他氨基酸共价键形成情况及相互作用的情况。
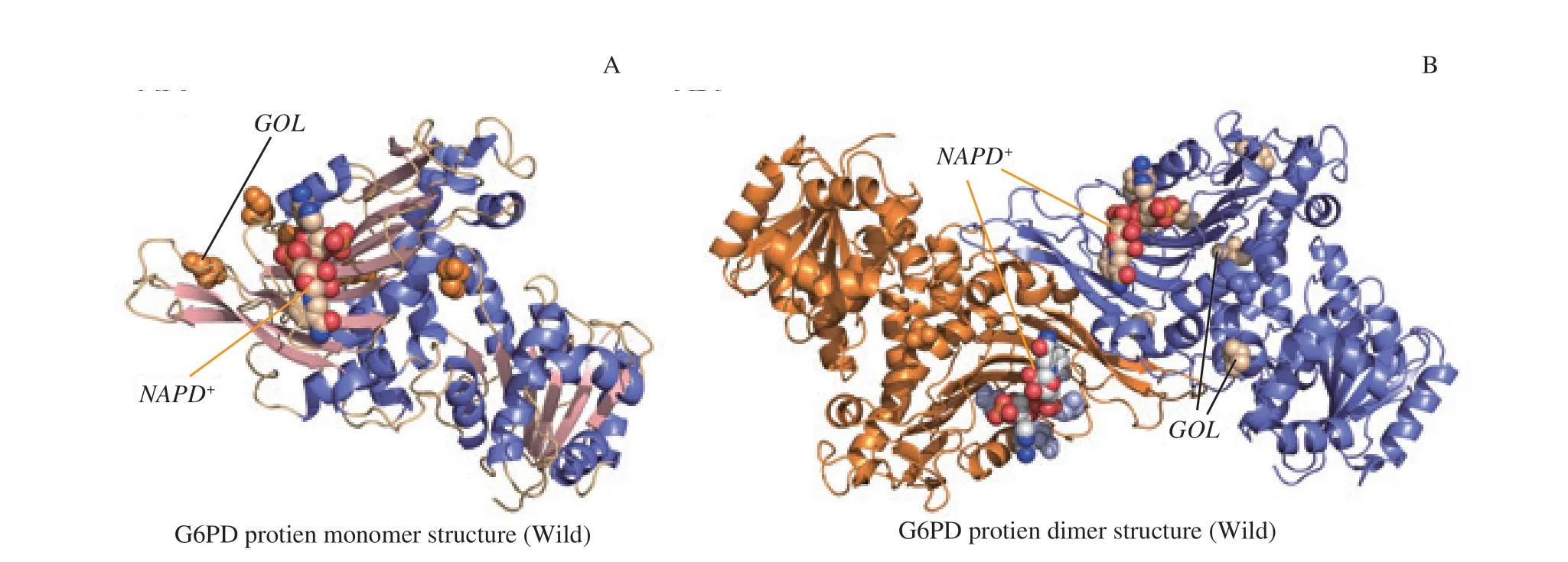
图2 G6PD 蛋白空间构型Fig 2 G6PD protein spatial configuration
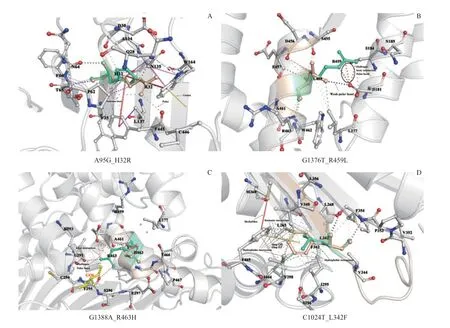
图3 4 种突变位点与相邻氨基酸的相互作用变化情况Fig 3 Interaction between four mutation sites and adjacent amino acids
从图中可以看到,H32 位于蛋白Rossman 结构区,H32R 突变前后部分残基之间的共价键断裂,成型环消失,此外也形成了新的阳离子链接区及极性键作用区(图3A)。R459 位于蛋白表面α 螺旋远离辅酶和底物结合区,用于稳定自身α 螺旋与相邻α 螺旋。与野生型相比,R459L 突变后与相邻α 螺旋间氢键、离子作用及极性作用均消失,与N181 号位氨基酸形成新的弱极性键(图3B)。R463 同样处于蛋白表面α 螺旋处,但与底物葡萄糖-6-磷酸结合位点相邻(图4),R463H 突变后部分氢键断裂、离子相互作用减弱,并且远离了配体甘油(GOL)的结合位点(图3C、图4),L342 位氨基酸处于α-β 结构域中的β-折叠区,L342F 突变导致该β 结构角度发生改变,另外形成了更多的疏水间连接,产生芳香基,出现成型环结构(图3D)。
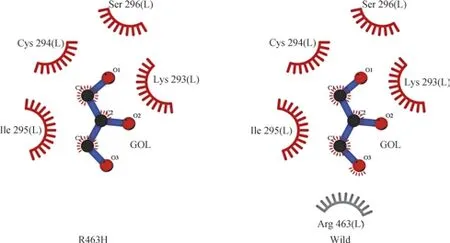
图4 配体GOL 与氨基酸相互作用情况Fig 4 Interaction between ligand GOL and amino acids
2.3.4 稳定性分析 采用6 种常用的蛋白稳定性预测方法对4 个突变后蛋白进行分析,综合结果显示4 种突变均可能造成不同程度蛋白稳定性的降低,尤其c.1388G>A 突变最为显著(表4)。
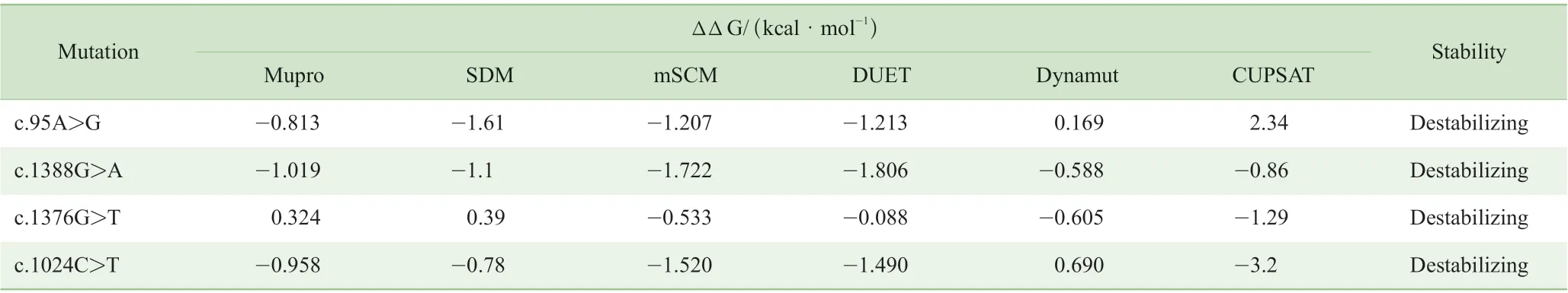
表4 6 种预测方法对4 种突变蛋白稳定性预测结果Tab 4 Six prediction methods for predicting the stability of four mutant proteins
2.3.5 氨基酸改变对蛋白功能的影响 采用PROVEAN分析软件对4 种氨基酸改变导致的蛋白功能改变情况进行预测,结果显示4 种突变的PROVEAN 得分均低于阈值-1.3,被判定为有害影响(表5)。
此现象表明,在蛋白质三维结构上,不同部分的4 个错义突变对G6PD 的表达具有负面影响。
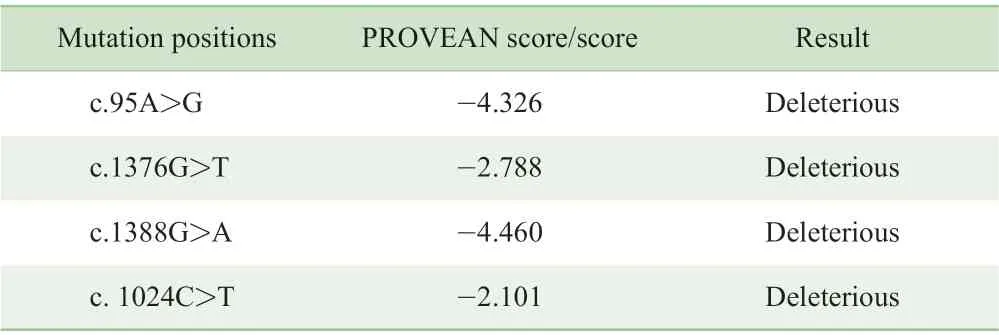
表5 4 种突变位点对蛋白功能影响的预测结果Tab 5 Prediction of the effect of four mutation positions on protein function
3 讨论
本研究回顾性分析了205 103 例新生儿G6PD 酶活性筛查记录,报道了其中的230 例酶活性检测G6PD 阳性样本,并对其中121 例进行了基因突变位点的检测;共检出8 种常见突变,其中3 种中国人群最常见突变被报道,发生率与其他研究相似,并有1 种突变c.1024C>T 在本研究中也表现出了高发生率。G6PD 基因的突变导致氨基酸的替换进而引起酶活性水平的降低。通过血液学研究,可根据红细胞中残留酶活性水平来判断G6PD 缺乏症症状严重程度。本研究的测定样本均来自新生儿滤纸干血片,以2.0 U/g 作为筛查临界值,所有突变体均显示出了比野生型酶蛋白低的活性(图1)。然而突变导致酶活性水平的降低可能是由于影响到G6PD 二聚体的形成、蛋白稳定性、NADP+结合区或者底物结合区等,比如F501 号位氨基酸突变成C501 改变了NADP+结合位点的大小,L205号位氨基酸与底物G6P 的结合及催化作用有关,不同的影响因素可能引起不同的临床表征。
通过对G6PD 的结构分析,发现α-螺旋及β-折叠氨基酸数量的改变可能会直接影响多肽链在空间上的折叠情况,造成固有结构域上原子或氨基酸之间的错位。c.95A>G 突变导致32 号位的组氨酸转变成精氨酸,H32位点位于蛋白Rossman 结构域,是β-α-β 组成的超二级结构单元,其序列为His-Ile-Phe-Ile-Ile-Met,由32 号组基酸开始,至37 号甲硫氨酸结束,共6 个氨基酸。Rossman折叠的结构域常含有功能性结合部位或活性部位,能调控整个酶的活性,因此在该位点上的突变可能会改变口袋的位置或大小,从而影响酶活性。此外,很多Rossman结构区具有磷酸化位点,磷酸化位点常出现在His 或Asp位置;而在该单元中32 号位恰好是His 位置,因此该位点也可能与蛋白的磷酸化/去磷酸化有关,该位点的突变可能导致了该位点无法去磷酸化,从而影响酶活性。c.1376G>T 突变导致精氨酸(R)突变成亮氨酸(L),亮氨酸是一个更为保守的氨基酸,氨基酸的保守突变在决定蛋白质三维结构与功能方面起重要作用。野生型R459 与D181 和N185 之间的螺旋间相互作用对于酶的催化活性和稳定性有重要影响,而突变型L459 导致螺旋间相互作用被削弱,从而使得相邻两螺旋的位移变大,螺旋结构变得松散。另外,L459 号位氨基酸的侧链与四聚体聚合有关,二聚体和四聚体的动态平衡对维持G6PD 蛋白具有重要作用。R463 与R459 同处一个α 螺旋,与L292、C294、I295、E460 形成了稳定的氢键、离子相互作用,稳定蛋白α 螺旋结构以及底物G6P 结合区。H463 突变导致该位点的侧链集团远离底物,与底物相互作用消失(图4),并且打断了与L292、C294、I295 等氨基酸的各种共价键与非共价键作用(图3C),与E460 号位的盐键崩溃,进而导致酶活性降低。L342 位于G6PD 中α+β 结构域的9 股不平衡β-折叠区,后与c.1024C>T 突变导致了亮氨酸突变成苯丙氨酸,F342 增大了该位点氨基酸所需的空间(图3D),而该位点的后一位氨基酸Y343 与底物GOL 接触,L342 突变可能在空间结构上会造成Y343 与底物的错位;L342 处于β 发夹结构并靠近β 转角(PDB 6e08),与F354 号位氨基酸形成氢键稳定发夹结构,F342 突变不仅会破坏稳定的发卡结构,还可能由于空间障碍阻碍β-转角的形成,使酶的三维结构改变。另外F342 突变增加了更多的疏水键,使得酶蛋白结构的刚性增加,而蛋白活性区的柔性与蛋白催化效率直接相关已经被证实,因此这一突变可能会导致蛋白催化活性的降低。4 种突变上述的结构错位、空间障碍、氢键断裂、盐键崩溃、柔性降低等影响,均可能导致酶蛋白的不稳定,从而降低突变蛋白的稳定性;如表4 中总结,4 种突变蛋白稳定性均表现出降低,并且对G6PD 蛋白均会造成不良影响,从而引起G6PD 蛋白的缺乏,造成新生儿的溶血等。
本研究详细地分析了G6PD 基因突变与酶活性降低的关系,根据基因突变引起的表达差异,从蛋白结构和功能上分析导致酶活性降低的可能原因。一方面对指导临床有重要作用;另一方面为我国G6PD 常见基因突变酶学结构与功能的研究提供了资料和理论依据,也为研究儿童遗传代谢类疾病提供了蛋白结构的分析方向。在G6PD 酶学结构及功能的研究和应用上,仍有许多未解决的问题有待探索。大多数已知的突变位点的蛋白结构仍然未被解析,酶活性降低是由于蛋白结构改变、稳定性降低、聚合能力的缺失还是配体结合能力的改变仍然不清楚,还有待未来研究人员探索。
参·考·文·献
[1] Cohen P, Rosemeyer MA. Subunit interactions of glucose-6-phosphate dehydrogenase from human erythrocytes[J]. Eur J Biochem, 1969, 8(1): 8-15.
[2] Au SW, Gover S, Lam VM, et al. Human glucose-6-phosphate dehydrogenase: the crystal structure reveals a structural NADP+molecule and provides insights into enzyme deficiency[J]. Structure, 2000, 8(3): 293-303.
[3] Minucci A, Moradkhani K, Hwang MJ, et al. Glucose-6-phosphate dehydrogenase (G6PD) mutations database: review of the “old” and update of the new mutations[J]. Blood Cells Mol Dis, 2012, 48(3): 154-165.
[4] Lin F, Lou ZY, Xing SY, et al. The gene spectrum of glucose-6-phosphate dehydrogenase (G6PD) deficiency in Guangdong Province, China[J]. Gene, 2018, 678: 312-317.
[5] 杜传书, 王菁, 陈路明, 等. 中国人中发现的6 种G6PD 基因点突变[J]. 中山大学学报(医学科学版), 1993, 14(4): 291.
[6] Yan JB, Xu HP, Xiong C, et al. Rapid and reliable detection of glucose-6-phosphate dehydrogenase (G6PD) gene mutations in Han Chinese using highresolution melting analysis[J]. J Mol Diagn, 2010, 12(3): 305-311.
[7] 林芬, 杨辉, 杨立业. 我国葡萄糖-6-磷酸脱氢酶缺乏症的分布特征和基因突变[J]. 分子诊断与治疗杂志. 2016, 8(2): 73-77.
[8] Hsu J, Fink D, Langer E, et al. PCR-based allelic discrimination for glucose-6-phosphate dehydrogenase (G6PD) deficiency in Ugandan umbilical cord blood[J]. Pediatr Hematol Oncol, 2014, 31(1): 68-75.
[9] Tseng CP, Huang CL, Chong KY, et al. Rapid detection of glucose-6-phosphate dehydrogenase gene mutations by denaturing high-performance liquid chromatography[J]. Clin Biochem, 2005, 38(11): 973-980.
[10] Xia ZM, Chen P, Tang N, et al. Rapid detection of G6PD mutations by multicolor melting curve analysis[J]. Mol Genet Metab, 2016, 119(1/2): 168-173.
[11] Scopes DA, Bautista JM, Naylor CE, et al. Amino acid substitutions at the dimer interface of human glucose-6-phosphate dehydrogenase that increase thermostability and reduce the stabilising effect of NADP[J]. Eur J Biochem, 1998, 251(1/2): 382-388.
[12] Hirono A, Kuhl W, Gelbart T, et al. Identification of the binding domain for NADP+of human glucose-6-phosphate dehydrogenase by sequence analysis of mutants[J]. PNAS, 1989, 86(24): 10015-10017.
[13] Jiang WY, Yu GL, Liu P, et al. Structure and function of glucose-6-phosphate dehydrogenase-deficient variants in Chinese population[J]. Hum Genet, 2006, 119(5): 463-478.
[14] WHO Working Group. Glucose-6-phosphate dehydrogenase deficiency[J]. Bull World Health Organ, 1989, 67(6): 601-611.
[15] Cappellini MD, Fiorelli G. Glucose-6-phosphate dehydrogenase deficiency[J]. Lancet, 2008, 371(9606): 64-74.
[16] Buchan DWA, Jones DT. The psipred protein analysis workbench: 20 years on[J]. Nucleic Acids Res, 2019, 47(W1): W402-W407.
[17] McGuffin LJ, Bryson K, Jones DT. The PSIPRED protein structure prediction server[J]. Bioinformatics, 2000, 16(4): 404-405.
[18] Geourjon C, Deléage G. SOPMA: significant improvements in protein secondary structure prediction by consensus prediction from multiple alignments[J]. Bioinformatics, 1995, 11(6): 681-684.
[19] Drozdetskiy A, Cole C, Procter J, et al. JPred4: a protein secondary structure prediction server[J]. Nucleic Acids Res, 2015, 43(W1): W389-W394.
[20] Waterhouse A, Bertoni M, Bienert S, et al. SWISS-MODEL: homology modelling of protein structures and complexes[J]. Nucleic Acids Res, 2018, 46(W1): W296-W303.
[21] ww PDB. Protein Data Bank: the single global archive for 3D macromolecular structure data[J]. Nucleic Acids Res, 2019, 47(D1): D520-D528.
[22] Berman HM, Westbrook J, Feng Z, et al. The Protein Data Bank[J]. Nucleic Acids Res, 2000, 28(1): 235-242.
[23] Cheng JL, Randall A, Baldi P. Prediction of protein stability changes for singlesite mutations using support vector machines[J]. Proteins, 2005, 62(4): 1125-1132.
[24] Pandurangan AP, Ochoa-Montaño B, Ascher DB, et al. SDM: a server for predicting effects of mutations on protein stability[J]. Nucleic Acids Res, 2017, 45(W1): W229-W235.
[25] Worth CL, Preissner R, Blundell TL. SDM: a server for predicting effects of mutations on protein stability and malfunction[J]. Nucleic Acids Res, 2011, 39(suppl): W215-W222.
[26] Parthiban V, Gromiha MM, Schomburg D. CUPSAT: prediction of protein stability upon point mutations[J]. Nucleic Acids Res, 2006, 34(Web Server): W239-W242.
[27] Pires DEV, Ascher DB, Blundell TL. mCSM: predicting the effects of mutations in proteins using graph-based signatures[J]. Bioinformatics, 2014, 30(3): 335-342.
[28] Pires DEV, Ascher DB, Blundell TL. DUET: a server for predicting effects of mutations on protein stability using an integrated computational approach[J]. Nucleic Acids Res, 2014, 42(W1): W314-W319.
[29] Rodrigues CH, Pires DE, Ascher DB. DynaMut: predicting the impact of mutations on protein conformation, flexibility and stability[J]. Nucleic Acids Res, 2018, 46(W1): W350-W355.
[30] Laskowski RA, Swindells MB. LigPlot+: multiple ligand-protein interaction diagrams for drug discovery[J]. J Chem Inf Model, 2011, 51(10): 2778-2786.
[31] Choi Y, Chan AP. PROVEAN web server: a tool to predict the functional effect of amino acid substitutions and indels[J]. Bioinformatics, 2015, 31(16): 2745-2747.
[32] Hanukoglu I. Proteopedia: Rossmann fold: a beta-alpha-beta fold at dinucleotide binding sites[J]. Biochem Mol Biol Educ, 2015, 43(3): 206-209.
[33] 徐芸, 罗建明. 我国G6PD 缺乏症基因突变的研究现状[J]. 中国小儿血液与肿瘤杂志, 2009, 14(3): 143-145.
[34] 董莹, 刘淑萍, 徐艳春, 等. 云南省1 例伯氨喹诱发溶血患者的G6PD 基因编码区突变分析及酶蛋白空间结构预测[J]. 中国寄生虫学与寄生虫病杂志, 2019, 37(4): 399-405.
[35] Rowland P, Basak AK, Gover S, et al. The three-dimensional structure of glucose 6-phosphate dehydrogenase from Leuconostoc mesenteroides refined at 2.0 A resolution[J]. Struct Lond Engl, 1994, 2(11): 1073-1087.
[36] Wang XT, Lam VMS, Engel PC. Marked decrease in specific activity contributes to disease phenotype in two human glucose 6-phosphate dehydrogenase mutants, G6PDUnionand G6PDAndalus[J]. Hum Mutat, 2005, 26(3): 284.
[37] Boonyuen U, Chamchoy K, Swangsri T, et al. A trade off between catalytic activity and protein stability determines the clinical manifestations of glucose-6-phosphate dehydrogenase (G6PD) deficiency[J]. Int J Biol Macromol, 2017, 104: 145-156.
[38] Bautista J, Mason PJ, Luzzatto L. Human glucose-6-phosphate dehydrogenase Lysine 205 is dispensable for substrate binding but essential for catalysis[J]. FEBS Lett, 1995, 366(1): 61-64.
[39] Lesk AM. NAD-binding domains of dehydrogenases[J]. Curr Opin Struct Biol, 1995, 5(6): 775-783.
[40] Hwang S, Mruk K, Rahighi S, et al. Correcting glucose-6-phosphate dehydrogenase deficiency with a small-molecule activator[J]. Nat Commun, 2018, 9(1): 4045.
[41] 邹承鲁. 酶活性部位的柔性[J]. 科学通报, 1989(5): 321-325.
[42] 邹承鲁. 活性部位的柔性[J]. 生理科学进展, 2001(1): 7-12.
