构建反转录病毒小向导RNA 表达载体用于小鼠T 细胞基因 功能研究
2020-03-19赵艳娜唐元家
赵艳娜,邱 荣,沈 南,唐元家
1.上海交通大学医学院附属仁济医院风湿病科,上海市风湿病学研究所,上海 200125;2.中国科学院大学上海营养与健康研究所,上海 200031
CRISPR/Cas9 系统作为一个强大的基因组编辑工具,最早发现于细菌和古细菌中。它是细菌和古细菌在长期不断进化的过程中产生的适应性免疫防御机制,用以保护自身的基因组免受外源核酸(如噬菌体、病毒等)的干扰和破坏[1]。CRISPR-Cas9 系统主要由2 个部分组成:一是CRISPR 相关核酸酶,目前基因编辑系统中用到的主要是Cas9 核酸酶;二是小向导RNA(small guide RNA,sgRNA),含有20 nt 能够与靶基因组互补的序列[2-3]。 Cas9 核酸内切酶可以在sgRNA 的引导下对靶基因组DNA 进行切割,导致DNA 双链断裂[4]。在哺乳动物细胞中由CRISPR/Cas9 系统引起的双链断裂主要通过易错的非同源末端连接(non-homologous end joining)机制修复,往往导致基因突变和功能丧失[5-6],因此被广泛应用于各种疾病治疗、基因功能鉴定、动物模型建立以及药物研发[6]。然而,如何有效递送CRISPR/Cas9 系统至目标细胞及靶器官仍是该技术面临的主要挑战之一。
辅助性CD4 T 细胞在适应性免疫系统中发挥着重要作用。在免疫应答过程中,初始CD4 T 细胞受到刺激后会分化成不同的效应T 细胞亚群,包括辅助性T 细胞1(T helper cell 1,Th1)、辅助性T 细胞2(T helper cell 2,Th2)、辅助性T 细胞17(T helper cell 17,Th17) 和调节性T 细胞(Regulatory T cells,Treg)[7]。不同的效应T 细胞亚群在免疫应答过程中发挥着不同的功能。例如,Th17及其分泌的效应细胞因子在宿主对抗各种感染(尤其是细胞外细菌感染)以及多种自身免疫疾病中发挥了重要作用[8]。Treg 细胞可以分泌转化生长因子β(transforming growth factor-β,TGF-β) 和 白 介 素-10(interleukin 10,IL-10)等抑制性细胞因子,不仅可以控制免疫耐受和免疫反应强度,而且在避免组织炎症损伤方面发挥重要作用[9]。因此,研究辅助性T 细胞的分化发育及其分子调控机制至关重要。
目前,CRISPR/Cas9 技术的发展,使得对免疫细胞进行大规模基因研究成为可能。如Shifrut 等[10]利用慢病毒sgRNA 文库和Cas9 蛋白,进行全基因组筛选影响人类T 细胞免疫反应的调控分子。但目前现有的CRISPR/Cas9文库大多基于慢病毒表达载体,很难感染小鼠T 细胞。因此,建立适合小鼠T 细胞基因功能研究的高通量筛选系统是亟待解决的问题。本研究构建了sgRNA 反转录病毒载体,结合Cas9 转基因小鼠,研究影响Th17 分化的调控分子,为小鼠T 细胞功能研究提供工具。
1 材料与方法
1.1 实验材料
1.1.1 实验动物 Rosa26-LSL-Cas9(024857)和CD4-Cre(022071)小鼠购自美国杰克森实验室,饲养于中国科学院上海生命科学研究院SPF 级动物房[实验动物生产许可证号SCXK(沪)2018-0002,使用许可证号SYXK(沪)2019-0001],饲养温度22 ~25 ℃,空气相对湿度40%~60%。实验获中国科学院上海生命科学研究院医学研究伦理委员会批准(伦理审批号为201903H486)。
1.1.2 主要试剂 抗小鼠IL17A 抗体(PE anti-mouse IL-17A antibody,TC11-18H10.1;BioLegend,美国);抗小鼠CD3e 抗体(145-2C11)、抗小鼠CD28 抗体(37.51)、抗小鼠γ 干扰素(interferon-γ,IFN-γ)抗体(XMG1.2)、抗小鼠IL-4 抗体(11-B11;eBioscience,美国);细胞固定/破膜试剂盒(BD Cytofix/Cytoperm ™ Kit;BD Biosciences,美国),小鼠CD4 T 细胞分选试剂盒(Miltenyi,德国);Xho1 内切酶、Sal1 内切酶、Taq DNA 聚合酶、NEBuider HiFi DNA Assembly Master Mix(NEB,美国);磷酸缓冲盐溶液(phosphate buffer saline,PBS)、DMEM 培养基、Opti-MEM 无血清培养基、RPM 1640、胎牛血清(fetal bovine serum,FBS;Gibco,美 国);TGF-β1、IL-6(R&D, 美 国);Lipo2000(Thermo Fisher Scientific, 美国),QIAamp DNA Mini Kit (Qiagen,中国)。
1.1.3 主要仪器和设备 超净工作台、CO2培养箱(Thermo Fisher Scientific, 美 国),Centrifuge 5417R 低温高速离心机(Eppendorf,德国),DNA Thermal Cycler 9700 PCR 仪(Applied Biosystems;Thermo Scientific, 美国),凝胶图像分析系统(Bio-Rad 3000,中国),普通光学显微镜(Olympus,日本),荧光显微镜(ZEISS,德 国),CytoFLEX LX 流 式 细 胞 仪(Beckman Coulter, 美国)。
1.2 方法
1.2.1 构建sgRNA 反转录病毒表达载体 利用内切酶Xho1 和Sal1 双酶切MSCV-LTR-miR30-PIG(LMP)反转录病毒载体,回收大片段得到反转录病毒载体骨架。以慢病毒载体pKLV-U6-sgRNA 为模版设计PCR 引物,引物序列如表1。利用NEBuider HiFi DNA Assembly Master Mix将U6-sgRNA-PGK-Puro-BFP 片段组装进反转录病毒载体骨架。经过转化,挑克隆,测序得到正确载体。

表1 U6-sgRNA-PGK-Puro-BFP 片段的PCR 引物序列Tab 1 Primer sequences for U6-sgRNA-PGK-Puro-BFP PCR
1.2.2 sgRNA 设计和重组反转录病毒包装 MIT CRISPR(http://crisp.mit.edu)网站设计sgRNA 序列,设计好的sgRNA 序列交由苏州金唯智生物科技公司合成。用Plat-E包装细胞产生反转录病毒,使用Lipo2000 进行质粒转染。以10 cm 培养皿为例,500 μLOpti-MEM 无血清培养基中加入20 μg sgRNA 反转录病毒载体;另取500 μL Opti-MEM 加入40 μL Lipo2000,将前2 步所得混合液混匀。室温放置15 min 后,加入培养皿中。转染6 h 后细胞换液,48 h 后收集上清(病毒),0.45 μm 细胞滤网过滤后冻存于-80 ℃冰箱中,备用。
1.2.3 T7E1 实验验证sgRNA 编辑效果 使用DNA 提取试剂盒提取细胞基因组DNA,Taq DNA 聚合酶进行PCR扩增。在Il17a sgRNA 靶向区域上下游设计引物,PCR 产物纯化,取200 ng DNA 按照T7E1 实验说明书进行操作。加入1 μL T7 核酸内切酶,37 ℃反应30 min,加入乙二胺四乙酸终止反应。产物在2%琼脂糖凝胶上进行凝胶电泳。T7E1 引物见表2。

表2 T7E1 PCR 引物序列Tab 2 Primer sequences for T7E1
1.2.4 分离Cas9 转基因小鼠CD4 T 细胞 分离Cas9 转基因小鼠脾脏细胞,裂解红细胞。按照小鼠CD4 T 细胞分选试剂盒说明书分离小鼠初始CD4 T 细胞。流式细胞仪检测细胞纯度(95%以上),进行后续实验。
1.2.5 CD4 T 细胞体外激活培养 CD3 和CD28 抗体包被在96 孔板,按照5 μg/mL 的终浓度加入抗小鼠CD3e抗体(145-2C11)和抗小鼠CD28 抗体(37.51),100 μL/孔包被96 孔板,并于4 ℃过夜。吸掉抗体悬液,加入200 μL 封闭液(PBS+ 1% FBS),于室温封闭30 min,封闭结束加入200 μL PBS 洗涤2 次。分离得到CD4 T 细胞,按照细胞浓度1×106/mL 重悬。每孔加入200 μL 细胞,放入37 ℃的CO2培养箱中进行培养。
1.2.6 反转录病毒感染和小鼠Th17 细胞诱导分化 小鼠CD4 T 细胞激活24 h 后进行反转录病毒的感染。以96孔板为例,准备病毒和聚凝胺(polybrene,5 μg/mL)的混合液并吸取培养基上清液,加入病毒和polybrene 混合液;32 ℃、1 000 mL 离心90 min。离心结束后,弃去病毒上清液,加入Th17 分化培养基。Th17 分化培养基为1640 完全培养基并添加相应细胞因子(mTGF-β1 1 ng/mL,mIL-6 10 ng/mL,抗小鼠IFN-γ 抗体10 μg/mL,抗小鼠IL-4 抗体10 μg/mL)。体外诱导3 d,进行后续检测。
1.2.7 细胞因子刺激和检测 小鼠Th17 细胞体外分化3 d 后,检测细胞因子Il17A 表达水平。收集细胞前,用50 ng/mL 佛波酯、500 ng/mL 离子霉素和蛋白转运抑制剂刺激4 h。收集细胞,用BD 细胞因子染色试剂盒进行染色,流式细胞仪检测。使用FlowJo 软件(Becton Dickinson 10.0.7)进行流式数据分析。
1.3 统计学方法
采用GraphPad Prism 5 软件进行数据分析,通过非配对t 检验 ,统计分析NC-sgRNA、Il17a-sgRNA、Irf4-sgRNA、Rorc-sgRNA 组sgRNA+Il17a+/sgRNA-Il17a+的数值。P<0.05 为差异有统计学意义。
2 结果
2.1 构建反转录病毒sgRNA 表达载体
为了建立用于小鼠T 细胞基因功能研究的CRISPR/Cas9 系统,实验构建了反转录病毒sgRNA 表达载体:用Xho1 和Sal1 双酶切反转录病毒载体LMP,通过胶回收得到反转录病毒载体骨架;以慢病毒载体pKLV-U6-sgRNA为模版,通过PCR 扩增获得U6-sgRNA-PGK-Puro-BFP 片段。利用同源重组将U6-sgRNA-PGK-Puro-BFP 片段组装进反转录病毒载体骨架得到反转录病毒sgRNA 表达载体。在该载体中,U6 启动子驱动sgRNA 表达,而PGK 启动子驱动嘌呤霉素抗性基因(puromycin,Puro)和蓝色荧光蛋白(blue fluorescent protein,BFP)表达(图1)。
2.2 表达sgRNA 反转录病毒包装和细胞感染
为了验证新构建的载体是否可以产生具有感染性的病毒颗粒,实验对载体进行病毒包装和细胞感染,结果见图2。 病毒包装细胞为Plat-E 细胞,只需要转染反转录病毒载体质粒即可。转染24 h 后,光学显微镜下观察细胞状态,细胞形态正常。转染48 h 后,荧光显微镜下观察到BFP的成功表达。为了验证反转录病毒是否具有感染活性,实验用产生的反转录病毒对NIH3T3 细胞进行感染。病毒感染48 h 后收集细胞,流式细胞仪检测BFP 阳性细胞的情况,结果显示BFP 阳性细胞比例为54.1%,说明反转录病毒sgRNA 表达载体构建成功并可产生有感染性的病毒颗粒,可用于后续实验。
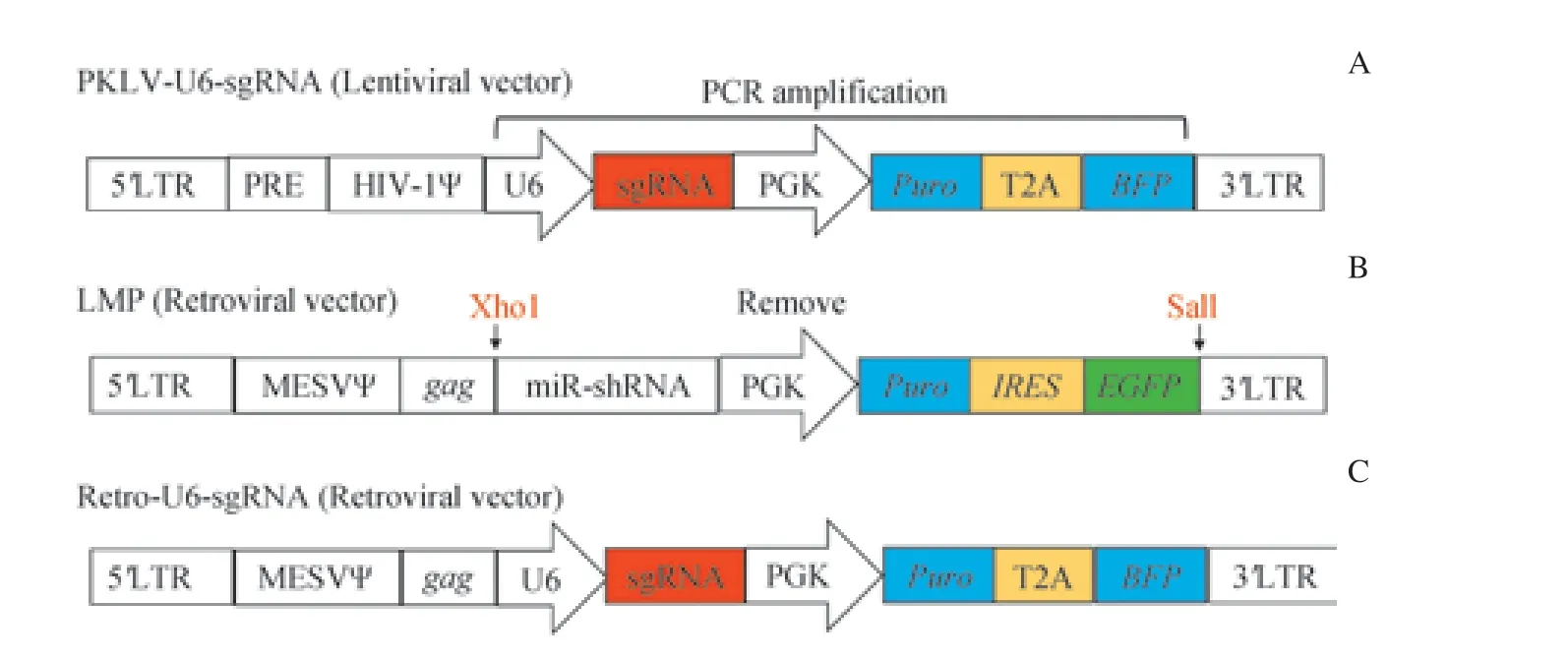
图1 反转录病毒sgRNA 表达载体构建策略Fig 1 Construction strategy of retroviral sgRNA expression vector
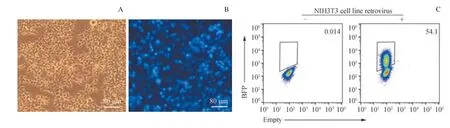
图2 反转录病毒包装和NIH3T3 细胞感染Fig 2 Retrovirus packaging and NIH3T3 cell infection
2.3 小鼠Th17 细胞体外分化系统建立
为了将CRISPR/Cas9 系统应用于小鼠Th17 细胞分化研究,实验首先建立了Th17 细胞体外诱导分化系统。利用小鼠CD4 T 细胞分选试剂盒分离Cas9 转基因小鼠初始CD4 T 细胞,流式细胞仪检测结果表明细胞纯度在95% 以上(图3)。CD4 T 细胞铺孔,观察发现最先分离得到的小鼠CD4 T 细胞体积较小,呈圆形。经过3 d 体外诱导培养后,细胞数目明显变多,形态逐渐变成椭圆形或者条形。结果说明,实验成功分离小鼠CD4 T 细胞并可以进行体外培养,可进行后续实验。

图3 小鼠Th17 细胞分化过程中的形态变化Fig 3 Morphological changes of mouse Th17 cells during differentiation
2.4 小鼠Th17 细胞中基因敲除
Th17 主要分泌Il17a 和Il17f 等促炎症细胞因子,在自身免疫病发病中发挥作用,而Th17 的分化受到很多转录因子的调控,如Irf4、Rorc、Batf 等。为了验证系统是否能用于基因敲除以及Th17 分化调控研究。实验分别设计了靶向Il17a、Irf4、Rorc,以及NC 的sgRNA,并进行克隆和病毒包装。分离Cas9 转基因小鼠初始CD4 T 细胞,经过CD3/28 抗体刺激24 h 后,进行反转录病毒感染。感染完成后,加入Th17 细胞培养基诱导分化3 d,流式细胞仪检测Th17 细胞内Il17a 表达。结果发现,在NC-sgRNA组中Il17a 阳性细胞在sgRNA+和sgRNA-2 个群体中的比例大致相等。而在Il17a-sgRNA 组中,Il17a 阳性细胞在sgRNA+群体较sgRNA-群体中的比例明显降低。结果说明在Il17a 孔中,sgRNA+群体Il17a 基因被成功敲除。同样 在Rorc 和Irf4 组 中,2 组sgRNA+群 体 和sgRNA-群体相比,IL17a 阳性细胞的比例明显降低。结果说明2 组sgRNA+群体中Rorc 和Irf4 基因被成功敲除。为了减小孔间差异对结果偏差的影响,实验进一步统计了同一个孔中sgRNA+组与sgRNA-组中Il17a 阳性细胞比例的比值,对数据进行标准化处理。对标准化后的比值进行统计分析(图4)。结果发现,与NC 组相比,Il17a、Rorc 和Irf4 组比值均显著降低。以上结果说明,利用该系统可以成功进行基因敲除。
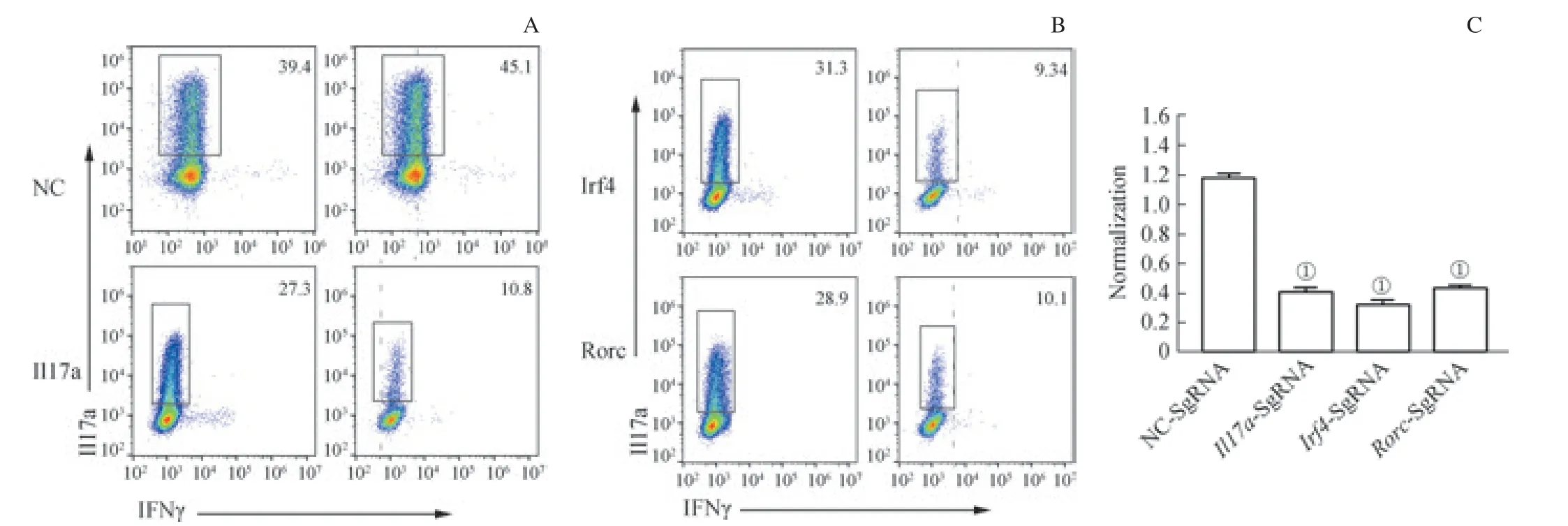
图4 小鼠Th17 细胞中基因敲除Fig 4 Gene knockout in mouse Th17 cells
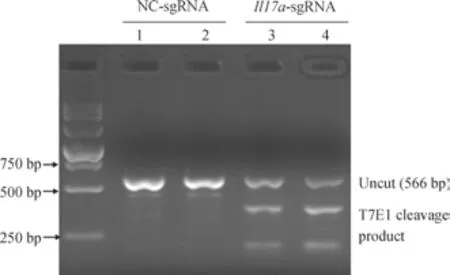
图5 T7E1 实验检测Il17a 位点DNA 突变Fig 5 Detecting DNA mutations of Il17a locus by T7E1
2.5 T7E1 实验检测Il17a 位点DNA 突变
为了进一步验证Il17a 基因组DNA 是否发生突变,实验提取编辑后细胞基因组DNA。在Il17a-sgRNA 靶向位置上下游,设计引物进行PCR 扩增,进行后续T7E1 实验。凝胶电泳结果显示,在NC-sgRNA 组中,条带完整未发生切割;而在Il17a-sgRNA 组中,条带被切割成约380 bp 和180 bp 的2 个条带,符合Il17a-sgRNA 靶向突变预期。结果表明,NC-sgRNA 组Il17a 基因未发生基因编辑;而Il17a-sgRNA 组IL17A 位点DNA 发生突变,Il17a 基因被敲除(图5)。通过检测基因组DNA 突变,进一步证明了系统的有效性。
3 讨论
初始CD4 T 细胞在不同细胞因子的刺激下,会分化成不同的细胞亚群,如Th1、Th2、Th17 和Treg[11]。但不同分化亚群在介导疾病发生中发挥着不同的作用,如Th17细胞主要在自身免疫病以及炎症中发挥重要作用,而Treg 细胞主要维持机体免疫稳态从而抑制自身免疫病的发生[12]。因此,研究T 细胞分化调控,有助于更好地理解自身免疫病的发病机制。
近年来,很多新技术应用于T 细胞分化的研究。Chen等[13]建立一套基于RNA 干扰体内筛选系统,用以筛选淋巴细胞脉络丛脑膜炎病毒(LCMV)感染状态下影响抗病毒CD4 和CD8 T 细胞分化的调控分子。CRISPR/Cas9 基因编辑技术的发展,使得全基因功能筛选成为可能。Shalem 等[14]建立了全基因组筛选影响人类肿瘤细胞增殖的基因。Parnas 等[15]基于慢病毒载体的全基因组sgRNA 文库和Cas9 转基因小鼠进行了全基因组筛选,用于研究树突状细胞在受到细菌脂多糖刺激产生肿瘤坏死因子-α(TNF-α)的调控分子。但是,由于Cas9 蛋白分子量大,很难包装进病毒载体,限制了CRISPR/Cas9 在小鼠免疫细胞中的广泛应用。因此,实验构建了Cas9 转基因小鼠。只需要构建表达sgRNA 的病毒载体即可在小鼠T 细胞中进行基因功能研究。近年来,慢病毒载体由于能整合到宿主基因组DNA 稳定遗传,被广泛应用于sgRNA 递送系统[16]。但是,慢病毒感染小鼠T 细胞效率较低,而且表达速度较慢[17]。所以实验构建了反转录病毒sgRNA 表达载体用于小鼠T 细胞分化和基因功能 研究。
综上,本研究利用反转录病毒sgRNA 表达载体和Cas9 转基因小鼠,成功在小鼠T 细胞中进行基因敲除,并将此系统应用于Th17 细胞分化调控研究,验证Rorc 和Irf4 是Th17 细胞分化中的关键调控分子。未来可以利用该系统构建反转录病毒sgRNA 全基因组筛选文库通量研究小鼠T 细胞分化调控分子,以期找到新的靶点。
参·考·文·献
[1] Horvath P, Barrangou R. CRISPR/Cas, the immune system of bacteria and archaea[J]. Science, 2010, 327(5962): 167-170.
[2] Jinek M, Chylinski K, Fonfara I, et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity[J]. Science, 2012, 337(6096): 816-821.
[3] Garneau JE, Dupuis MÈ, Villion M, et al. The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA[J]. Nature, 2010, 468(7320): 67-71.
[4] Ran FA, Hsu PD, Lin CY, et al. Double nicking by RNA-guided CRISPR Cas9 for enhanced genome editing specificity[J]. Cell, 2013, 154(6): 1380-1389.
[5] Hwang WY, Fu YF, Reyon D, et al. Efficient genome editing in zebrafish using a CRISPR-Cas system[J]. Nat Biotechnol, 2013, 31(3): 227-229.
[6] Jinek M, East A, Cheng A et al. RNA-programmed genome editing in human cells[J]. Elife, 2013, 2 (6121): e00471.
[7] Luckheeram RV, Zhou R, Verma AD, et al. CD4+T cells: differentiation and functions[J]. Clin Dev Immunol, 2012, 2012: 1-12.
[8] Ouyang WJ, Kolls JK, Zheng Y. The biological functions of T helper 17 cell effector cytokines in inflammation[J]. Immunity, 2008, 28(4): 454-467.
[9] Sharma A, Rudra D. Emerging functions of regulatory T cells in tissue homeostasis[J]. Front Immunol, 2018, 9: 883.
[10] Shifrut E, Carnevale J, Tobin V, et al. Genome-wide CRISPR screens in primary human T cells reveal key regulators of immune function[J]. bioRxiv, 2018, 175 (7): 1958-1971 e1915.
[11] Zhu JF, Yamane H, Paul WE. Differentiation of effector CD4 T cell populations[J]. Annu Rev Immunol, 2010, 28(1): 445-489.
[12] Lee G. The balance of Th17 versus treg cells in autoimmunity[J]. Int J Mol Sci, 2018, 19(3): 730.
[13] Chen RQ, Bélanger S, Frederick MA, et al. In vivo RNA interference screens identify regulators of antiviral CD4+and CD8+T cell differentiation[J]. Immunity, 2014, 41(2): 325-338.
[14] Shalem O, Sanjana NE, Hartenian E, et al. Genome-scale CRISPR-Cas9 knockout screening in human cells[J]. Science, 2014, 343(6166): 84-87.
[15] Parnas O, Jovanovic M, Eisenhaure TM, et al. A genome-wide CRISPR screen in primary immune cells to dissect regulatory networks[J]. Cell, 2015, 162(3): 675-686.
[16] O'Keefe EP. Nucleic acid delivery: lentiviral and retroviral vectors[J]. Mater Methods, 2013, 3:174-177
[17] Baumann JG, Unutmaz D, Miller MD, et al. Murine T cells potently restrict human immunodeficiency virus infection[J]. J Virol, 2004, 78(22): 12537-12547.
