大熊猫源致病大肠杆菌CCHTP全基因组测序及耐药和毒力基因分析
2019-12-24邓雯文李才武赵思越李仁贵何永果吴代福杨盛智黄炎张和民邹立扣
邓雯文,李才武,赵思越,李仁贵,何永果,吴代福,杨盛智,黄炎,张和民,邹立扣
大熊猫源致病大肠杆菌CCHTP全基因组测序及耐药和毒力基因分析
邓雯文1,李才武1,赵思越2,李仁贵1,何永果1,吴代福1,杨盛智2,黄炎1,张和民1,邹立扣2
1. 中国大熊猫保护研究中心,大熊猫国家公园珍稀动物保护生物学国家林业和草原局重点实验室,都江堰 611830 2. 四川农业大学资源学院,成都 611130
致病性大肠杆菌是引起动物泌尿系统感染的重要病原菌,本研究对泌尿生殖道感染出现潜血的大熊猫尿液中分离的一株致病性大肠杆菌(CCHTP)进行全基因组测序,检测其中耐药基因和毒力因子的情况,同时对基因岛上耐药和毒力基因及其基因环境进行研究。研究发现,大肠杆菌CCHTP中存在多种类型的耐药基因,其中外排泵系统基因数量最多,包括A、E和N等介导多重耐药外排泵的基因。此外,该菌还携带166种毒力因子及563个相关毒力基因,其中属于黏附与侵袭类的毒力因子及相关基因数量最多。对19个基因岛分析发现,基因岛GIs011和GIs017中各有一段包含耐药和毒力基因的序列,两侧与可移动遗传元件(转座酶和插入序列)相连,这些结构可能介导耐药及毒力基因水平转移。本研究通过全基因组测序分析了大熊猫源致病性大肠杆菌中存在的耐药及毒力基因情况,对大熊猫相关疾病的科学治疗、合理用药有重要意义。
大熊猫;大肠杆菌;全基因组测序;耐药基因;毒力因子
大肠杆菌()通常存在于人和动物的胃肠道,属于条件性致病菌,在宿主健康的情况下,可起到保护宿主、防止病原菌定植的作用,但在特定情况下会产生致病性[1]。此外,致病性大肠杆菌还可在胃肠道以外,如尿道、血液中生存并引起严重的疾病,它们具有广泛的宿主谱,其防治仍是当今世界性的难题,对大熊猫感染性疾病的发生有重要影响[2,3]。
目前,用于治疗大熊猫细菌感染的药物主要为抗生素,抗生素通过直接杀死或抑制病原菌的生长来达到治疗效果,这种作用机理使细菌具有选择压力,并催生病原菌耐药性的产生[4]。同时,由于耐药基因的水平转移特性使耐药性可在不同细菌种属间进行传播,加速了耐药水平的提高。研究表明大熊猫源大肠杆菌已对不同大类抗生素,如四环素类、磺胺类和β-内酰胺类抗生素呈现出不同程度耐药[5]。此外,毒力因子是病原菌具有的能够使其在宿主环境中定植、繁殖和致病的基因产物[6]。大肠杆菌的致病性是由多种毒力相关因子共同决定,这些毒力因子相互协调,直接或间接参与与致病过程,进而引发宿主有害炎症反应。近年来随着基因组学的飞速发展,可从分子水平对毒力因子进行研究,揭示病原菌致病机理[7]。
目前关于大熊猫源大肠杆菌的研究主要集中在对大熊猫肠道内大肠杆菌的分离鉴定、耐药性检测等[3,5,8],但有关大熊猫肠外大肠杆菌相关的研究报道还较少。本实验通过对1株从泌尿生殖道感染出现潜血的大熊猫尿液中分离的大肠杆菌进行全基因测序,研究耐药及毒力因子情况,分析耐药及毒力相关基因的环境,可对大肠杆菌致病机理研究提供理论基础,为合理采用抗生素治疗大熊猫大肠杆菌感染提供依据。
1 材料与方法
1.1 菌株来源
2017年5月从中国大熊猫保护研究中心一只泌尿生殖道感染出现潜血的大熊猫尿液中分离得到的大肠杆菌。
1.2 菌株纯化鉴定
大肠杆菌使用EMB培养基和TSA划线纯化,将纯化菌株腹腔注射小白鼠进行致病性实验[9],确认后按文献[1]的方法进行形态学和16S rDNA测序再次鉴定确认,鉴定结果为大肠杆菌(CCHTP)。挑取单菌落,使用TSB培养基培养18~20 h,取1.8 mL过夜孵育的菌液到一个2 mL无菌离心管中,离心后吸掉上清培养基,获取大肠杆菌沉淀用于后续DNA提取。
1.3 抗生素药敏试验
根据美国临床和实验室标准协会(Clinical and Laboratory Standards Institute, CLSL) 2019年发布的《抗菌药物敏感性试验执行标准》[10],采用K-B药敏纸片法,选择15种抗生素测定菌株的敏感性。利用无菌生理盐水稀释菌落制备菌悬浮液至0.5麦氏浓度,用无菌棉签蘸取菌液涂布于MHA琼脂平板上,镊子灭菌后分别夹取药敏纸片,按一定间隔贴在平板的不同区域,37℃恒温培养16~18 h。按CLSI标准判定耐药性。选择ATCC®25922,ATCC®35218作为质控菌株。
1.4 DNA提取
根据UltraClean®Microbial DNA Isolation Kit试剂盒手册,提取细菌DNA。取60 ng DNA样品1.0%琼脂糖凝胶电泳,在80V电压电泳30 min,检测DNA浓度。
1.5 全基因组测序
将DNA送往北京诺禾致源生物信息科技有限公司(Novogene)进行3代Pacbio平台测序。经电泳检测合格的DNA样品用Covaris g-TUBE打断成构建文库所需大小的目的片段,经DNA损伤修复及末端修复后,使用DNA黏合酶将发卡型接头连接在DNA片段两端,使用AMpure PB磁珠对DNA片段进行纯化选择,构建SMRT Bell文库。纯化后的片段经buffer回溶后,使用BluePipin片段筛选特定大小的片段,并使用AMpure PB磁珠对DNA片段进行纯化。构建好的文库经Qubit浓度定量,并利用Agilent 2100检测插入片段大小,随后用PacBio平台进行测序。
1.6 数据处理
对原始数据进行过滤处理,得到有效数据(clean data)。使用SMRT Link v5.0.1软件对reads进行组装[11,12],得到初步组装结果,把reads比对到组装序列上,统计测序深度的分布情况。将得到的初步组装结果进行比对分析,并将染色体序列组装成为一个环状基因组,即最终的0 gap完成图序列。使用GeneMarkS软件(http://topaz.gatech.edu/)进行细菌的编码基因预测基因。注释使用Blastn数据库(https:// blast.ncbi.nlm.nih.gov/Blast.cgi)以及RAST[13]。采用毒力因子数据库(Virulence Factors of Pathogenic Bacteria, VFDB)和抗生素耐药基因数据库(the Comprehensive Antibiotic Research Database, CARD)[14]对菌株的基因组中所包含的毒力基因和耐药基因进行比对,使用IslandPath-DIOMB软件预测基因岛。
2 结果与分析
2.1 药敏实验结果
药敏实验结果显示,获得的大肠杆菌CCHTP对阿莫西林、氨苄西林、阿莫西林/克拉维酸、头孢克洛、头孢曲松、头孢噻肟、庆大霉素、红霉素、阿奇霉素、氧氟沙星、诺氟沙星、恩诺沙星和四环素耐药,对氨曲南和卡那霉素敏感(附表1)。
2.2 大肠杆菌CCHTP全基因组测序结果
大肠杆菌CCHTP经PacBio测序平台进行全基因组测序后,通过对所测序列进行组装,最终获得1个contig,组装的contigs总长度为5120 663 bp,N50为5120 663 bp。通过组装获得一个长度为5106 047 bp的环状染色体序列(图1),鸟嘌呤–胞嘧啶(GC)含量为50.49%对基因组编码基因预测,注释得到的基因总数为4896,注释得到的基因总长度为4470 000 bp。对基因组非编码RNA (ncRNA)预测,得到61个sRNA、22个rRNA (包括5S、16S、23S)和89个tRNA。
经预测,共有4431个基因具有COG功能分类,其中信息储存和处理基因共745个,细胞过程和信号转导基因共1194个,代谢类基因共1945个,功能未知基因547个。菌株共有13 862个基因在GO数据库3大分支数据库中被注释到。共有43种功能注释结果,其中细胞学组件类有12个分支,共3360个注释结果,生物学途径类有23个分支,共7432基因注释结果,分子功能类存在10个分支共4107个相关注释结果。KEGG分析显示,共1544 个基因富集在 192条代谢通路中,其中涉及基因最多的通路主要有ABC转运蛋白代谢通路(ko02010) (200个基因)、双组分调节系统代谢通(ko02060) (142个基因)和嘌呤代谢通路(ko00230) (83个基因)。
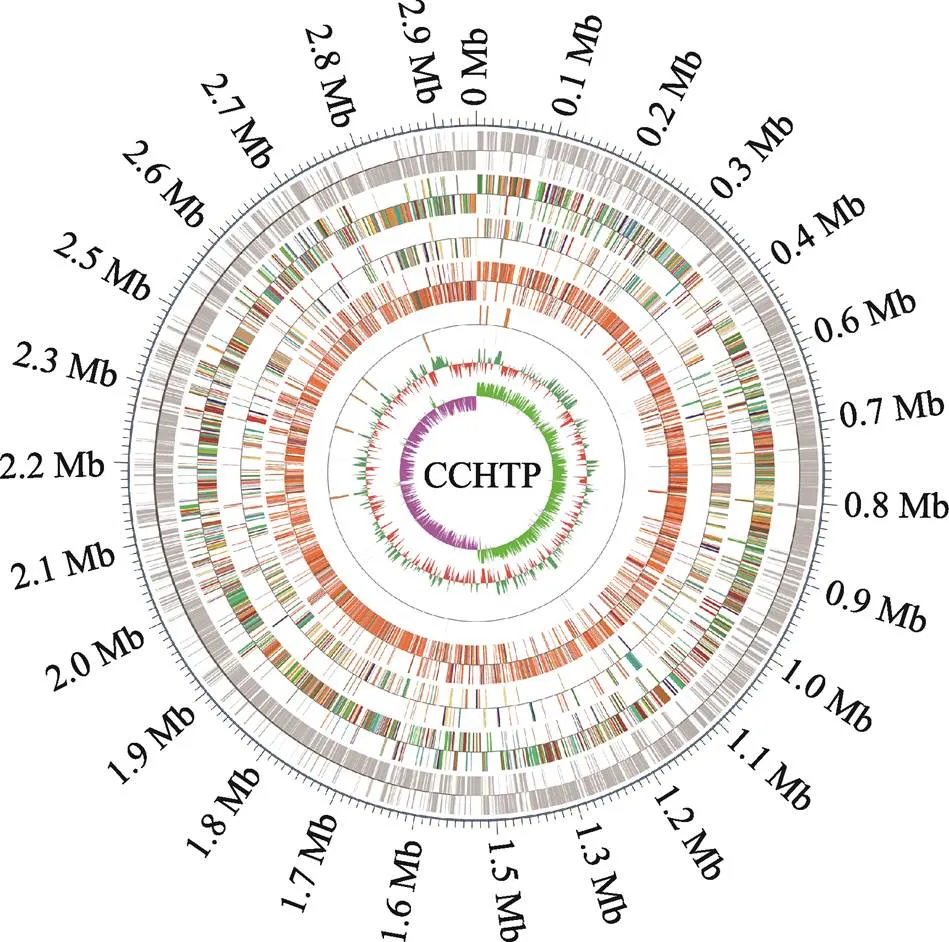
图1 大肠杆菌CCHTP基因组图谱
从外圈至内圈:1、2圈为编码基因;3、4圈为eggNOG;4、5圈为KEGG;7、8圈为GO;9、10圈为ncRNA;11、12圈为基因组GC含量;13、14圈为基因组GC skew值分布。
2.3 大肠杆菌CCHTP中抗生素耐药基因分析
通过对染色体基因序列进行比对,发现大肠杆菌携带了多种类型的抗生素耐药基因,包括14大类161个抗生素耐药基因。从表1中可知,外排泵系统基因数量最多,主要包括B (13个)、L (6个)、A (6个)、S (5个)等115个耐药基因。其次为介导糖肽类抗生素(万古霉素)耐药的TG/ TC/RI等10个耐药基因、介导多肽类抗生素(多粘菌素)耐药的-3、F/E/C等9个耐药基因和介导四环素类抗生素(四环素)耐药的tetT/B(60)/A(48)/ 34。此外,少量介导喹诺酮类、磺胺类和β-内酰胺类等抗生素耐药的基因也在基因组中识别到。
2.4 大肠杆菌CCHTP中毒力因子分析
经VFDB数据库比对,发现大肠杆菌CCHTP共携带了166种毒力因子及563个相关毒力基因。按毒力因子大类(黏附与侵袭、分泌系统、铁转运和细菌毒素)来分析,发现属于黏附与侵袭类的毒力因子及相关基因数量最多,共有47种毒力因子及239个相关毒力基因,毒力基因数量前15的黏附与侵袭类毒力因子如图2所示,主要黏附因子包括5种菌毛类:Type I fimbriae (Ⅰ型菌毛)、Type Ⅳ pili (Ⅳ型菌毛)、Pix pilus (Pix菌毛)、Pap pilus (盂肾炎相关菌毛/P菌毛)和common pilus (大肠杆菌共生菌毛)和3种鞭毛类:Peritrichous flagella (周生鞭毛)、S fimbriae (S鞭毛)和P fimbriae (P鞭毛)。研究发现Peritrichous flagella相关毒力基因数量最多,包括Z/Y/T/S、A/B/C/D、N/M/L等51个毒力基因,其次为LOS (脂寡糖)基因族中的F/E/D、K/H/ D/C等25个基因,Capsule (荚膜)基因族中的A、F、A等15个基因和TypeⅠ fimbriae基因族中的A/B/C等12个基因。

表1 大肠杆菌CCHTP中耐药基因分析结果
分泌系统共发现16种毒力因子及76个相关毒力基因(图3)。按照分泌系统类型来看,共识别到5类,属于Ⅱ型分泌系统的毒力因子类型最多,包括gspT2SSoutpulGspA和exe,其次为Ⅵ型分泌系统,包括ACE T6SSSCI-I T6SSCTS2和T6SS。其余3类分泌系统各识别到两种毒力因子,分别为Trehalose-recycling ABC transporter和ABC transporter (Ⅰ型分泌系统)、T3SS和IcsP (SopA) (Ⅲ型分泌系统)、CTS2和T6SS (Ⅵ型分泌系统)。与ACE T6SS相关的毒力基因数量最多,主要为假定蛋白编码基因17/18/22/23/26等19个基因,其次为SCI-I T6SS基因族中的3402、2814和82_ 443等17个基因。
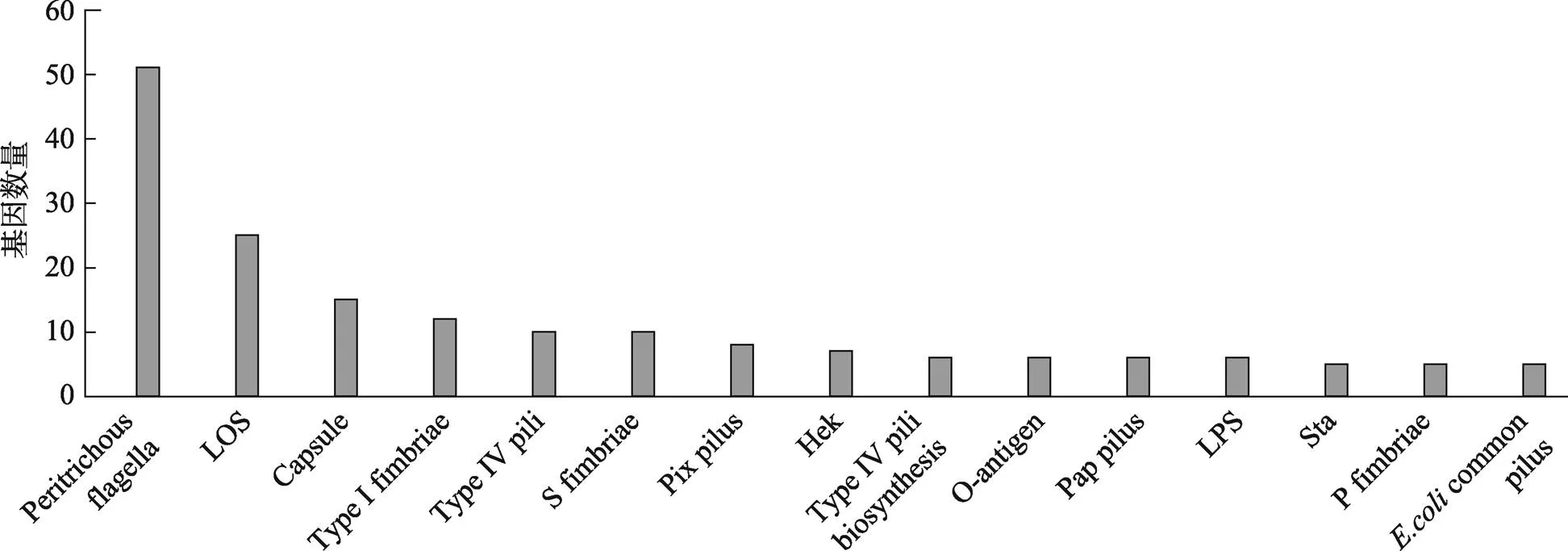
图2 黏附与侵袭类主要毒力因子
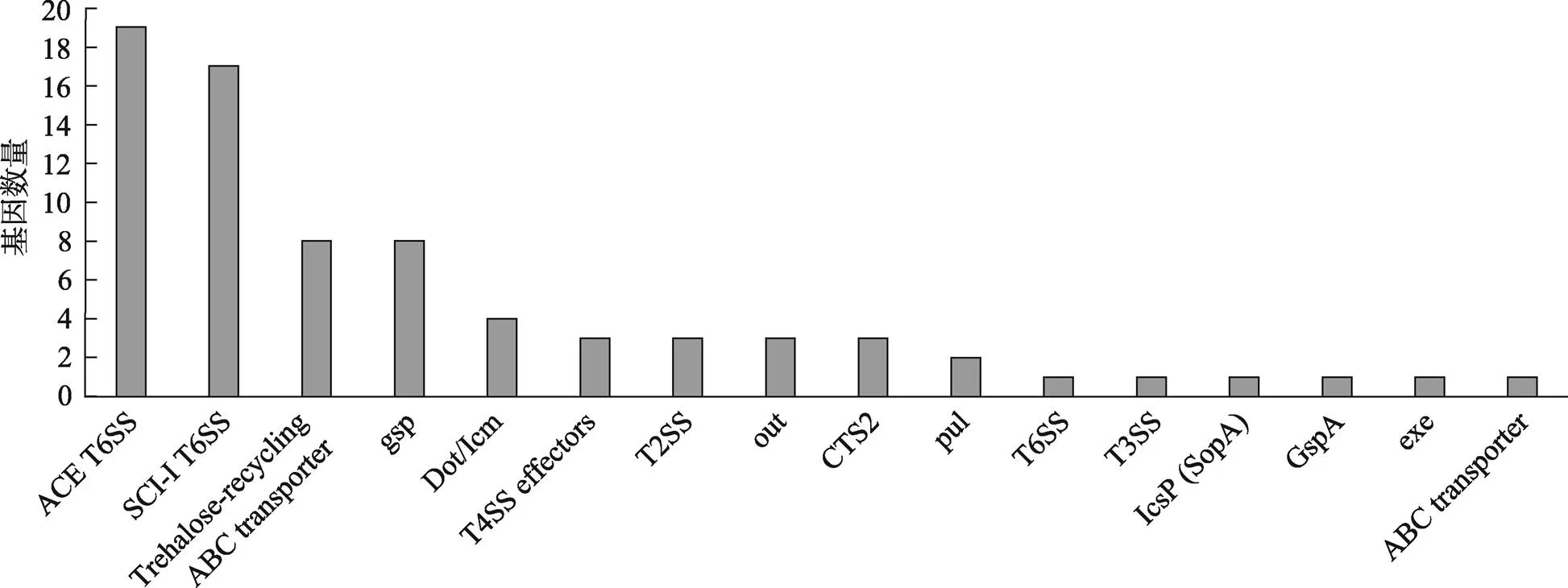
图3 分泌系统类主要毒力因子
本研究共识别12种毒力因子及59个相关毒力基因属于铁转运系统。如图4所示,与毒力因子Enterobactin (肠细菌素)和血红素合成(Heme biosynthesis)相关的基因数量最多。其次为Yersiniabactin siderophore (耶尔森菌素合成)、Yersiniabactin (耶尔森菌素)及Salmochelin (沙门菌素)。
如图5,通过比对得到细菌毒素类毒力因子11种,毒力基因36个。毒力因子主要包括5种溶血素:Alpha-hemolysin (α溶血素)、cytolysin A (溶血素A)、Hemolysin, HlyA (HlyA溶血素)、Hemolysin Ⅲ (Ⅲ型溶血素)和Beta-hemolysin (β溶血素)。研究发现Colibactin (聚酮肽基因毒素)相关毒力基因数量最多, 包括R/Q/P/O/N等19个基因。
2.5 大肠杆菌E. coli CCHTP中耐药和毒力基因环境分析
在染色体序列共发现19个基因岛,总长在5187~74 031 bp之间,其中发现两个基因岛GIs011和GIs017上各有一段含可移动遗传元件(插入序列)、毒力因子和耐药基因的序列,基因环境绘图如图6所示,在基因岛GIs011上发现一段序列包含外排泵基因调控因子(R)和四环素类耐药基因(A(60)),F1C菌毛(YH)和S菌毛(G/F/E/D/A/B/C) 相关基因,在这些基因两侧存在可移动遗传元件IS、转座酶基因及IS。GIs017上也发现在一段包含外排泵基因、α-溶血素基因A/B/C/D及细菌毒素CNF-1,两侧分别与转座酶基因、插入序列IS以及IS相连,大肠杆菌中的耐药或毒力基因可由可移动遗传元件介导传播。

图4 铁转运主要毒力因子

图5 细菌毒素主要毒力因子

图6 抗生素耐药基因和毒力基因环境
此外,对多肽类耐药基因-3及其侧翼环境进行分析(图7),发现基因-3一侧与假定抗性蛋白(putative resistance protein, PRP),并具有假定蛋白(hypothetical protein, HP)G。另一侧与假定蛋白相连,并发现Capsule类毒力基因F与ABC转运器和假定蛋白相连,另外在基因-3周围序列中还发现chu类毒力基因A和T以及外排泵基因L、X、W和E。

图7 mcr-3基因环境
PRP: putative resistance protein,假定抗性蛋白;HP: hypothetical protein,假定蛋白。
3 讨论
许多已知的病原菌都能引起尿路感染,如白色念珠菌()、肺炎克雷伯氏菌()、铜绿假单胞菌()、奇异变形杆菌()、金黄色葡萄球菌()和大肠杆菌等[15,16]。然而,目前对大熊猫泌尿生殖道感染的研究较少,仅发现肺炎克雷伯氏菌可引起大熊猫的泌尿生殖道血尿症出现[17]。本研究从一只泌尿生殖道感染出现潜血的大熊猫尿液中分离出大肠杆菌CCHTP。通过全基因组测序,发现该菌携带多种类型的抗生素耐药基因,并以外排泵基因为主,表明外排泵系统可能为大肠杆菌CCHTP产生耐药性的主要因素。此外,本研究还发现A、E、K和M等多重耐药外排泵系统基因。目前已有大量研究表明多重耐药外排泵能够介导对抗生素、消毒剂、洗涤剂和染料等有毒化合物的内在抗性[18],阻止药物在细菌体内积聚,是细菌多重耐药性产生的主要机制之一[19]。其中,A外排泵能够外排环丙沙星,卡那霉素、新霉素及季铵盐类消毒剂等[20,21],K基因可表达细菌对诺氟沙星、阿霉素及吖啶黄的外排,E编码的外排泵能外排四环素、红霉素、结晶紫及染色剂溴化乙锭[22]。此外,研究检测到四环素类、磺胺类和β-内酰胺类耐药基因,这与部分学者在大熊猫粪便源大肠杆菌检测到耐药基因结果相似[5,23]。四环素类抗生素广泛用于动物疾病治疗中,目前已发现40多种不同类型的四环素耐药基因,主要介导3种耐药机制,分别为外排泵机制、核糖体保护机制和酶降解机制[24],其中基因A和B可编码主要易化子超家族(MFS)外排泵蛋白将四环素药物泵出细胞外,降低细胞内药物浓度,四环耐药基因T通过编码核糖体保护机制,使耐药细菌可以产生核糖体保护蛋白与核糖体结合引起构型改变,从而弱化了对二甲胺四环素和强力霉素的抑制作用,导致耐药性的产生[25]。研究还发现多粘菌素耐药基因3,这种新型基因在2017年被Yin等[26]首次发现于大肠杆菌的IncHI2质粒中,其在大肠杆菌中的表达可有效阻止多粘菌素所引发的活性氧形成,并一定程度地对宿主细菌造成一定的代谢压力。研究表明基因-3的氨基酸序列与前期发现的多粘菌素基因-1和2差异较大,仅有32.5%和31.7%的一致性[27],具有多种亚型(如-3.3、-3.5和-3.7),宿主范围广,主要发现于大肠杆菌、肺炎克雷伯氏菌和鼠伤寒沙门氏菌中[28]。基因-3可由染色体和质粒介导,通过质粒、插入序列、转座子等方式介导传播[27,29]。Ling等[29]对气单胞菌中有染色体介导的-3.3基因环境进行分析,与本实验中-3基因环境对比发现具有部分相同的序列结构,基因两侧均有假定蛋白,且其中一侧与假定蛋白相连。但仍存在差异,表现为基因-3.3周围序列中发现插入序列遗传标记IS。此外,-3基因可与其他耐药基因共存[28,30,31],Liu等[27]发现由-3位于IncP质粒中的TnAs3转座子上,并与IncHI2质粒携带的基因-1和IncX3质粒携带的基因NDM-5共存于一株大肠杆菌中,引起多重耐药,进一步增强宿主细菌的耐药性。
近年来随着人们对于各种毒力因子的逐步了解,一些毒力因子的致病机理也逐渐被揭示出来。通过与VFDB数据库比对,发现大肠杆菌CCHTP携带了大量毒力因子及相关毒力基因。例如属于黏附与侵袭类毒力因子的Ⅰ型菌毛,其普遍存在于大肠杆菌中,主要在大肠杆菌致病的第一阶段起作用[32,33]。Vizcarra等[33]研究显示Ⅰ型菌毛还能产生屏蔽效应增强细胞内细菌对抗生素的抵抗能力,造成慢性感染发生,表明Ⅰ型菌毛对细菌抗生素耐药性的产生有一定影响。研究检测到了A/B/C/D/H等12个编码Ⅰ型菌毛的基因,其中C和D主要在协调菌毛装配及细菌致病过程中起作用,H介导 粘附作用[32]。此外,Ⅰ型菌毛和P菌毛都能介导大肠杆菌黏附于上尿道,也是尿道感染发生的重要因素[33,34]。此外,细菌毒素类包含多种溶血素,其中α溶血素毒力基因A具有广谱杀细胞作用,可作用于红细胞、肾脏上皮细胞和免疫细胞,诱导细胞凋亡[35]。Reijer等[36]研究表明溶血素在金黄色葡萄球菌生物膜形成等致病过程中也起着重要作用,增加了金葡菌的危害性。编码毒素Colibactin相关毒力的基因数量最多,该毒素可导致慢性有丝分裂、染色体畸变和基因突变频率增加,能够引发肿瘤[37]。本研究通过对基因岛分析发现,大量抗生素耐药及毒力基因两侧存在插入序列。研究表明插入序列是染色体特殊组成部分,可携带耐药基因转移,同时插入序列可通过自身携带的转座子提高耐药基因表达量从而提高细菌耐药程度[38,39]。因此,研究结果提示大肠杆菌中耐药或毒力基因可能通过可移动遗传元件介导传播。
分泌系统和铁转运系统虽未直接涉及致病过程,但对细菌致病性的产生也至关重要。目前在革兰氏阴性菌中已经发现并命名9种不同类型的分泌系统(Ⅰ~Ⅸ型),致病菌借助分泌系统将特异蛋白直接注入宿主细胞内,可导致细菌的定殖和感染[40]。在本研究共识别到5类(Ⅰ~Ⅳ型),其中Ⅲ型分泌系统广泛存在于革兰氏阴性致病菌,如耶尔森氏菌属、沙门氏菌属、大肠杆菌、副溶血性弧菌等中[7,41]。沙门菌素和肠杆菌素是肠杆菌科病原菌中常见的铁载体,其中肠杆菌素主要通过于血浆转铁蛋白中的铁结合来促进细菌生长而引起感染,与其他已知的铁载体蛋白相比具有最高的铁亲和力[42]。
综上所述,本研究通过全基因组测序技术首次对泌尿生殖道感染出现潜血的大熊猫尿液中分离的大肠杆菌进行检测,主要研究了其中毒力因子及耐药基因情况,可较为全面地预测大肠杆菌中耐药基因、耐药谱、毒力因子及相关毒力基因等重要致病特性。研究结果可为圈养大熊猫临床治疗和合理用药提供指导,为后续开展圈养大熊猫源病原菌中毒力因子和耐药基因研究提供依据。
附录:
附表1详见文章电子版www.chinagene.cn。
附表1 大肠杆菌CCHTP耐药情况

Supplementary Table 1 Antibiotic resistance of E. coli CCHTP
[1] Li B, Li XL, Guo LJ, Long M, Guo F, He XM, Luo Y, Zou LK. Isolation and drug resistance analysis onin intestines from giant pandas., 2012, 25(3): 1109–1113.李蓓, 李旭林, 郭丽娟, 龙梅, 郭放, 何雪梅, 罗燕, 邹立扣. 大熊猫肠道大肠杆菌的分离、鉴定及其耐药性分析. 西南农业学报, 2012, 25(3): 1109–1113.
[2] Johnson TJ, Wannemuehler Y, Kariyawasam S, Johnson JR, Logue CM, Nolan LK. Prevalence of avian-pathogenicstrain O1 genomic islands among extraintestinal and commensalisolates., 2012, 194(11): 2846–2853.
[3] Guo LJ, He XM, Deng WW, Wu GY, Long M, Li B, Zhang AY, Wang HN, Zou LK. Disinfectant resistance ofandisolated from giant panda., 2014, 33(6): 801–807.郭莉娟, 何雪梅, 邓雯文, 吴国艳, 龙梅, 李蓓, 张安云, 王红宁, 邹立扣. 大熊猫源大肠杆菌及肺炎克雷伯氏菌对消毒剂耐药性研究. 四川动物, 2014, 33(6): 801–807.
[4] Chen FF, Di HX, Lan LF. Small molecules targetingvirulence., 2013, 58(36): 3743–3572.陈菲菲, 狄红霞, 蓝乐夫. 金黄色葡萄球菌重要毒力因子的功能及其抑制剂研究进展. 科学通报, 2013, 58(36): 3743–3572.
[5] Guo LJ, Long M, Huang Y, Wu GY, Deng WW, Yang X, Li B, Meng Y, Cheng L, Fan LQ, Zhang HM, Zou LK. Antimicrobial and disinfectant resistance ofisolated from giant pandas., 2015, 119(1): 55–64.
[6] Webb SA, Kahler CM. Bench-to-bedside review: Bacterial virulence and subversion of host defences., 2008, 12(6): 234.
[7] Yin L, Qi KZ, Song XJ, Tu J. Type III secretion system 2 pathogenicity islands of., 2017, 44(12): 3031–3037.尹磊, 祁克宗, 宋祥军, 涂健. 大肠杆菌Ⅲ型分泌系统2毒力岛研究进展. 微生物学报, 2017, 44(12): 3031–3037.
[8] Zou WC, Li CW, Yang X, Wang YX, Cheng GY, Zeng JX, Zhang XZ, Chen YP, Cai R, Huang QR, Feng L, Wang HN, Li DS, Zhang GQ, Chen YX, Zhang ZZ, Zhang HM. Frequency of antimicrobial resistance and integron gene cassettes inisolated from giant pandas () in China., 2018, 116: 173–179.
[9] Ma ZJ, Rui P, Lu CX, Yang CR, Liu XR, Wang QY, Liu YZ, Zhang JW. Isolation and identification of extraintestinal pathogenic, 2015, 31(2): 130–134.马增军, 芮萍, 逯春香, 杨彩然, 刘谢荣, 王秋悦, 刘曜综, 张建文. 猪源肠外致病性大肠杆菌的分离与鉴定. 中国人兽共患病学报, 2015, 31(2): 130–134.
[10] CLSI. 2019. Performance Standards for Antimicrobial Susceptibility Testing. 29th ed. CLSI supplement M100, Clinical and Laboratory Standards Institute, Wayne, PA.
[11] Li RQ, Zhu HM, Ruan J, Qian WB, Fang XD, Shi ZB, Li YR, Li ST, Shan G, Kristiansen K, Li SG, Yang HM, Wang J, Wang J. De novo assembly of human genomes with massively parallel short read sequencing., 2010, 20(2): 265–272.
[12] Li RQ, Li YR, Kristiansen K, Wang J. SOAP: short oligonucleotide alignment program., 2008, 24(5): 713–714.
[13] Brettin T, Davis JJ, Disz T, Edwards RA, Gerdes S, Olsen GJ, Olson R, Overbeek R, Parrello B, Pusch GD, Shukla M, Thomason JA, Stevens R, Vonstein V, Wattam AR, Xia F. RASTtk: a modular and extensible implementation of the RAST algorithm for building custom annotation pipelines and annotating batches of genomes., 2015, 5: 8365.
[14] Thomas M, Fenske GJ, Antony L, Ghimire S, Welsh R, Ramachandran A, Scaria J. Whole genome sequencing- based detection of antimicrobial resistance and virulence in non-typhoidalisolated from wildlife., 2017, 9: 66.
[15] Linhares I, Raposo T, Rodrigues A, Almeida A. Frequency and antimicrobial resistance patterns of bacteria implicatedin community urinary tract infections: a ten-year surveillance study (2000-2009),, 2013, 13: 19.
[16] Kulkarni R, Dhakal BK, Slechta ES, Kurtz Z, Mulvey MA, Thanassi DG. Roles of putative type II secretion and type IV pilus systems in the virulence of uropathogenic., 2009, 4(3): e4752.
[17] Wang CD, Lan JC, Luo L, Yang Z, Zhang ZR.-pathogen of urogenital hematuria of giant panda., 2006, 25(1): 83–85.王成东, 兰景超, 罗娌, 杨智, 张再蓉. 大熊猫感染性泌尿生殖道血尿症病原一肺炎克雷伯氏杆菌, 四川动物, 2006, 25(1): 83–85.
[18] Sulavik MC, Houseweart C, Cramer C, Jiwani N, Murgolo N, Greene J, DiDomenico B, Shaw KJ, Miller GH, Hare R, Shimer G. Antibiotic susceptibility profiles ofstrains lacking multidrug efflux pump genes., 2001, 45(4): 1126–1136.
[19] Xue Y, Zhang XY, Wang GX, Huan YP. Progress on active efflux system inconferring multidrug resistance., 2009, 31(6): 493–496.薛原, 张秀英, 王贵霞, 华育平. 大肠杆菌主动外排系统的研究进展. 中国预防兽医学报, 2009, 31(6): 493– 496.
[20] Zou LK, Meng JH, McDermott PF, Wang F, Yang QR, Cao GJ, Hoffmann M, Zhao SH, Presence of disinfectant resistance genes inisolated from retail meats in the USA., 2014, 69(10): 2644–2649
[21] Edgar R, Bibi E. MdfA, anmultidrug resistance protein with an extraordinarily broad spectrum of drug recognition., 1997, 179(7): 2274–2280.
[22] Ma C, Chang G. Structure of the multidrug resistance efflux transporterE from., 2004, 101(109): 2852–2857.
[23] Zou WC, Li CW, Yang X, Wang YX, Cheng GY, Zeng JX, Zhang XZ, Chen YP, Cai R, Huang QR, Feng L, Wang HN, Li DS, Zhang GQ, Chen YX, Zhang ZZ, Zhang HM. Frequency of antimicrobial resistance and integron gene cassettes inisolated from giant pandas () in China., 2018, 116: 173–179.
[24] Jurado-Rabadán S, de la Fuente R, Ruiz-Santa-Quiteria JA, Orden JA, de Vries LE, Agersø Y. Detection and linkage to mobile genetic elements of tetracycline resistance gene(M) inisolates from pigs., 2014, 10: 155–161.
[25] Nguyen F, Starosta AL, Arenz S, Sohmen D, Dönhöfer A, Wilson DN, Tetracycline antibiotics and resistance mechanisms., 2014, 395(5): 559–575.
[26] Yin WJ, Li H, Shen YB, Liu ZH, Wang SL, Shen ZQ, Zhang R, Walsh TR, Shen JZ, Wang Y. Novel plasmid- mediated colistin resistance gene-3 in, 2017, 8(3): e00543–17.
[27] Liu L, Feng Y, Zhang X, McNally A, Zong Z. New variant of mcr-3 in an extensively drug-resistantclinical isolate carrying-1 andNDM-5., 2017, 61(12): e01757–17.
[28] Xu YC, Zhong LL, Srinivas S, Sun J, Huang M, Paterson DL, Lei S, Lin JX, Li F, Tang ZC, Feng SY, Shen C, Tian GB, Feng YJ. Spread of-3 colistin resistance in China: An epidemiological, genomic and mechanistic study., 2018, 34: 139–157.
[29] Ling ZR, Yin WJ, Li H, Zhang QD, Wang XM, Wang Z, Ke YB, Wang Y, Shen JZ. Chromosome-mediated-3 variants infrom chicken meat., 2017, 61(11): e01272–17.
[30] Xiang R, Ye X, Tuo H, Zhang X, Zhang A, Lei C, Yang Y, Wang H, Co-occurrence of-3 andNDM-5 genes in multidrug-resistantST709 from a commercial chicken farm in China., 2018, 52(4): 519–520.
[31] Hernández M, Iglesias MR, Rodríguez-Lázaro D, Gallardo A, Quijada NM, Miguela-Villoldo P, Campos MJ, Píriz S, López-Orozco G, de Frutos C, Sáez JL, Ugarte-Ruiz M, Domínguez L, Quesada A. Co-occurrence of colistin- resistance genes-1 and-3 among multidrug- resistantisolated from cattle, Spain, September 2015., 2017, 22(31): 1–5.
[32] Liu HY, Wang JW, Wang J, Li YQ, Huang XM, Qu ZN. Research progress of avianvirulence factors., 2013, 30(10): 25–28, 29.刘红玉, 王君玮, 王娟, 李玉清, 黄秀梅, 曲志娜. 禽大肠杆菌毒力因子的研究进展. 中国动物检疫, 2013, 30(10): 25–28, 29.
[33] Vizcarra IA, Hosseini V, Kollmannsberger P, Meier S, Weber SS, Arnoldini M, Ackermann M, Vogel V. How type 1 fimbriae helpto evade extracellular antibiotics., 2016, 6: 18109.
[34] Guo X, Kong LC, Liu SM, Ma HX. Progress on virulence factors in extraintestinal pathogenic.,2015, 37(5): 405–408.郭霞, 孔令聪, 刘树明, 马红霞. 动物源肠外致病性大肠杆菌毒力因子研究进展. 中国预防兽医学报, 2015, 37(5): 405–408.
[35] Landraud L, Gibert M, Popoff M, Boquet P, Gauthier M. Expression ofbyJ96 involves a large upstream DNA region including theCABD operon, and is regulated by the RfaH protein., 2003, 47(6): 1653–1667.
[36] den Reijer PM, Haisma EM, Lemmens-den Toom NA, Willemse J, Koning RI, Demmers JAA, Dekkers DHW, Rijkers E, Ghalbzouri AE, Nibbering PH, van Wamel W. Detection of alpha-toxin and other virulence factors in biofilms ofon polystyrene and a human epidermal model., 2016, 11(1): e0145722.
[37] Yang YT, Qu CM, Li LY. Effect and mechanism of intestinal bacteria flora on colorectal cancer., 2016, 21(5): 315–318.杨永涛, 屈昌民, 李连勇. 肠道菌群在结直肠癌中的作用及其机制. 胃肠病学, 2016, 21(5): 315–318.
[38] Mojica FJM, Díez-Villaseñor C, García-Martínez J, Soria E. Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements., 2005, 60(2): 174–182.
[39] Wang HT, Liu NN, Ling BD. The effect of the insert sequenceon the expression of carbapenemase gene in multidrug-resistant, 2014, 39(3): 229–233.王同慧, 刘男男, 凌保东. 插入序列对多重耐药鲍曼不动杆菌碳青霉烯酶基因表达的影响. 中国抗生素杂志, 2014, 39(3): 229–233.
[40] Mu LL, Niu B, Zhao Y. Role ofsecretion system in pathogenicity., 2019, 59(4): 621–631.穆丽丽, 牛犇, 赵勇. 副溶血性弧菌分泌系统在致病力中作用的研究进展. 微生物学报, 2019, 59(4): 621–631.
[41] Wang L, Wang YM, Yue JJ, Liang L, Huang PT. Progress in type III secretion system., 2004, 44(6): 840–844.汪莉, 王玉民, 岳俊杰, 梁龙, 黄培堂. Ⅲ型分泌系统分子伴侣研究进展. 微生物学报, 2004, 44(6): 840–844.
[42] Chen Y, Liu JL, Qu HP. Mechanistic research on the role of siderophores in enhancing the virulence of, 2016, 16(6): 804–807.陈杨, 刘嘉林, 瞿洪平. 铁载体对肺炎克雷伯菌毒力增强的机制研究. 中国感染与化疗杂志, 2016, 16(6): 804– 807.
Whole genome sequencing reveals the distribution of resistance and virulence genes of pathogenicCCHTP from giant panda
Wenwen Deng1, Caiwu Li1, Siyue Zhao2, Rengui Li1, Yongguo He1, Daifu Wu1, Shengzhi Yang2, Yan Huang1, Hemin Zhang1, Likou Zou2
Pathogenicis the most common pathogen causing urinary tract infection in animals. We investigated the antibiotic resistance and virulence genes of pathogenicCCHTP derived from urine with occult blood of the giant panda by whole genome sequencing. The flanking sequencing of resistance and virulence genes in genomic islands were also analyzed. Our results demonstrate thatCCHTP contains different families of antibiotic resistance genes, most of which are efflux pump relatedgenes, including multiple drug resistance efflux pump genesA,E, andN. A total of 166 virulence factors and 563 virulence genes were identified, and the most virulence factors and related genes are involved in host cell attachment and invasion processes. Furthermore, sequence analysis of 19 genomic islands revealed that antibiotic and virulence genes are associated with mobile genetic elements (transposon and insertion sequence) in GIs011 and GIs017. These structures can mediate horizontal transfer of antibiotic and virulence genes. Our work described the distribution of antibiotic resistance genes and virulence genes inCCHTP, which may provide an important guidance for treatment and rational drug use ofCCHTP infection in the giant panda.
giant panda;; whole genome sequencing; resistance gene; virulence gene
2019-09-17;
2019-11-25
中国大熊猫保护研究中心项目(编号:CCRCGP181918)和国家自然科学基金项目(编号:31400066)资助[Supported by the Project of China Conservation and Research Center for the Giant Panda (No. CCRCGP181918) and the National Natural Science Foundation of China (No. 31400066)]
邓雯文,硕士研究生,研究方向:大熊猫分子遗传学。E-mail: dwenwen130@163.com李才武,硕士研究生,高级工程师,研究方向:大熊猫临床兽医。E-mail: 83330019@qq.com邓雯文和李才武并列第一作者。
邹立扣,博士,教授,博士生导师,研究方向:微生物资源利用、细菌耐药性。E-mail: zoulikou@sicau.edu.cn
10.16288/j.yczz.19-243
2019/11/28 11:20:00
URI: http://kns.cnki.net/kcms/detail/11.1913.R.20191127.1143.004.html
(责任编委: 谢建平)
