牛源肺炎克雷伯菌菌毛类型鉴定及fimA、mrkA基因序列分析
2022-06-09聂青青单文杰
聂青青,张 璇,黄 攀,单文杰
(1.赣州职业技术学院,江西赣州 341000;2.垫江县市政服务中心,重庆 408300;3.巴南区人民政府莲花街道农业服务中心,重庆 401321;4.共青城市农业农村水利局,江西九江 332020)
肺炎克雷伯菌(Klebsiellapneumoniae,K.pneumoniae)属肠杆菌科(Enterobaeteriaceae)、克雷伯菌属(Klebsiella),为革兰氏阴性短杆菌,是1种人畜共患病病原,也是引起多种动物患病的条件性致病菌,可引起肺炎、败血症、化脓性感染、支气管炎、尿路感染及脑膜炎等[1-3]。现有研究表明,菌毛是肺炎克雷伯菌的1种主要的毒力因子[4-5]。在感染过程中,细菌通过菌毛接近宿主黏膜表面或者吸附于宿主细胞,进而使机体致病[6]。肺炎克雷伯菌的菌毛可分为Ⅰ型和Ⅲ型菌毛[7]。Ⅰ型菌毛属于甘露糖敏感性(mannose-sensitive hemagglutination,MSHA)菌毛,即甘露糖存在时会阻断菌毛与红细胞的凝集现象,主要附着于尿道上皮细胞;Ⅲ型菌毛具有甘露糖抵抗血凝特性(mannose-resistant hemagglutination,MRHA),又称为甘露糖抵抗血凝特性菌毛,主要附着于呼吸道上皮细胞[8]。上述两种菌毛的主要结构亚单位基因为fimA和mrkA[9]。Ⅰ型菌毛由一组fim(B、E、A、I、C、D、F、G、H)基因群所表达,fimA是负责表达Ⅰ型菌毛柄的结构基因;Ⅲ型菌毛由一组mrk(E、A、B、C、D、F)基因群所表达,mrkA是负责表达Ⅲ型菌毛柄的结构基因。
本研究对近年在重庆、四川部分肉牛场分离到的20株肺炎克雷伯菌进行了MSHA/MRHA试验,鉴定其菌毛类型,并对20株菌的fimA基因和mrkA基因进行了测序及氨基酸序列分析。研究旨在揭示川渝地区克雷伯菌菌毛类型与fimA基因和mrkA基因的变异特点,为深入研究其致病机制提供参考。
1 材料与方法
1.1 试验材料
通过棉拭子的方法采集重庆、四川部分肉牛场的牛上呼吸道黏液,分离细菌;通过生化试验、16S rRNA鉴定出20株肺炎克雷伯菌,分别编号为K.pn1~K.pn20,其中K.pn4、K.pn11、K.pn20分离自四川,其余分离自重庆。SPF豚鼠购自重庆市中药研究院。
1.2 试剂与培养基
D-甘露糖(上海国药集团化学试剂有限公司)、DNA回收试剂盒(广州飞扬生化有限公司)、PCR试剂盒(宝生物工程(大连)有限公司);小剂量DNA提取试剂盒、DL2000DNA Marker(生工生物工程(上海)有限公司);改良Minka液体培养基、2%豚鼠红细胞悬液等。
1.3 主要仪器
AR1140/C电子分析天平(上海奥豪斯公司)、LDZX-50KBS立式压力蒸汽灭菌器(上海申安医疗器械)、DYY-6C电泳仪(北京六一生物科技有限公司)、S10000PCR仪(Thermal Cycler)、TGL-16高速冷冻离心机等。
1.4 MSHA/MRHA试验
按照参考文献[10]进行。将各菌株分别经改良Minka液体培养基37℃静置培养48 h,同样培养条件下连续传代3次,即为菌毛化菌体。菌毛化菌体制成约50亿CFU/mL细菌悬液,每株菌分为不加D-甘露糖和加D-甘露糖(终浓度为0.5%)的两个样本,加入等量2%豚鼠红细胞悬液,设生理盐水空白对照;振荡后4℃保存2 h,观察血球凝集反应和D-甘露糖是否抑制凝集。将甘露糖抵抗血凝试验阳性的菌株,4℃条件下完全凝集,37℃静置30 min,检查凝集现象是否被解脱。凝集解脱后,重新混匀4℃保存1.5 h,观察是否再现凝集反应。能够凝集豚鼠红细胞的菌株可判定MSHA反应阳性;若37℃出现解脱,4℃又重现凝集反应,可判定为MRHA反应阳性。
1.5 fimA和mrkA基因的PCR扩增、测序
20株菌毛化菌体经麦康凯培养基培养后,核酸提取试剂盒提取其DNA,PCR扩增fimA和mrkA基因序列,引物序列见表1。

表1 引物序列Tab.1 Primer sequence
PCR反应体系(25μL):上游引物0.5μL、下游引物0.5μL、ddH2O 9.5μL、DNA模板2μL、2×mix DNA聚合酶预混液12.5μL。
PCR反应条件:94℃5 min;94℃30 s,56.5℃30 s,72℃40 s,30个循环;72℃10 min。
Ⅰ型菌毛菌株的PCR反应体系与Ⅲ型相同,但反应条件为:94℃5 min;94℃30 s,55℃30 s,72℃35 s,30个PCR循环;72℃10 min。
反应结束后取5.0μL PCR产物经1.5%琼脂糖凝胶电泳检测,观察结果。DNA回收试剂盒对目的片段进行回收与纯化,由上海生物工程有限公司测序。
1.6 序列分析
测序结果利用MEGA软件与GenBank中登录的fimA和mrkA基因序列进行同源性比较,绘制系统进化树。同时将基因翻译成氨基酸序列,进行氨基酸序列分析。
2 结果与分析
2.1肺炎克雷伯菌MSHA/MRHA试验结果(见图1)
由图1可知,将20株菌毛化肺炎克雷伯菌进行MSHA/MRHA试验后,结果显示,有5株菌株不能抵抗D-甘露糖的抑制作用,其余均能够抵抗D-甘露糖的抑制作用,且在37℃下均可被解脱,在4℃下重现完全凝集,符合MRHA阳性判定标准,阴性对照组无自凝现象。
上述结果表明,该20株肺炎克雷伯菌中产生Ⅰ型菌毛的有5株,为:K.pn16~K.pn20;产生Ⅲ型菌毛的有15株,为:K.pn1~K.pn15。
2.2 肺炎克雷伯菌基因的PCR鉴定(见图2、图3)
由图2、图3可知,20株供试菌株中,有5株菌针对Ⅰ型菌毛主要结构亚单位基因(fimA)序列的引物扩增出549 bp的目的片段;15株菌针对Ⅲ型菌毛的主要结构亚单位基因(mrkA)序列的引物扩增出609 bp的目的片段,与预期的大小相符,且PCR鉴定结果的肺炎克雷伯菌Ⅰ型菌毛菌株和Ⅲ型菌毛菌株与MSHA/MRHA试验结果相符。
2.3 序列同源性及系统进化树分析(见图4)
研究中的5株fimA基因和15株mrkA基因与GenBank中登录的fimA和mrkA基因序列进行同源性比较,并构建系统进化树。结果显示,fimA基因核苷酸序列的同源性为82.6%~98.9%,推导氨基酸序列同源性为90.9%~97.7%,mrkA基因核苷酸序列的同源性为80.7%~99.7%,推导氨基酸序列同源性84.1%~100%。5个fimA基因与代表株NZ_CP009461.1的核苷酸序列同源性为83.1%~99.0%,推导氨基酸序列同源性为90.3%~97.7%;15个mrkA基因与代表株AOGO01000023.1的核苷酸序列同源性80.7%~99.7%,推导氨基酸序列同源性为84.1%~100.0%。
由图4可知,分离株菌毛类型有Ⅰ型和Ⅲ型,Ⅰ型菌毛共5株,Ⅲ型菌毛菌株居多(15株)。
Ⅲ型菌毛菌株可分为4个分支,其中6个分离株(K.pn1~K.pn6)与AOGO01000023.1株同属一个分支。
2.4 重庆、四川部分肉牛场的肺炎克雷伯菌氨基酸突变位点比对分析(见表2、表3)
由表2可知,测序的5个fimA氨基酸序列(菌毛类型均为Ⅰ型)与代表株NZ_CP009461.1比较,可分为2个分支,K.pn20与K.pn19为同一支,均仅在95位氨基酸位点存在特异性变异。K.pn16、K.pn17和K.pn18存在12个氨基酸差异,K.pn16和K.pn17均在19、95、97、101、104、118、129、133、142、151、170位氨基酸位点存在特异性变异,K.pn18在19、74、95、97、118、129、151、170氨基酸位点存在特异性差异,并在74位出现特有的变异。
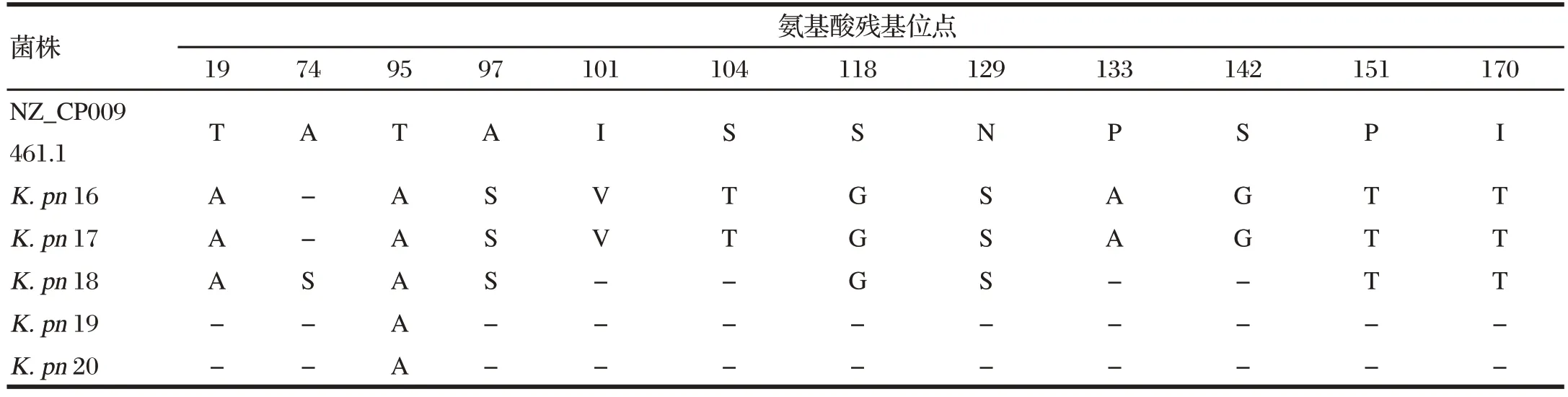
表2 重庆、四川部分肉牛场的肺炎克雷伯菌fimA氨基酸突变位点比对分析Tab.2 Comparative analysis of fimA amino acid mutation sites of Klebsiellapneumoniae in some beef cattle farms in Chongqing and Sichuan
由表3可知,测序的15个mrkA氨基酸序列(菌毛类型均为Ⅲ型)与代表株AOGO01000023.1比较,存在多个位点存在差异,分离株两分支间有32个氨基酸存在差异。其中K.pn3分离株在20位出现特有变异,K.pn12分离株在17位出现特有变异,K.pn10分离株在70、187位出现特有变异,K.pn11分离株在132、181、185位出现特有变异,K.pn15分离株在115、119、144、146、175位出现特有变异。
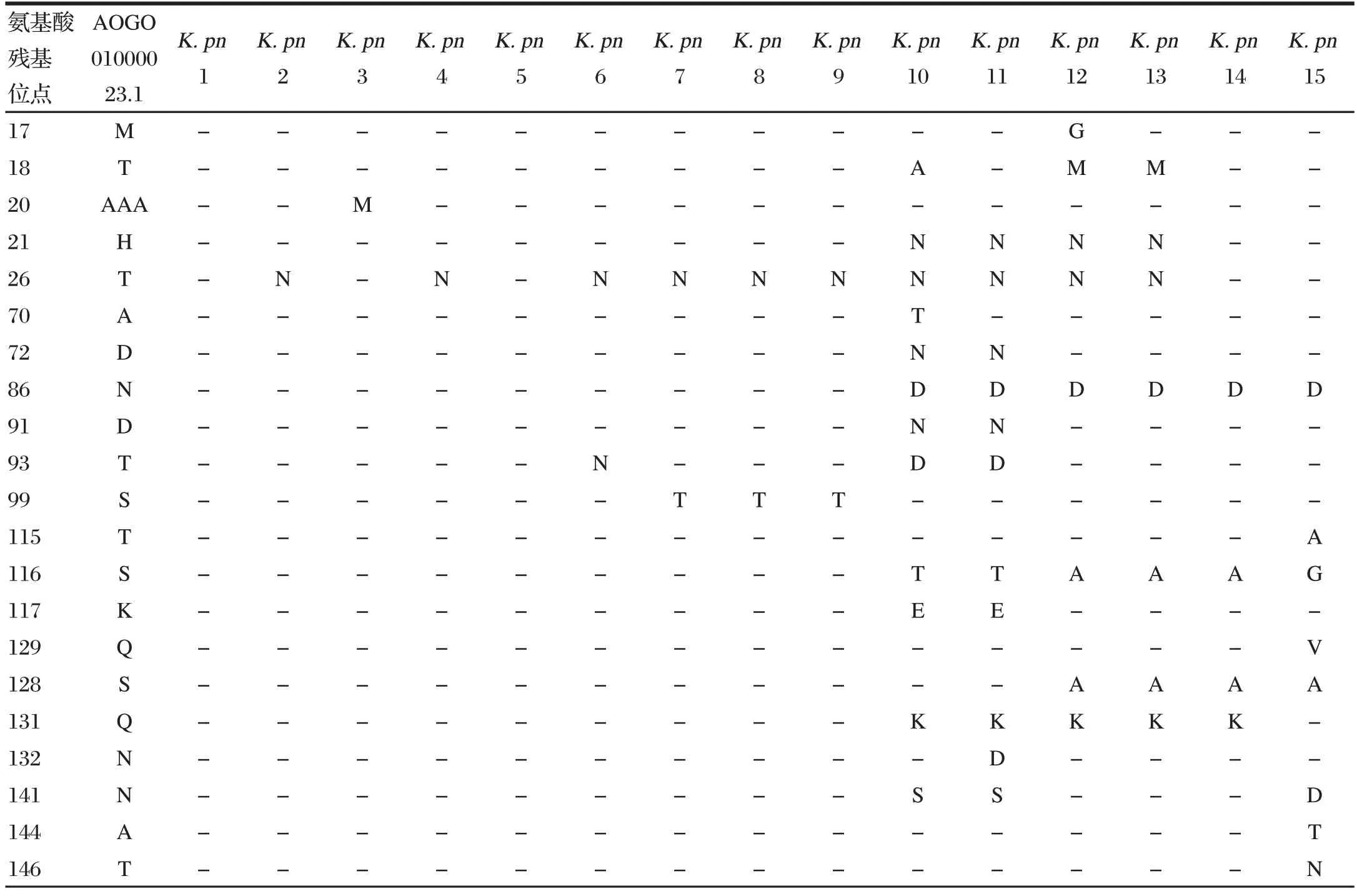
表3 重庆、四川部分肉牛场的肺炎克雷伯菌mrkA氨基酸突变位点比对分析Tab.3 Comparative analysis of mrkA amino acid mutation sites of Klebsiellapneumoniae in some beef cattle farms in Chongqing and Sichuan
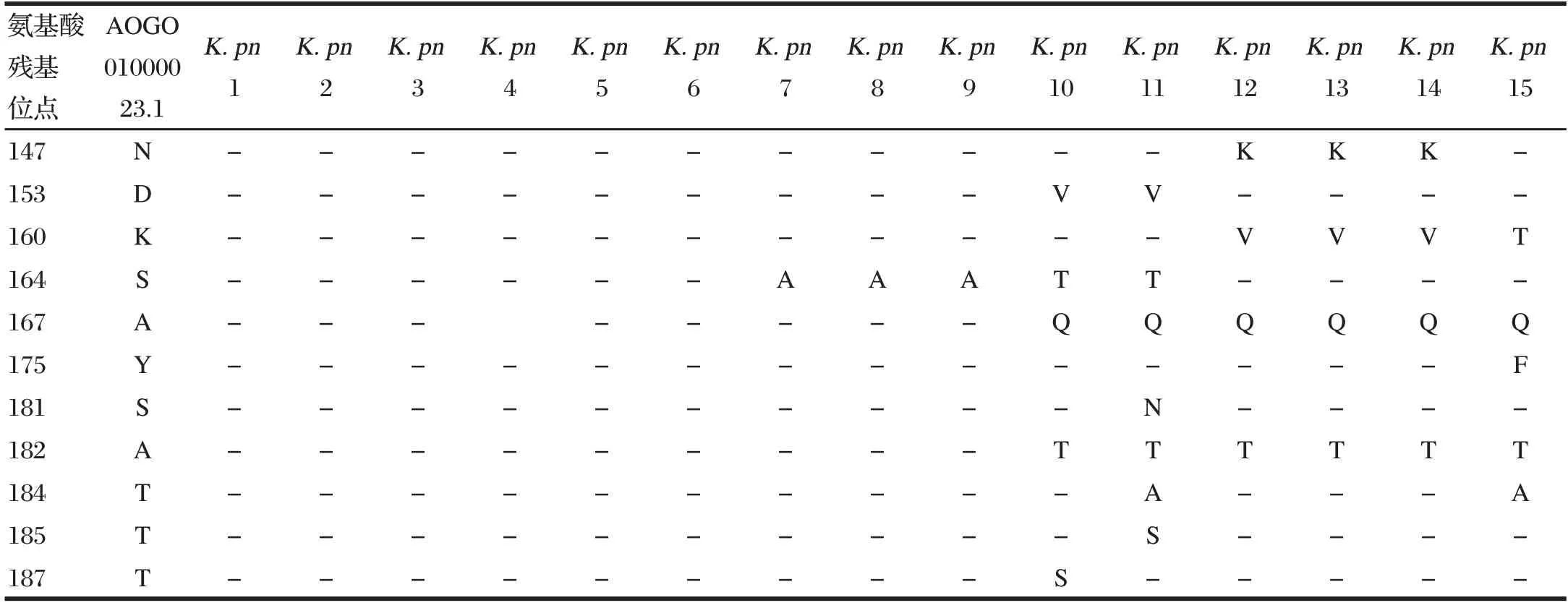
续表3 重庆、四川部分肉牛场的肺炎克雷伯菌mrkA氨基酸突变位点比对分析Tab.3(continue)Comparative analysis of mrkA amino acid mutation sites of Klebsiellapneumoniae in some beef cattle farms in Chongqing and Sichuan
3 讨论
本研究通过MSHA/MRHA试验和PCR方法,鉴定出近年重庆、四川部分肉牛场的20株肺炎克雷伯菌的菌毛类型。其中有5株为Ⅰ型菌毛,占25%;15株为Ⅲ型菌毛,占75%。来自四川的3株菌株中的1株是Ⅰ型菌毛,2株是Ⅲ型菌毛。分离自重庆的17株,其中4株是Ⅰ型菌毛,占23.5%;13株属于Ⅲ型菌毛,占76.5%。本研究结果表明,重庆、四川肉牛所感染的肺炎克雷伯菌主要产Ⅲ型菌毛,进行肺炎克雷伯菌感染的防控应该更重视Ⅲ型菌毛。
MSHA/MRHA试验法确定菌毛类型较烦琐、耗时。何礼洋等[11]、刘选梅等[12]报道,采用PCR方法可快速区分鉴别Ⅰ型、Ⅲ型菌毛肺炎克雷伯菌,与MSHA/MRHA试验相比更加敏感、快速、方便。本研究结果表明,运用PCR对菌毛进行分型,试验结果与MSHA/MRHA试验一致:MSHA/MRHA试验法鉴定出Ⅰ型菌毛的5株菌株均扩增出了约549 bp大小的特异片段,而MSHA/MRHA试验法鉴定出Ⅲ型菌毛的15株菌株均扩增出了约609 bp大小的特异片段。PCR法鉴定菌株菌毛类型可避免MSHA/MRHA试验的局限性,且更加快速、准确、方便。
进化树是描述生物体形成或进化顺序的拓扑树结构,一般由一系列节点和分支组成,节点间的连线代表物种之间的亲缘关系[13]。为阐明fimA基因在各Ⅰ型菌毛菌株间及mrkA基因在各Ⅲ型菌毛菌株的进化关系,采用邻接法作进化树构建。本试验结果显示,15株Ⅲ型菌毛的菌株可以分成4个分支,说明各区县间的肺炎克雷伯菌菌毛类型进化关系存在差异。
根据fimA和mrkA氨基酸序列比对结果显示,本研究菌株的fimA和mrkA氨基酸序列存在多处变异;与参考菌株比较,fimA中12处位点存在变异,marA中32处氨基酸位点存在变异。四川株的fimA氨基酸序列相对于重庆株变异较少,如菌株K.pn20仅在95位氨基酸位点存在变异;重庆株除K.pn19与K.pn20相同,仅在95位氨基酸位点存在变异,其余分离株存在8~11个氨基酸存在差异。mrkA氨基酸序列的分离株有32个氨基酸存在差异,K.pn1~K.pn9氨基酸变异少,发生氨基酸变异位点0~3之间,其余6株菌氨基酸变异8~18个。
氨基酸的变异不仅表示基因的多态性,还可能表示蛋白质的功能、菌株的致病性等特征发生变化。肺炎克雷伯菌Ⅰ型菌毛菌株aa95发生了氨基酸替代并由亲水性氨基酸变为疏水性氨基酸,K.pn16和K.pn17在aa19、aa118和aa142位点均由亲水性氨基酸变为疏水性氨基酸,K.pn18在aa19和aa118由亲水性氨基酸变为疏水性氨基酸。Ⅰ型菌毛黏附作用的关键在于疏水能力的大小[14],排斥水分子的能力增加时细菌菌毛的黏附能力更强,宿主机体更易致病。Ⅲ型菌毛的表达与细菌表面疏水性、致病性相关[15],当菌毛主要亚单位组成的疏水性氨基酸的比例增高时,Ⅲ型菌毛可提高肺炎克雷伯菌表面疏水性。
本研究中Ⅲ型菌毛菌株K.pn11~K.pn15中aa86、aa167、aa182均发生了氨基酸替代,mrkA氨基酸187个位点中28.13%的氨基酸由亲水性变为疏水性;因此,预测相应菌株的黏附能力可能增强,毒力更强。
4 结论
MSHA/MRHA试验法对本研究的20株肺炎克雷伯菌敏感性不高。通过PCR方法,鉴定出20株肺炎克雷伯菌中有5株为Ⅰ型菌毛,15株为Ⅲ型菌毛,重庆、四川肉牛所感染的肺炎克雷伯菌主要产Ⅲ型菌毛,且各区县之间的肺炎克雷伯菌菌毛类型进化关系存在差异;分析fimA和mrkA氨基酸序列后发现,fimA中12处位点存在变异,mrkA中32处氨基酸位点存在变异,肺炎克雷伯菌Ⅰ型菌毛菌株aa95发生了氨基酸替代并由亲水性氨基酸变为了疏水性氨基酸,Ⅲ型菌毛菌株K.pn7、K.pn8和K.pn9在164位氨基酸上均发生了S164A。因此,预测相应菌株的黏附能力可能增强,毒力更强。
