lncRNA TCONS_00815878对猪骨骼肌卫星细胞分化的影响
2019-12-24黄子莹李龙李倩倩刘向东李长春
黄子莹,李龙,李倩倩,刘向东,2,李长春,2
lncRNA TCONS_00815878对猪骨骼肌卫星细胞分化的影响
黄子莹1,李龙1,李倩倩1,刘向东1,2,李长春1,2
1. 华中农业大学,农业动物遗传育种与繁殖教育部重点实验室,武汉 430070 2. 广西扬翔股份有限公司生产中心,贵港 537131
猪骨骼肌发育是一个复杂的生物学过程,其中骨骼肌卫星细胞分化是影响骨骼肌发育的重要环节。近年来发现长链非编码RNA (long non-coding RNA, lncRNA)在骨骼肌卫星细胞分化中具有重要作用。为探究lncRNA TCONS_00815878对猪骨骼肌卫星细胞分化的影响,本研究利用qRT-PCR技术检测出生7 d内大白仔猪6种组织(心脏、脾脏、肺脏、肾脏、背肌和腿肌)及从胚胎期到出生后5个不同时间点(35 d、45 d、55 d胚胎及产后第7 d和第200 d后腿肌肉组织) TCONS_00815878的表达情况;利用反义核苷酸(antisense oligonucleotides, ASO)在猪骨骼肌卫星细胞中敲低TCONS_00815878,检验分化标记基因和表达情况;通过生物信息学分析预测TCONS_00815878靶基因,并利用DAVID软件在线预测其靶基因的功能与通路。结果表明:TCONS_00815878在猪心肌和腿肌中高表达;仔猪出生后7 d内,TCONS_00815878在猪肌肉组织中表达量不断升高,第7 d达到高峰;在猪骨骼肌卫星细胞增殖和分化过程中,TCONS_00815878在分化期表达量不断上升,且在分化30 h表达量达到峰值;敲低TCONS_00815878后,和基因表达量降低,其中表达量显著下降(0.05)。此外,功能预测结果发现,其靶基因富集到糖酵解和丙酮酸代谢等与骨骼肌卫星细胞分化相关的多个生物学过程。本研究推测,lncRNA TCONS_00815878可能对猪骨骼肌卫星细胞的分化起促进作用。
lncRNA TCONS_00815878;猪骨骼肌卫星细胞;分化;敲低;靶基因
猪()骨骼肌生长发育与其产肉量高低直接相关。此外,因与人具有相似的解剖学和生理学特征,研究猪骨骼肌的发育规律对人类相关研究也大有裨益[1]。骨骼肌发育是一个复杂且有序的过程,是各种遗传因素及调控因子共同作用的结果。骨骼肌卫星细胞是位于肌肉组织基膜和肌细胞膜之间的骨骼肌干细胞,在骨骼肌发育与再生过程中发挥着重要作用[2,3]。骨骼肌卫星细胞被激活后首先形成单核成肌细胞,增殖分化,随后融合成多核肌细胞,进一步相互连接形成肌纤维,最终并排形成肌肉组织[4,5]。
长链非编码RNA (long non-coding RNA, lncRNA)是一类长度大于200 nt的非编码RNA。研究证实,lncRNA在骨骼肌发育、肌细胞增殖与分化过程中发挥关键调控作用[6~8]。有关lncRNA参与骨骼肌发育的研究已有很多,但多集中对小鼠()的研究[9,10]。近年来,随着生物信息学及生物检测技术的发展,已在猪上获得大量与肌肉发育潜在相关的lncRNA,但多数lncRNA作用还尚不明确[11]。TCONS_ 00815878是Tang等[12]对贵州小型猪9个组织和3个骨骼肌样本进行高通量测序鉴定得到的肌肉组织特异性lncRNA,位于15号染色体,全长1089 nt,包含2个外显子。为进一步探究TCONS_00815878在猪骨骼肌发育过程中的作用,本研究检测了出生7 d内大白仔猪6种组织及从胚胎期到出生后5个不同时间点TCONS_00815878的表达情况,进一步在猪骨骼肌卫星细胞中对TCONS_00815878进行敲低处理,检测其对猪骨骼肌卫星细胞分化产生的影响。最后通过生物信息学分析,预测TCONS_ 00815878的靶基因及其靶基因可能参与的生物学过程和通路。通过以上研究,推测TCONS_00815878可能对猪骨骼肌卫星细胞分化过程产生的影响,旨在为猪骨骼肌生长发育相关lncRNA的功能研究提供更有价值的参考。
1 材料与方法
1.1 猪骨骼肌卫星细胞分离和培养
出生7 d内大白仔猪(购自华中农业大学种猪场),取两后腿全部肌肉,置于装有20~30 mL PBS (美国GIBCO公司)的培养皿中,在PBS中漂洗3次去除肌肉表面筋膜,剪碎组织肉样至肉糜状;将肉糜移至T75培养瓶,吸净上清,向肌肉组织中加入2倍体积0.2% Ⅰ型胶原酶(美国GIBCO公司)工作液;37℃、1200r/min水浴震荡消化2.5 h后加等体积终止培养基终止消化;过100目筛、200目筛及400目筛,2000 r/min离心10 min,除上清,加培养基重悬细胞,溶液移至10 cm培养皿;37℃、5% CO2贴壁培养2.5 h,将培养皿溶液上清移至新培养皿中继续培养;卫星细胞在37℃、5% CO2培养箱(美国Thermo Scientific公司)培养24 h,用PBS清洗2遍并更换培养基。
1.2 样品采集
取3头出生7 d内大白仔猪(购自华中农业大学种猪场),采集心脏、脾脏、肺脏、肾脏、背肌和后腿肌肉6种组织,用于检测TCONS_00815878在不同组织中的表达量。
采集大白猪35 d、45 d、55 d胚胎及产后第7 d和第200 d的后腿肌肉组织,用于检测TCONS_ 00815878从猪胚胎期到出生后不同时间点的表达量。上述所有组织样品采集后,经液氮速冻,保存于−80℃备用。
收集增殖期12 h、24 h和分化期12 h、24 h的猪骨骼肌卫星细胞,检测增殖期和分化期和的表达量,以确定猪骨骼肌卫星细胞的增殖分化效果。
收集增殖期6 h、12 h、18 h、30 h、36 h和42 h及分化期0 h、6 h、12 h、24 h、30 h、36 h和42 h的猪骨骼肌卫星细胞,检测TCONS_00815878在猪骨骼肌卫星细胞增殖与分化不同时期的表达量。
收集诱导分化24 h的猪骨骼肌卫星细胞,分离其细胞质与细胞核RNA,用于检测TCONS_00815878在猪骨骼肌卫星细胞的细胞质和细胞核的表达量。
1.3 总RNA提取
组织总RNA提取:取–80℃冰箱保存的组织样品,从冻存管中取出适量迅速放入液氮预冷的研钵中,加入适当液氮后研磨,待研磨成粉后转移到1.5 mL无RNA酶的离心管中,按照RNA提取试剂盒(德国Epigentek公司)说明书提取上述组织样本中的总RNA,取1 μL样品于NanoDrop 2000 (美国Thermo Scientific公司)分光光度计检测浓度,260/280值在1.8~2.0之间则表示RNA纯度较高。
细胞总RNA提取:将六孔板内的猪骨骼肌卫星细胞弃掉培养基后,用PBS冲洗两次,每孔直接加入1 mL Trizol,用移液枪吹打数次,室温裂解5 min,然后吸入1.5 mL无RNA酶离心管,于−80℃冰箱储存,后续RNA提取步骤与组织样品RNA提取方法一致。
细胞核与细胞质RNA分离与提取:猪骨骼肌卫星细胞用预冷的PBS冲洗两次,2187 r/min弃上清液。将沉淀重悬于0.2 mL裂解缓冲液(50 mmol/L Tris-HCl pH 8.0、140 mmol/L NaCl、1.5 mmol/L MgCl2、0.5% IGEPAL、1 mmol/L DTT、1 U/μL RNase抑制剂)。将混合物冰上放置5 min,之后4℃、2187 r/min细胞悬液离心3 min。收集上清液到1.5 mL离心管(细胞质部分),14 000 r/min离心4 min。剩余沉淀用0.2 mL裂解液冲洗2次(细胞核部分)。最后用1 mL Trizol提取所有细胞核和细胞质中RNA。
1.4 引物及反义核苷酸(antisense oligonucleotides, ASO)片段的设计、合成
lncRNA TCONS_00815878序列参考文献[9];和18S rRNA的mRNA序列由NCBI数据库获得。本研究采用Primer Premier 5软件设计引物。引物由生工生物工程(上海)股份有限公司合成。引物信息详见表1。
通过网站(http://sfold.wadsworth.org/cgi-bin/index.)在线设计敲低TCONS_00815878的ASO片段,由生工生物工程(上海)股份有限公司进行全链硫代修饰。ASO序列为GGATCTGGATTATGAACTTG。
1.5 cDNA合成和qRT-PCR扩增
参照SMARTerTMPCR cDNA Synthesis Kit试剂盒(日本TaKaRa公司)操作说明书反转录合成所有组织和猪骨骼肌卫星细胞中的cDNA,反转录产物于−20℃保存备用,以18S rRNA为内参,qRT-PCR检测TCONS_00815878表达量。反应总体系为10 μL:SYBR Green 5 μL,ddH2O 3.6 μL,引物各0.2 μL,cDNA 1 μL。反应条件:95℃ 2 min;95℃ 15 s, 56℃/60℃ 20 s,72℃ 15 s,40个循环;95℃ 5 s,58℃ 5 s,95℃ 50 s,1个循环。扩增结束后进行溶解曲线分析。
1.6 TCONS_00815878靶基因及功能预测
根据参考文献[9]测序结果中的蛋白编码基因表达量数据,计算TCONS_00815878与蛋白编码基因之间的Pearson相关系数,预测TCONS_00815878靶基因。选择0.05、Pearon相关系数0.95的基因作为靶基因[13](附表1)。利用DAVID功能注释生物信息学芯片分析预测靶基因功能与通路,取结果0.05为有效功能预测结果。
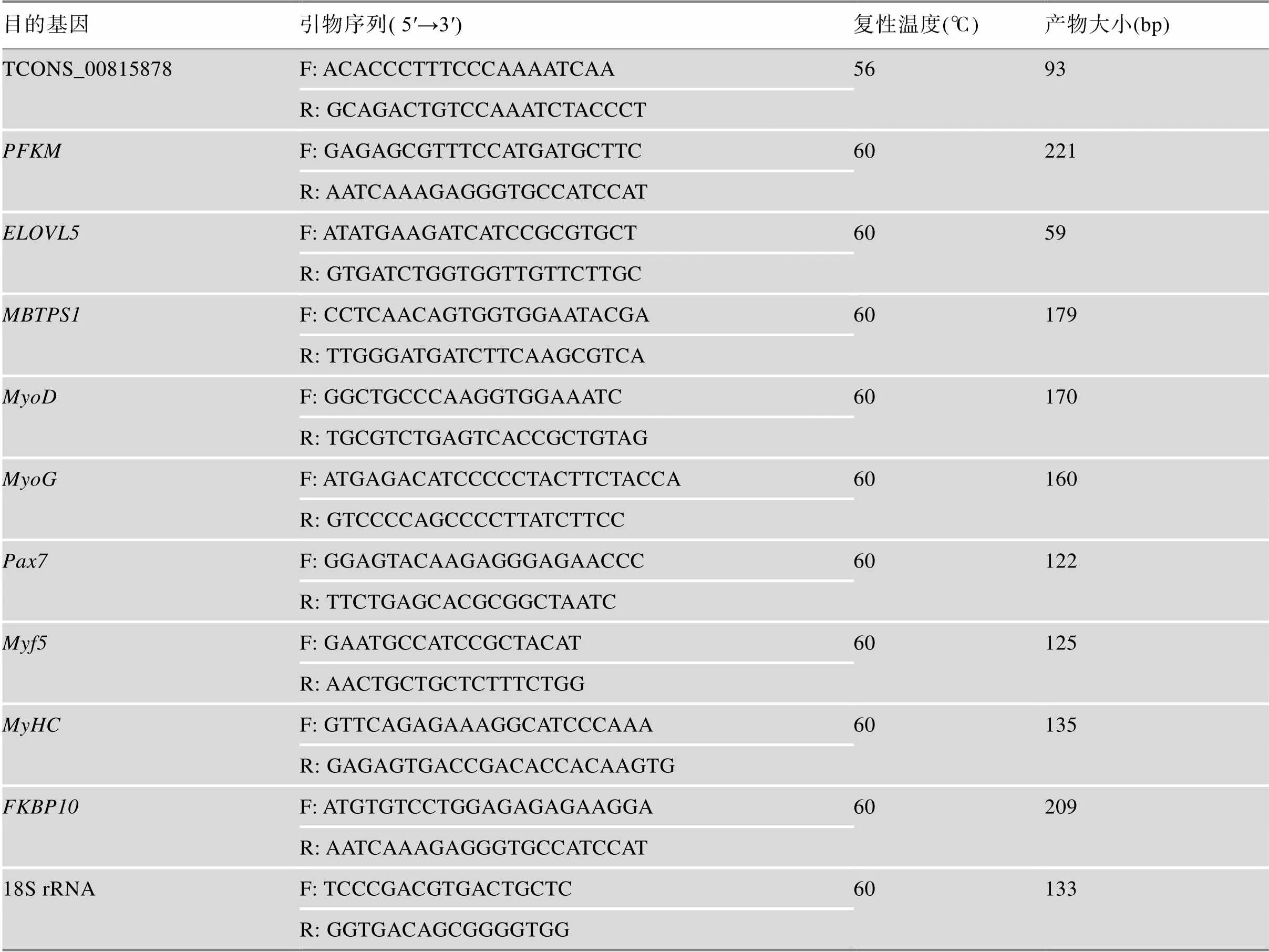
表1 本文所用引物信息
1.7 lncRNA TCONS_00815878敲低实验及靶基因验证
1.7.1 猪骨骼肌卫星细胞转染及TCONS_ 00815878敲低实验
在10 cm培养皿中培养猪骨骼肌卫星细胞,接种至六孔板,ASO敲低组(ASO)和对照组(NC)均设3个重复,待细胞密度达到80%左右进行转染实验,每孔ASO转染量为200 pmol。待细胞密度达到90%~ 100%更换分化培养基,分别于诱导分化后18 h、30 h收取细胞,进行RNA提取。qRT-PCR检测TCONS_00815878及和在不同分化时期表达量。
1.7.2 免疫荧光分析
将细胞用预冷的PBS洗涤2次,用预冷的4%多聚甲醛(广州赛国生物科技有限公司)室温静置固定15 min。PBS洗涤后,用预冷的0.25% TritonX-100 (广州赛国生物科技有限公司)室温静置处理10 min。PBS洗涤,封闭液4℃封闭1~2 h (封闭液:3%BSA, 0.3%T TritonX-100, 10%FBS,PBS)。PBS洗涤细胞,4℃孵育一抗过夜(抗体稀释液:3%BSA,0.3% TritonX-100, PBS)。之后37℃孵育二抗1 h。最后使用DAPI对细胞进行染色,荧光显微镜下观察荧光。
1.7.3 靶基因功能验证
使用敲低TCONS_00815878后的细胞样品。以18S rRNA为内参,qRT-PCR检测靶基因和的表达量。反应总体系为10 μL:SYBR Green 5 μL,ddH2O 3.6 μL,引物各0.2 μL,cDNA 1 μL。反应条件:95℃ 2 min;95℃ 15 s,60℃ 20 s,72℃ 15 s,40个循环;95℃ 5 s,58℃ 5 s,95℃ 50 s,1个循环。扩增结束后进行溶解曲线分析。
1.8 统计分析
qRT-PCR扩增结果参照2–ΔΔCt方法[14]评估TCONS_00815878及和相对表达量,以18S rRNA作为内参基因,0.05表示为差异显著,0.01表示为差异极显著。每项检测设置3个生物学重复,每个生物学重复设置3个技术重复,由GraphPad Prism 6.0软件完成统计并作图。生物信息学分析结果由Excel软件统计,利用R软件分析作图。
2 结果与分析
2.1 TCONS_00815878表达特征及核质定位
利用qRT-PCR检测出生7 d内大白仔猪心脏、脾脏、肺脏、肾脏、背肌和后腿肌肉6种组织中TCONS_00815878的表达量(图1A)。结果表明,TCONS_00815878在心脏、背肌和后腿肌肉中表达量较高,在其他组织中不表达。取35 d、45 d、55 d胚胎及产后第7 d和第200 d大白仔猪后腿肌肉组织,检测TCONS_00815878表达量(图1B)。结果表明,TCONS_00815878表达量在胚胎期35 d和45 d呈缓慢下降趋势,从45 d开始呈上升趋势,直至仔猪出生后第7 d达到峰值。

图1 TCONS_00815878表达特征及核质定位结果
A:lncRNA TCONS_00815878在猪不同组织中的表达量;B:TCONS_00815878从猪胚胎期到出生后不同时间点表达量;C:TCONS_00815878核质定位结果。
提取分化24 h猪骨骼肌卫星细胞中细胞质和细胞核RNA,qRT-PCR检测TCONS_00815878表达量(图1C)。结果表明,TCONS_00815878在细胞核中高表达,推测其可能在猪骨骼肌卫星细胞的细胞核中发挥作用。
2.2 TCONS_00815878对猪骨骼肌卫星细胞分化的影响
收集增殖期12 h、24 h和分化期12 h、24 h猪骨骼肌卫星细胞,qRT-PCR检验骨骼肌细胞体外增殖及诱导分化效果。结果表明,增殖期标记基因和在猪骨骼肌卫星细胞增殖期呈上升趋势,至增值期24 h达到峰值,之后在分化期表达量不断下降(图2A;附图1A)。分化期标记基因和表达量在猪骨骼肌卫星细胞增殖期呈下降趋势,分化初期表达量上升,末期表达量下降。分化期标记基因在猪骨骼肌卫星细胞中的表达量,从增殖期到分化期12 h呈上升趋势,之后下降(图2B;附图1,B和C)。表明猪骨骼肌卫星细胞体外诱导分化成功。
收集增殖期6 h、12 h、18 h、30 h、36 h和42 h及分化期6 h、12 h、24 h、30 h、36 h和42 h的猪骨骼肌卫星细胞,提取总RNA,反转录得到cDNA,qRT-PCR检测猪骨骼肌卫星细胞增殖期和分化期不同时间点TCONS_00815878的表达量。结果表明,在猪骨骼肌卫星细胞增殖期,TCONS_00815878表达量普遍较低,分化初期呈上升趋势,分化30 h达到峰值,说明TCONS_00815878可能作用于猪骨骼肌卫星细胞分化过程(图2C)。因此,本研究通过网站在线设计ASO片段,在分化期猪骨骼肌卫星细胞中敲低TCONS_00815878。检测结果表明,在分化期18 h和30 h的猪骨骼肌卫星卫星细胞中,ASO敲低组与对照组相比TCONS_00815878表达量显著降低(<0.001),表明ASO片段具有明显敲低效果,可 用于后续TCONS_00815878表达调控分析(图3,A和B)。
在分化18 h和30 h收集细胞,qRT-PCR检测猪骨骼肌卫星细胞分化标记基因和的表达量。结果表明,在分化18 h和30 h,基因表达量显著下降(<0.05),和基因呈下降趋势,推测TCONS_00815878可能促进猪骨骼肌卫星细胞分化(图3,C和D)。通过免疫荧光检测敲低后细胞样品中基因表达量,发现基因表达量显著下降(<0.01) (图3E)。以上结果初步说明TCONS_00815878可能影响猪骨骼肌卫星细胞的分化过程。
2.3 TCONS_00815878靶基因功能预测及验证
通过计算TCONS_00815878与其他蛋白编码基因的表达量相关性,预测得到TCONS_00815878的多个靶基因(附表1)。利用DAVID软件在线预测靶基因功能,推测TCONS_00815878可能通过调控其靶基因,参与生长因子合成、糖酵解和代谢等与骨骼肌发育有关的多个生物学过程(图4A,附表2)。其中和等基因参与糖酵解 代谢(glycolytic process,glycolysis和glycolysis/ gluconeogenesis)过程,和等基因参与生长因子结合(growth factor binding和GTPase activator activity)等过程。最后,本研究选取与TCONS_00815878表达呈正相关性的和基因,利用qRT-PCR在敲低TCONS_00815878的细胞中检测其表达量。结果表明,和基因表达量显著下降(0.05),验证了靶基因预测结果的准确性(图4B)。通过以上结果,提示TCONS_ 00815878可能通过调控其靶基因进而影响骨骼肌发育过程。
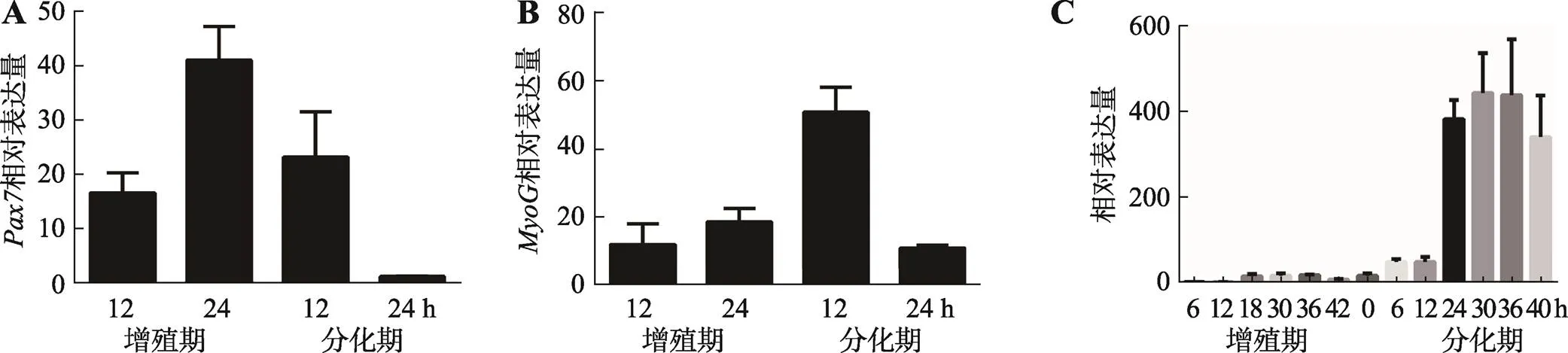
图2 猪骨骼肌卫星细胞分化效果及TCONS_00815878在猪骨骼肌卫星细胞不同时期表达量
A,B:增殖期基因与分化期基因的表达;C:TCONS_00815878在猪骨骼肌卫星细胞增殖和分化不同时间点的表达量。

图3 TCONS_00815878对猪骨骼肌卫星细胞分化影响
A,B:诱导分化18 h和30 h后TCONS_00815878表达量;C,D:敲低TCONS_00815878分化18 h和30 h后分化标记基因的表达情况;E:敲低TCONS_00815878后免疫荧光图片(400Í)及基因表达量。比例尺为20 μm。*表示<0.05,差异显著;**表示<0.01,***表示<0.001,差异极显著;NC:正常对照。
3 讨论
运用高通量测序和生物信息学分析技术,目前已鉴定得到了大量的功能基因[15]。研究证实,人类基因组75%以上的基因被选择性地转录,但只有少部分被翻译,其余不具有任何编码能力的转录物被标注为非编码RNA(non-coding RNA, ncRNA)[16],其中长度大于200 nt的为lncRNA[17]。越来越多的研究表明,lncRNA参与多种细胞和组织的发育过程,如基因组印记、干细胞维持、胚胎发育及成肌过程等[18~22]。然而,目前lncRNA的功能研究,在猪上仍处于探索的初级阶段[23]。
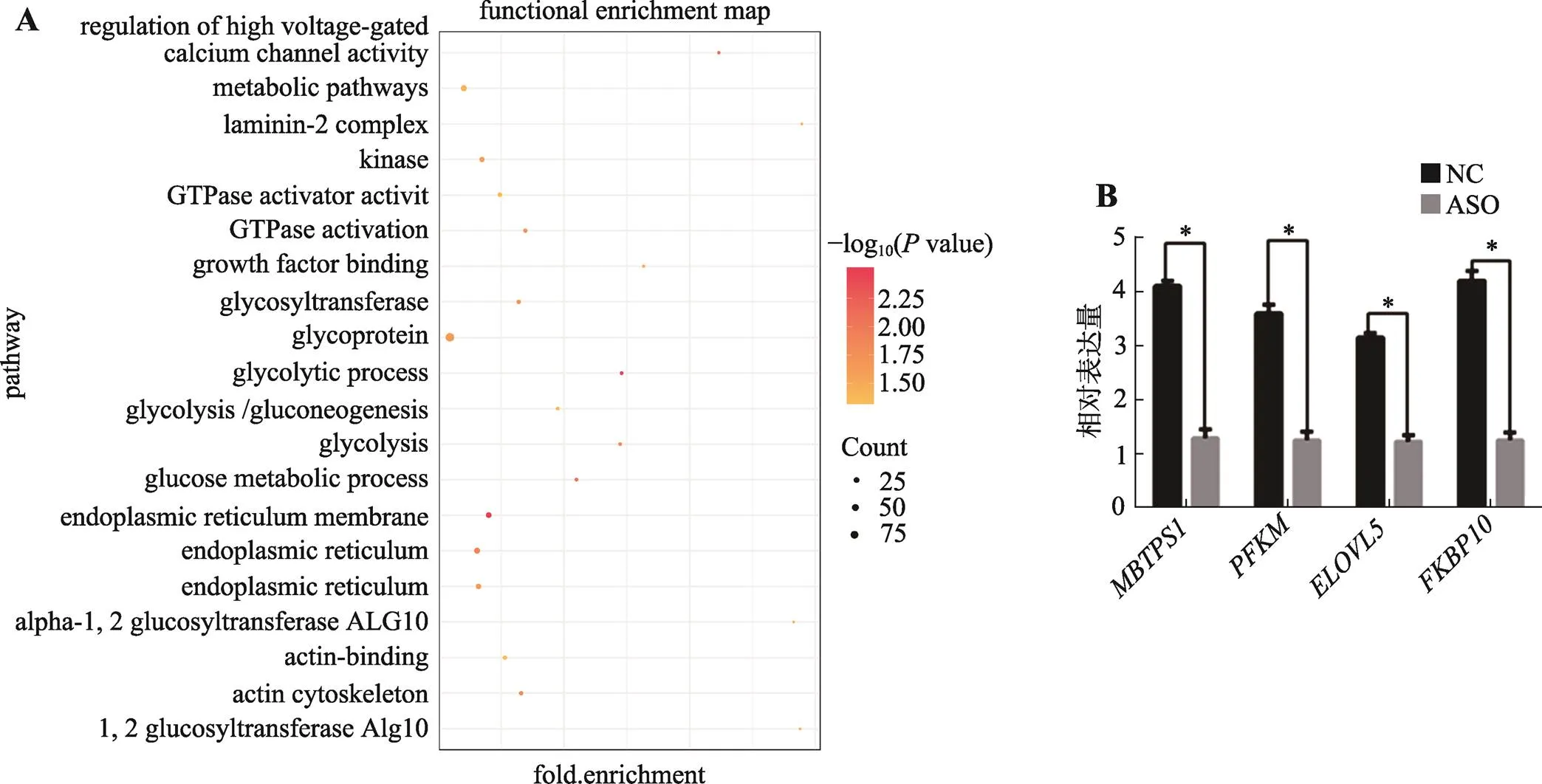
图4 TCONS_00815878靶基因功能预测及验证结果
A:GO富集分析和KEGG通路分析结果图;B:ASO敲低样品中靶基因表达量检测结果。*表示<0.05,差异显著。
猪骨骼肌的发育主要取决于肌纤维的生成,胚胎期35~90 d的两次生长波以及出生后1~60 d肌纤维类型的转变,对骨骼肌最终的发育起关键作用[24,25]。本研究利用qRT-PCR检测大白猪胚胎期35~55 d及出生后7 d和200 d不同时间点后腿肌肉组织中TCONS_ 00815878表达量,结果推测该lncRNA可能在猪胚胎期和出生早期发挥作用。
猪骨骼肌卫星细胞是研究骨骼肌发育规律进行研究的理想材料[26],本研究利用猪骨骼肌卫星细胞进行TCONS_00815878核质定位实验,结果表明TCONS_00815878主要分布在猪骨骼肌卫星细胞的细胞核中。另外,在猪骨骼肌卫星细胞增殖与分化期不同时间点检测TCONS_00815878表达量,结果推测该基因主要在猪骨骼肌卫星细胞分化期发挥作用。在分化期猪骨骼肌卫星细胞中敲低TCONS_ 00815878,qRT-PCR和免疫荧光实验结果均表明,相较于对照组,敲低组中基因的表达量显著下降。以上结果推测TCONS_00815878可能促进猪骨骼肌卫星细胞的分化。
LncRNA作用范围广泛,调控机制复杂。根据lncRNA不同的作用模式分为顺式(cis)和反式(trans)两种调控模式,其中反式调控模式可以通过计算lncRNA与蛋白编码基因的表达量相关性预测lncRNA靶基因[27]。基于Tang等[12]分析得到的各基因表达量,本研究通过计算TCONS_00815878与其他蛋白编码基因的表达量相关性,找到其可能参与调控的多个靶基因。基因功能富集分析发现其靶基因富集到多个与肌肉发育相关的通路,其中和基因参与糖酵解代谢过程,和基因参与生长因子结合过程。已有研究表明,糖酵解过程可以通过促进成肌细胞增殖与分化来支持胚胎肌肉生长[28]。Mangano等[29]研究表明,基因可以通过促进多不饱和脂肪酸的合成间接促进肌肉的生长发育。而和基因作为糖酵解或丙酮酸代谢的关键基因,在肌肉生长发育中起促进作用[30]。为验证靶基因预测准确性,本研究利用qRT-PCR在敲低后细胞样品中检测与TCONS_00815878表达呈正相关性的和基因表达量,发现和基因表达量与对照组相比下降显著(0.05),由此推测TCONS_00815878可能通过调控其靶基因间接参与骨骼肌的发育。本研究初步探讨了lncRNA TCONS_ 00815878对猪骨骼肌卫星细胞分化可能起到的调控作用,为深入揭示lncRNA TCONS_00815878在猪骨骼肌卫星细胞分化和骨骼肌发育中的作用机制提供参考。
附录:
附表1、附表2和附图1见文章电子版www.chinagene.cn。
附表1 靶基因列表
Supplementary Table 1 List of target genes

附图1 猪骨骼肌卫星细胞增殖期和分化期标记基因的表达
Supplementary Fig. 1 Expression level of marker genes in proliferative and differentiation phases of porcine skeletal muscle satellite cells
A:基因表达量;B:基因表达量;C:基因表达量。
[1] Groenen MAM, Archibald AL, Uenishi H, Tuggle CK, Takeuchi Y, Rothschild MF, Rogel-Gaillard C, Park C, Milan D, Megens HJ, Li ST, Larkin DM, Kim H, Frantz LAF, Caccamo M, Ahn H, Aken BL, Anselmo A, Anthon C, Auvil L, Badaoui B, Beattie CW, Bendixen C, Berman D, Blecha F, Blomberg J, Bolund L, Bosse M, Botti S, Bujie Z, Bystrom M, Capitanu B, Silva DC, Chardon P, Chen C, Cheng R, Choi SH, Chow W, Clark RC, Clee C, Crooijmans RPMA, Dawson HD, Dehais P, De Sapio F, Dibbits B, Drou N, Du ZQ, Eversole K, Fadista J, Fairley S, Faraut T, Faulkner GJ, Fowler KE, Fredholm M, Fritz E, Gilbert JGR, Giuffra E, Gorodkin J, Griffin DK, Harrow JL, Hayward A, Howe K, Hu ZL, Humphray SJ, Hunt T, Hornshøj H, Jeon JT, Jern P, Jones M, Jurka J, Kanamori H, Kapetanovic R, Kim J, Kim JH, Kim KW, Kim TK, Larson G, Lee K, Lee KT, Leggett R, Lewin HA, Li YR, Liu WS, Loveland JE, Lu Y, Lunney JK, Ma J, Madsen O, Mann K, Matthews L, McLaren S, Morozumi T, Murtaugh MP, Narayan J, Nguyen DT, Ni PX, Oh SJ, Onteru S, Panitz F, Park EW, Park HS, Pascal G, Paudel Y, Perez-Enciso M, Ramirez-Gonzalez R, Reecy JM, Zas SR, Rohrer GA, Rund L, Sang YM, Schachtschneider K, Schraiber JG, Schwartz J, Scobie L, Scott C, Searle S, Servin B, Southey BR, Sperber G, Stadler P, Sweedler JV, Tafer H, Thomsen B, Wali R, Wang J, Wang J, White S, Xu X, Yerle M, Zhang GJ, Zhang JG, Zhang J, Zhao SH, Rogers J, Churcher C, Schook LB. Analyses of pig genomes provide insight into porcine demography and evolution., 2012, 491(7424): 393–398.
[2] Almada AE, Wagers AJ. Molecular circuitry of stem cell fate in skeletal muscle regeneration, ageing and disease., 2016, 17(5): 267–279.
[3] Sacco A, Doyonnas R, Kraft P, Vitorovic S, Blau HM. Self-renewal and expansion of single transplanted muscle stem cells., 2008, 456(7221): 502–506.
[4] Allen RE, Merkel RA, Young RB. Cellular aspects of muscle growth: myogenic cell proliferation., 1979, 49(1): 115–127.
[5] Buckingham M. Myogenic progenitor cells and skeletal myogenesis in vertebrates., 2006, 16(5): 525–532.
[6] Jathar S, Kumar V, Srivastava J, Tripathi V. Technological developments in lncRNA biology., 2017, 1008: 283–323.
[7] Yang F, Yi F, Cao HQ, Liang ZC, Du Q. The emerging landscape of long non-coding RNAs., 2014, 36(5): 456–468.杨峰, 易凡, 曹慧青, 梁子才, 杜权. 长链非编码RNA研究进展. 遗传, 2014, 36(5): 456–468.
[8] Lu C, Huang YH. Progress in long non-coding RNAs in animals., 2017, 39(11): 1054–1065.路畅, 黄银花. 动物长链非编码RNA研究进展. 遗传, 2017, 39(11): 1054–1065.
[9] Weikard R, Demasius W, Kuehn C. Mining long noncoding RNA in livestock., 2017, 48(1): 3–18.
[10] Li H, Feng JC, Li GL, Wang X, Li MZ, Liu HF. The effect of lnc-RAP3 on 3T3-L1 preadipocyte differentiation in mouse., 2018, 40(9): 758–766.李欢, 冯晋川, 李贵林, 王讯, 李明洲, 刘海峰. Lnc- RAP3对小鼠3T3-L1前脂肪细胞分化的影响. 遗传, 2018, 40(9): 758–766.
[11] Xie SH. Integrated analysis of miRNA and mRNA expression profiles of muscle development in Changbai and Lantang pigs[Dissertation]. Sun Yat-Sen University, 2017.谢水华. 长白猪和蓝塘猪肌肉发育差异miRNA和mRNA表达谱的整合分析[学位论文]. 中山大学, 2017.
[12] Tang ZL, Wu Y, Yang YL, Yang YCT, Wang ZS, Yuan JP, Yang Y, Hua CJ, Fan XH, Niu GL, Zhang YB, Lu ZJ, Li K. Comprehensive analysis of long non-coding RNAs highlights their spatio-temporal expression patterns and evolutional conservation in., 2017, 7: 43166.
[13] Zhou ZY, Li AM, Adeola AC, Liu YH, Irwin DM, Xie HB, Zhang YP. Genome-Wide identification of long intergenic noncoding RNA genes and their potential association with domestication in pigs., 2014, 6(6): 1387–1392.
[14] Yun L, Liu JP, Zhuang ZX, Yang LQ, Zhang RL, Ye XM, Cheng JQ. Real-time RT-PCR gene expression relative quantification REST©software analysis compared with 2(–ΔΔCT)method., 2007, 7(10): 956–958.庾蕾, 刘建平, 庄志雄, 杨淋清, 张仁利, 叶小明, 程锦泉. 实时RT-PCR基因表达相对定量REST©软件分析与2(–ΔΔCT)法比较. 热带医学杂志, 2007, 7(10): 956–958.
[15] Chen G, Shi TL, Shi LM. Characterizing and annotating the genome using RNA-seq data., 2017, 60(2): 116–125.
[16] Djebali S, Davis CA, Merkel A, Dobin A, Lassmann T, Mortazavi A, Tanzer A, Lagarde J, Lin W, Schlesinger F, Xue C, Marinov GK, Khatun J, Williams BA, Zaleski C, Rozowsky J, Röder M, Kokocinski F, Abdelhamid RF, Alioto T, Antoshechkin I, Baer MT, Bar NS, Batut P, Bell K, Bell I, Chakrabortty S, Chen X, Chrast J, Curado J, Derrien T, Drenkow J, Dumais E, Dumais J, Duttagupta R, Falconnet E, Fastuca M, Fejes-Toth K, Ferreira P, Foissac S, Fullwood MJ, Gao H, Gonzalez D, Gordon A, Gunawardena H, Howald C, Jha S, Johnson R, Kapranov P, King B, Kingswood C, Luo OJ, Park E, Persaud K, Preall JB, Ribeca P, Risk B, Robyr D, Sammeth M, Schaffer L, See LH, Shahab A, Skancke J, Suzuki AM, Takahashi H, Tilgner H, Trout D, Walters N, Wang H, Wrobel J, Yu Y, Ruan X, Hayashizaki Y, Harrow J, Gerstein M, Hubbard T, Reymond A, Antonarakis SE, Hannon G, Giddings MC, Ruan Y, Wold B, Carninci P, Guigó R, Gingeras TR. Landscape of transcription in human cells., 2012, 489(7414): 101–108.
[17] Deniz E, Erman B. Long noncoding RNA (lincRNA), a new paradigm in gene expression control., 2017, 17(2–3): 135–143.
[18] Jandura A, Krause HM. The new RNA world: growing evidence for long noncoding RNA functionality., 2017, 33(10): 665–676.
[19] Li YY, Chen XN, Sun H, Wang HT. Long non-coding RNAs in the regulation of skeletal myogenesis and muscle diseases., 2018, 417: 58–64.
[20] Flynn RA, Chang HY. Long noncoding RNAs in cell-fate programming and reprogramming., 2014, 14(6): 752–761.
[21] Qin CY, Cai H, Qing HR, Li L, Zhang HP. Recent advances on the role of long non-coding RNA H19 in regulating mammalian muscle growth and development., 2017, 39(12): 1150–1157.秦辰雨, 蔡禾, 卿涵睿, 李利, 张红平. 长链非编码RNA H19对哺乳动物肌肉生长发育的调控. 遗传, 2017, 39(12): 1150–1157.
[22] Zhou R, Wang YX, Long KR, Jiang AA, Jin L. Regulatory mechanism for lncRNAs in skeletal muscle development and progress on its research in domestic animals., 2018, 40(4): 292–304.周瑞, 王以鑫, 龙科任, 蒋岸岸, 金龙. LncRNA调控骨骼肌发育的分子机制及其在家养动物中的研究进展. 遗传, 2018, 40(4): 292–304.
[23] Li MX, Huang T, Ma LP, Liu Y, Li T, Gong HB, Qiu MY, Xie S, Sun XM. Cloning of porcine lncRNA-ENSSSCT00000018610 and its expression pattern in porcine ovarian follicles., 2018, 49(9): 1830–1839.李梦寻, 黄涛, 马力鹏, 刘乙, 李涛, 公红斌, 邱梅玉, 谢苏, 孙晓梅. 猪lncRNA-ENSSSCT00000018610的克隆及其在猪卵泡中的表达. 畜牧兽医学报, 2018, 49(9): 1830–1839.
[24] Picard B, Lefaucheur L, Berri C, Duclos MJ. Muscle fibre ontogenesis in farm animal species., 2002, 42(5): 415–431.
[25] Lefaucheur L, Vigneron P. Post-natal changes in some histochemical and enzymatic characteristics of three pig muscles., 1986, 16(3): 199–216.
[26] Zammit PS, Heslop L, Hudon V, Rosenblatt JD, Tajbakhsh S, Buckingham ME, Beauchamp JR, Partridge TA. Kinetics of myoblast proliferation show that resident satellite cells are competent to fully regenerate skeletal muscle fibers., 2002, 281(1): 39–49.
[27] Liao Q, Liu CN, Yuan XY, Kang SL, Miao RY, Xiao H, Zhao GG, Luo HT, Bu DC, Zhao HT, Skogerbø G, Wu ZD, Zhao Y. Large-scale prediction of long non-coding RNA functions in a coding-non-coding gene co-expression network., 2011, 39(9): 3864–3878.
[28] Sanoudou D, Haslett JN, Kho AT, Guo SQ, Gazda HT, Greenberg SA, Lidov HGW, Kohane IS, Kunkel LM, Beggs AH. Expression profiling reveals altered satellite cell numbers and glycolytic enzyme transcription in nemaline myopathy muscle., 2003, 100(8): 4666–4671.
[29] Mangano KM, Sahni S, Kerstetter JE, Kenny AM, Hannan MT. Polyunsaturated fatty acids and their relation with bone and muscle health in adults., 2013, 11(3): 203–212.
[30] Lambadiari V, Triantafyllou K, Dimitriadis GD. Insulin action in muscle and adipose tissue in type 2 diabetes: The significance of blood flow., 2015, 6(4): 626–633.
The effect of lncRNA TCONS_00815878 on differentiation of porcine skeletal muscle satellite cells
Ziying Huang1, Long Li1, Qianqian Li1, Xiangdong Liu1,2, Changchun Li1,2
Porcine skeletal muscle development is a complex biological process, and differentiation of skeletal muscle satellite cells is an important part of skeletal muscle development. In recent years, it has been found that lncRNA plays an important role in the differentiation of skeletal muscle satellite cells. Here we investigate the effect of lncRNA TCONS_00815878 on the differentiation of porcine skeletal muscle satellite cells. We first used qRT-PCR to detect the expression levels of TCONS_00815878 in six tissues (heart, spleen, lung, kidney, back muscles and leg muscles) of Yorkshire piglets within seven days of birth.At the same time, the expression levels of TCONS_00815878 at five different time points from the embryonic stage to the postnatal stage (35 d, 45 d, 55 d of embryos, and 7 d, 200 d of postpartum leg muscles) were examined. The expression of the differentiation marker genes,andwas examined by knocking down TCONS_00815878 in porcine skeletal muscle satellite cells using antisense oligonucleotides (ASO). The target gene of TCONS_00815878 was predicted by bioinformatics analysis, and the function and pathway of its target gene were predicted online using DAVID software. The results showed that TCONS_00815878 had the highest expression level in pig myocardium and leg muscles. Within seven days after birth, TCONS_00815878 increased in the muscle tissue of pigs, and reached the peak of expression level on the 7th day. During the process of proliferation and differentiation of porcine skeletal muscle satellite cells, the expression level of TCONS_00815878 increased during the differentiation stage and peaked at 30 h of differentiation. After knocking down TCONS_00815878, the expression levels ofandwere decreased, but the expression level ofwas significantly decreased (<0.05). In addition, functional predictions revealed that the target gene of TCONS_00815878 is enriched in multiple biological processes, such as glycolysis and pyruvate metabolism, related to skeletal muscle satellite cell differentiation. This study speculates that lncRNA TCONS_00815878 may promote the differentiation of porcine skeletal muscle satellite cells.
lncRNA TCONS_00815878; porcine skeletal muscle satellite cells; differentiation; knockdown; target gene
2019-05-21;
2019-11-19
国家自然科学基金项目(编号:31872322)和中央高校创新研究基金项目(编号:2662017PY030)资助[Supported by the National Natural Science Foundation of China (No. 31872322) and Central University Innovation Research Fund (No. 2662017PY030)]
黄子莹,在读硕士研究生,专业方向:动物遗传育种大数据分析。E-mail: 327620138@qq.com
李长春,博士生导师,研究方向:猪重要经济性状的表观遗传学基础。E-mail: lichangchun@mail.hzau.edu.cn
10.16288/j.yczz.19-146
2019/12/7 17:08:48
URI: http://kns.cnki.net/kcms/detail/11.1913.R.20191206.0936.004.html
(责任编委: 李明洲)
