龙眼体细胞胚胎发生早期SDG基因家族的全基因组鉴定与表达分析
2019-12-14申序陈晓慧徐小萍霍雯李晓斐蒋梦琦张婧林玉玲赖钟雄
申序 陈晓慧 徐小萍 霍雯 李晓斐 蒋梦琦 张婧 林玉玲 赖钟雄


摘 要 SET domain group(SDG)基因家族控制的組蛋白赖氨酸甲基化,是参与染色质功能调控和表观遗传调控基因表达的重要部分。为了解龙眼(DlSDG)家族的生物学功能,采用生物信息学分析方法进行龙眼全基因组SDG家族成员的鉴定,并对蛋白质结构域、保守基序、基因结构、启动子顺势作用元件、进化树、相关基因的蛋白互作和体胚发生早期的基因表达情况进行预测和分析。结果显示,龙眼SDG家族基因包括32个成员,分为Suv、Ash、ATXR5/ATXR6、Trx、E(z)、SMYD和SETD 7个亚家族,其中Suv亚族成员数量最多;外显子数量在1~22个不等,蛋白结构域保守,都含有一个SET结构域,DlSDG蛋白保守motif不同亚家族间差异较大;DlSDG启动子包含许多诸如低温、干旱和压力等非生物胁迫响应元件和激素响应元件;龙眼SDG成员在体胚发生早期均能不同程度表达,其中DlSUVR4b在EC和ICpEC阶段可能发挥着较为重要的作用;DlSUVH4可以和DNA甲基转移酶MET1发生互作。本研究表明,龙眼DlSDG32个组蛋白赖氨酸甲基转移酶基因可能除了在生物钟的调节、植物发枝数量、根的生长发育、种子的萌发、花的发育和植株的形态建成等方面发挥重要功能外,也可能直接参与胁迫响应和体细胞胚胎发生的调控,并且还可能在参与DNA甲基化、染色质重塑的过程中形成体细胞胚胎发生的协作调控网络,表现其具有功能上的多样性、复杂性。
关键词 龙眼;组蛋白赖氨酸甲基转移酶;成员鉴定;功能分析;表观遗传中图分类号 S667.2 文献标识码 A
Genome-wide Identification and Expression Analysis of SDG Gene Family During Early Somatic Embryogenesis in Dimocarpus longan Lour.
SHEN Xu, CHEN Xiaohui, XU Xiaoping, HUO Wen, LI Xiaofei, JIANG Mengqi, ZHANG Jing, LIN Yuling, LAI Zhongxiong*
Institute of Horticultural Biotechnology, Fujian Agriculture and Forestry University, Fuzhou, Fujian 350002, China
Abstract The SET domain group (SDG) gene family controls histone lysine methylation, which is an important part of the regulation of chromatin function and epigenetic regulation of gene expression. To understand the biological function of longan (DlSDG) family, the wholeSDGfamily members of longan genome were identified by bioinformatics analysis, and the protein domain, conserved motif, gene structure, promoter homeopathic elements, evolutionary tree, protein interaction of related genes and gene expression at the early stage of somatic embryogenesis were predicted and analyzed. There were 32 members of longanDlSDGgene family, which could be divided into seven subfamilies,Suv,Ash,ATXR5/ATXR6,Trx,E(z),SMYDandSETD, andSuvhad the largest subfamily members. The number of exons varied from 1 to 22, and protein domains were conserved, all containing a SET domain. DlSDG protein conserved motif had large differences among different subfamilies.DlSDGpromoters contained many abiotic stress response elements such as low temperature, drought, stress and hormone response elements.SDGmembers could be expressed to different degrees in the early stage of somatic embryogenesis.DlSUVR4bmight play an important role in EC and ICpEC stages. DlSUVH4 could interact with DNA methyltransferase MET1. The study showed that the longanDlSDG32 histone lysine methylation transferase genes maight directly involved in the regulation of stress response and somatic embryogenesis, possibly in DNA methylation, chromatin remodeling, formation in the process of somatic embryogenesis of collaborative control network in addition to the regulation of the circadian clock, branch number, root growth and development, seed germination, the growth of flowers and plants of morphogenesis with diversity and complexity.
Keywords longan; histone lysine methyltransferase; identification of members; functional analysis; epigenetic
DOI10.3969/j.issn.1000-2561.2019.10.002
在植物表观遗传学快速的发展背景下,植物组蛋白修饰作为重要的表观遗传调控手段已日益成为研究的热点。组蛋白修饰同DNA甲基化、染色质重塑和非编码RNA调控一样是表观遗传调控的重要机制之一。真核生物中基因组DNA和组蛋白组装在一起形成染色体。首先,组蛋白H2A、H2B形成二聚体,H3、H4形成四聚体,而染色质的核心组成单位八聚体由2个H2A、H2B二聚体和1个H3、H4四聚体组成,经146 bp的DNA缠绕形成染色体的基本组成单元核小体[1-3]。作为调控核小体结构的主要方式,组蛋白翻译后修饰主要包括甲酰化(Formylation)、乙酰化(Acetylation)、甲基化(Methylation)、磷酸化(Phosphorylation)、泛素化(Ubiquity lation)、抑制基因表达的SUMO(small ubiquitin-like modifier modification,SUMOylation)化、ADP(ADP-ribosylation)核糖基化等修饰方式[4-7],且多发生在N端,多种组蛋白修饰结合与排列在一起构成了组蛋白密码[8],组蛋白与组蛋白、组蛋白与DNA的互作受到组蛋白密码的影响从而调控基因的转录与表达[9]。组蛋白甲基化修饰是最为复杂的植物组蛋白修饰方式,表现出修饰位点多样性和不同程度的修饰。植物组蛋白赖氨酸甲基化共价修饰主要依赖赖氨酸甲基转移酶来完成,含有SET结构域的SDG(SET domain group)是植物中唯一存在的具有组蛋白甲基转移酶活性的蛋白家族。赖氨酸甲基转移酶(histone lysine mathylatransferases,HKMTs)是两类组蛋白甲基转移酶的一大类,在植物中共发现17个组蛋白赖氨酸甲基化位点和7个精氨酸甲基化位点。组蛋白甲基化常发生在组蛋白H3、H4的赖氨酸(K)和精氨酸(R)的残基上,赖氨酸残基上发生最为常见。组蛋白赖氨酸甲基化修饰主要通过改变赖氨酸残基甲基化状态和程度,介导转录沉默与染色质的活化而不改变修饰位点携带的电荷量,从而提高组蛋白疏水性,改变组蛋白分子内部和分子间的相互作用,也可通過含有识别甲基化信号特定结构域的阅读器来实现[10]。组蛋白赖氨酸甲基化可以发生单甲基化、双甲基化和三甲基化3种不同程度的甲基化形式,其甲基化程度可以通过赖氨酸甲基转移酶和赖氨酸去甲基转移酶调控维持动态平衡[10]。H3和H4组蛋白中分别有5个(K4、K9、K27、K36、K79)赖氨酸残基和1个(K20)赖氨酸残基可被甲基化。一般H3K4、H3K36和H3K79甲基化通常和参与基因转录的激活有关,H3K9、H3K27和H4K20甲基化则和参与基因的转录抑制相关[11]。
组蛋白赖氨酸甲基转移酶在植物的生长发育中发挥着重要作用。组蛋白赖氨酸甲基化可以通过调控春化途径从而参与开花、促进花粉发育的过程[12-13];还可以参与启动植物种子萌发、调节根的生长发育、调节生物钟以及与拟南芥的发枝数量等[14-17]。组蛋白赖氨酸甲基化能够介导植物体细胞胚胎发生相关基因的表达,从而参与对体细胞胚胎生长发育过程的调控,并且植株的形态建成等完整生命过程均有组蛋白甲基化修饰的参与[18]。此外,组蛋白赖氨酸甲基转移酶常通过调控基因的表达而参与各种胁迫应答反应。综上,提示组蛋白赖氨酸甲基化几乎参与了植物生长发育的所有过程。
龙眼(Dimocarpus longanLour.)是我国重要的热带、亚热带常绿木本果树,果实富含蛋白质、氨基酸、类黄酮和多酚多种物质,营养价值和药用价值极高[19],其胚胎发育情况很大程度上影响果实产量、品质和可食用率,但在龙眼生长发育的过程中仍然存在核大、可食率低的问题。赖钟雄等[20-21]以龙眼幼胚为材料建立了龙眼松散型胚性愈伤组织系,进行长期保持,并以此为基础开展了大量与龙眼体细胞胚胎发生相关的研究。但在龙眼体细胞胚胎发生的进程中关于组蛋白赖氨酸甲基转移酶的研究未见报道。据其他植物研究报道,组蛋白赖氨酸甲基转移酶基因MEA在胚胎和胚乳中表达,并通过基因组印迹参与胚胎和胚乳的发育[22-23]。因此,鉴于SDG在高等植物尤其是木本果树胚胎发育过程中潜在的生物学功能,进一步的分析鉴定很有必要。在本实验室龙眼基因组破译的基础上,基于基因组开展龙眼体胚发生早期组蛋白赖氨酸甲基转移酶基因家族的生物学功能研究,不仅对龙眼生物技术的发展具有极大的意义,也对无患子科甚至所有木本植物体胚发生过程中组蛋白甲基转移酶的研究具有重要借鉴意义。
1 材料与方法
1.1材料
采用本实验室最新精确组装的龙眼基因组及全转录组数据库(待发表)进行分析。拟南芥(Arabidopsis thaliana)、甜橙(Citrus sinensis)基因序列、氨基酸序列下载于https://phytozome. html;水稻(Oryza sativa)氨基酸序列下载于Rice Annotation project Database(RAP-DB)[24]。
1.2 方法
1.2.1 龙眼DlSDG基因家族成员鉴定 为对龙眼DlSDG家族进行鉴定,首先选取模式植物拟南芥SDG氨基酸序列,经龙眼数据库同源比对,结合NCBI Blast分析,初步筛选确定37条具有完整开放阅读框的龙眼DlSDG候选序列。采用DNAMAN 6.0进行gDNA、CDS、氨基酸序列比对,其中Dlo032596、Dlo027491注释一样,经DNAMAN同源比对,Dlo032596、Dlo027491的CDS序列Identity为52.51%,氨基酸为42.38%,视为2条基因,分别命名为SUVH4a、SUVH4b;Dlo020952、Dlo020987的氨基酸同源比对显示Identity为97.43%,CDS为97.96%,且5?端完全重合,选取Dlo020952进行后续分析;Dlo014820、Dlo034485的氨基酸和CDS同源比对显示Identity均为100%,后续选取Dlo014820进行分析。结合SMART(http://smart.embl-heidelberg.de)和Pfam结构域预测分析和Softberry基因全长预测分析,最终确定了龙眼基因组中存在32条DlSDG序列。
1.2.2 龙眼DlSDG基因染色体定位与进化树构建 根据拟南芥SDG成员在龙眼基因组中的查找注释,参考拟南芥的命名方法对龙眼DlSDG基因家族成员进行命名。采用MEGA7的ClustalW对32个DlSDG基因家族成員氨基酸序列进行多序列比对分析,用进化树构建软件的Neighbor- joining method临近法对龙眼、拟南芥、水稻、甜橙4个物种140条SDG基因家族成员的氨基酸序列进行系统进化树构建,Bootstrap值设置为1000。采用TBtools[25]进行龙眼SDG基因染色体位置示意图绘制。
1.2.3 龙眼DlSDG基因结构及蛋白结构域分析 采用TBtools[25]软件,利用龙眼gff文件和genome文件对龙眼DlSDG家族32个成员进行基因结构预测和分析;采用ExPASy对32条DlSDG氨基酸序列进行蛋白的等电点(pI)、分子量(Mw)、氨基酸数目等数据的分析;通过MEME(multiple expectation maximization for motif elicitafion)(http://meme-suite.org/tools/meme)进行龙眼DlSDG的保守基序分析,采用TBtools的可视化分析与motif图片绘制。采用SMART和Pfam对龙眼DlSDG进行蛋白结构域在线预测,利用TBtools进行蛋白结构域示意图绘制。
1.2.4 龙眼DlSDG启动子分析 采用TBtools进行龙眼DlSDG基因起始密码子ATG上游2000 bp序列的提取。采用PlantCARE(http://bioinform a
tics.psb.ugent.be/webtools/plantcare/html/)网站在线预测分析龙眼DlSDG不同成员的启动子特征和顺势作用元件功能特点,采用TBtools进行可视化绘图。
1.2.5 龙眼DlSDG成员蛋白互作预测分析 利用STRING(https://string-db.org)网站进行蛋白质互作预测、利用k均值聚类算法进行4个聚类分析,选用研究最深入的拟南芥作为参考,最低互动评分要求选择highest confidence(0.900)探究DlSDG成员自身之间、与其他蛋白之间的互作情况。
1.2.6 龙眼DlSDG家族体胚发生早期阶段表达分析 为更深入的了解DlSDG在体细胞胚胎和不同组织器官中可能具有的功能特点,利用龙眼转录组数据库提取DlSDG基因在不同体胚发生阶段胚性愈伤组织(EC)、不完全胚性紧实结构(IcpEC)、球形胚(GE)特异表达的FPKM值,分析家族各成员的表达情况。利用TBtools绘制表达热图。
2 结果与分析
2.1龙眼DlSDG家族基因鉴定与蛋白特性分析
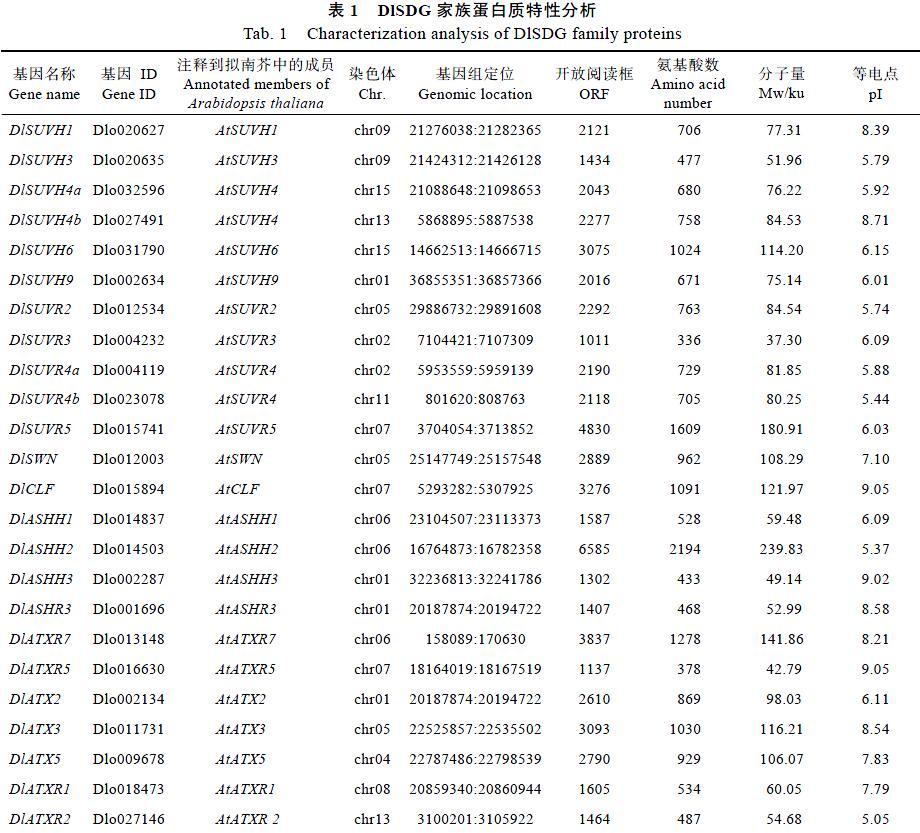
在龙眼中获得了32个SDG家族成员。通过NCBI Blast双向比对,与模式植物拟南芥、单子叶子植物水稻和木本果树甜橙家族成员进行系统进化分析,将其命名情况见表1。SDG家族蛋白特性分析发现,该基因家族氨基酸长度相差较大,有336~2426 aa,大部分在1000 aa以下,蛋白分子量在36.33~275.37 ku之间,等电点(pI)为4.82~9.05(表1)。
2.2龙眼DlSDG家族基因染色体定位分析
DlSDG家族基因染色体定位预测发现,赖氨酸甲基转移酶家族基因成员能精确定位在组装好的染色体上,在龙眼基因组组装好的15条染色体中,染色体3、10、12没有DlSDG成员分布(图1)。DlSDG基因在染色体上分布不均匀,其中1、5、6、7、9、13、15号染色体均分布3个以上的赖氨酸甲基转移酶家族成员,以6号染色体最多,存在6个成员的分布,4、11、14号染色体最少,均只有1个成员分布。为分析DlSDG基因家族成员数量的庞大是否因为基因的串联重复导致,参考Huang等[26]的方法进行串联重复分析,结果表明DlSDG家族成员无串联复制现象存在(表1)。
2.3龙眼DlSDG基因结构与蛋白结构域分析
为进一步了解龙眼组蛋白赖氨酸甲基转移酶SDG基因家族成员的生物学功能,对32条基因的结构进行内含子、外显子的数目及位置进行分析(图1)。结果表明,所有SDG基因中有15条基因序列完整,具有基因结构的上下游,4条基因序列具有上游或下游,13条序列缺少完整上下游。DlSDG内含子数为1~22个之间,内含子第1个均落在成熟编码序列内部,有利于将信号序列与编码序列整齐地分开。基因结构图明显看出,龙眼赖氨酸甲基转移酶SDG基因家族成员基因长度存在较大的差异,可能是内含子数量差异较大的原因,同时也可能影响其基因功能的分化。
为了解龙眼DlSDG基因家族的蛋白保守基序特点和分布情况,通过MEME软件搜索出15个保守motif(图2)。由图2可知,整个龙眼SDG基因家族中最为保守的基序是motif2,所有32个成员中只有DlSUVH1缺少该保守基序,其次是motif1和motif3,均有27个成员具有这2个保守基序,大多数基因还具有motif4和motif7。进化分析可以看出,同一个分支上的保守基序位置大致相同,这可能与它们功能的保守性有关。
SMART和Pfam蛋白结构域预测分析,DlSDG家族均属于含SET结构域超家族赖氨酸甲基转移酶活性的蛋白。总体而言,DlSDG家族成员均至少含有1个SET结构域,其中E(z)亚族的DlCLF蛋白最为特殊,含有3个SET结构域,且为间断排列,多数SET结构域位于SET结构域蛋白的末端,少数位于SET结构域蛋白的前端和中部。图3结构域分析显示,主要包括8个组别,分别为:含PreSET和SET结构域的DlSUVH和DlSUVR;含有AWS、SET和PostSET的DlASHR3、DlASHH2,DlATXR7与其结构域最为相似,归为同一类;含有2个及以上PHD和1个SET结构域的DlATX2、DlATX3、DlATX5;含有1个PHD和1个SET结构域的DlATXR5;含有特殊SANT結构域的DlSWN和DlCLF;2条含有AWS和SET的短序列DlASHH1和DlASHH3;只含有1个结构域的DlASHR1、DlASHR2、DlATXR1、DlATXR2、DlATXR3、DlATXR4、DlSDG41和DlSETD10;以及含Rubis-subs-bind和SET的DlSDG40和DlSETD3。
2.4植物SDG家族的系统进化树分析
为更深入的了解龙眼DlSDG家族的生物学功能,采用MEGA7对拟南芥、水稻、甜橙和龙眼4个物种140条氨基酸序列进行系统进化树的构建(图4)。参考拟南芥、水稻中基因的分类,并结合聚类分析,大致可以将龙眼DlSDG家族分为7类。第1类为Suv中的DlSUVH1、DlSUVH3、DlSUVH4a、DlSUVH4b、DlSUVH6、DlSUVH9以及DlSUVR2、DlSUVR3、DlSUVR4a、DlSUVR4b、DlSUVR5;第2类为Ash中的DlASHH1、DlASHH2、DlASHH3、DlASHR3;第3类主要包括DlATXR5;第4类为Trx中的DlATX5、DlATX3和DlATX2;第5类主要包括E(z)中的DlSWN和DlCLF;第6类主要包括SMYD中的DlATXR1、DlATXR2、DlATXR3、DlATXR4、DlASHR2;第7类主要为SET domain contain protein (SETD),包括DlSDG41、DlSDG40和SETD3、SETD10。
2.5龙眼DlSDG基因家族启动子分析
为进一步的分析龙眼DlSDG家族基因启动子的功能,通过PlantCARE在线预测分析DlSDG基因的启动子顺式作用元件发现(图5),DlSDG
家族成员启动子序列均含有较多的CAAT-box和TATA-box,说明家族DlSDG基因均能进行正常的转录。所有龙眼DlSDG家族基因启动子含有较多的光响应、厌氧诱导响应元件,90%以上基因启动子含有茉莉酸甲酯激素应答元件,50%基因启动子含有乙烯、水杨酸和赤霉素应答元件,少数基因响应脱落酸和生长素应答。此外,DlSDG家族基因还响应低温、干旱、胁迫、种子特异调控和胚乳表达元件,提示DlSDG家族基因可能在抗逆、耐寒、种子生长和胚胎成熟发育过程中发挥重要的作用。
2.6龙眼DlSDG家族蛋白互作网络预测
利用String蛋白互作在线数据库对32个DlSDG蛋白成员之间的功能关系进行预测(通过拟南芥与甜橙、杨树互作结果比对,选择研究较为深入的拟南芥作为模式植物,图6)。蛋白互作聚类预测显示,绿色和黄色区域的蛋白质间互作关系强,SDG家族除自身成员之间会发生互作之外,还会与其他蛋白互作。SDG家族主要有ASHH1、EFS(ASHH2)、ATXR5、ATXR6互作和SWN、CLF、MEA之间的互作。说明这两组基因之间功能的关联性强。
2.7龙眼DlSDG家族基因组织特异表达分析
结合龙眼3代转录组数据库注释中不同体胚发生阶段的FPKM值,制作聚类分析热图(图7)。结果表明:在不同体胚发生过程中,大致有4种表达模式:GE阶段上调,EC和ICpEC阶段下调(DlSUVH4b、DlATXR7、DlSWN、DlCLF);EC阶段上调,GE和ICpEC阶段下调(DlASHH2、DlATXR2、DlASHR2、DlSDG40);ICpEC阶段上调,GE和EC阶段下调(DlSUVH1、DlSUVH3、DlSUVH9、DlSUVH4a、DlSUVR5、DlASHR3、DlATX2、DlATXR5);EC和ICpEC阶段上调,GE阶段下调(DlSUVH6、DlSUVR2、DlSUVR3、DlSUVR4a、DlSUVR4b、DlASHR3、DlATX2、DlASHH2、DlATXR4、DlASHR1、DlATXR1、DlASHH1、DlSETD10、DlSETD3、DlSDG41)。总之,龙眼赖氨酸甲基转移酶家族在体胚发生不同阶段均发生不同程度的表达,总体来看,与EC和ICpEC阶段相比,大部分龙眼SDG家族成员在GE中表达较低,其中DlSUVR4b在EC和ICpEC中出现高表达,DlCLF较EC和ICpEC相比,在GE中高表达,可能预示着DlSUVR4b在EC和ICpEC中、DlCLF在GE中发挥着较为重要的作用。
3 讨论
3.1龙眼组蛋白甲基转移酶家族成员可能具有功能上的多样性
组蛋白赖氨酸甲基转移酶介导组蛋白的赖氨酸甲基化共价修饰的完成,绝大多数含有一个具有甲基转移活性,130~150个氨基酸的SET保守结构域。在拟南芥、水稻和玉米中至少有47、37和35个SDG基因[27],其他在葡萄和杨树中有报道[28-29],关于无患子科植物组蛋白甲基转移酶SDG家族的研究尚未见报道。系统进化分析将含有SET结构域的龙眼组蛋白甲基转移酶基因分为7个大类。鉴定龙眼基因组有32个完整SET结构域的SDG成员,少于拟南芥的47个、水稻的35个,可能是因为DlSDG在龙眼基因组中无串联重复或染色体片段复制导致的基因复制相对较少有关。与之前的研究相似,系统发育分析将龙眼SDG基因分为了7个主要的类群[27,30](图4)。近年来的研究揭示,拟南芥组蛋白甲基转移酶ATX1可通过调节水杨酸(salicylic acid,SA)和茉莉酸(jasmonic acid,JA)信号途径的关键转录因子WRKY70启动子的H3K4的甲基化,来激活WRKY70,进而上调水杨酸信号途径基因PR1的表达以及下调茉莉酸信号途径基因THI2.1的表达,参与到拟南芥对假单胞菌(PstDC3000)侵染的防御反应[31-32]。植物病原抵御基因能够被转录激活标志H3K36me3修饰诱导并大量表达,从而抵御病菌的侵害[33]。也有研究显示ASHH2通过介导ERF1、MYC2、PDF1.2a和VSP2等JA/ET信号途径基因位点,诱导这些基因的快速转录,增强拟南芥对真菌(黑斑病菌和灰霉病菌)的抗性[33]。这些研究表明H3K36甲基化修饰正调控植物对细菌及真菌的防御反应。在龙眼中没有鉴定出ATX1基因,但鉴于SDG亚族间功能上有冗余性的特点,龙眼中与ATX1结构域相似的DlATX2、DlATX3和DlATX5具有同DlASHH2一样抵御病原菌侵害的潜在功能。SDG基因还参与了花粉的发育、根的生长发育、种子萌发、生物钟的调节等生物过程[14-17]和控制拟南芥根尖分生组织和静止中心的细胞分裂[34]。
结合蛋白结构域分析,龙眼SDG同其他植物一样都具有保守的SET结构域。E(z)亚家族在拟南芥中包括CURLYLEAF(CLF)、MEDEA(MEA)和SWINGER(SWN)3个成员,除SET结构域外,还含有SWl3、ADA2、NCoR、TFIIIBDNA. Binding(SANT)和cysteine. rich(CXC)2种结构域,龙眼E(z)家族的DlSWN和DlCLF 2个成员均含有SANT结构域,只有DlCLF还含有CXC,可能龙眼dlswn与拟南芥atswn功能存在差异。Trx亚家族中ATXR7不含有除SET和PostSET以外的结构域,其他成员的PHD结构域是一类含类C4HC3型锌指基序结构域的统称,具有与DNA、RNA、蛋白质、组蛋白共价修饰等多种生物大分子结合的能力,同一个蛋白质上的不同PHD结构域,甚至同一PHD结构域的不同基序都可能具有识别不同生物大分子的功能[35];PWWP結构域可以特异地与甲基化的赖氨酸残基结合[36];可能龙眼中DlATX2、DlATX3、DlATX5具有识别生物大分子并结合形成复合物发挥相关功能。此外FYRN和FYRC结构域存在于染色质相关的蛋白质中,具体功能未知[37],DlATX2具有完整的FYRN和FYRC结构域,暗示其可能与染色质生物学功能有关。Suv亚家族是SDG中最大的亚族,可以细分Suvhomologs(SUVH)亚族和Suv-related homolog(SUVR)亚族。SUVH亚族成员含有一个保守的SET和RING finger associated(SRA)结构域,可以特异地与DNA序列中甲基化的胞嘧啶结合[38],暗示龙眼DlSUVH可能与DNA甲基化共同参与某一生物学问题。研究证明SMYD亚家族的AtASHRl与H3K4me2甲基化相关[39],研究认为SETD亚家族成员不作用于组蛋白的甲基化[27]。
3.2龙眼DlSDG可能影响体细胞胚胎发生的进程
在植物中,组蛋白赖氨酸甲基转移酶直接参与体细胞胚胎发生的研究报道很少。已见报道E(z)家族的MEA在胚胎和胚乳中表达,参与胚胎和胚乳的发育。对于同亚族SWN,有报道称萌发后的clf和swn双突变体中有LEC1异位表达[40]。也有报道表明SWN与MEA存在功能冗余,SWN突变增强了MEA突变体的胚胎致死性[41]。SWN可能抑制与胚胎发生无关的基因,并且是营养阶段抑制LEC1的重要因子。在鉴定的龙眼组蛋白赖氨酸甲基转移酶SDG家族中,存在2个E(z)家族成员DlSWN和DlCLF成员,且在龙眼早期胚胎发生GE阶段上调表达,表明龙眼DlSWN和DlCLF可能直接参与植物体细胞胚胎发育进程。
組蛋白修饰酶是参与染色质重塑最重要的因子[42]。在拟南芥中,已经证实了染色质重塑与体细胞胚胎之间发生的关系,据报道,一种染色质重塑因子PICKLE(PKL)影响胚胎特异性基因的表达,如拟南芥中的LEC1[43]。许多研究报道证明,组蛋白甲基化在表观遗传控制基因的表达和染色质重塑与包装方面发挥着重要的作用[44],尤其是在分化方面[45]。越来越多证据表明植物体胚发生过程中基因表达的时空特异性除了受特异的DNA序列控制,一定程度的DNA甲基化有利于植物体细胞胚胎的正常发育[46]。对拟南芥MET和CMT3(维持甲基化酶)无效等位基因的突变体进行研究,发现其胚胎无法正常发育且生活力下降,且胚胎发育相关的基因未出现表达,说明DNA甲基化对拟南芥的胚胎发生起着非常关键的作用[47]。早期的研究发现,H3K9甲基化对DNA甲基化是必须的,对拟南芥kyp(suvh4)突变株功能分析证实了这一关系,和cmt3的突变体相似,kyp突变体中CNG区域的DNA甲基化水平降低[48],表明CMT3介导的DNA甲基化位于H3K9甲基化的下游。另外的研究表明CMT3的Chromo结构域可以与K9和K27同时被甲基化的组蛋白H3K9K27的N-端结合[49],表明H3K9和H3K27甲基化可能共同形成了被CMT3识别的信号导致CNG的甲基化。这些研究表明,组蛋白赖氨酸转移酶可能通过参与染色质重塑和DNA甲基化来影响体细胞胚胎发生基因的表达,从而对体细胞胚胎的生长发育进程产生重要的影响。在龙眼基因组中鉴定出32个组蛋白赖氨酸甲基转移酶,表达分析提示组蛋白赖氨酸甲基转移酶可能在参与DNA甲基化和染色质重塑与组装的过程中对体细胞胚胎发生的进程产生重要的影响,也可能通过形成DNA甲基化、组蛋白修饰和染色质重塑更为复杂的调控网络,协同参与体细胞胚胎生长发育的调控。蛋白质互作预测也显示出SDG家族成员MEA、CLF、SWN、SUVH4与DNA甲基转移酶MET1之间具有互作关系,进一步说明组蛋白赖氨酸甲基转移酶可能通过与DNA甲基化和染色质重塑关联参与植物体细胞胚胎调控。
3.3龙眼DlSDG可能响应非生物胁迫
龙眼SDG家族顺势作用元件分析(图5)发现,除DlSUVH3、DlSUVH4a、DlSUVR3、DlSUVR4b、DlASHR3、DlATXR2、DlATXR5、DlSWN、DlASHH3、DlASHR1、DlASHH1、DlASHR2、DlSETD3、DlSDG40、DlSDG41以外启动子均含有低温响应元件,9个成员DlSUVH1、DlSUVR2、DlSUVR4a、DlSUVR4b、DlASHR3、DlATXR7、DlATX3、DlATXR1、DlASHH1预测到干旱胁迫响应元件,还有DlSUVH1、DlSUVH3、DlSUVH9、DlSUVR3、DlSUVR4a、DlASHR3、DlASHH2、DlATXR2、DlASHR1、DlATXR1、DlASHH1、DlSDG40、DlSDG41、DlSETD3共14个成员具有抵御和压力胁迫响应元件,预示龙眼中SDG家族可能与龙眼干旱、低温和盐胁迫等非生物胁迫响应有关。组蛋白赖氨酸甲基化共价修饰在响应低温、干旱胁迫、盐胁迫和生物胁迫等方面具有一定的作用,而与高温胁迫响应无密切关系[50]。在干旱条件下,ABA合成关键酶NINE-CIS-EPOXYCAROTENOIDDIOXYGENASE3(IVCED3)基因座能特异结合ATX1,上调其表达,ABA依赖的干旱响应信号通路被启动;同时也上调表达RESPONSIVETODESICCATION 29A(RD29A)和RD29B等基因,参与其他干旱响应信号通路[51-52]。多次干旱处理,RD29B和RESPONSIVETOABA18(RABl8)等基因的基因座上H3K4me3水平在水分充足的情况下同样维持在较高水平,表明ATXl参与了干旱胁迫的记忆过程[53]。CLF引导的H3K27me3同样参与了干旱胁迫过程,但并不形成胁迫记忆[54]。龙眼SDG中未能鉴定到ATX1赖氨酸甲基转移酶基因,有一个CLF基因,鉴于同一家族结构保守和可能存在功能冗余性,除上述具有干旱、低温和压力胁迫响应的基因外,龙眼SDG家族中DlATX2、DlATX5和DlCLF也可能具有抗旱、抗寒和耐盐胁迫的潜在功能。此外,ASHH2也被证明在重复的机械刺激造成的对触碰诱导基因TOUCH3(TCH3)的胁迫记忆中发挥作用[55],顺式作用响应元件分析表明,龙眼DlASHH2可能具有这方面的功能。
參考文献
[1]Kornberg R D. Chromatin structure: a repeating unit of histones and DNA[J]. Science, 1974, 184(4139): 868-871.
[2] Luger K, M?der A W, Richmond R K,et al.Crystal structure of the nucleosome core particle at 2.8 ? resolution[J]. Nature, 1997, 389(6648): 251-260.
[3] Wolffe A P, Guschin D. Chromatin structural features and targets that regulate transcription[J]. Journal of Structural Biology, 2000, 129(2-3): 102-122.
[4] Sura W, Kabza M, Karlowski W M,et al.Dual role of the histone variant H2A. Z in transcriptional regulation of stress-response genes[J]. The Plant Cell, 2017, 29(4): 791-807.
[5] Strahl B D, Allis C D. The language of covalent histone modifications[J]. Nature, 2000, 403(6765): 41-45.
[6] Zhang Y, Reinberg D. Transcription regulation by histone methylation: interplay between different covalent modifications of the core histone tails[J]. Genes & Development, 2001, 15(18): 2343-2360.
[7] Iizuka M, Smith M M. Functional consequences of histone modifications[J]. Current Opinion in Genetics & Development, 2003, 13(2): 154-160.
[8] Jenuwein T, Allis C D. Translating the histone code[J]. Science, 2001, 293(5532): 1074-1080.
[9] Chinnusamy V, Zhu J K. Epigenetic regulation of stress responses in plants[J]. Current Opinion in Plant Biology, 2009, 12(2): 133-139.
[10] Liu C, Lu F, Cui X, et al. Histone methylation in higher plants[J]. Annual Review of Plant Biology, 2010, 61(1): 395-420.
[11] Martin C, Zhang Y. The diverse functions of histone lysine methylation[J]. Nature Reviews Molecular Cell Biology, 2005, 6(11): 838-849.
[12] Yang H, Mo H, Fan D,et al. Overexpression of a histone H3K4 demethylase, JMJ15, accelerates flowering time inArabidopsis[J]. Plant Cell Reports, 2012, 31(7): 1297-1308.
[13] Cartagena J A, Matsunaga S, Seki M, et al. The Arabidopsis SDG4 contributes to the regulation of pollen tube growth by methylation of histone H3 lysines 4 and 36 in mature pollen[J]. Developmental Biology, 2008, 315(2): 355-368.
[14] Dong G, Ma D P, Li J. The histone methyltransferase SDG8 regulates shoot branching in Arabidopsis[J]. Biochemical and Biophysical Research Communications, 2008, 373(4): 659-664.
[15] Yao X, Feng H, Yu Y,et al.SDG2-mediated H3K4 methylation is required for proper Arabidopsis root growth and development[J]. PLoS One, 2013, 8(2): e56537.
[16] Malapeira J, Khaitova L C, Mas P. Ordered changes in histone modifications at the core of theArabidopsis circadianclock[J]. Proceedings of the National Academy of Sciences, 2012, 109(52): 21540-21545.
[17] Müller K, Bouyer D, Schnittger A,et al.Evolutionarily conserved histone methylation dynamics during seed life-cycle transitions[J]. PLoS One, 2012, 7(12): e51532.
[18] Alvarez-Venegas R, Pien S, Sadder M,et al.ATX-1, an Arabidopsis homolog of trithorax, activates flower homeotic genes[J]. Current Biology, 2003, 13(8): 627-637.
[19] Lin Y L, Min J M, Lai R L,et al. Genome-wide sequencing of longan (Dimocarpus longan Lour.) provides insights into molecular basis of its polyphenol-rich characteristics[J]. GigaScience, 2017, 6(5): 1-14.
[20] 賴钟雄, 陈振光. 龙眼胚性愈伤组织的高频率体细胞胚胎发生[J]. 福建农业大学学报, 1997(3): 271-276.
[21] 陈春玲, 赖钟雄. 龙眼胚性愈伤组织体胚发生同步化调控及组织细胞学观察[J]. 福建农林大学学报(自然科学版), 2002, 31(2): 192-194.
[22] Autran D, Huanca-Mamani W, Vielle-Calzada J P. Genomic imprinting in plants: the epigenetic version of anOedipus complex[J]. Current Opinion in Plant Biology, 2005, 8(1): 19-25.
[23] Leroy O, Hennig L, Breuninger H,et al. Polycomb group proteins function in the female gametophyte to determine seed development in plants[J]. Development, 2007, 134(20): 3639-3648.
[24] Ohyanagi H, Tanaka T, Sakai H,et al.The rice annotation project database (RAP-DB): hub forOryza sativassp. japonica genome information[J]. Nucleic Acids Research, 2006, 34(S1): D741-D744.
[25] Chen C, Xia R, Chen H,et al. TBtools, a toolkit for biologists integrating various biological data handling tools with a user-friendly interface[J/OL]. BioRxiv, 2018: 289660. http://dx.doi.org/10.1101/289660.
[26] Huang S, Gao Y, Liu J,et al. Genome-wide analysis ofWRKYtranscription factors inSolanum lycopersicum[J]. Molecular Genetics and Genomics, 2012, 287(6): 495-513.
[27] Ng D W K, Wang T, Chandrasekharan M B,et al. Plant SET domain-containing proteins: structure, function and regulation[J]. Biochimica et Biophysica Acta (BBA)-Gene Structure and Expression, 2007, 1769(5-6): 316-329.
[28] Aquea F, Vega A, Timmermann T,et al.Genome-wide analysis of the SET DOMAIN GROUP family inGrapevine[J]. Plant Cell Reports, 2011, 30(6): 1087-1097.
[29] Lei L, Zhou S L, Ma H,et al.Expansion and diversification of the SET domain gene family following whole-genome duplications inPopulus trichocarpa[J]. BMC Evolutionary Biology, 2012, 12(1): 51.
[30] Baumbusch L O, Thorstensen T, Krauss V, et al.TheArabidopsis thalianagenome contains at least 29 active genes encoding SET domain proteins that can be assigned to four evolutionarily conserved classes[J]. Nucleic Acids Research, 2001, 29(21): 4319-4333.
[31] Alvarez-Venegas R, Abdallat A A, Guo M, et al.Epigenetic control of a transcription factor at the cross section of two antagonistic pathways[J]. Epigenetics, 2007, 2(2): 106-113.
[32] Alvarez-Venegas R, Sadder M, Hlavacka A,et al. The Arabidopsis homolog of trithorax,ATX1, binds phosphatidylinositol 5-phosphate, and the two regulate a common set of target genes[J]. Proceedings of the National Academy of Sciences, 2006, 103(15): 6049-6054.
[33] Berr A, McCallum E J, Alioua A, et al. Arabidopsis histone methyltransferase SET DOMAIN GROUP8 mediates induction of the jasmonate/ethylene pathway genes in plant defense response to necrotrophic fungi[J]. Plant Physiology, 2010, 154(3): 1403-1414.
[34] Kumpf R, Thorstensen T, Rahman M A,et al.The ASH1-RELATED3 SET-domain protein controls cell division competence of the meristem and the quiescent center of the Arabidopsis primary root[J]. Plant Physiology, 2014, 166(2): 632-643.
[35] Sanchez R, Zhou M M. The PHD finger: a versatile epigenome reader[J]. Trends in Biochemical Sciences, 2011, 36(7): 364-372.
[36] Wang Y, Reddy B, Thompson J,et al. Regulation of Set9-mediated H4K20 methylation by a PWWP domain protein[J]. Molecular Cell, 2009, 33(4): 428-437.
[37] García-Alai M M, Allen M D, Joerger A C,et al. The structure of the FYR domain of transforming growth factor beta regulator 1[J]. Protein Science, 2010, 19(7): 1432-1438.
[38] Johnson L M, Bostick M, Zhang X,et al. The SRA methyl-cytosine-binding domain links DNA and histone methylation[J]. Current Biology, 2007, 17(4): 379-384.
[39] De-La-Pe?a C, Rangel-Cano A, Alvarez-Venegas R. Regulation of disease-responsive genes mediated by epigenetic factors: interaction of Arabidopsis-Pseudomonas[J]. Molecular Plant Pathology, 2012, 13(4): 388-398.
[40] Makarevich G, Leroy O, Akinci U,et al. Different polycomb group complexes regulate common target genes in Arabidopsis[J]. EMBO Reports, 2006, 7(9): 947-952.
[41] Wang D, Tyson M D, Jackson S S,et al. Partially redundant functions of two SET-domain polycomb-group proteins in controlling initiation of seed development inArabidopsis[J]. Proceedings of the National Academy of Sciences, 2006, 103(35): 13244-13249.
[42] Loidl P. A plant dialect of the histone language[J]. Trends in Plant Science, 2004, 9(2): 84-90.
[43] Ogas J, Kaufmann S, Henderson J,et al. PICKLE is a CHD3 chromatin-remodeling factor that regulates the transition from embryonic to vegetative development inArabidopsis[J]. Proceedings of the National Academy of Sciences, 1999, 96(24): 13839-13844.
[44] Cao R, Zhang Y. The functions of E (Z)/EZH2-mediated methylation of lysine 27 in histone H3[J]. Current Opinion in Genetics & Development, 2004, 14(2): 155-164.
[45] Tariq M, Paszkowski J. DNA and histone methylation in plants[J]. Trends in Genetics, 2004, 20(6): 244-251.
[46] Nic-Can G I, De la Pena C. Epigenetic advances on somatic embryogenesis of agronomical and important crops[M]// Epigenetics in Plants of Agronomic Importance: Fundamentals and Applications. Cham: Springer International Publishing, 2014: 91-109.
[47] Xiao W, Custard K D, Brown R C,et al.DNA methylation is critical for Arabidopsis embryogenesis and seed viability[J]. The Plant Cell, 2006, 18(4): 805-814.
[48] Jackson J P, Lindroth A M, Cao X,et al.Control of CpNpG DNA methylation by the KRYPTONITE histone H3 methyltransferase[J]. Nature, 2002, 416(6880): 556-560.
[49] Lindroth A M, Shultis D, Jasencakova Z, et al.Dual histone H3 methylation marks at lysines 9 and 27 required for interaction with CHROMOMETHYLASE3[J]. The EMBO Journal, 2004, 23(21): 4286-4296.
[50] Kim J M, Sasaki T, Ueda M,et al. Chromatin changes in response to drought, salinity, heat, and cold stresses in plants[J]. Frontiers in Plant Science, 2015, 6: 114.
[51] Kim J M, To T K, Ishida J,et al.Alterations of lysine modifications on the histone H3 N-tail under drought stress conditions inArabidopsis thaliana[J]. Plant and Cell Physiology, 2008, 49(10): 1580-1588.
[52] Kim J M, To T K, Ishida J,et al.Transition of chromatin status during the process of recovery from drought stress inArabidopsis thaliana[J]. Plant and Cell Physiology, 2012, 53(5): 847-856.
[53] Ding Y, Fromm M, Avramova Z. Multiple exposures to drought ‘train transcriptional responses in Arabidopsis[J]. Nature Communications, 2012, 3: 740.
[54] Liu N, Fromm M, Avramova Z. H3K27me3 and H3K4me3 chromatin environment at super-induced dehydration stress memory genes ofArabidopsis thaliana[J]. Molecular Plant, 2014, 7(3): 502-513.
[55] Cazzonelli C I, Nisar N, Roberts A C,et al.A chromatin modifying enzyme, SDG8, is involved in morphological, gene expression, and epigenetic responses to mechanical stimulation[J]. Frontiers in Plant Science, 2014, 5: 533.
