基于线粒体D-loop区和COI基因序列研究2个禾花鲤群体和野生鲤群体的遗传多样性与系统进化关系
2019-11-27潘贤辉周康奇杜雪松覃俊奇文露婷潘志忠
潘贤辉,周康奇,陈 忠,杜雪松,黄 姻,覃俊奇,文露婷,潘志忠, 邓 潜,罗 辉,叶 华,林 勇
(1.广西壮族自治区水产科学研究院,广西水产遗传育种与健康养殖重点实验室, 广西水产良种南繁基地,南宁 530021; 2.西南大学,鱼类繁育与健康养殖研究中心,重庆 402460)
禾花鲤(Procyprismerus)又名禾花鱼,在原产地因以稻田的禾花为食而得名。原产于广西桂林、灌阳、全州等地区的禾花鲤,因体色乌黑,又被称为禾花乌鲤或乌鲤[1]。这种禾花鲤的体形粗短、个体小、细叶鳞、皮薄、腹部呈紫褐色且隐约可见内脏。而产于广西融水、三江等地区的禾花鲤,较其他普通鲤鱼而言,其脊背处两侧各有金黄色的长条纹,又名为金边禾花鲤。以上两种禾花鲤都具有肉多刺少,肉质细嫩清香,无腥味的特点,一般上市个体在50~250 g之间,从外部形态和体色上明显区别于广西野生鲤鱼。
线粒体DNA(mtDNA)属于核外遗传物质,具有分子小、结构简单、进化速度快、不发生重组且遵循母系遗传等特点,已成为研究群体遗传结构、近缘物种进化关系、基因流动、物种起源的重要遗传标记[2,3]。mtDNACOI基因具有进化速率适中的特点。而mtDNA D-loop多态性丰富,进化速度快,是遗传高变区,两者都被广泛应用于种属系统进化关系和遗传多样性的研究中[4,5]。目前,禾花鲤是“十三五”广西桂西北农民扶贫攻坚中主导养殖品种,也是广西重要的地理标志产品。因此为防止禾花鲤种质资源退化、混杂现象的发生,进而影响其产品质量和经济效益,本研究拟采用mtDNA D-loop区和COI基因序列共同对全州禾花鲤(QuanzhouP.merus)、融水禾花鲤(RongshuiP.merus)和野生鲤鱼的遗传多样性与遗传结构进行分析,并初步探讨禾花鲤种质资源鉴定和评价方法,以及阐述2个地区禾花鲤与野生鲤群体的分子系统进化关系。从而为禾花鲤的种质资源保护、人工选育及资源的合理开发利用提供有效的科学依据。
1 材料与方法
1.1 材料
32尾全州禾花鲤(简称QZ)采自广西桂林绿淼生态农业有限公司七星乡禾花鲤稻田养殖基地,体长为(14.1±0.2)cm,体重为(8.3±0.2)g;32尾融水金边禾花鲤(简称RS)采自广西融水县融荣水产品养殖专业合作社金边禾花鲤科技繁育基地,体长为(34.3±1.9)cm,体重为(231.2±3.6)g;32尾野生鲤(简称YS)采自广西那龙镇广道水产养殖场,体长为(22.1±1.2)cm,体重为(201.0±4.1)g。分别取鳍条组织,置于95%无水乙醇中储存,放于-20 ℃保存备用。以上样品分别用于mtDNA D-loop区和COI基因分析。
1.2 DNA提取
每个个体剪取50 mg鳍条用于提取DNA,用mtDNA D-loop区和COI基因扩增的提取方法按照天根DNA提取试剂盒说明书进行,最终获得的DNA溶液置于-20 ℃保存备用测序。mtDNA D-loop区扩增引物序列:
D-loop Pro:5′-TCCCAAAGCTAGGATTCTAAAC TAAAC-3′
D-loop Pre:5′-TTCATCTTAACATCTTCAGTGTT ATGC-3′[4]
每个反应体系都为50 μL,含25 μL 2×Es Taq Master Mix(Dye),10 μmol/L的上、下游引物各2 μL,DNA模板3 μL,ddH2O 18 μL。PCR反应程序为:94.0 ℃预变性3 min;94.0 ℃变性30 s,55.0 ℃退火30 s,72.0 ℃延伸1 min,30个循环;最后72.0 ℃延伸10 min,4 ℃保存。PCR产物用1%的琼脂糖进行凝胶电泳检测,凝胶成像系统观察并拍照保存。COI基因扩增引物序列:
L5956-F:5′-CACAAAGACATTGGCACCCT-3′
H6855-R:5′-AGTCAGCTGAAKACTTTTAC-3′[6]。
PCR反应体系、反应程序和产物检测与扩增D-loop区相同。以上两对引物都由生工生物工程(上海)股份有限公司合成。PCR产物纯化后,委托生工生物工程(上海)股份有限公司进行双向测序,测序所用引物为PCR扩增引物。
1.3 序列分析
所获得测序结果运用Vector NTI软件进行拼接,用ClustalX 1.83软件对测序结果进行比对与校正。用DnaSP 6.12.01软件计算单倍型数、单倍型多样性(Hd)指数以及核苷酸多样性(π)、单倍型间核苷酸差异数(k)等遗传多样性指数,并进行遗传分化和中性检测分析。用MEGA5.0软件对不同序列间的变异位点、碱基组成和简约信息位点等进行分析,并计算群体内和群体间遗传距离[7],以及基于Kimura双参数法(Kimura 2-parameter,K2p)模型采用邻接法(Neighbor Joining,NJ)构建系统进化树。用Arlequin3.0软件对群体内和群体间方差进行分子方差(AMOVA)分析[8]。用Network软件中的中介法(Median joining,MJ)构建单倍型网络图。
2 结果
2.1 序列分析
将测序结果经ClustalX软件比对和人工校正后,获得93条mtDNA D-loop区序列长度为927~930 bp(QZ为30尾,RS为32尾,YS为31尾)及95条mtDNACOI基因序列长度为897~899 bp(QZ为32尾,RS为31尾,YS为32尾)。基于mtDNA D-loop区序列分析结果显示(表1),在序列中A、T、G、C碱基平均含量分别为33.4%、32.9%、14.0%和19.7%,其中A+T(66.3%)明显高于G+C(33.7%)。而基于COI基因序列分析结果表明,序列中A、T、G、C碱基平均含量为26.7%、28.5%、17.8%和27%,同样A+T(55.2%)高于G+C(44.8%)。而COI基因序列中的4种碱基在第1、2 和3位密码子分布不均匀,出现较为明显的偏倚性,其中碱基A明显偏向第3位密码子(37.6%),碱基T明显偏向于第2位密码子(40.5%),碱基G则在第1位密码子上出现频率(30.2%)最低。
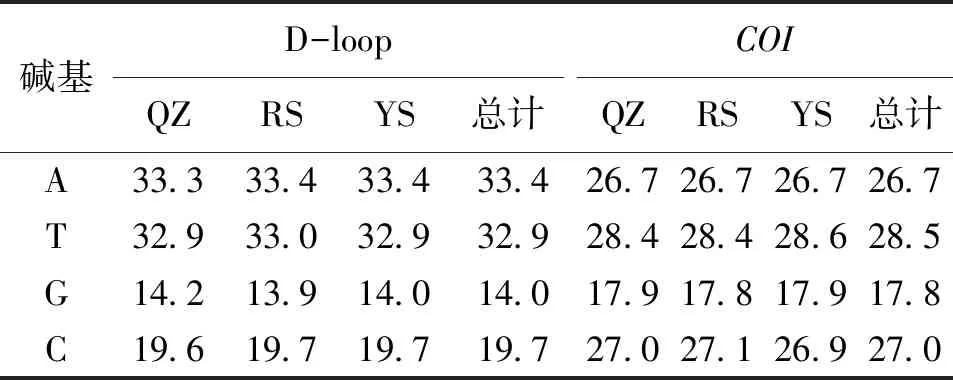
表1 基于线粒体D-loop区和COI基因序列的碱基组成Tab.1 Nucleotide composition based on mtDNA D-loop region and COI sequences %
2.2 遗传多样性分析
基于mtDNA D-loop序列分析获得3个群体的遗传多样性参数,从表2可以看出QZ的单倍型数(h)为17个,分别多于YS(15个)和RS(13个)的单倍型数量,QZ的单倍型多样性(Hd)为0.938,也大于YS(0.86)和RS(0.889)的单倍型多样性指数,而在核苷酸多样性(π)指数上,YS(0.009 31)>QZ(0.008 62)>RS(0.004 84)。基于mtDNACOI基因序列分析发现在变异位点、简约信息位点和单一突变位点上YS均大于其余两个群体。在遗传多样性参数信息上可以看出,YS的单倍型数(14个)、单倍型多样性(0.893)、核苷酸多样性(0.003 76)和平均核苷酸差异数(3.371)4个参数上都高于其余两个群体;其次在QZ中的单倍型数(14个)和单倍型多样性(0.857)2个参数要高于RS(6,0.789),但余下核苷酸多样性和平均核苷酸差异数2个参数QZ(0.002 5,2.244)低于RS(0.002 9,2.605)。综合D-loop区和COI基因序列分析结果可知,3个群体的单倍型多样性和核苷酸多样性都较高。
2.3 遗传距离和遗传分化分析
利用K2p法计算3个群体内和群体间遗传距离与遗传分化指数(表3)。基于D-loop序列的分析结果显示,3个群体的群体内平均遗传距离分别为0.004~0.009,大小顺序为YS>QZ>RS;各群体间遗传距离为0.007~0.01,其中QZ与YS群体间遗传距离最大(0.01)。基于COI基因序列的计算结果表明,RS与QZ的群体内平均遗传距离都为0.003,YS的群体内距离为0.004;RS和QZ与YS群体间的遗传距离都为0.004,而RS与QZ群体间的遗传距离为0.003。用DnaSP软件进行遗传分化和基因流分析结果揭示,基于D-loop区和COI基因序列分析总体3个群体的近邻统计(Snn)值分别为0.948 92(P<0.001)和0.829 26(P<0.001),表明3个群体已出现极显著分化现象;3个群体的基因流(Nm)为0.149 95~0.309 53和0.153 04~0.252 8,基因流均处在0~1之间,说明种群间基因交流较弱,群体间分化程度较大[9]。此外,3个群体间遗传分化指数(Fst)分别为0.150 03~0.309 67和0.153~0.250 86,遗传分化指数均大于0.15,达到显著水平(P<0.05),表明3个群体间出现显著分化(表3)。

表2 基于线粒体D-loop区和COI基因序列进行遗传多样性分析Tab.2 Parameters of genetic diversity based on mtDNA D-loop and COI sequences

表3 基于线粒体D-loop区和COI基因序列的 遗传距离和遗传分化分析Tab.3 The genetic distance and genetic differentiation based on mtDNA D-loop region and COI sequences
注:左下角表示群体间遗传距离;对角线上表示群体内遗传距离;右上角表示群体间遗传分化;*表示P<0.05。
将3个群体基于D-loop区和COI基因序列进行Tajima′s D test和Fu′s Fs test中性检验,结果均不显著,表明3个群体均符合中性进化假设,且3个群体未发生种群扩张。基于D-loop区序列的AMOVA分析显示,3个群体主要变异来源群体内(77.54%),其遗传变异高于群体间(22.46%);而基于COI基因序列AMOVA分析结果与D-loop区相似,3个群体内变异(80.86%)高于群体间遗传变异(19.14%)。
2.4 单倍型分布与分子系统进化树分析
将D-loop区和COI基因测序结果经ClustalX软件比对和Dnasp软件进行序列定义,分别获得了40与25个单倍型。在D-loop区序列的单倍型中,所有单倍型都为1个群体特有,未发现共享单倍型;根据不同单倍型在群体中的比例显示,QZ的优势单倍型有Hap11和Hap17,占QZ单倍型数的20%和16.7%;RS的优势单倍型Hap12较为明显,其占比46.9%,其次是Hap6占比为12.5%;YS的优势单倍型为Hap31,占比32.3%。在COI基因序列的单倍型中,除Hap9和Hap13为QZ和RS群体的共享单倍型外,其余群体间未存在共享单倍型;QZ的优势单倍型有Hap2、Hap3、Hap9和Hap13,占比分别为18.8%、15.6%、31.2%和15.6%;RS的优势单倍型为Hap5、Hap13和Hap17,占比分别为16.1%、54.8%和19.4%;YS的优势单倍型为Hap4和Hap16,占比分别为25%和18.8%。由此看出,各群体间拥有较少的共享单倍型,在3个群体内独有的单倍型占有量非常丰富,而独有单倍型大量的存在说明3个群体间出现一定程度的分化。
基于K2p模型分析D-loop序列的40个单倍型间遗传距离为0.021~0.336,总体平均遗传距离为0.246;COI基因序列的25个单倍型间遗传距离为0.038~0.449,总体平均遗产距离为0.186。采用NJ法构建进化树分析结果显示,在D-loop的进化树上发现YS中是Hap25、26、27、28、29单倍型聚为一支,QZ中的Hap17、18、19、20、24、35、36、38单倍型聚为一小支。而含有RS单倍型的分支中都存在着其它群体的单倍型,并没有形成该群体的单系(图1-a)。此外发现QZ单倍型Hap24与YS单倍型Hap30分别单独成一支。在COI基因的进化树上未发现3个群体单独形成的分支,每条分支内都分布着2个或2个以上不同群体来源的单倍型(图1-b)。

图1 基于线粒体D-loop区(a)和COI基因(b)序列构建的NJ系统进化树Fig.1 Neighbor-joining tree based on mtDNA D-loop and COI sequences ■为YS,▲为QZ,●为RS,存在2个图标表示共享单倍型。
2.5 单倍型网络图分析
运用Network软件中的Median-joining法,基于D-loop区的40个单倍型序列构建单倍型网络图(图2-a)显示,3个群体都以各自群体主要的单倍型为中心向其他单倍型发散,其中QZ是以Hap17单倍型为中心,YS是以Hsp25和Hap31两个单倍型为中心,RS是以Hsp12为中心。此外,在网络图上还发现QZ中的优势单倍型Hap11是由RS的Hap12单倍型突变形成。基于COI基因25个单倍型序列构建单倍型网络图(图2-b)发现未呈现单一星状散射分布,不同群体单倍型的来源都由共享单倍型Hap9(QZ和RS)直接或间接突变后形成。另外,在图中明显观察到2个回路结构特征,一是Hap9→Hap16→Hap26→Hap7→Hap12→Hap4→Hap9,二是Hap9→Hap3→Hap4→Hap9。

图2 基于线粒体D-loop区(a)和COI基因(b)序列构建禾花鲤单倍型网络图Fig.2 The haplotype network of mtDNA D-loop region and COI sequences of P.merus populations 圆圈面积大小表示单倍型频率,缩写代码同表2,红色圆圈表示缺失单倍型。
3 讨论
3.1 D-loop和COI基因序列的碱基组成
将本研究所获得的3个群体的93条D-loop区和95条COI基因序列放入NCBI数据库进行BLAST比对结果显示,所得3个群体的2种序列与鲤鱼相对应序列相似度均达到99%,说明所获得的D-loop区和COI基因序列为本次研究对象序列。从获取的3个群体D-loop区和COI基因序列的碱基组成可以看出,D-loop区和COI基因序列都表现出很强的碱基组成偏向性,即在A、T、G、C共4种碱基中,G的含量明显低于其他3种碱基的含量,这与廖健等[10]研究结果相似;另外,3个群体的D-loop区和COI基因序列中A+T(66.3%,55.2%)含量都较C+G(33.7%,44.8%)高,其他硬骨鱼类mtDNA D-loop区和COI基因序列同样具有类似情况,说明3个群体的碱基组成符合脊椎动物线粒体基因组中4种碱基分布不均这一特点[11,12]。
3.2 群体遗传多样性
一般而言,物种遗传多样性与其生存、适应能力和进化潜力紧密相关,物种的遗传变异是适应生境的必要条件[13]。同时,遗传多样性也是生物多样性形成的基础和物种进化潜能的保证[14]。而物种遗传多样性评价主要是以单倍型多样性(Hd)、核苷酸多样性(π)和单倍型平均遗传距离(P)3个内容来作为衡量指标[15],数值越大,说明种群或群体的遗传多样性越高。从mtDNA D-loop区和COI基因单倍型序列的P值上看,3个群体P值分别为0.241和0.186,而当P值大于0.01时,则表明群体间变异较大[16]。根据Grant等[17]的研究结果,将Hd=0.5、π=0.005作为分界点,在基于mtDNA-loop区研究发现3个群体的单倍型性多样性和核苷酸多样性均较高,而基于COI基因分析发现3个群体则为高单倍型多样性和低核苷酸多样性。COI基因分析结果与D-loop区存在差异,一方面是受COI基因本身特性所影响;另一方面是COI基因为编码蛋白序列,而D-loop区为非编码序列,两者在进化速率上前者要比后者慢,所以导致在遗传多样分析上COI基因的灵敏度要低于D-loop区[10,18]。在本研究中,从D-loop区和COI基因序列分析所获得的各遗传多样性参数(表2)也能看出相似的结果。因此,本研究将以D-loop区遗传多样性分析结果为准。
高单倍型多样性和高核苷酸多样性的类型在台湾铲颌鱼[19]、鲢[20]、松江鲈[21]、斑点叉尾鮰[22]等淡水鱼类上普遍存在。已有学者依据单倍型多样性和核苷酸多样性数值的大小划分为低单倍型多样性和低核苷酸多样性、高单倍型多样性和低核苷酸多样性、低单倍型多样性和高核苷酸多样性以及高单倍型多样性和高核苷酸多样性共4个模式[17]。按其分类本研究结果应属于第4类模式,预示着3个群体是由一个大而稳定的种群经过长时间演化所产生。这与全州和融水两地县志中记载的关于全州禾花鲤和融水金边禾花鲤的稻田养殖发展历史相符,2个群体的出现时间可追溯到清代乾隆年间或更早。基于mtDNA D-loop区和COI基因序列的遗传多样性分析结果可以看出3个群体遗传丰富度的大小顺序为YS>QZ>RS,说明YS对环境适应能力、生存能力和突变频率都要强于QZ和RS。总体而言,目前稻田养殖的2个禾花鲤群体的遗传多样性较YS有所下降,因此在后续人工选育工作中应加强亲本的选择,以保证在稻田养殖过程中禾花鲤种群遗传多样性丰富度,从而提高禾花鲤的种质。
3.3 种群遗传分化
遗传分化指数(Fst)又称固定系数,是衡量群体间遗传分化的重要指标,当Fst在0~0.05为极小遗传分化;在0.05~0.15为示中度遗传分化;在0.15~0.25为较大遗传分化;大于0.25,则为极大遗传分化[23,24]。本研究基于mtDNA D-loop区和COI基因序列进行AWOVA分析所得总体Fst值分别为0.224 59和0.191 43,处于0.15~0.25范围内,表明3个群体间出现较大的遗传分化。此外,遗传距离是群体间分类的一个重要依据,结合mtDNA D-loop区和COI基因序列分析所得群体内和群体间的遗传距离范围分别处于0.003~0.009和0.003~0.01之间,都远小于种群(0.05)遗传距离的分类水平[25],且群体内与群体间遗传距离差异不明显,说明3个群体间未呈现明显的种群分化。虽然在D-loop区构建的系统进化树中发现部分YS和QZ各自聚在一起,形成一支独立的单系类群,但RS、QZ与YS共同构成1个单系群,说明3个群体为3种生态型种群,并非3种有效物种。
根据mtDNA D-loop区40个单倍型序列建立的单倍型网络图可以看出在所有的单倍型中共享单倍型仅存在同一个群体内,群体间均不存在共享单倍型。出现这种分布方式可能是由于3个群体样本采样点距离较远,产生一定的地理隔离导致3个群体之间基因交流较少,另外,迁徙能力较短可能也是影响基因流的一个原因。但是来自3个群体的单倍型之间存在亲缘关系较近,突变步数1~4步不等,且存在一个群体内单倍型直接来源于另一个群体的现象,如QZ的Hap1和Hap11单倍型分别由YS的Hap2单倍型与RS的Hap12单倍型突变而来。此现象在COI基因单倍型网络图中也普遍存在。而依据COI基因单倍型序列构建的单倍型网络图显示,QZ和RS共享2个YS没有的单倍型(Hap9,Hap13),说明QZ和RS两个群体的亲缘关系较近,这与2个群体的遗传距离分析结果相同。此外,依据Tenpleton等[26]对内支(inierior clade)与末支(tip clade)的定义,可判定2个共享单倍型都为内支。而因Hap9发出的连线最多,并与其它单倍型的联系最为密切,所以推测该单倍型为古老单倍型,其进化时间要早于Hap13。同时,在COI基因单倍型网络图还存在回路结构特征,包含来自3个群体单倍型。这是由于回路中的所有单倍型都发生了趋同演化的结果,说明群体的生活环境条件与所面对环境选择压力相似,这可能是致使3群体间的遗传分化只发生在种群内的原因之一。
目前稻田养殖的全州禾花鲤和融水金边禾花鲤从体型、体态、体色等外部形态方面与野生鲤存在明显的差异,推测这可能一方面是由全州县和融水县当地人们在人工选育过程中对鲤鱼外部形态的评价标准不一致所造成的;另一方面3个群体所面临的生境相似,使得3个各群体间机体结构未发生太大变化。综合以上分析结果表明,3个群体的遗传变异主要还是来源于种群内变异(以外部形态变异为主),未达到种群或以上变异程度,表明2个地区禾花鲤群体与野生鲤同属于鲤科鱼类。另外,依据表3群体间的遗传距离大小可以看出QZ与RS亲缘关系较近,其次是QZ与YS,而RS与YS亲缘关系较远,推测2个禾花鲤群体都是由YS演变而来,且QZ出现的时间要早于RS。
