高温胁迫下香菇基因组甲基化差异分析*
2019-11-08王守现宋庆港
宋 爽 ,刘 宇 **,高 琪 ,赵 爽 ,王守现 ,宋庆港
(1.北京市农林科学院 植物保护环境保护研究所,北京 100097;2.北京市食用菌工程技术研究中心,北京 100097;3.农业部都市农业(北方)重点实验室,北京 100097)
DNA甲基化是目前研究较为透彻的表观遗传修饰,常发生在胞嘧啶的第5位碳原子上,在DNA甲基转移酶的作用下,将S -腺苷甲硫氨酸上甲基转移到胞嘧啶上[1]。作为一种重要并相对稳定的表观遗传修饰策略,DNA甲基化通过稳定染色质结构、调控基因表达、参与基因重组等方式,在原核生物、动物、植物等多种生物的基本生物学过程中发挥重要作用[2]。在真菌中,DNA甲基化可能参与形态建成、逆境胁迫和次级代谢过程,但对其具体功能研究较少。目前,对基因组甲基化水平分析手段主要采用甲基化敏感扩增多态性(methylation sensitive amplification polymorphism,MSAP)技术,利用同尾酶HpaⅡ和MspⅡ对CCGG序列中的胞嘧啶甲基化反应的不同,对基因组范围内的甲基化状态变化进行分析[3]。
根据中国食用菌协会统计,2017年我国香菇总产量为986.51万吨,位居所有食用菌品种之首[4]。香菇为中低温型变温结实性菇类,菌丝最适生长温度为24℃~27℃,在生产中如遇到较高的环境温度,常会引起菌丝死亡和菌棒腐烂[5]。由于全球变暖,高温胁迫已经影响到我国许多地区的农业生产,因此耐高温能力成为食用菌抗逆性评价的重要指标,而食用菌抵御高温胁迫的机制也成为研究热点。目前,已经有研究报道了香菇受高温胁迫后生理响应和耐热相关基因表达变化,但关于香菇高温条件下基因组表观遗传变化还未见报道[5]。本研究利用MSAP技术分析了耐高温和高温敏感香菇菌株在热胁迫处理后基因组DNA甲基化水平和甲基化模式变化,为表观遗传修饰在香菇逆境应答中的作用研究提供参考。
1 材料与方法
1.1 试验材料
1.1.1 供试菌株
供试材料为课题组前期筛选的耐热香菇菌株(JZB2102050)和热敏感香菇菌株(JZB2102030),由北京市农林科学院植物保护环境保护研究所保存。
1.1.2 引物与试剂
所用引物均由生工生物工程(上海)股份有限公司合成;DNA Marker、DNA聚合酶、限制性内切酶HpaⅡ和MspⅡ, 购自宝生物工程(大连)有限公司;基因组DNA提取试剂盒,购自德国QIAGEN公司。
1.2 试验方法
1.2.1 菌丝培养
利用打孔器将活化好的香菇JZB2102050和JZB2102030菌丝体分别定量接种在PDA培养基中心,25℃恒温培养箱中培养5 d后,继续置于35℃培养箱中培养4 d,刮取100 mg的菌丝,利用试剂盒提取基因组DNA。25℃恒温培养9 d的香菇菌丝为对照组。
1.2.2 基因组MSAP分析
双酶切:分别用各10 U的HpaⅡ/EcoRⅠ和MspⅡ/EcoRⅠ双酶切400 ng香菇基因组DNA,酶切体系50 μL,37℃酶切12 h。
接头连接:2条EcoRⅠ接头和HpaⅡ/MspⅡ接头分别等量混合,95℃变性5 min合成双链接头。连接体系:双链EcoRⅠ(5 pmol·μ L-1)接头0.5 μL,双链 HpaⅡ /Msp Ⅱ(5 pmol·μ L-1)接头 5 μL,酶切产物 12 μL,10×T4连接酶 buffer 2 μL,T4连接酶 0.5 μL,16℃连接过夜。
预扩增:反应体系为连接产物2 μ L,10 × Ex Taq Buffer 5 μ L,dNTP 5 μ L,预扩增引物各 1 μ L,Ex Taq酶0.2 μL,加ddH2O补至50 μL。反应条件:95℃预变性3 min;95℃变性30 s,55℃退火40 s,72℃延伸1 min,共30个循环;72℃延伸10 min; 4℃保存。
选择性扩增:将预扩增产物稀释50倍后作为选择性扩增模板,扩增引物序列见表1,共64对。反应体系为稀释后预扩增产物5 μL,10 × Ex Taq buffer 5 μ L,dNTP 5 μ L,EcoR Ⅰ选择性扩增引物 1 μ L,HpaⅡ/MspⅡ选择性扩增引物1 μ L,Ex Taq酶0.2 μ L,ddH2O 补至 50 μ L。反应条件:95℃预变性5 min;95℃变性30 s,65℃退火30 s,72℃延伸1 min,10个循环,每个循环退火温度下降1℃;95℃变性30 s,55℃退火30 s,72℃延伸1 min,共25个循环;72℃延伸10 min,4℃保存。
聚丙烯酰胺凝胶电泳:选择性扩增产物进行6%聚丙烯酰胺凝胶电泳,银染观察并拍照。CCGG位点未发生甲基化,HpaⅡ/EcoRⅠ和MspⅡ/EcoRⅠ酶切均有条带;Ⅱ型(1,0)表示CCGG位点发生半甲基化,前者酶切有条带,后者酶切无条带;Ⅲ型(0,1)表示CCGG位点发生全甲基化,前者酶切无条带,后者酶切有条带;Ⅳ型(0,0)表示两者酶切均无条带,CCGG位点发生超甲基化。

表1 引物及接头序列Tab.1 Sequence of primers and adaptors
2 结果与分析
2.1 高温胁迫下香菇基因组DNA甲基化水平变化
采用64对选择性扩增引物对高温胁迫处理及对照组的香菇JZB2102050和JZB2102030基因组DNA进行MSAP分析,见表2。
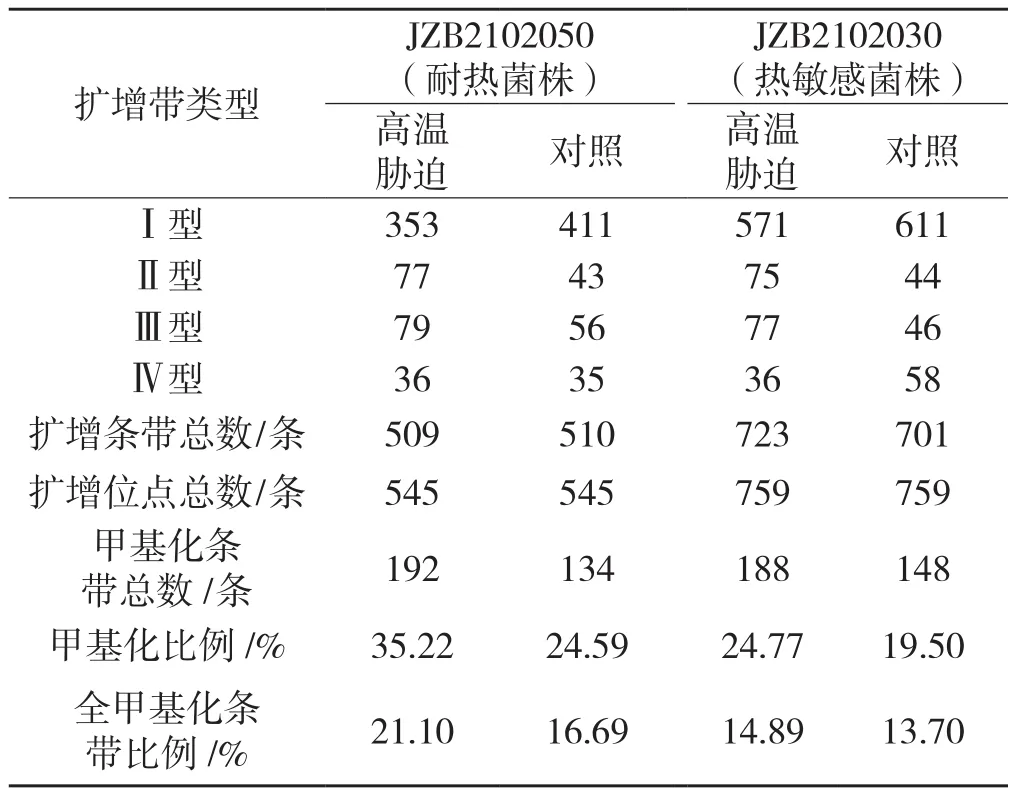
表2 高温胁迫下香菇基因组DNA甲基化水平Tab.2 DNA methylation level of Lentinula edodes after heat stress
由表2可以看出,耐热菌株和热敏感菌株对照组DNA甲基化比例分别为24.59%和19.50%,高温胁迫后甲基化比例分别上升了43.22%和27.03%,全甲基化比例分别上升了26.42%和8.69%,说明高温可诱导香菇菌株甲基化水平提高,且耐热菌株甲基化水平变化大于热敏感菌株。
2.2 高温对香菇基因组DNA甲基化模式变化的影响
进一步对2个香菇菌株处理组和对照组的扩增条带带型变化进行分析,归纳为以下13种带型,其中A型表示处理组与对照组之间甲基化模式无变化,B型表示处理组发生甲基化,C型表示处理组发生去甲基化,见表3。
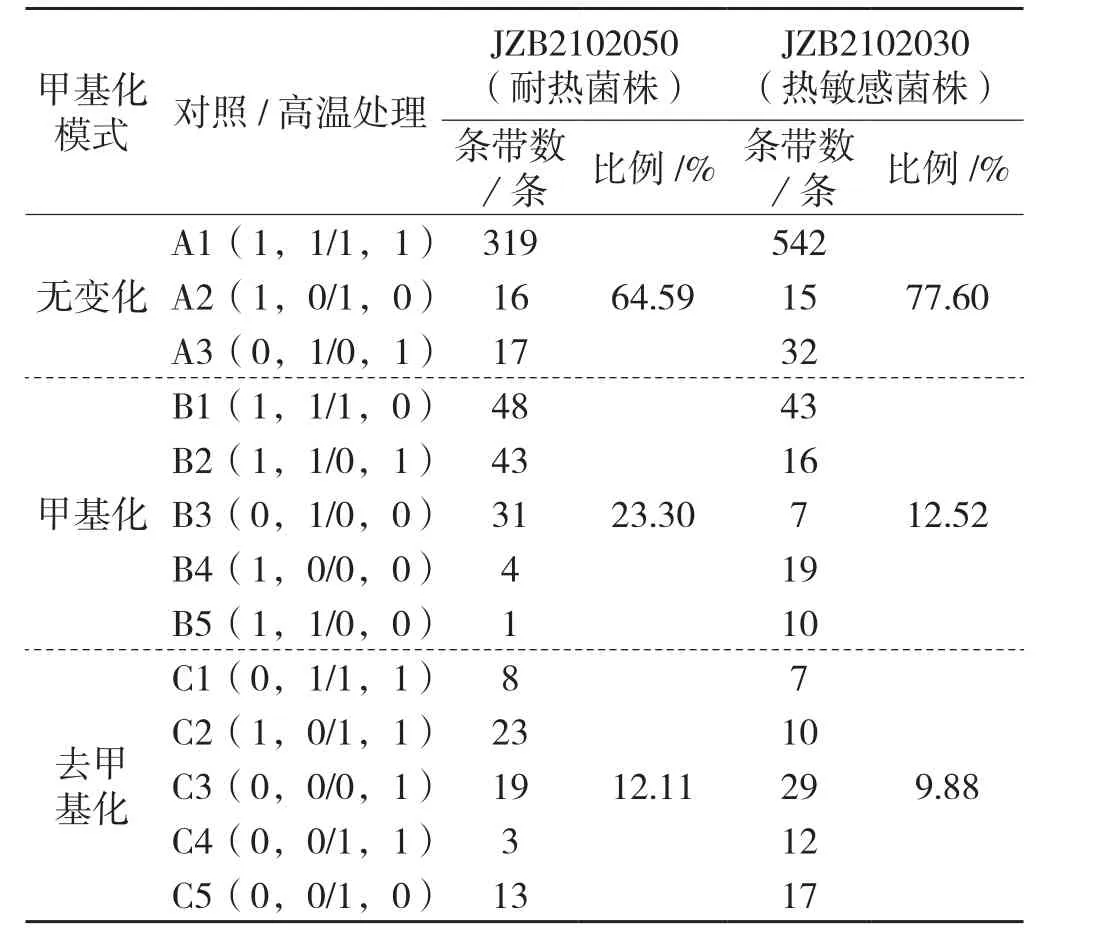
表3 高温胁迫下香菇基因组DNA甲基化模式变化Tab.3 Methylation pattern variation of Lentinula edodes after heat stress
由表3可以看出, 扩增条带最多的带型为A1(1,1/1,1),即处理组与对照组在该位点未发生甲基化。在所有甲基化条带中,耐热菌株JZB 2102050发生甲基化和发生去甲基化条带比例分别占56.19%和29.20%,热敏感菌株JZB2102030发生甲基化和发生去甲基化条带比例分别占43.78%和34.56%(见图1),表明在受到高温胁迫后,耐热菌株主要发生甲基化,而热敏感菌株主要发生去甲基化。

图1 热敏感菌株JZB2102030发生甲基化(B5)和去甲基化(C3)位点电泳条带图Fig.1 Methylation and demethylation bands of heat-sensitive strain JZB2102030 after heat stress
3 讨论
食用菌生长发育需要适宜的温度、湿度、营养、光照、pH等条件,在遇到环境胁迫时,为保持基因组稳定,DNA甲基化模式改变为时常发生的生理反应。植物在胁迫后甲基化变异研究较多,殷欢等[6]研究发现黄瓜幼苗在高温胁迫后叶片甲基化水平提高,曾子入等[5]研究发现热胁迫后耐热萝卜材料主要发生去甲基化过程,而不耐热材料发生超甲基化频率较高,但在食用菌中关于DNA甲基化在胁迫过程中的响应研究较少。肖冬来等[6]利用F-MSAP技术分析了光照对广叶绣球菌甲基化的影响,结果表明,光照可诱导全甲基化率上调,半甲基化率下调。蓝丽雯等[7]研究发现,抑制灵芝DNA甲基化可促进灵芝酸的生物合成,这种作用是通过提高灵芝酸合成酶基因的表达来实现的。本研究以香菇耐热菌株JZB 2102050和热敏感菌株JZB2102030为材料,研究了菌丝在高温胁迫前后的DNA甲基化变化,结果显示,2个香菇菌株在胁迫处理后甲基化水平都表现为上升,但耐热菌株甲基化水平变化大于热敏感菌株;香菇在受到高温胁迫后,甲基化和去甲基化同时发生,耐热菌株主要发生甲基化,而热敏感菌株主要发生去甲基化,这对深入研究胁迫环境下香菇适应性进化提供了一定参考。
