羊传染性脓疱病毒OVIFNR 蛋白结构分析与B 细胞表位预测
2019-11-04成伟伟陈伯祥赵子惠常攀峰
成伟伟,陈伯祥,杨 明,赵子惠,常攀峰
(甘肃省畜牧兽医研究所,甘肃平凉 744000)
羊传染性脓疱(俗称羊口疮)是由羊传染性脓疱病毒(ORFV)引起的一种以山羊和绵羊嘴唇和舌鼻等部位形成水泡、脓疱和结成厚痂的一种人兽共患传染病[1]。该病毒主要感染2~6 月龄的羔羊和部分免疫力下降的成年羊,被感染羔羊常因继发感染而死亡,被感染成年羊可因进食困难导致减产甚至死亡[2]。该病在规模化羊场流行广泛,给养羊业造成了严重经济损失。
OVIFNR 蛋白是ORFV 的抗性蛋白[3],具有抗干扰素功能。OVIFNR 蛋白依靠其羧基末端的一段保守性基序,竞争性结合dsRNA 和病毒复制中间体,从而抑制干扰素(IFN)诱导的蛋白激酶PKR 的激活,达到阻止宿主细胞发挥抑制病毒蛋白翻译的作用[4-5]。目前,对OVIFNR 蛋白的结构和功能还不是很清楚。所以,本研究以从羊口疮确诊病例的嘴唇结痂扩增所得的OVIFNR基因序列为研究对象,应用生物信息学方法进行OVIFNR基因序列比较和ORFV 遗传进化分析,以及OVIFNR蛋白的理化特性、二级结构、三级结构和B 细胞表位预测分析,以期为OVIFNR 蛋白的功能研究和ORFV 检测方法的建立提供理论依据。
1 材料与方法
1.1 材料
甘肃省某羊场羊口疮确诊病羊的嘴唇结痂。ExTaqDNA聚合酶、dNTP、IPTG、X-gal、pMD18-T,均购自宝生物工程有限公司;DNA 提取试剂盒,购自QIAGEN 公司;DH5α 感受态细胞,购自天根生化有限公司;胶回收和质粒小提试剂盒,购自AXYGEN 公司。
1.2 结痂组织DNA 提取
取结痂组织100 mg 置于研钵中,加入液氮研成粉末;参照组织基因组DNA 提取试剂盒(QIAGEN)说明书的要求和步骤,提取总DNA;经紫外分光光度计检测浓度和纯度,-80 ℃保存。
1.3 引物设计、基因扩增
参照GenBank公布的ORFVOVIFNR(JN565695.1)基因序列,应用Primer 5.0软件设计引物。上引物:CCCAAGCTTATGGCCTGCGAGTGCGCGTCT(下划线部分为HindIII 酶切位点);下引物:CGCGGATCCTTAGAAGCTGATGCCGCAGTTGTTGATG(下划线部分为BamHI 酶切位点)。引物均由上海桑尼有限公司合成。以1.2 提取的基因组DNA为模板,加入上下游引物进行PCR 反应。反应体系为:模板5 μL、premix ExTaq25 μL,上下引物各1 μL,ddH2O 加至50 μL。反应条件:94 ℃预变性5 min;94 ℃变性45 s,59 ℃退火45 s,72 ℃延伸1 min,35 个循环;最后72 ℃延伸10 min,4 ℃保存。扩增结束后,取反应液5~10 μL,用1%的琼脂糖凝胶电泳检测。参照PCR 产物纯化试剂盒(AXYGEN)说明,将PCR 产物切胶回收纯化,与pMD18-T 载体连接,连接产物转化到大肠杆菌DH5α 感受态细胞;涂布含氨苄青霉素、X-gal 和IPTG 的选择平板上培养12~16 h,然后进行蓝白斑筛选;挑取白色单菌落接种于LB 液体培养基,摇床培养6 h 后进行菌液PCR 鉴定。扩增条件与反应参数与上述PCR 反应相同。
1.4 序列测定和遗传进化分析
将上述PCR 鉴定为阳性的菌株,送上海桑尼测序公司测序;应用DNAstar 软件,对测序得到的基因序列和GenBank 公布的参考序列,分别进行核苷酸和氨基酸水平的同源性分析和遗传进化树构建[6-7]。
1.5 OVIFNR 蛋白理化特性和二、三级结构预测分析
运用在线工具ProtParam(网址为https://web.expasy.org/protparam/),分析OVIFNR 蛋白的理化特性[8];利用DNAstar 软件的KyteDolittle 方法,预测OVIFNR 蛋白的亲水性和疏水性;利用在线软件TMHMM 和SingnalIP 分析工具(网址为http://www.cbs.dtu.dk/services/TMHMM/和http://www.cbs.dtu.dk/services/SignalP/),预测OVIFNR蛋白分子的跨膜区和信号肽[9];利用DNAstar 软件,进行OVIFNR 蛋白的二级结构预测分析[10]。将OVIFNR 蛋白的氨基酸序列提交至在线工具Swiss-Model(网 址 为https://swissmodel.expasy.org/),构建OVIFNR 蛋白的三级结构模型[11-12]。
1.6 OVIFNR 蛋白的B 细胞表位预测
以OVIFNR 蛋白二级结构预测结果为参数,运用在线工具ABCpred(网址为http://crdd.osdd.net/raghava/abcpred/),综合分析预测OVIFNR 蛋白B 细胞表位[13-14]。
2 结果
2.1 序列测定及遗传进化分析
PCR 扩增得到大小约552 bp 的基因片段,与预期结果一致。序列测定结果显示,OVIFNR基因大小为552 bp,编码184 个氨基酸,GC 含量为60.51%。将该基因序列提交GenBank,获得序列号为KF916586,将所含该基因的毒株命名为orfvchGanSu。将GenBank 上公布的参考毒株(挑选了14 株,具体信息见表1)以及orfv-chGanSu 的OVIFNR基因进行核酸序列的多序列比对。结果(表2)显示:有两对毒株的OVIFNR基因序列同源性为100%,分别是Ena 和Matsumoto 株、Hoping 和Nantou 株。orfv-chGanSu 的OVIFNR基因序列与Hoping 和Nantou 株的同源性最高,为99.6%;orfv-chGanSu 与Hoping 和Nantou 株 在198 位和273 位上分别有2 个单核苷酸不同(在198 位 上orfv-chGanSu 是C,Hoping 和Nantou是T,在273 位 上orfv-chGanSu 是T,Hoping 和Nantou 是C),但都没有氨基酸序列的不同。orfvchGanSu 和D1701 株的同源性最低,为95.3%;orfv-chGanSu 和NZ2、OV-SA00、OV-IA82 株的同源性分别为96.0%、97.5%、96.7%。这些数据说明ORFV 各毒株的OVINR基因发生了突变(包括orfv-chGanSu)。
这14株参考毒株和orfv-chGanSu 株OVIFNR 蛋白的氨基酸序列比对结果见表2 左下部分。在所有105 个比对组合中,有7 个同源性为100%,其中Hoping、Nantou、ShanX 和orfvchGanSu 株的OVIFNR 蛋白氨基酸序列完全同源,Ena 和Matsumoto 完全同源。orfv-chGanSu与已公布的参考株OVIFNR 蛋白氨基酸序列的同源性为94.1%~100%,其中与D1701、NZ2、OV-IA82、OV-SA00 的同源性分别为94.1%、95.1%、95.1%、97.8%。通过遗传进化树可以发现,本次克隆的OVIFNR基因毒株与Hoping、ShanX、Nantou 株的关系最近,处于同一分支(图1)。这一点也为病毒存在的地理差异提供了证据(orfv-chGanSu、Hoping、ShanX、Nantou 都是国内毒株)。
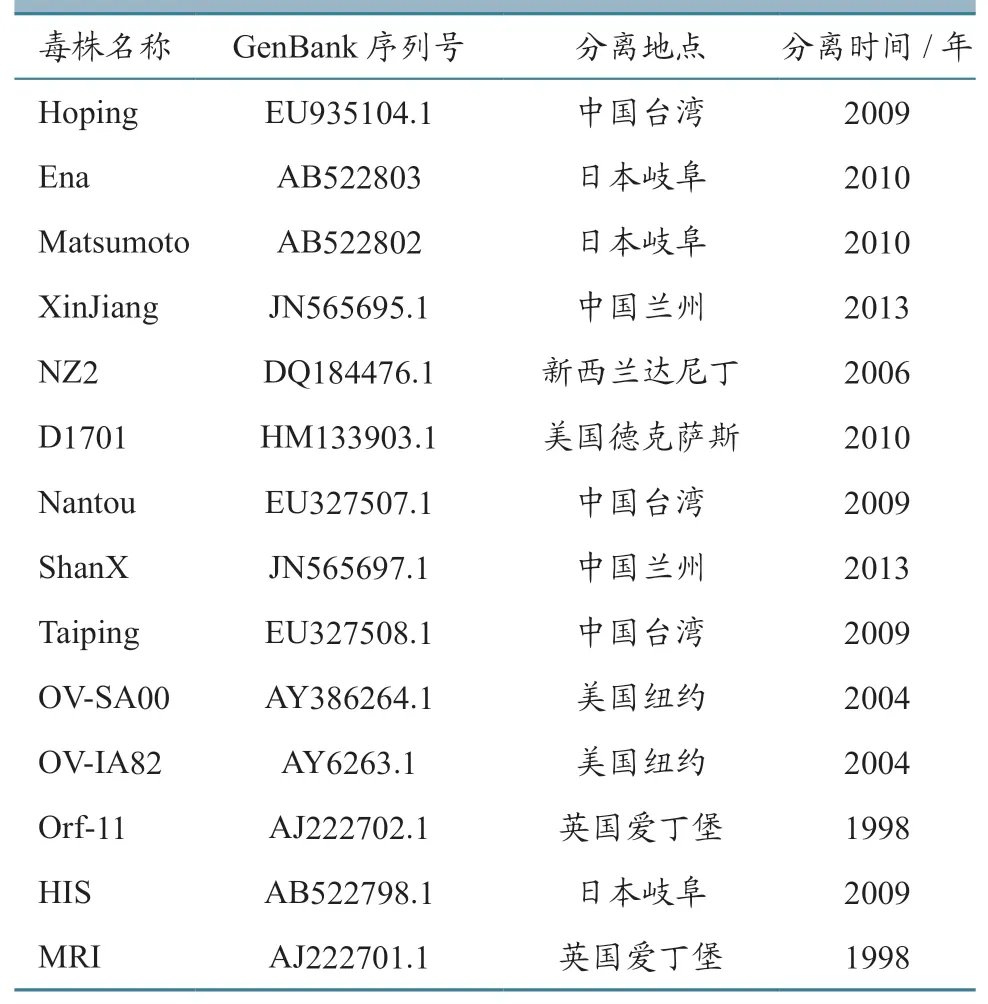
表1 14 株ORFV 参考毒株信息

表2 ORFV 各毒株的OVIFNR 基因核酸和氨基酸同源性比对结果
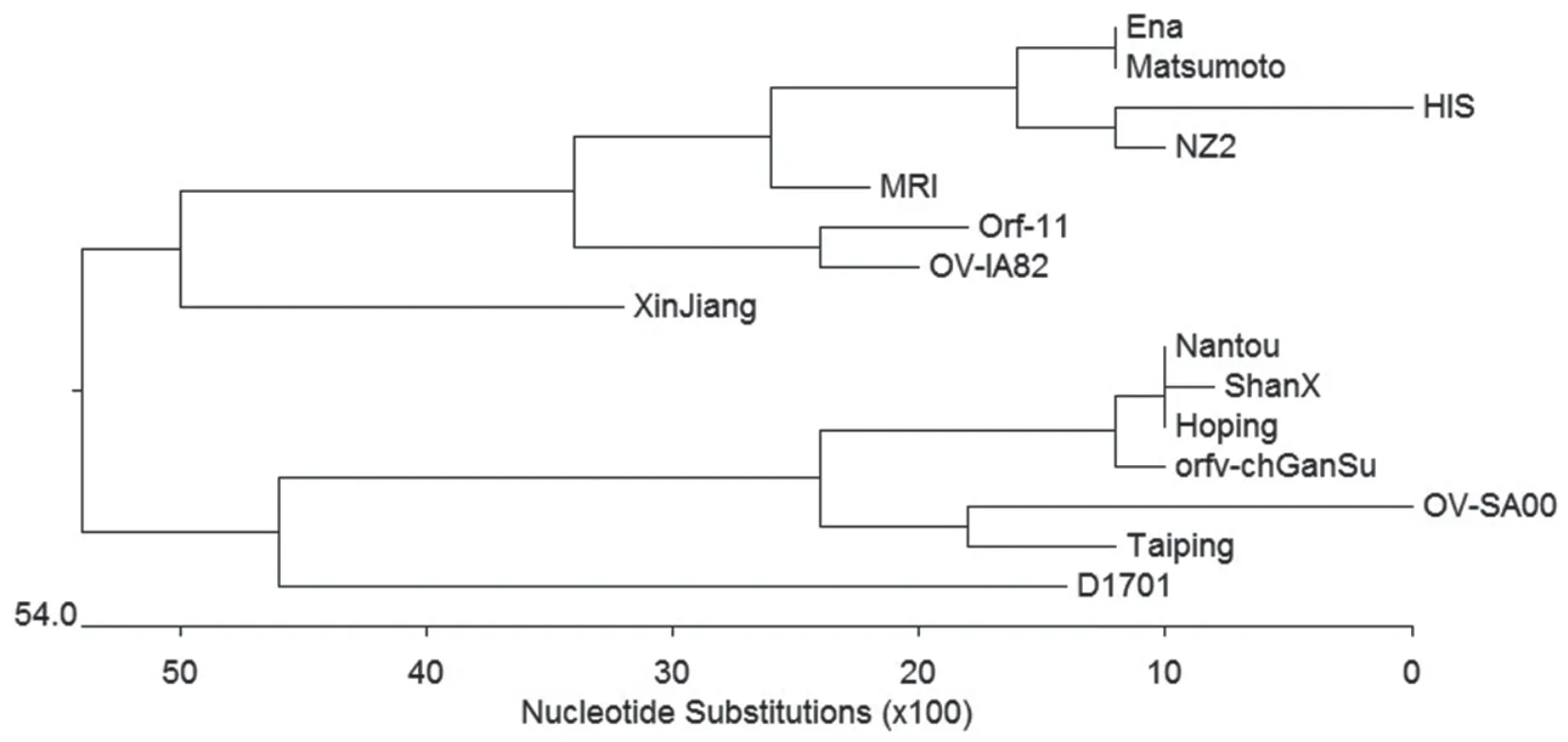
图1 所扩增的OVIFNR 基因毒株与参考毒株遗传进化树
2.2 OVIFNR 蛋白的理化特性和二、三级结构预测分析
orfv-chGanSu 的OVIFNR蛋白理化特性如表3 所示。用KyteDolittle 法进行亲疏水性分析发现,OVIFNR 蛋白具有较强的亲水性,OVIFNR 蛋白亲水性区主要集中在12~29、30~46、47~64、65~85、120~140、156~165 等6 个区段(图2);使用TMHMMH 和SOSUI服务器分析发现,OVIFNR 蛋白无跨膜区和信号肽。
OVIFNR 蛋白的二级结构预测结果如图3 所示:OVIFNR 蛋白的二级结构主要包括α 螺旋、β 折叠、β 转角和无规卷曲4 种类型,所占比重分别为68.64%、7.56%、15.13%和9.18%;OVIFNR蛋白的α 螺旋主要位于1~41、67~90、94~105、111~130、150~174 区段,β 折叠主要位于42~43、47~50、59~61、141~144、174~177、182~184 区段,β 转角分别位于43~48、51~54、56~59、61~63、91~93、105~106、131~137、138~141、146~147、177~181 区段,无规卷曲分别位于56~58、63~67、93~95、106~112、147~149、181~182 区 段。从 构建的OVIFNR 蛋白的三级结构(图4)可以看出,OVIFNR 蛋白结构比较简单,在OVIFNR 蛋白三级结构中α 螺旋占据较多的构象。
2.3 OVIFNR 蛋白B 细胞表位预测
利用ABCpred 预测的B 细胞抗原表位见表4。以预测的OVIFNR 蛋白二级结构的β 转角和无规卷曲为参数,综合筛选出OVIFNR 蛋白的优势B 细胞抗原表位位于41~48、51~54、56~67、91~95、105~112、131~141、146~149、177~181 区段。

表3 OVIFNR 蛋白的理化特性预测结果

图2 OVIFNR 蛋白的亲疏水性预测结果

图3 OVIFNR 蛋白二级结构预测结果

图4 OVIFNR 蛋白三级结构建模结果
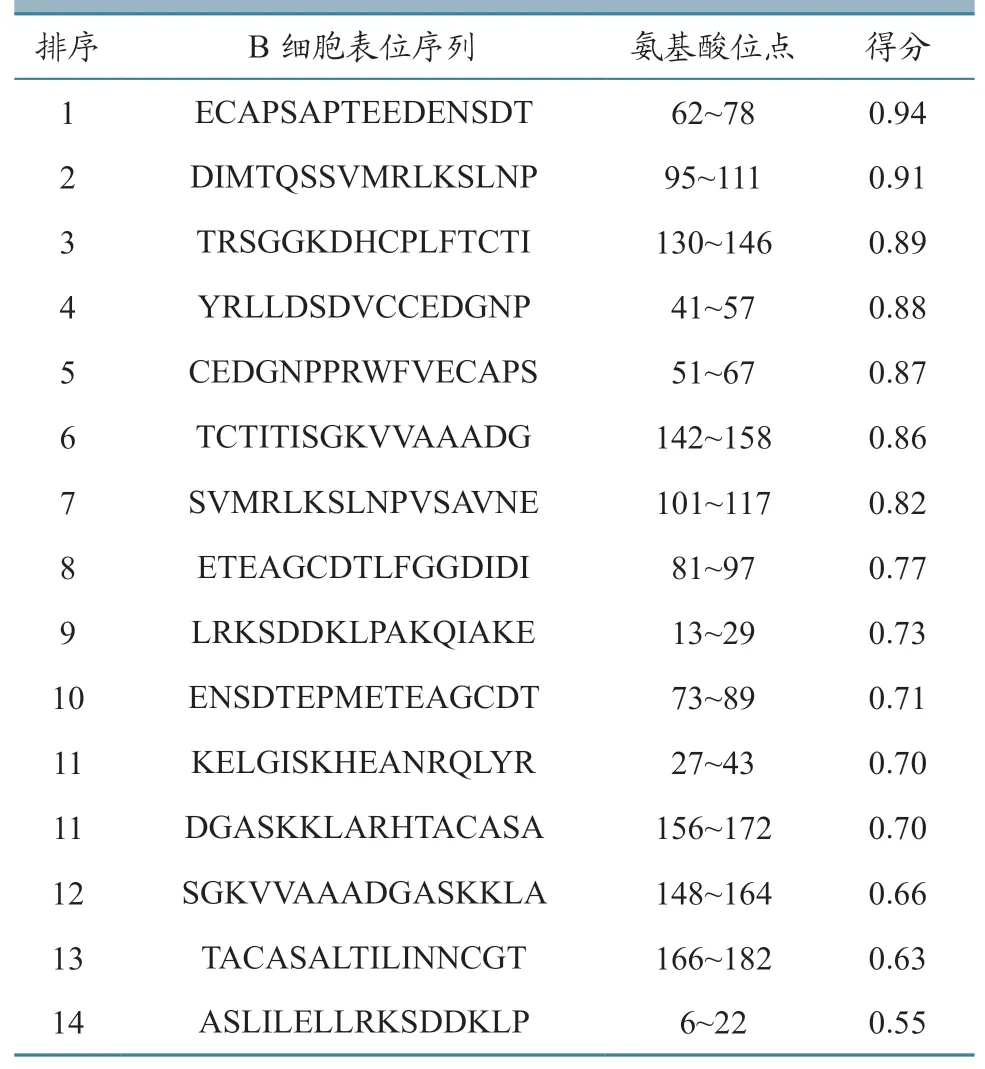
表4 OVIFNR 蛋白B 细胞表位预测结果
3 讨论
将本研究中扩增所得的OVIFNR基因序列与14 株ORFV 的OVIFNR基因序列比较发现,没有同源性达到100%的毒株,与Hoping 和Nantou 株的同源性最高达到了99.6%。氨基酸序列比对发现,Hoping、Nantou、ShanX 和orfv-chGanSu 等 毒 株的OVIFNR 蛋白氨基酸序列完全同源。通过遗传进化树可以发现,本次克隆的OVIFNR基因毒株与Hoping、ShanX、Nantou 等毒株的关系最近,处于同一分支。这可能与地理分类有关,因为orfvchGanSu、Hoping、ShanX、Nantou 都是国内毒株。OVIFNR 蛋白理化特性预测表明,OVIFNR 蛋白半衰期长达30 h。如果将该蛋白作为免疫抗原,那么其在宿主体内能够稳定存在较长时间。OVIFNR 蛋白有较强的亲水性,无跨膜区,推测该蛋白可以实现高表达量的可溶性表达。预测的OVIFNR 蛋白二级结构含有丰富的α 螺旋结构和β 转角,推测出OVIFNR 蛋白比较稳定,含有较多的B 细胞抗原表位。三级结构的构建可以较直观地展现OVIFNR蛋白形态,从而为OVIFNR 蛋白功能的进一步研究提供可视化依据。
目前,基于生物信息学的B 细胞抗原表位预测方法发展迅速,已出现了多种方法,如基于多项物理、化学参数预测的Bcepred 方法,基于模拟肽的EpiSearch 在线方法和基于人工神经网络的ABCpred 方法,其中ABCpred 是应用比较广泛和具有较高准确率的预测方法[15-16]。本研究运用ABCpred 方法预测OVIFNR 蛋白B 细胞抗原表位,并以蛋白质二级结构预测结果为参数进行综合筛选,得到了具有较高可信度的优势B细胞抗原表位。
