猪繁殖与呼吸综合征病毒3'-UTR碱基缺失对病毒复制影响的初步研究
2019-10-10熊俊垚杨勇博田志军蔡雪辉安同庆
熊俊垚,赵 款,于 芳,杨勇博,田志军,蔡雪辉,安同庆
(中国农业科学院哈尔滨兽医研究所兽医生物技术国家重点实验室,黑龙江哈尔滨150069)
猪繁殖与呼吸综合征(Porcine reproductive and respiratory syndrome,PRRS)是由PRRS 病毒(PRRS virus,PRRSV)引起的以妊娠母猪繁殖障碍和仔猪呼吸症状为主要特征的传染病。该病流行范围广,对世界养猪业造成了严重的危害。我国于1996 年首次分离到PRRSV,即CH-1a 株[1]。随后几年中,PRRSV 蔓延到我国各主要养猪省份。2006 年,我国暴发了高致病性PRRS (Highly pathogenic PRRS,HP-PRRS),其具有传播迅速、发病率和死亡率高等特点,给我国养猪业造成了重大的经济损失[2]。
PRRSV 属于动脉炎病毒科、动脉炎病毒属成员。其基因组为不分节段的单股正链RNA,全长约15 kb,5' 端有帽子结构,3' 端有poly(A)尾巴结构,基因组至少编码9 个开放性阅读框(Open reading frames, ORFs), 包括 ORF1a、 ORF1b、 ORF2、ORF2a、ORF3~ORF7[3-4]。在PRRSV 复制过程中,首先合成自身的RNA 依赖性RNA 聚合酶(RNA-dependent RNA polymerase,RdRp)结合到3'-UTR(Untranslated region,UTR)上,才能开始病毒负链基因组的合成,随后以负链基因组为模板,合成子代病毒的基因组[5-7]。已有研究表明,与经典PRRSV 相比,HP-PRRSV 复制效率在非洲绿猴胚胎肾细胞(Marc-145)和猪肺泡巨噬细胞(PAM)中高出10~100倍左右[8]。
本研究对HP-PRRSV 和经典PRRSV 进行序列比对,发现前者3'-UTR 中存在规律性的碱基缺失。但目前关于PRRSV 3'-UTR 的研究较少,该缺失对病毒复制的影响尚不可知。本研究基于HP-PRRSV HuN4 株感染性克隆,构建突变体病毒,进一步研究缺失碱基对PRRSV 复制的影响,为深入研究3'-UTR 在PRRSV 复制过程中的作用提供了实验依据。
1 材料与方法
1.1 主要实验材料 HP-PRRSV HuN4 株感染性克隆pOK-HuN4 由本实验构建并保存;Marc-145 细胞由本实验室保存。Q5 超保真DNA 聚合酶、限制性内切酶和T4 DNA 连接酶购自NEB 公司;X-treme GENE HP DNA Transfection Reagent 购自Roche 公司;PRRSV M 蛋白单克隆抗体(MAb)由田志军研究员惠赠;羊抗鼠IgG-FITC 购自Sigma-Aldrich 公司。胶回收试剂盒购自OMEGA 公司;质粒大提试剂盒购自Invitrogen 公司。
1.2 PRRSV 3'-UTR 基因序列的比对分析 为了研究HP-PRRSV 和经典PRRSV 在3'-UTR 序列上的差异,从NCBI 中下载了40 个PRRSV 3'-UTR 基因序列,其中HP-PRRSV 34 个,经典PRRRSV 6 个,且病毒范围广,覆盖我国18 个省份区域。利用DNAStar (Megalign)对40 个PRRSV 3'-UTR 基因序列进行序列比对。
1.3 病毒RNA 二级结构预测 通过在线软件Mfold 预测RNA 二级结构,使用服务器默认参数:37 ℃,1 mol/L NaCl。通过预测得到CH-1、HuN4、HuN4-(17+G)和HuN4-△SL2 4 株病毒的3'-UTR 二级结构,使用RNAviz 2.0 软件编辑上述二级结构。
1.4 引物设计和合成 根据PRRSV HuN4 株基因组序列(EF635006), 使用 Oligo 7 设计 PRRSV 3'-UTR 区域的引物。引物由吉林库美生物技术有限公司合成(表1)。
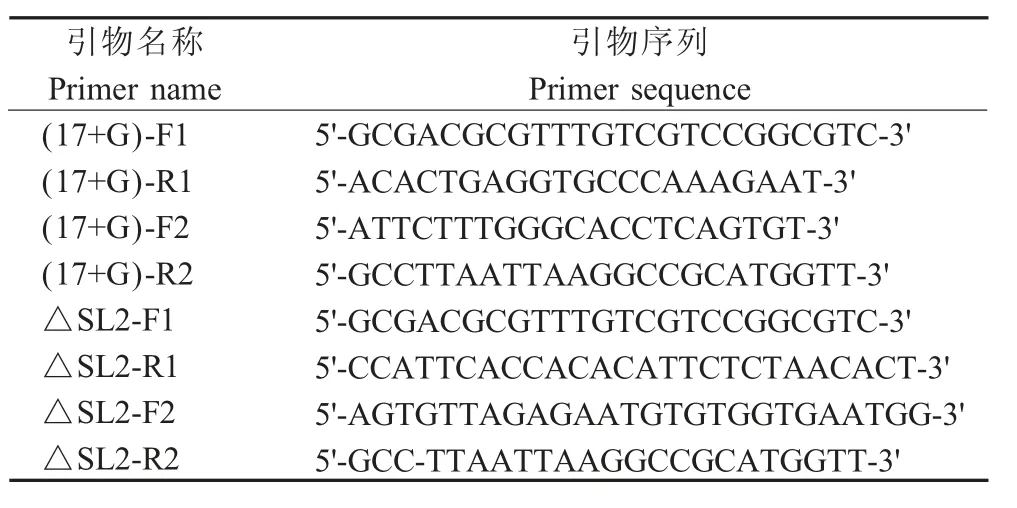
表1 本研究所用引物序列Table 1 Primers used in this study
1.5 突变质粒的构建和鉴定 以pOK-HuN4 株感染性克隆质粒为模板,采用(17+G)-F1/(17+G)-R1、(17+G)-F2/(17+G)-R2 为引物分别扩增并命名为A、B 片段,△SL2-F1/△SL2-R1、△SL2-F2/△SL2-R2为引物分别扩增并命名为C、D 片段(图1)。PCR 反应条件为:98 ℃30 s;98 ℃10 s、54 ℃30 s、72 ℃1 min,共35 个循环;72 ℃10 min。经电泳鉴定并胶回收A、B、C 和D 片段后,以A 和B 片段为模板,(17+G)-F1/(17+G)-R2 为引物进行融合PCR,融合PCR 产物命名为AB 片段,反应条件同上;以C和D 片段为模板,△SL2-F1/△SL2-R2 为引物进行融合PCR,融合PCR 产物命名为CD 片段,反应条件同上。电泳鉴定后分别胶回收AB 和CD 片段,以AscⅠ和MluⅠ双酶切AB 和CD 片段,分别克隆至pOK-HuN4,构建突变质粒pOK-HuN4-3'-UTR-(17+G),pOK-HuN4-3'-UTR-△SL2。将亲本质粒pOKHuN4 以及突变质粒pOK-HuN4-3'-UTR-(17+G)和pOK-HuN4-3'-UTR-△SL2 分别以(17+G)-F1/(17+G)-R2、(17+G)-F1/(17+G)-R2、△SL2-F1/△SL2-R2 为引物进行PCR 鉴定。同时将突变质粒pOK-HuN4-3'-UTR-(17+G)和pOK-HuN4-3'-UTR-△SL2 送于吉林库美生物技术有限公司测序鉴定。
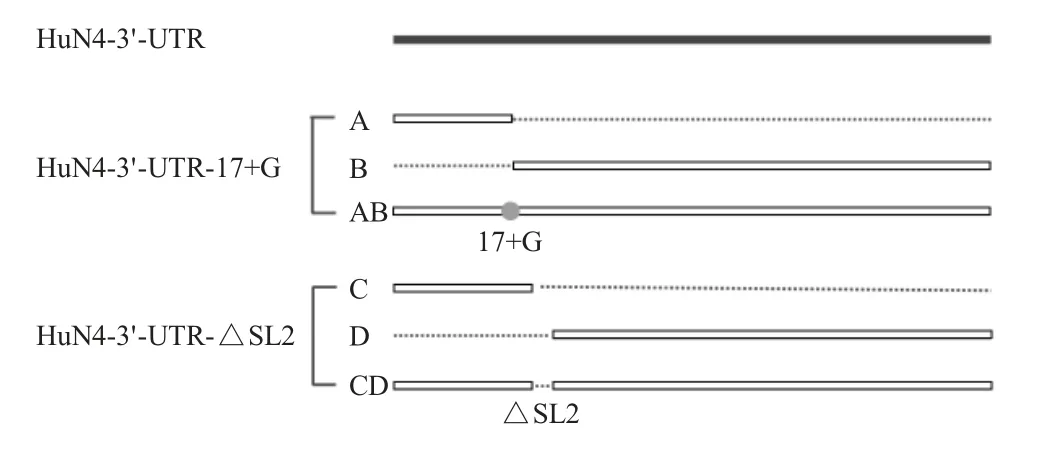
图1 突变体HuN4-3'-UTR-(17+G)和HuN4-3'-UTR-△SL2 的构建示意图Fig.1 Schematic representation of the construction of mutants HuN4-3'-UTR-17+G and HuN4-3'-UTR-△SL2
1.6 突变病毒的拯救及间接免疫荧光试验(IFA)检测 将Marc-145 细胞均匀铺于6 孔板中,待细胞形成80 %单层时,将质粒pOK-HuN4、pOK-HuN4-3'-UTR-(17+G)和pOK-HuN4-3'-UTR-△SL2 根据X-tremeGENE HP DNA Transfection Reagent 试剂说明分别转染Marc-145 细胞,5 d 后收获病毒,作为病毒P0 代,拯救病毒分别命名为HuN4、HuN4-3'-UTR-(17+G)和HuN4-3'-UTR-△SL2。将P0 代病毒接种于汇合度为80 %的Marc-145 细胞中,72 h 后收获病毒作为P1 代,以该方法直至获取P3 代病毒液,分装后冻于-80 ℃以备后用。将质粒pOK-HuN4、pOK-HuN4-3'-UTR-(17+G)和pOK-HuN4-3'-UTR-△SL2转染Marc-145 细胞,5 d 后取下层细胞加入1 mL 无水乙醇室温固定30 min,以PRRSV M 蛋白MAb(1∶100)为一抗,以山羊抗小鼠IgG-FITC (1∶100)为二抗,采用IFA 检测各拯救病毒HuN4、HuN4-3'-UTR-(17+G)和HuN4-3'-UTR-△SL2。
1.7 复制动力学曲线 将亲本病毒HuN4、突变体病毒HuN4-3'-UTR-(17+G)和HuN4-3'-UTR-△SL2 以MOI 0.01 接种于生长至80 %单层Marc-145 细胞的6 孔板中,分别在转染后0、24 h、48 h、72 h 和96 h收集病毒,分装后-80 ℃保存。将不同病毒不同时间点的病毒接种于生长至80 %单层Marc-145 细胞的96 孔板中,观察并记录细胞产生细胞病变(CPE)的孔数,参照Reed-Muench 法计算病毒TCID50值,以Graphpad 软件绘制复制动力学曲线。
1.8 统计学分析 以上实验的3 次重复数据采用多重t 检验进行统计学分析,数据由平均值±标准方差表示,p<0.05 表示差异显著,p<0.01 表示差异极显著。
2 结 果
2.1 P RRSV 3'-UTR 序列的变异分析结果 将34条HP-PRRSV 3'-UTR 和6 条经典PRRSV 3'-UTR 基因以Megalign 进行多序列比对。结果显示,与经典PRRSV 相比,HP-PRRSV 在3'-UTR 第17 位缺失G。其中34 株HP-PRRSV 均在3'-UTR 第17 位缺失G,表明该处缺失普遍存在于HP-PRRSV 中。
2.2 突变病毒二级结构的预测 通过在线软件Mfold 预测CH-1、HuN4、HuN4-(17+G)和HuN4-△SL2 病毒的3'-UTR 二级结构。结果显示HuN4 和CH-1 在二级结构形成上有明显差异;但HuN4-(17+G)、HuN4-△SL2 与HuN4- 在二级结构形成上无差异(图2)。表明HuN4 3'-UTR 中第17 位G 和第二个茎环SL2 的缺失并不改变HuN4 二级结构。
2.3 突变质粒的鉴定 将质粒pOK-HuN4 以及突变质粒pOK-HuN4-3'-UTR-(17+G)和pOK-HuN4-3'-UTR-△SL2 以相应引物PCR 扩增,结果显示扩增片段与预期片段大小一致,分别为647 bp、648 bp 和640 bp(图3),同时质粒测序结果与目的序列一致。表明正确构建突变质粒pOK-HuN4-3'-UTR-(17+G)和pOKHuN4-3'-UTR-△SL2。

图3 突变质粒HuN4-3'-UTR-(17+G)和HuN4-3'-UTR-△SL2 的PCR 鉴定Fig.3 Identification of mutated HuN4-3'-UTR-(17+G)and HuN4-3'-UTR-△SL2 plasmids by PCR
2.4 突变病毒拯救的IFA 检测结果 通过IFA 检测病毒HuN4、HuN4-3'-UTR-(17+G)和HuN4-3'-UTR-△SL2,结果显示突变体病毒HuN4-3'-UTR-(17+G)和HuN4-3'-UTR-△SL2 均能够被拯救,其中HuN4阳性对照病毒也能够被拯救,未加入质粒的阴性对照不能够被拯救(图4)。表明HuN4 3'-UTR 中第17位碱基G 和第二个茎环SL2 的缺失不影响病毒的拯救,同时第二个茎环SL2 的缺失可以作为标记疫苗的分子标签。
2.5 突变病毒的复制动力学曲线 通过一步生长曲线研究HuN4 3'-UTR 中第17 位碱基G 和SL2 的缺失对病毒复制的影响。复制动力学曲线结果显示,与亲本病毒HuN4 相比,突变体病毒HuN4-3'-UTR-(17+G)病毒滴度较低,在感染后48 h 和96 h 二者差异显著(p<0.05);同样,与亲本病毒HuN4 相比,突变体病毒HuN4-3'-UTR-△SL2 的滴度也有所降,且在感染后48 h、72 h 和96 h 二者差异极显著(p<0.01)(图5)。表明HuN4 3'-UTR 中第17 位碱基G 的缺失促进了HP-PRRSV 在Marc-145 细胞中的复制水平,但第二个茎环SL2 的缺失则降低HP-PRRSV 在Marc-145 细胞中的复制水平。
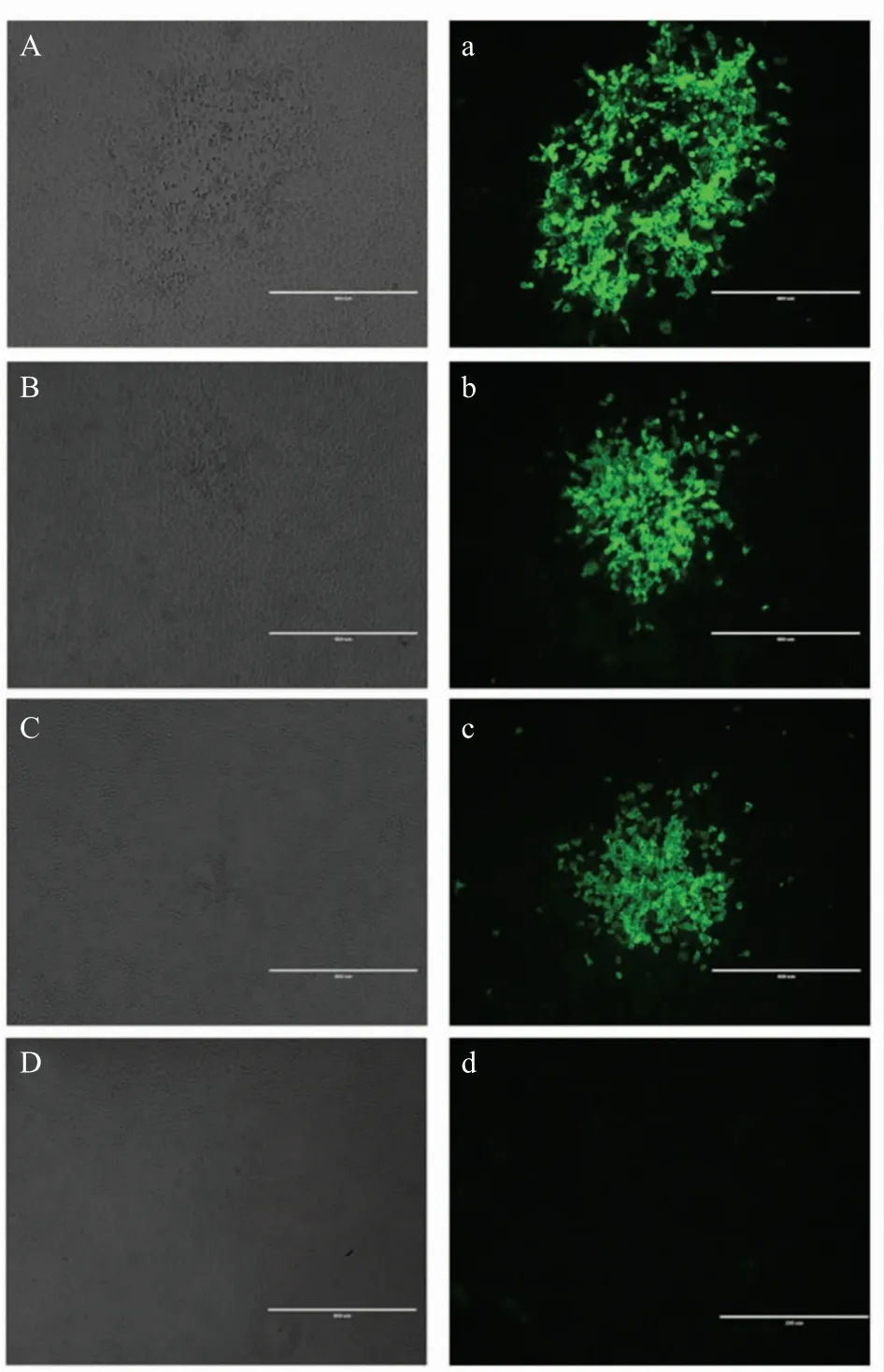
图4 突变体病毒HuN4-3'-UTR-(17+G)和HuN4-3'-UTR-△SL2 的IFA 检测Fig.4 Identification of mutated HuN4-3'-UTR-(17+G)and HuN4-3'-UTR-△SL2 viruses by IFA
3 讨 论
HP-PRRSV 以致病性强、传播迅速和死亡率高为特点,在我国流行范围极广,给我国养猪业造成了重大的经济损失。通过对HP-PRRSV 和经典PRRSV 序列比对发现,HP-PRRSV 基因组具有一些新的特征,其中最显著的特点是相比于经典PRRSV,HP-PRRSV 的NSP2 区域缺失了30 个氨基酸,但在感染性克隆的基础上补平该缺失之后,显示NSP2 区域的30 个氨基酸的缺失与HP-PRRSV 的复制无关[9];研究还表明NSP9 和NSP10 共同决定HP-PRRSV 的致病性[10]。然而调控PRRSV 复制的关键基因需进一步研究。
图5 HuN4-3'-UTR-(17+G)和HuN4-3'-UTR-△SL2 突变病毒的复制动力学曲线Fig.5 The growth curve of mutated HuN4-3'-UTR-(17+G)and HuN4-3'-UTR-△SL2 viruses
在PRRSV 感染细胞过程中,首先以其基因组为基础,合成自身的RdRp,随后结合到3'-UTR 上,以负链RNA 不连续转录模型开始病毒负链RNA 的合成,随后再以负链基因组为模板,启动亚基因组的转录和病毒基因组的复制。有研究证明PRRSV 3'-UTR 末端序列5'-AAUU-3' 和5'-AACCA-3' 对于维持病毒亚基因组合成和感染性至关重要[7];PRRSV 3'-UTR 和ORF7 区域形成的“相吻结构”是病毒复制的必要因素[11];人工合成的干扰RNA 结合的3'-UTR 基因序列,可以有效抑制PRRSV 的复制,降低病毒基因滴度[12]。由此可见,3'-UTR 在PRRSV的复制过程中起到了非常关键的调控作用。但是目前对PRRSV 3'-UTR 的研究较少,也未能找到3'-UTR 中调控PRRSV 复制的关键基因,因此对PRRSV 3'-UTR 的研究变的尤为重要。
本研究基于本实验室构建的HP-PRRSV HuN4株感染性克隆平台,开展了对HP-PRRSV 3'-UTR 区域的研究。首先HP-PRRSV 和经典PRRSV 3'-UTR序列比对发现HP-PRRSV 3'-UTR 中第17 位缺失碱基G,同时该处缺失普遍存在于HP-PRRSV 中。这与之前研究证明PRRSV 3'-UTR 中存在高度保守的一级序列结果一致[13]。随后的突变质粒构建和病毒拯救实验结果显示HuN4-3'-UTR-(17+G)和HuN4-3'-UTR-△SL2 突变体病毒可以被拯救,且二级结构预测结果表明HuN4 3'-UTR 第17 位碱基G 和第二个茎环SL2 的缺失并不改变HuN4 3'-UTR 二级结构。最后的复制动力学试验结果显示与亲本病毒HuN4比较,突变体病毒HuN4-3'-UTR-(17+G)呈现较低的病毒滴度,在感染细胞后48 h 和96 h 二者差异显著(p<0.05)。表明第17 位碱基G 的缺失增强了HP-PRRSV 的复制能力,这可能由于3'-UTR 第17位碱基的缺失改变了其对应区域第二个茎环SL2 的碱基组成,进而增加了复制转录复合体(Replication and transcription complex,RTC)与3'-UTR 的结合水平,最终引起HP-PRRSV 复制能力的增强。有研究证明PRRSV 5'-UTR 中近5' 端茎环结构影响病毒亚基因组的转录和病毒基因组的复制[14]。还有研究显示Ⅱ型PRRSV APRRSV 株3'-UTR 末端部分碱基的改变会影响病毒的复制效率[7]。同时本实验结果还表明HuN4 3'-UTR 中第二个茎环SL2 的缺失不影响病毒拯救,可以作为标记疫苗的分子标签。
本研究初步揭示了我国HP-PRRSV 与经典PRRSV 3'-UTR 在一级序列组成和二级空间结构形成上的差异,明确了第17 位碱基G 的缺失可以增强HP-PRRSV 的复制能力。本研究为深入探究PRRSV 3'-UTR 的作用机制和功能奠定了基础。
