碱基编辑系统研究进展
2019-09-24宗媛高彩霞
宗媛,高彩霞
碱基编辑系统研究进展
宗媛,高彩霞
中国科学院遗传与发育生物学研究所,植物细胞与染色体工程国家重点实验室,基因组编辑中心,北京 100101
碱基编辑技术(base editing)是基于CRISPR/Cas系统发展起来的新型靶基因修饰技术,目前依据碱基修饰酶的不同可分为胞嘧啶碱基编辑器(cytosine base editor, CBE)和腺嘌呤碱基编辑器(adenine base editor, ABE)。这两类碱基编辑系统利用胞嘧啶脱氨酶或人工进化的腺嘌呤脱氨酶对靶位点进行精准的碱基编辑,最终可以分别实现C-T (G-A)或A-G (T-C)的碱基替换。碱基编辑技术自2016年被开发以来,因其高效、不依赖DNA双链断裂产生、无需供体DNA参与等优势,已经成功应用在各种动物、植物及其他生物中,为基因治疗及精准作物育种等领域提供了重要技术支撑。本文从碱基编辑技术的特点、开发过程、优化、应用、脱靶效应及改善策略等方面进行了系统介绍,最后对未来需要迫切解决的一些问题进行了分析和展望,以期为相关领域的科研人员进一步了解、使用及优化碱基编辑系统提供参考。
CRISPR/Cas;碱基编辑技术;胞嘧啶碱基编辑器(CBE);腺嘌呤碱基编辑器(ABE)
如何精准、高效地对基因组进行修饰是生命科学领域研究的重要目标。近年来,基因组编辑技术尤其是CRISPR/Cas9 (clustered regularly interspaced short palindromic repeats/CRISPR associated 9)介导的基因组编辑系统成为实现该目标的最强工具[1~7]。CRISPR/Cas9系统是在sgRNA引导下招募Cas9蛋白靶向基因组DNA,从而在靶点处产生DNA双链断裂(double strand break, DSB),诱发细胞内非同源末端连接(non-homologous end joining, NHEJ)和同源重组(homologous recombination, HR)修复途径,进而实现对基因组DNA的定点敲除、替换、插入等精准修饰[8]。其中,NHEJ途径发生频率较高,但通常是一种不精确的修复方式,往往会在靶点处产生少量碱基的插入或缺失(insertions/deletions, Indels),是一种有效地创制基因敲除突变体的途径。HR途径是一种更为精确的修复方式,需要人为提供与基因组DNA具有同源序列的供体DNA,以供体DNA为模板进行重组修复,最终实现精准的基因定点替换或定点插入[8]。
单核苷酸变异会导致约2/3人类疾病的发生[9],也是许多作物重要农艺性状变异的遗传基础[10],因此如何开发一种精准且高效实现碱基替换的技术尤为重要。理论上,HR途径可实现任意碱基之间的改变,但该途径受细胞类型及细胞周期的限制,且如何将供体DNA高效递送到细胞中也是一大难题,这些弊端导致HR在动植物中的发生频率及应用范围均受到了一定的限制[11]。另外,NHEJ途径会与HR途径竞争发生,因此往往会造成靶点处不必要的编辑产物的产生。因此,DSB引发的HR很难实现高效的、稳定的单碱基突变。
2016年,碱基编辑系统(base editing)的开发将CRISPR/Cas系统从切割DNA的“剪刀”变为能改写特定碱基的“修正器”,打开了精准基因组编辑的大门。该系统目前已被广泛地应用于农业、基因治疗、作物育种等各个领域的研究[12~17]。2017年,杂志将碱基编辑技术评为年度十大科学技术突破之一。
1 碱基编辑系统的概况及特点
碱基编辑系统将失去催化活性的Cas蛋白(如deactivated Cas,简称dCas)或只有切割一条链活性的Cas蛋白(如nickase Cas,简称nCas)和可作用于单链DNA (single-stranded DNA, ssDNA)的脱氨酶进行融合,从而实现对靶点的碱基替换。目前碱基编辑系统依据融合的不同碱基修饰酶分为胞嘧啶碱基编辑器(cytosine base editors, CBE)和腺嘌呤碱基编辑器(adenine base editors, ABE)[12]。这两种碱基编辑器能在不产生DSB的情况下,分别利用胞嘧啶脱氨酶或经过改造的腺嘌呤脱氨酶对靶位点上一定范围的胞嘧啶(C)或腺嘌呤(A)进行脱氨基反应,最终经DNA修复或复制,实现精准的C-T或A-G的替换(图1)。该系统与传统的基于HR途径实现点突变的方法相比,主要优势如下(表1):
(1)不依赖DSB的产生。HR途径必须依赖DSB的产生才有可能实现基因定点替换,并且在该过程中会不可避免的激发NHEJ途径产生的非必要Indels产物。有效DSB的发生通常需要科研人员花费较大的精力去找寻并筛选出足够高活性的sgRNA或Cas核酸酶。另一方面,当细胞内产生大量甚至过量的DSB后会对细胞带来较大的毒性,从而会对生物体造成一定的影响。而碱基编辑系统由于利用失去切割活性的或只有一条链切割活性的Cas蛋白来实现靶位点处碱基的定点替换,因此不依赖DSB的产生。尽管有相关报道证明CBE系统会产生一 定的Indels,但效率远远低于其介导的单碱基替换效率[12]。
(2)无需供体DNA的参与。HR途径必须在提 供外源供体DNA的前提下才能实现碱基的定点替换。供体DNA形式目前主要包括环形双链DNA (double-stranded DNA, dsDNA)、线性dsDNA和单链DNA (single-stranded DNA, ssDNA) 3种形式。而对于选择何种形式的供体DNA以及确定供体DNA形式后又应该设计多少长度的同源序列才能够达到最佳的同源重组效果,这在不同的动物细胞系或不同的植物体中均不相同,目前还没有明确的统一的方法[4]。此外,如何将足量的供体DNA有效地递送到细胞中也是目前的一大难题。碱基编辑系统无需提供供体DNA,有效地避免了上述各种不确定性。该系统借助于胞嘧啶脱氨酶或腺嘌呤脱氨酶的作用,通过sgRNA的引导直接对靶标碱基进行脱氨,从而高效地实现C-T或A-G的突变。
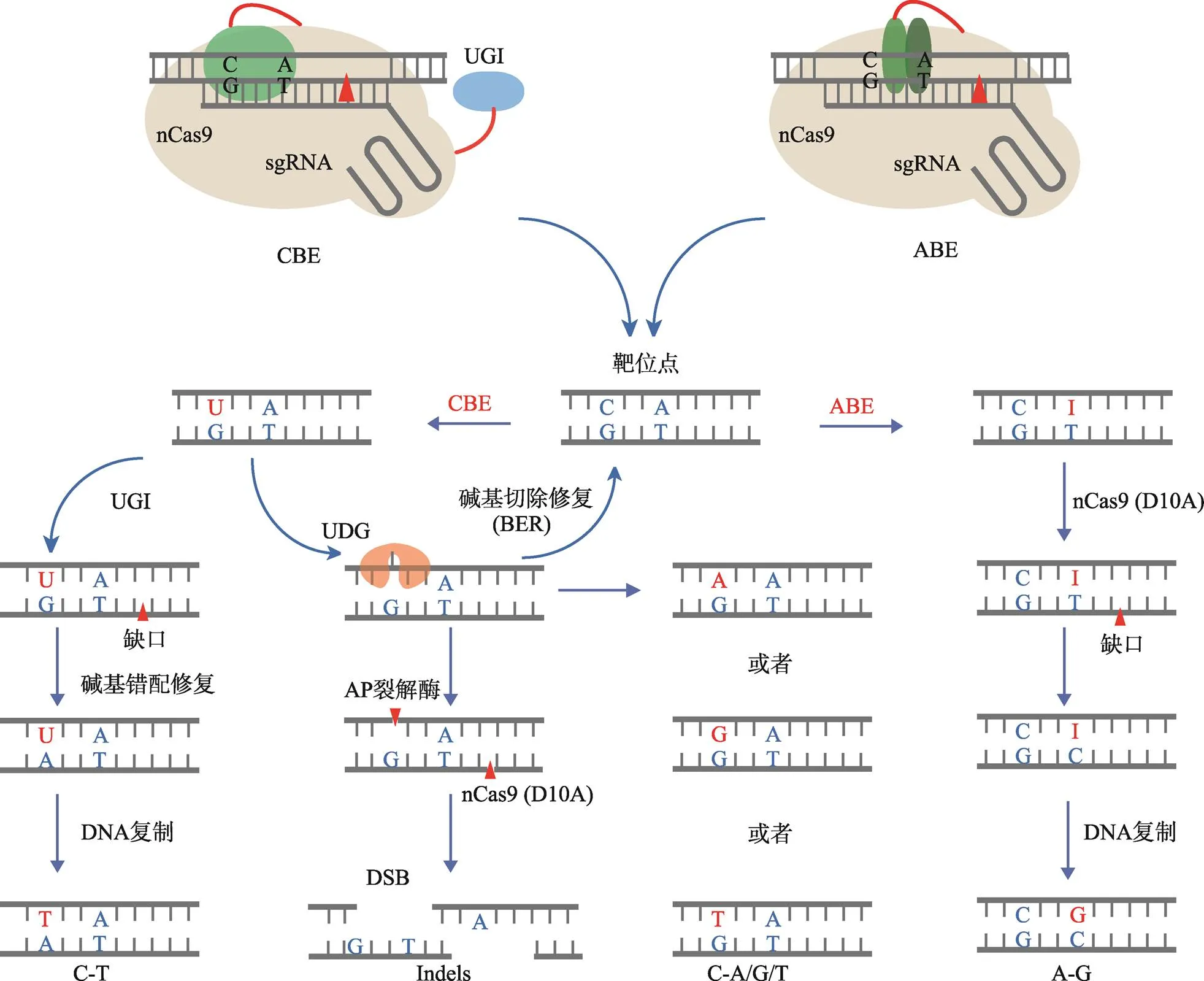
图1 CBE和ABE工作模式图
CBE系统在胞嘧啶脱氨酶及UGI的作用下将靶位点处一定活性窗口内的C脱氨变为U,经DNA修复和复制后实现C-T的突变(左);CBE系统经胞嘧啶脱氨酶、UDG、AP裂解酶等作用下会产生非C-T或Indels的突变(中);ABE系统在腺嘌呤脱氨酶的作用下将A脱氨变为I,经DNA修复和复制后实现A-G的突变(右)。
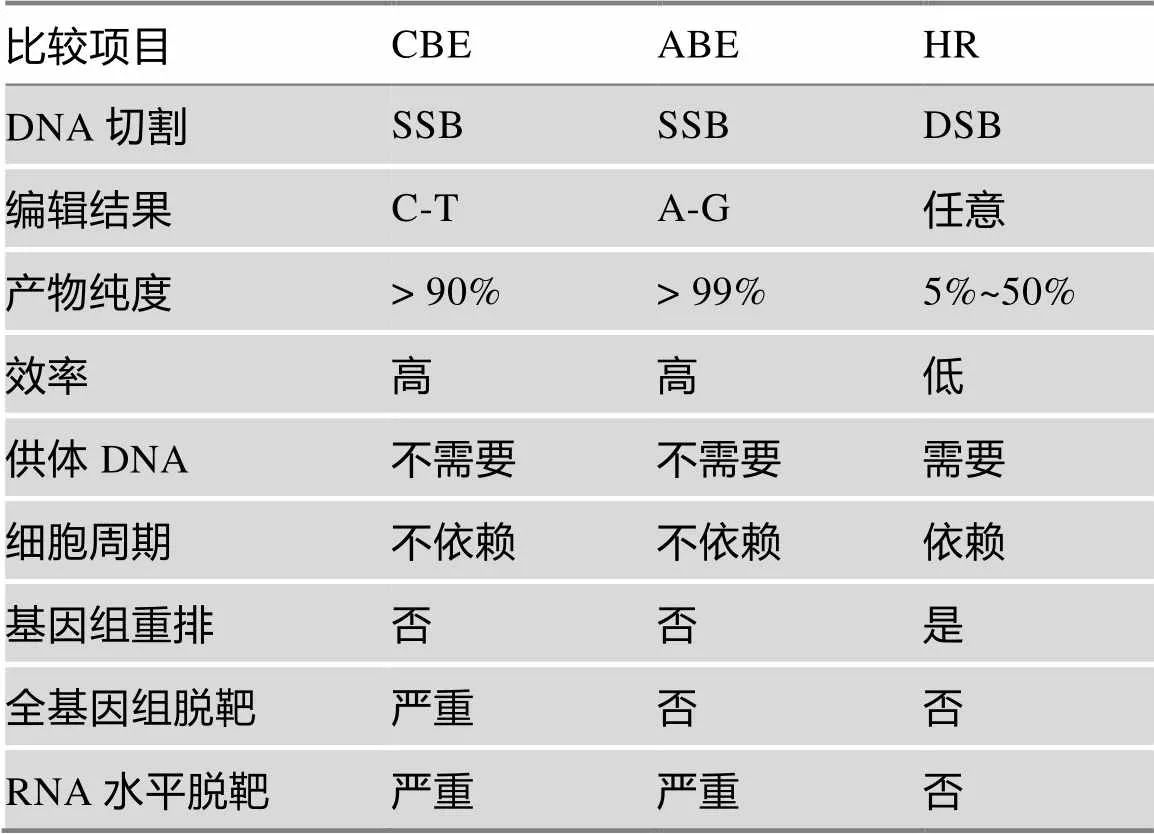
表1 CBE、ABE和HR的比较
SSB代表DNA单链断裂。
(3)高效性和广适性。HR一般发生在分裂的细胞,并且只能发生在细胞的S期和G2期[11],这使其在动植物中发生的频率较低,尤其在植物中HR的阳性筛选往往需要依赖除草剂抗性基因或靶标基因附近的抗生素基因来富集提高效率,因此大多数报道仅仅局限于对某些特殊的基因如和等的研究[4,11],使其很难被广泛地应用。而碱基编辑系统介导的碱基替换在动植物中均表现出高效的优势,且已被广泛地应用于各种动物细胞系、人类胚胎、各种哺乳动物、非哺乳动物、单双子叶植物、细菌、真菌等中[12,13,15,16]。
但是相比HR,碱基编辑系统目前只能实现碱基之间的转换(transition)而不能实现碱基之间的颠换(transversion),并且其在全基因组范围和RNA水平存在较高的脱靶效应(下文将详细介绍)。但从整体来看,碱基编辑系统的开发及不断的优化发展为生命科学领域带来了新的曙光。
2 CBE碱基编辑系统
2.1 CBE系统的开发
CBE一般由胞嘧啶脱氨酶、nCas9或dCas9组成[12,18]。具体工作原理是当CBE融合蛋白在sgRNA的引导下靶向基因组DNA时,胞嘧啶脱氨酶可结合到由Cas9蛋白、sgRNA及基因组DNA形成的R-loop区的ssDNA处,将该ssDNA上一定范围内的胞嘧啶(C)脱氨为尿嘧啶(U),进而通过DNA修复或复制将U转变为胸腺嘧啶(T),最终实现C至T (C-T)或G至A (G-A)的直接替换[18](图1)。
自然界中存在的胞嘧啶脱氨酶大多数作用于RNA,而APOBEC (apolipoprotein B mRNA editing enzyme, catalyticpolypeptide-like)家族是目前报道的少数可作用于单链DNA的胞嘧啶脱氨酶,该类脱氨酶共同特点是都有一个能对碱基C进行脱氨的CDA (cytidine deaminase)保守结构域,通过与CRISPR系统相结合,该类胞嘧啶脱氨酶由传统的“突变器”被开发为新型的“编辑器”[18]。
2016年,美国哈佛大学David R. Liu实验室率先建立了3种基于融合来源大鼠胞嘧啶脱氨酶rAPOBEC1的碱基编辑器(base editors, BE)[18]。第一代碱基编辑器BE1 (rAPOBEC1-XTEN-dCas9)由rAPOBEC1和完全失去切割活性的dCas9组成,可在体外实现有效的C-T碱基的编辑(25%~40%),编辑的活性窗口可覆盖sgRNA的第4~8位(PAM计为第21~23位),但该碱基编辑器在哺乳动物细胞内的编辑效率却大大下降(0.8%~7.7%)。这在很大程度上与细胞体内存在的尿嘧啶DNA糖基化酶(uracil DNA glycosylase, UDG)有关,UDG可识别U•G错配,并切割尿嘧啶和脱氧核糖骨架之间的糖苷键,通过细胞内的碱基错配修复途径(base excision repair, BER)逆转由BE1产生的U•G中间产物返回到C•G碱基对(图1)。为了抑制UDG的作用,David R. Liu实验室在BE1基础上融合了来自噬菌体PBS的尿嘧啶DNA糖基化酶抑制剂(uracil DNA glycosylase inhibitor, UGI),开发了第二代碱基编辑器BE2 (rAPOBEC1-XTEN-dCas9-UGI)。UGI能够抑制人类和细菌中的UDG作用,在一定程度上增加C-T的编辑效率,经测试BE2比BE1编辑效率提高了3倍。随后,该实验室结合细胞的内源修复机制开发了第三代碱基编辑器BE3 (rAPOBEC1-XTEN-nCas9-UGI)。BE3将BE2中的dCas9替换为nCas9 (D10A),可特异性的将非编辑链产生一个缺口,进而刺激细胞内碱基错配修复途径(mismatch repair, MMR)以含有C的编辑链作为模板进行修复,从而增加碱基编辑效率。研究结果表明,BE3在人类细胞的6个位点中可实现平均约37%的编辑效率,比BE2提高了2~6倍,其编辑窗口仍为第4~8位,该碱基编辑器也是目前使用较为广泛的CBE版本。
另外,日本神户大学Akihiko Kondo实验室采用上述类似策略将来源于七鳃鳗()的胞嘧啶脱氨酶PmCDA1用于开发新的碱基编辑器—Targeted-AID[19]。研究人员通过该系统成功在酵母及哺乳动物细胞中实现了C-T的定点替换,效率分别高达80%及10%,编辑窗口为sgRNA的第5~9位,相比BE3的编辑窗口略大。
同年,基于融合人类胞嘧啶脱氨酶hAID的碱基编辑系统也被建立[20,21]。其中,中国科学院上海生命科学研究所健康所常兴实验室开发的TAM系统融合了dCas9及UGI[20];美国斯坦福大学Michael C. Bassik实验室开发的CRISPR-X同样融合了dCas9,不同的是该碱基编辑器未融合UGI并且通过MS2招募hAID脱氨酶[21]。这两种系统在转入多个sgRNA的情况下,可以在哺乳动物细胞中实现较大范围(编辑范围宽达100 nt)以及较多类型的碱基编辑。
另外,美国哈佛大学George M. Church实验室开发了基于融合ZF或TALE特异性识别单元与hAID的碱基编辑系统[22]。相比上述基于融合Cas9变体的碱基编辑系统,这两种碱基编辑器无PAM的需求,但在细菌及人类细胞中的编辑效率明显下降,猜测原因是由于ZF或者TALE识别DNA特定区域后仍以dsDNA形式存在,未能够提供胞嘧啶脱氨酶工作时所需要的ssDNA底物,从而降低了脱氨酶的催化效率。
2.2 CBE系统的限制及优化
2.2.1 编辑产物纯度
多项研究表明,经CBE系统编辑后的编辑产物纯度并不高,主要表现在两方面:(1) CBE除将C编辑为T之外,也有一定几率将C编辑为G或A[12]。这是由于UDG可将碱基U切除形成无嘧啶位点(apyrimidinic site, AP),经过跨损伤合成(translesion synthesis, TLS)聚合酶作用及DNA复制等,也有一定的几率将该位置原始碱基C替换为其他碱基(图1);(2)在编辑过程中会产生少量的Indels[12,18],这是由于形成的AP位点会在AP裂解酶或自发断裂下产生一个缺口,进而与nCas9在非编辑链产生的缺口刚好形成一个DSB,经过NHEJ修复途径后会产生非必要的Indels产物(图1)。David R. Liu实验室在BE3的基础上融合了第二个拷贝的UGI,构建了第四代碱基编辑器BE4。测试结果表明,BE4不仅提高了碱基编辑效率,并且非C至T的替换效率相比BE3降低了2.3倍[23]。上海科技大学陈佳实验室采取类似策略,通过优化UGI的表达量,将UGI增至4个拷贝,建立的eBE-S3在哺乳动物细胞中同样提高了编辑产物纯度[24]。因此,通过添加更多拷贝UGI的策略对提高编辑产物纯度具有重要作用。另外,David R. Liu实验室进一步建立的BE4-Gam碱基编辑器可有效降低Indels的发生,这是由于融合的来源于噬菌体Mu的Gam蛋白可结合于DSB的末端防止其降解,进而阻止了NHEJ途径的发生[23]。而基于融合环化的Cas9的4种CP-CBEmax编辑器也可一定程度的降低副编辑产物的发生[25]。
2.2.2 碱基编辑效率
研究发现,细胞内碱基编辑器的表达水平是影响其编辑效率的一个重要因素。David R. Liu实验室通过增加不同个数的核定位信号(nuclear localization signal, NLS)以及使用不同公司优化的密码子序列等方法构建了BE4max和AncBE4max,这两种碱基编辑器可在各种哺乳动物细胞进行高效编辑[26]。Zafra等[27]通过增加NLS个数、改变NLS位置以及将nCas9密码子优化为哺乳动物细胞所偏好的密码子序列,开发的FNLS-BE3的编辑效率相比BE3平均提高了15倍。另外,本实验室开发的基于融合人类APOBEC3A胞嘧啶脱氨酶的A3A-PBE可在植物中实现比BE3平均高约13倍的碱基编辑效率[28]。
2.2.3 碱基编辑范围
上述碱基编辑器均是融合SpCas9,因此只能靶向NGG序列的PAM形式,大大限制了CBE在基因组中的靶向范围。为此,研究人员将SpCas9替换为其他Cas蛋白或相应变体,通过不同Cas蛋白识别不同PAM,进而扩大CBE系统在基因组中的编辑覆盖度。目前SpCas9的不同变体(VQR、EQR和VRER)、SaCas9和SaCas9的变体SaKKH与胞嘧啶脱氨酶的融合成功将CBE由NGG的PAM拓展为相应的NGA、NGCG、NNGRRT和NNNRRT等多种PAM形式[29,30]。而dCpf1和Sp-macCas9的融合将CBE由识别富含GC序列拓宽至识别富含AT序列的的PAM位点[31~33]。值得一提的是,基于融合xCas9及SpCas9-NG变体的两种碱基编辑器的开发使CBE系统可实现直接靶向NG PAM形式的位点[34,35]。
2.2.4 碱基编辑活性窗口
不同类型的CBE的碱基编辑窗口有略微的不同,依据不同的研究目的需要对编辑活性窗口做相应的改变。当被用于精准改变某个特定的碱基C时,过大的脱氨化窗口会导致窗口内非靶标碱基C的编辑,使得CBE系统被用于基因治疗等领域时存在一定风险。David R. Liu实验室通过突变rAPOBEC1胞嘧啶脱氨酶上与DNA作用的关键酶活位点,开发了YE1-BE3、YE2-BE3、EE-BE3和YEE-BE3碱基编辑器,进而可将脱氨窗口由5 nt缩小至1~2 nt,但同时对靶标C的编辑效率有所降低[29]。德国马普分子植物生理所Ralph Bock实验室开发的BE3-PAPAPAP以及通过缩短PmCDA1脱氨酶羧基端序列而建立的一系列nCDA1-BE3版本,能够在保证对靶标C编辑效率依然高效的同时,可有效将编辑的活性窗口缩小到1~2 nt[36]。但如何进一步缩小突变窗口,真正做到只改变单一碱基而不影响旁侧序列,将是碱基编辑系统今后重点攻克的方向。
另外,当CBE系统被用于基因功能筛选、大规模饱和突变、编辑调控元件、引入提前终止密码子或进行可变剪接等时,更大的编辑活性窗口是更为有利的。Jiang等[37]采取SunTag系统策略开发了BE-PLUS系统,从而将BE3的突变窗口由5 nt (4~8)拓宽至13 nt (4~16)。基于融合hA3A胞嘧啶脱氨酶的碱基编辑器在人类细胞及植物中脱氨化的窗口可分别宽达14 nt (2~13)和17 nt (1~17)[28,38]。而CP1020- CBEmax和CP1028-CBEmax的编辑活性窗口可覆盖sgRNA的8~9 nt[25]。Liu等[39]通过改造开发的DBE- A3A和DBE-AIDmono可将突变窗口拓宽为2~17位,并且依赖DBE系统可在体外培养的细胞中实现抗体亲和成熟。
2.2.5 序列背景依赖性
基于融合rAPOBEC1的BE3具有强烈偏好TC序列,而对GC序列基本无编辑活性的特点[18]。而基于融合hAID、PmCDA1和hA3A的碱基编辑系统均可在GC序列背景下有效地编辑[23,28,40],且hA3A- BE3可对高甲基化区域有较强的编辑活性[38]。而美国麻省总医院Keith Joung实验室通过加强碱基编辑效率依赖序列背景的特性,开发了eA3A-BE3,该碱基编辑器在保证其偏好TC序列背景的前提下,对其他序列背景下C的编辑活性大大下降,也进一步降低了对邻近非靶标C的编辑[41]。类似的,基于融合人类hAPOBEC3G的hA3G-BE3只特定的编辑CC序列的第二个C或CCC序列的后两个C[42]。
总之,针对胞嘧啶碱基编辑系统的各种不足之处,科研人员们不断开发和优化出新的碱基编辑器,极大地丰富了碱基编辑工具盒。上述各种碱基编辑器具体构建及编辑特性见表2。
2.3 CBE系统的应用
胞嘧啶碱基编辑系统自开发以来,被迅速的广泛应用于各种动物细胞系、人类胚胎、动物、植物及细菌等基础体系的建立(图2,表3),其中包括酵母()、真菌、小鼠()、斑马鱼()、兔子()、家蚕(L.)、海胆()、非洲爪蟾()、猪(L.)、羊(L.)、水稻(L.)、小麦(L.)、玉米(L.)、番茄(Mill.)、拟南芥()、马铃薯(L.)、西瓜(L.)、棉花(L.)和细菌等[12,13,15,16,43~50]。除此之外,该系统也被用于基因治疗、动物疾病模型建立、植物重要农艺性状创制等方面[12,13,15](图2,表3)。
2.3.1 基因治疗及动物疾病模型的建立
CBE系统的开发为精准基因治疗提供了强有力的技术手段。通过CBE在各种细胞系水平上修正相应致病位点的研究已经很多,其中包括Komor等[18]利用BE3在小鼠星形胶质细胞中修正了与阿尔兹海默症相关的基因的突变;Zara等[27]在小鼠肝脏细胞中通过递送密码子优化的FNLS-BE3纠正了与癌症相关的S45F突变;Komor等[18]在人类乳腺癌细胞中纠正了Y163C突变;Koblan等[26]在来源于病人的原发性纤维细胞中实现了、等位基因的纠正等。而在哺乳动物体内及人类胚胎中实现致病基因的定向修正也有相关报道,如Chadwick等[51]在成年小鼠中通过慢病毒递送BE3,对基因进行提前终止突变,可大幅度降低小鼠血浆中PCSK9蛋白表达水平及胆固醇水平;Yang等[52]对组蛋白相关基因进行点突修饰,探索了表观调控在小鼠胚胎发育中的重要性。Zeng等[53]在人类细胞和杂合子胚胎中纠正了马凡综合征致病性突变;中山大学黄军就实验室在重构的人类胚胎中修复了引起β-地中海贫血病的突变[54];中国科学院上海生命科学研究院神经科学研究所杨辉实验室研究发现在人类胚胎的二细胞期注射BE3比在一细胞期注射时对基因的编辑效率更高,这为研究人类胚胎发育中的基因功能提供了一种强有力的方法[55]。
第一,往大处想——定义一个更加宽泛的问题,不断地问“这个问题背后更大的问题是什么?”和“我们要达到的更好的效果是什么?”让思维获得更多的自由,从而想出更多的解决方案。第二,换位思考——进入你想模仿的人或公司的思维模式,重新审视遇到的问题,放开平常的限制,会发掘出新的、未想到过的解决方法。第三,强加约束——给现状引入一个强有力的限制,探索这样做能带来什么样的好处。第四,选择构架——怎么创造一种环境,让人们在这种环境中选择我们想要他们采取的行动。
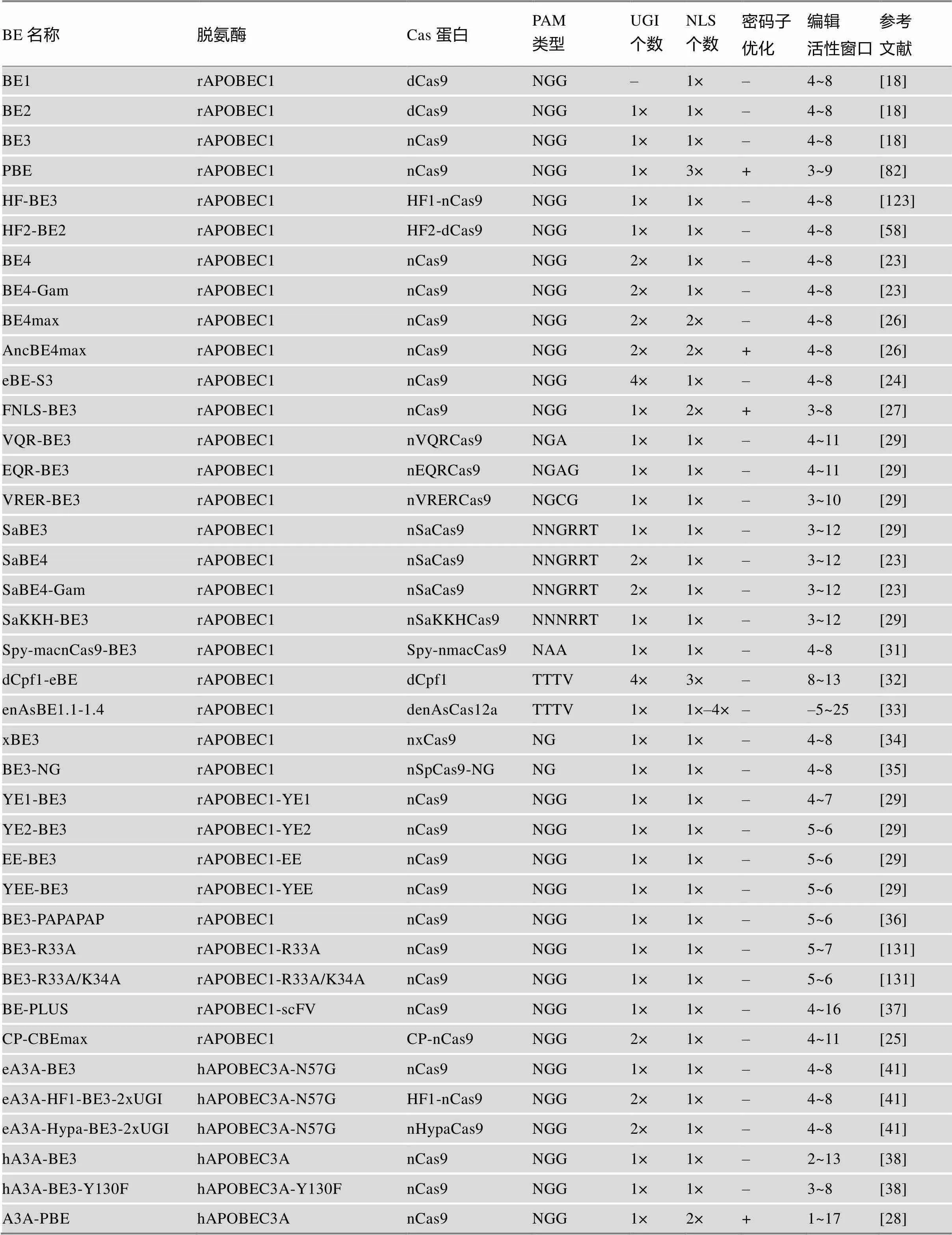
表2 各种碱基编辑器(BE)的特点
续表
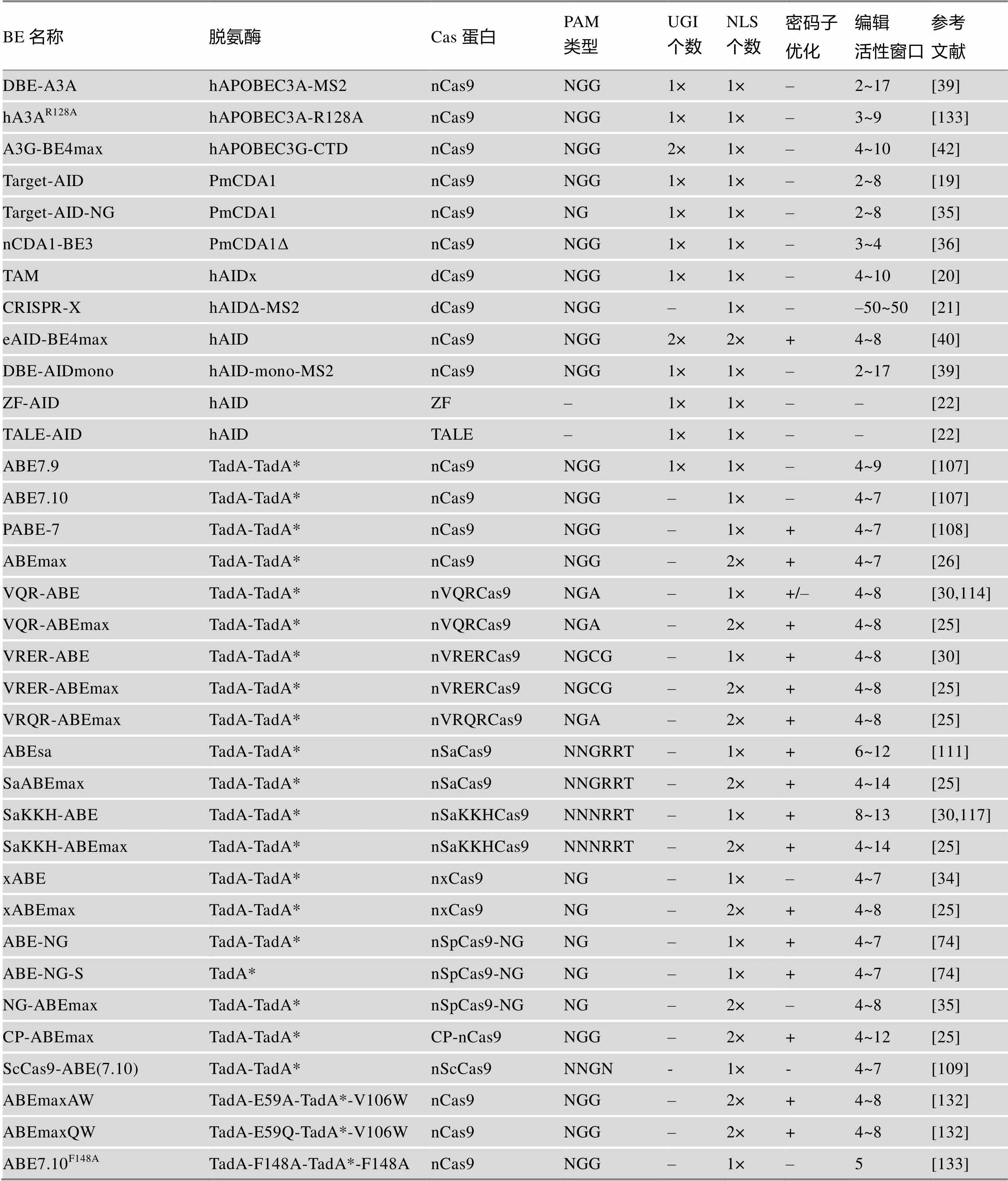
BE名称脱氨酶Cas蛋白PAM类型UGI个数NLS个数密码子优化编辑活性窗口参考文献 DBE-A3AhAPOBEC3A-MS2nCas9NGG1×1×–2~17[39] hA3AR128AhAPOBEC3A-R128AnCas9NGG1×1×–3~9[133] A3G-BE4maxhAPOBEC3G-CTDnCas9NGG2×1×–4~10[42] Target-AIDPmCDA1nCas9NGG1×1×–2~8[19] Target-AID-NGPmCDA1nCas9NG1×1×–2~8[35] nCDA1-BE3PmCDA1ΔnCas9NGG1×1×–3~4[36] TAMhAIDxdCas9NGG1×1×–4~10[20] CRISPR-XhAIDΔ-MS2dCas9NGG–1×––50~50[21] eAID-BE4maxhAIDnCas9NGG2×2×+4~8[40] DBE-AIDmonohAID-mono-MS2 nCas9NGG1×1×–2~17[39] ZF-AIDhAIDZF–1×1×––[22] TALE-AIDhAIDTALE–1×1×––[22] ABE7.9 TadA-TadA*nCas9NGG1×1×–4~9[107] ABE7.10 TadA-TadA*nCas9NGG–1×–4~7[107] PABE-7TadA-TadA*nCas9NGG–1×+4~7[108] ABEmax TadA-TadA*nCas9NGG–2×+4~7[26] VQR-ABETadA-TadA*nVQRCas9NGA–1×+/–4~8[30,114] VQR-ABEmaxTadA-TadA*nVQRCas9NGA–2×+4~8[25] VRER-ABETadA-TadA*nVRERCas9NGCG–1×+4~8[30] VRER-ABEmaxTadA-TadA*nVRERCas9NGCG–2×+4~8[25] VRQR-ABEmaxTadA-TadA*nVRQRCas9NGA–2×+4~8[25] ABEsaTadA-TadA*nSaCas9NNGRRT–1×+6~12[111] SaABEmaxTadA-TadA*nSaCas9NNGRRT–2×+4~14[25] SaKKH-ABETadA-TadA*nSaKKHCas9NNNRRT–1×+8~13[30,117] SaKKH-ABEmaxTadA-TadA*nSaKKHCas9NNNRRT–2×+4~14[25] xABETadA-TadA*nxCas9NG–1×–4~7[34] xABEmaxTadA-TadA*nxCas9NG–2×+4~8[25] ABE-NGTadA-TadA*nSpCas9-NGNG–1×+4~7[74] ABE-NG-STadA*nSpCas9-NGNG–1×+4~7[74] NG-ABEmaxTadA-TadA*nSpCas9-NGNG–2×–4~8[35] CP-ABEmaxTadA-TadA*CP-nCas9NGG–2×+4~12[25] ScCas9-ABE(7.10)TadA-TadA*nScCas9NNGN-1×-4~7[109] ABEmaxAWTadA-E59A-TadA*-V106WnCas9NGG–2×+4~8[132] ABEmaxQWTadA-E59Q-TadA*-V106WnCas9NGG–2×+4~8[132] ABE7.10F148ATadA-F148A-TadA*-F148AnCas9NGG–1×–5[133]
点突变动物模型的建立可通过HR实现,但该途径效率低且周期长,并不能被广泛的应用。目前通过CBE系统已在多种动物中建立了相应的疾病模型(图2A,表3)。在小鼠中建立的疾病模型类型最多,包括白化病、肌营养不良、肝病和雄性激素不敏感综合征等[56],其中韩国首尔国立大学Jin-Soo Kim实验室通过BE3 mRNA编辑小鼠胚胎基因和而建立了相应的小鼠白化病和肌营养不良疾病模型[57];中山大学松阳洲实验室利用HF2-BE2也建立了小鼠白化病模型[58];Sasaguri等[59]利用BE3和Targeted-AID系统在小鼠中引入了与早衰症相关的基因的突变;Villiger等[60]在成年小鼠中利用AAV慢病毒载体递送分割的BE3系统,修正了突变,治疗了代谢肝病;Li等[61]利用hA3A-BE3- Y130F编辑富含GC的区域,获得了雄性激素不敏感综合征的小鼠。在大型哺乳动物如兔子、猪和羊中也有相关疾病模型的建立。兔子中,利用BE3、BE4-Gam、AID-nCas9和hA3A-eBE-N57G等不同的CBE碱基编辑器建立了白化病、早衰症、双肌臀、X-连锁扩张性心肌病等疾病模型[62~65]。在猪中,Li等[66]利用BE3在猪胚胎成纤维细胞中编辑基因和基因,而后通过体细胞核移植的方法建立了无脸巨口综合征和皮肤白化病的疾病模型;而中国科学院广州生物医药与健康研究院赖良学实验室在猪中首次实现了多个基因靶点的同时C-T突变,并建立了相应的疾病模型[67]。在羊中,利用BE3编辑和基因,成功获得了体重、尺寸和毛长等生长指标增加的突变体羊[68,69]。除了在哺乳动物中,在非哺乳动物斑马鱼和非洲爪蟾中建立模拟人类疾病的模型也有相关报道,其中Zhang等[70]通过编辑斑马鱼胚胎的基因建立了模拟人类眼白化病的疾病模型;Tanaka等[71]利用Targeted-AID编辑了斑马鱼基因和,并观察到了相应突变体表型。在非洲爪蟾中,Park等[72]通过转化BE3的RNP形式建立了白化病疾病模型;Shi等[73]利用BE3建立了模拟人类Holt-Oram综合征和眼皮白化病1A型的疾病模型。上述致病基因的点突动物模型的建立为进一步真正进行临床疾病治疗提供了坚实的基础。
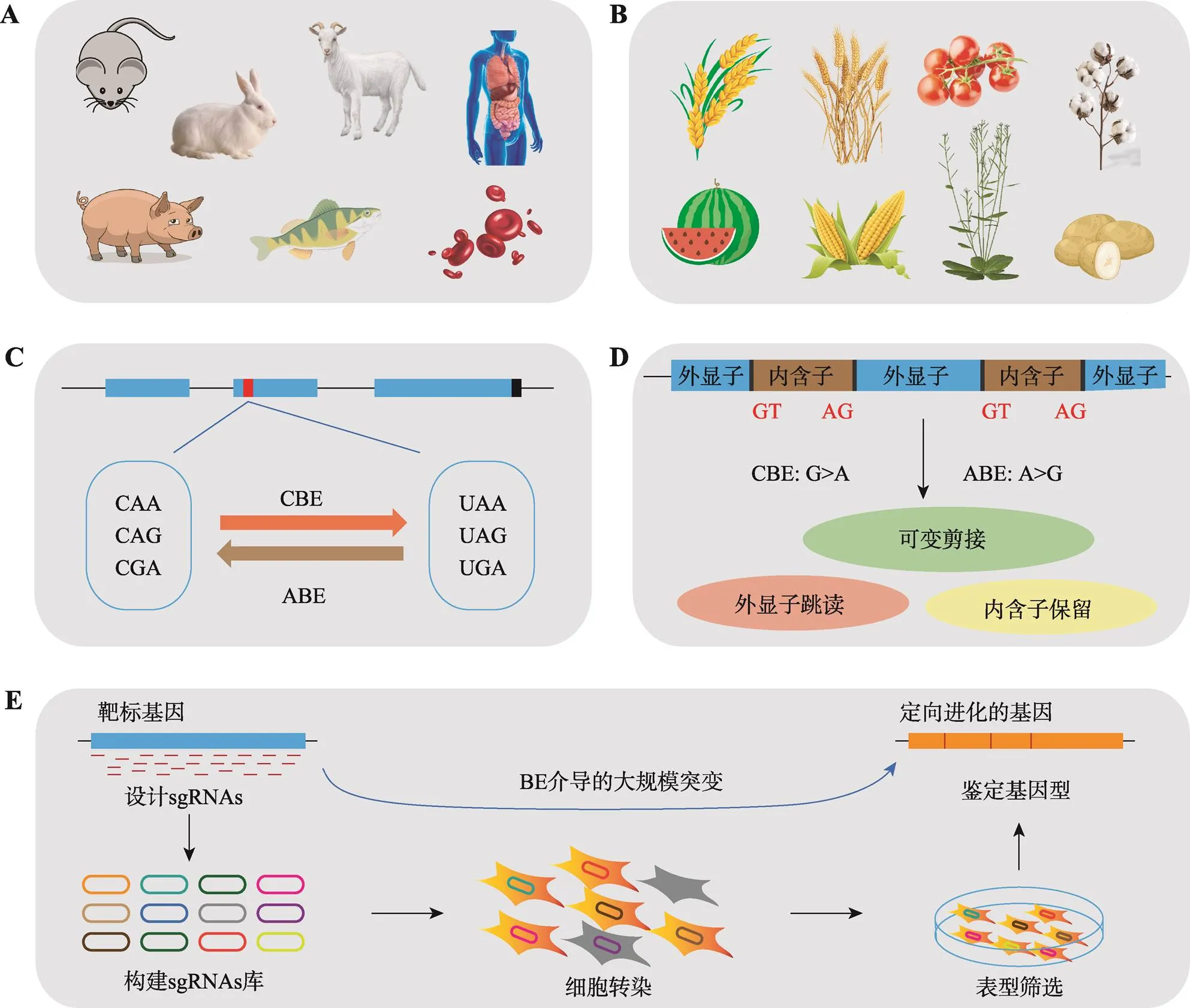
图2 碱基编辑系统的应用
A:基因治疗及动物疾病模型建立;B:植物中的应用;C:提前终止密码子的引入及改变;D:mRNA剪接;E:蛋白定向进化。
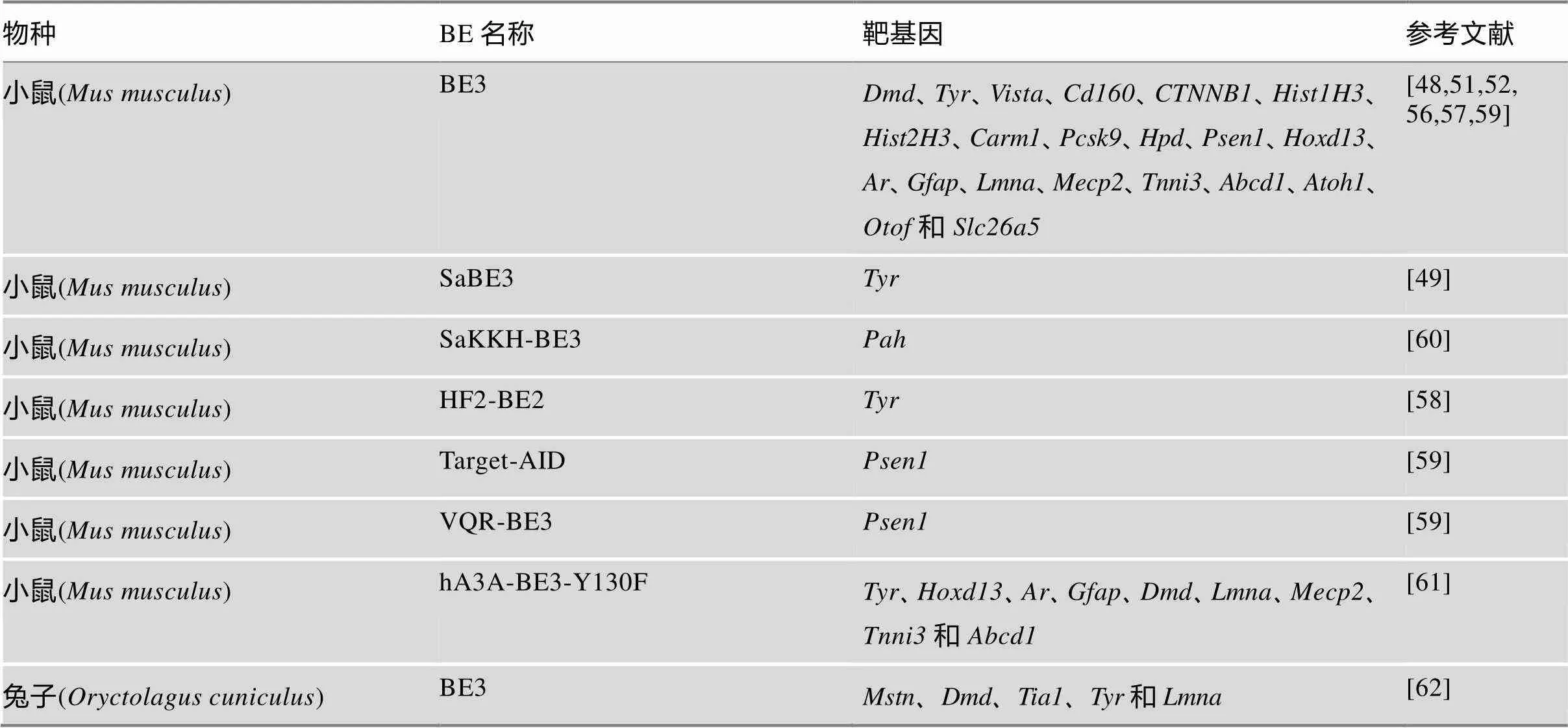
表3 碱基编辑系统在动物、植物和细菌中的应用
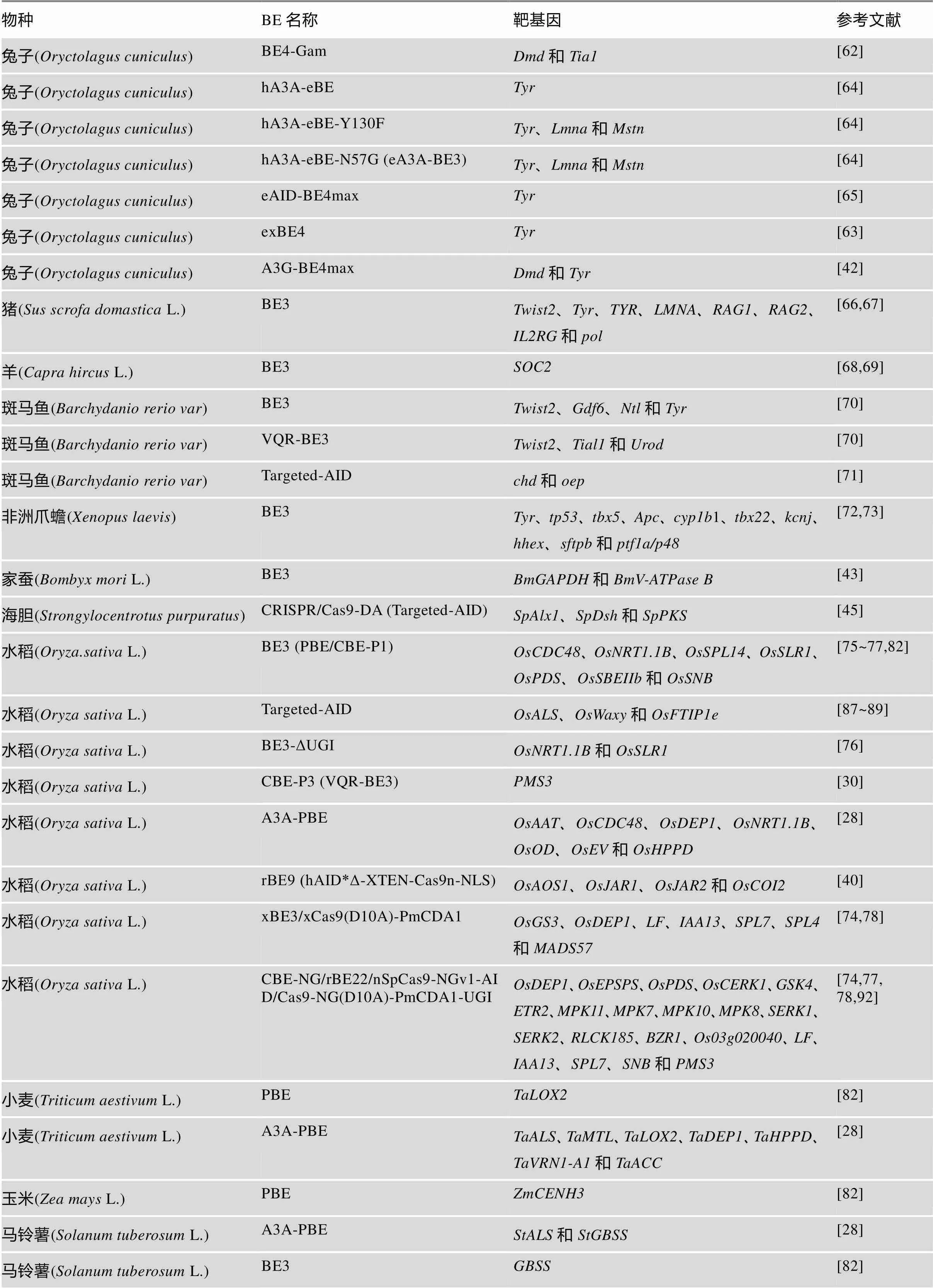
续表
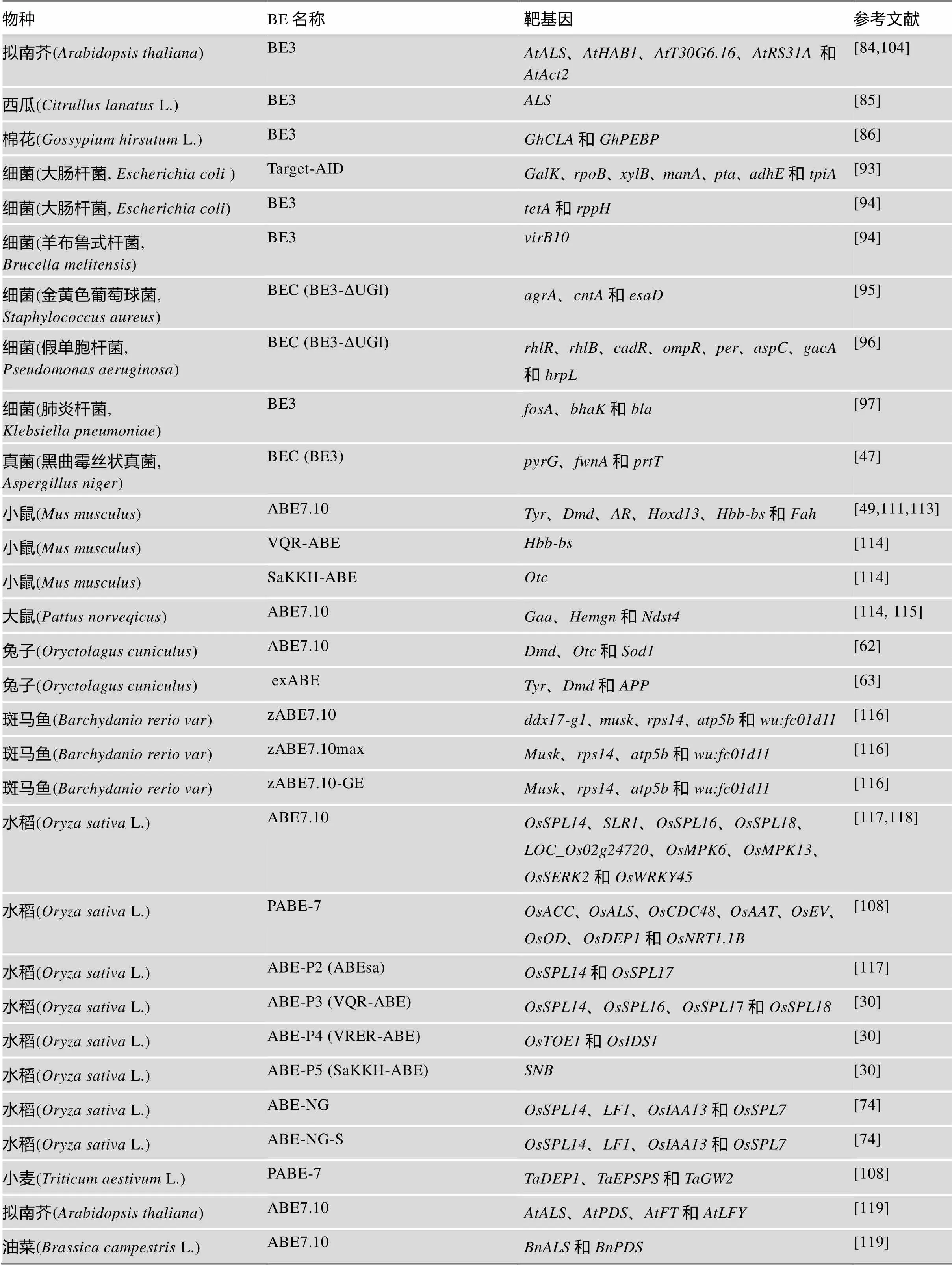
续表
2.3.2 在植物中的应用
植物中许多重要农艺性状是由单个或少数几个碱基突变引起,基于传统化学诱变的TILLING (targeting induced local lesions in genomes)可获得植物中的点突,但该方法耗时、耗力、随机性强、突变效率低,而CBE系统的开发为在植物中快速、高效且精准的创制突变体提供了有力的工具(图2B,表3)。其中,基于融合rAPOBEC1的BE3系统在植物中的应用最多。自2016年以来,来自国内外的多个研究团队利用BE3及基于融合SpCas9-VQR、SaKKH、SpCas9-NG和xCas9变体的BE3碱基编辑器在水稻中创制了多个基因的突变体[30,74~81]。本实验室利用基于融合rAPOBEC1的PBE系统在水稻、小麦和玉米原生质体中实现了多个基因的编辑,并获得了突变效率高达45.8%的相应突变体植株[82];并利用PBE系统编辑小麦和基因,创制了一系列抗除草剂小麦新种质,为麦田杂草防控提供了育种新材料及技术路径[83]。另外,基于此本实验室还在小麦中建立了无外源选择标记、直接筛选碱基编辑事件的方法[83]。Chen等[84]通过对基因的碱基替换,创制了抗除草剂拟南芥材料。Tian等[85]利用BE3获得了世界首例碱基编辑抗除草剂西瓜。另外,BE3介导的碱基编辑也已成功用于四倍体棉花基因组中[86]。
基于融合其他类型胞嘧啶脱氨酶的CBE系统在植物中的应用也有相关报道。基于融合PmCDA1脱氨酶的Target-AID系统已成功用于水稻、番茄和马铃薯相关基因的编辑,并创制了抗咪唑类除草剂水稻材料[87~90]。并且,该系统后续被用于建立植物抵御RNA病毒的免疫系统[91]。另外,SpCas9-NGv1与PmCDA1的融合进一步拓宽了胞嘧啶碱基编辑系统在水稻基因组中的靶向范围[78,92]。而本实验室利用基于融合人类APOBEC3A的A3A-PBE系统在水稻、小麦和马铃薯原生质体实现了非常高效的碱基编辑并且创制了抗除草剂小麦材料[28]。基于融合hAID的碱基编辑系统在水稻中表现出能高效编辑GC序列背景的特点[40]。
2.3.3 在细菌中的应用
CRISPR/Cas系统来源于原核生物的免疫系统,可被用于细菌的编辑,同样BE系统已被测试也可用于细菌的碱基编辑(表3)。Banno等[93]利用Targeted- AID编辑了大肠杆菌()等多个基因,更重要的是该系统在只转入4个sgRNA的情况下可在多拷贝的转座子元件中诱导41个突变的发生。Zheng等[94]利用BE3对大肠杆菌基因引入了提前终止密码子。除在大肠杆菌中,科研人员利用BE3或者BE3-ΔUGI在羊布鲁式杆菌()、金黄色葡萄球菌()、假单胞杆菌()和肺炎球菌()等中也成功实现了相关基因的碱基编辑[94~97],这表明碱基编辑系统可被广泛的应用于多种原核生物的编辑。
2.3.4 引入提前终止密码子
CBE系统通过C-T或G-A突变可以将非编码链的CAA、CAG、CGA以及TGG共4种密码子中的任意一种突变为终止密码子,从而在不产生DNA双链断裂的情况下达到敲除基因的效果(图2C)。Jin-Soo Kim实验室率先利用BE3在基因中引入提前终止密码子,建立了肌营养不良的小鼠疾病模型[57]。随后报道的CRISPR-Stop和iSTOP方法利用同样的思路建立了大规模BE3介导的基因失活体系。其中,CRISPR-Stop相关研究发现,通过BE3产生的基因失活效率与Cas9介导的敲除效率相当,并且BE3对细胞的死亡损伤明显降低[98]。Billon等[99]建立的iSTOP可覆盖人类基因组读码框的98.6%以上,并且该实验室提供了可免费设计人类、小鼠和拟南芥等8个物种iSTOP sgRNA的在线数据库。而当同时使用多个sgRNA时,可显著增加BE3介导的iSTOP效率[100]。
2.3.5 调控内源mRNA的可变剪接
mRNA可变剪接是转录后基因调控的重要过程,典型真核内含子连接有5ʹ-GT(供体序列)和AG-3ʹ (受体序列),它们在剪接中起着重要作用[101],而碱基编辑系统的出现对特定mRNA剪接过程的功能研究提供了重要的技术工具(图2D)。Gapinske等[102]率先利用BE3介导的CRISPR-SKIP剪接体系,对小鼠和人类不同细胞系的mRNA的可变剪接位点进行了编辑,导致外显子跳读。常兴实验室利用TAM系统在人类细胞中实现了目前存在的4种mRNA剪接形式的改变(外显子跳读、改变可变剪接位点的选择、调节互斥外显子的选择和诱导小的内含子的包含),并且通过编辑基因的剪接位点,实现了目标外显子的完全跳读,在所有表达TAM的细胞中修复了心肌细胞的缺陷[103]。植物中,本实验室利用BE3在拟南芥中对位于内含子5'端的剪接保守位点“GU”以及基因5' UTR区的内含子剪接位点处进行突变,结果发现利用该方法可高效的删除或改变mRNA特定剪接形式或组成型剪接形式[104]。中山大学李剑峰实验室同样利用BE3在拟南芥和水稻中建立了有效的mRNA可变剪接体系[105]。
2.3.6 蛋白定向进化
碱基编辑系统除可以进行单个氨基酸位点的基因编辑以外,理论上还可以通过氨基酸位点的遗传筛选进行蛋白结构和功能的研究(图2E)。常兴实验室利用TAM系统在慢性粒细胞白血病细胞中靶标基因,有效鉴定了赋予细胞伊马替尼抗性的已知突变和新突变,这表明CBE系统可以在单碱基水平上筛选功能获得性变异[20]。Michael C. Bassiks实验室利用基于MS2招募hAID脱氨酶的CRISPR-X系统在哺乳动物细胞中突变了癌症治疗药物万珂(bortezomib)的靶标基因,从中找到了引发bortezomib耐药性的已知和全新的突变[21]。中国科学院上海生命科学研究院生物化学与细胞生物学研究所李劲松实验室利用优化的BE3向小鼠“人造精子”中导入了一个靶向基因的一个sgRNA慢病毒文库(含77个sgRNA),通过两轮的遗传筛选,他们发现并验证DND1蛋白的E59K、V60M、P76L和G82R对PGCs的发育具有重要作用[106]。该项研究是首次利用碱基编辑技术实现了个体水平蛋白质关键氨基酸功能位点的遗传筛选,理论上可同样筛选人类疾病相关基因的功能位点。目前,在植物中碱基编辑系统用于基因大规模突变并没有相关报道,分析原因主要有3个方面:(1)由于组织培养产生的大量再生植物没有快速且高效的阳性筛选体系,为鉴定突变体带来了很大的困扰;(2)缺乏高效的植物原生质体再生方法,限制了突变类型的饱和度;(3)将碱基编辑系统与多个sgRNA同时递送到植物细胞中是目前植物遗传转化的一大困难。因此,在植物中利用碱基编辑系统实现蛋白功能定向进化是目前重要研究方向之一。
3 ABE碱基编辑系统
3.1 ABE系统的开发
2017年,David R. Liu实验室开创性地开发了腺嘌呤碱基编辑系统(ABE),该系统由人工定向进化的腺嘌呤脱氨酶及nCas9组成,可实现A至G (A-G)或T至C (T-C)的替换[107]。其作用原理与CBE系统类似,即腺嘌呤脱氨酶可将靶位点处一定范围的腺嘌呤(A)脱氨变为肌苷(I),肌苷在DNA水平会被当作鸟嘌呤(G)进行读码与复制,最终实现A-G的改变。ABE的开发打破了之前碱基编辑系统仅能编辑C或G的限制,为碱基之间的相互转变提供了更多的可能性(图1)。
由于目前已知的腺嘌呤脱氨酶不能以DNA为底物对碱基A进行脱氨,这导致ABE系统的开发比CBE系统更具有挑战性。David R. Liu实验室最初也尝试将来自大肠杆菌TadA、大鼠ADA、人类ADAR2和人类ADAT2 4种腺嘌呤脱氨酶分别直接与nCas9融合,但均没有编辑活性,这表明必须将现有腺嘌呤脱氨酶进行定向改造,才有可能实现对碱基A的编辑[107]。对此,该实验室选取大肠杆菌TadA为改造对象,将随机突变的TadA序列融合dCas9构建成随机突变文库,通过ABE恢复氯霉素、卡那霉素、壮观霉素等抗性基因的功能,结合易错PCR、DNA重排等定向进化策略,进行相应的抗生素筛选, 先后经过7轮进化与改造后,成功筛选到了能直 接作用于ssDNA的ABE系统。在人类细胞中建立了目前效率最高且应用最广的ABE版本ABE7.10 (ecTadA-ecTadA*-nCas9)。ABE7.10将nCas9与野生型腺嘌呤脱氨酶ecTadA和经过定向进化的腺嘌呤脱氨酶ecTadA* (与野生型相比引入了W23R、H36L、P48A、R51L、L84F、A106V、D108N、H123Y、S146C、D147Y、R152P、E155V、I156F和K157N共14个位点的突变)二聚体相融合,在人类细胞中编辑效率约为50%,编辑活性窗口可覆盖sgRNA的第4~9位[107]。相比CBE,ABE不需要抑制烷基腺嘌呤DNA糖基化酶(alkyl adenine DNA glycosylase, AAG)的活性,这暗示着肌苷的损伤修复机制可能与AAG无关。
3.2 ABE系统的优化
研究发现ABE无论在哺乳动物还是在植物等中的编辑,相比CBE系统可保证高精确度的碱基替换和较低的Indels发生,因此科研人员对ABE系统的优化主要表现在对碱基编辑效率的提高、扩大编辑活性窗口和碱基编辑范围方面[12,13,15]。
对于碱基编辑效率的提高,本实验室率先通过优化密码子、调整腺嘌呤脱氨酶二聚体的位置和NLS的位置、增加NLS个数以及使用3种不同sgRNA形式等手段优化的PABE-7碱基编辑器相比原始的ABE7.10的编辑效率在小麦和水稻中提高了1.1倍[108]。David R. Liu实验室通过不同公司优化的密码子序列和增加NLS的个数而建立的ABEmax也提高了对碱基A的替换效率[26]。另外,CP1012- ABEmax、CP1028-ABEmax、CP1041-ABEmax和CP1249-ABEmax碱基编辑器在保证效率与ABEmax相当的情况下,可在一定程度上将4~8位的碱基编辑窗口拓展为4~12位[25]。在扩大PAM选择范围方面,VQR-SpCas9 (PAM: NGA)、VRER-SpCas9 (PAM: NGCG)、VRQR-SpCas9 (PAM: NGA)、SaCas9 (PAM: NNGRRT)、SaKKH (PAM: NNNRRT)、ScCas9 (PAM:NNGN)、xCas9 (PAM:NG)和SpCas9-NG (PAM: NG)的融合扩大了ABE在动植物基因组可编辑位点的范围[25,30,74,109]。
3.3 ABE系统的应用
ABE系统的开发为创制更多的动植物突变体、治疗更多的点突引起的疾病以及创制优异的农作物性状等提供了更加全面有力的工具(表3)。
在动物中,目前ABE系统已成功用于各种动物细胞系、人类胚胎、小鼠、大鼠、兔子和斑马鱼等中[12,13,15,110]。ABE可修改提前终止密码子突变,当靶向非编码链时可将编码链的TAA、TAG和TGA修改为CAA、CAG和CGA (图2C)。Jin-Soo Kim实验室率先利用ABE7.10对小鼠基因引入了错义突变,并将ABE7.10包裹到双反式剪接的AAV病毒系统中,对基因进行了无义突变,建立了杜氏肌营养不良的小鼠疾病模型[111]。上海科技大学黄行许实验室通过编辑和基因,建立了ABE介导的相关小鼠疾病模型,同时该研究使用不同的Cas9同源蛋白实现了ABE和CBE的同时编辑[49]。另外,该实验室利用ABEmax-NG在动物细胞内实现了精准的mRNA剪接位点的编辑[112]。Liang等[113]在小鼠中编辑了基因和基因的可变剪接位点,并在含有突变的小鼠中观察到了相应的杜氏肌营养不良的表型。Yang等[114]利用ABE7.10建立了由突变引起的Ⅰ型酪氨酸血症小鼠模型,并且该研究也证实SaKKH-ABE和VQR-ABE两种编辑器同样可在小鼠中产生有效的A-G的突变。在大鼠中,Yang等[114]和Ma等[115]实现了大鼠基因的编辑,并且Yang等[114]在被ABE7.10编辑的D645G/ I646V F1后代的大鼠多个组织中发现了充满糖原的大溶酶体异常积累,建立了庞贝氏症大鼠疾病模型。在兔子中,Liu等[62]利用ABE7.10高效地编辑了兔子中的、和基因,并且T279A突变体表现出与人类家族性X-连锁扩张型心肌病相似的典型临床症状。除了哺乳动物,Qin等[116]利用斑马鱼密码子优化的zABE7.10成功在斑马鱼、、和中引入了突变,并且突变体有着相应的表型,该实验室进一步将zABE7.10max成功用于斑马鱼基因的编辑。
在植物中,ABE系统已成功被用于水稻、小麦、拟南芥和油菜(L.)等相关基因突变提的创制及相应性状的创制(表3)。中国科学院上海植物逆境生物学研究中心朱健康实验室利用ABE-P1 (ABE7.10)和ABE-P2 (ABEsa)编辑了水稻的6个基因,并在后续研究中实现了ABE-P3 (VQR-ABE)、ABE-P4 (VRER-ABE)和ABE-P5 (SaKKH-ABE)、ABE-NG和ABE-NG-S (只融合人工进化的TadA变体TadA*)的进一步拓展,且在水稻中可同时实现C-T和A-G的同时编辑[30,74,117]。Yan等[118]同样证明ABE在水稻中可高效工作。而本实验室利用经过多种元件优化后的PABE-7版本编辑了水稻和小麦的多个基因,并且获得了相应的小麦突变体和抗除草剂的水稻基因突变材料[108]。Jin-Soo Kim实验室利用RPS5A启动子驱动ABE,在双子叶拟南芥和油菜中实现了高效的编辑,并创制了拟南芥早花的突变体材料以及实现了油菜基因mRNA的可变剪接[119]。
4 碱基编辑系统的脱靶效应及改善策略
碱基编辑系统最重要的用途在于实现基因的碱基替换,从而达到治疗疾病及作物改良的目的。因此,其编辑的特异性备受关注,也尤为重要。碱基编辑系统的脱靶分为3个方面(图3):
第一、传统的可预测的脱靶。该类脱靶是指碱基编辑系统除了编辑靶位点以外,还可以编辑与靶位点相似性很高的其他位点,而这些位点往往与靶位点只有几个碱基的错配。这类的脱靶位点一般可用目前的软件预测(如Cas-OFFinder、E-CRISP和CRISPR-P等),后续结合分子生物学手段来判断是否存在脱靶效应。导致该类脱靶发生的原因主要有两方面:一是sgRNA序列的容错性,脱靶效应的高低与sgRNA和靶标DNA之间错配碱基的数目有关[120,121];二是Cas9核酸酶自身的非特异性、脱氨酶与Cas9融合蛋白的高浓度及长时间持续表达都容易造成对非靶标位点的编辑。目前降低此类脱靶效应的策略主要有以下几种[122]:(1) sgRNA的修饰。利用缩短的sgRNA (gX18或gX17)或延长的sgRNA (gX20或ggX20)均可在保持BE3或ABE系统效率的前提下降低它们的脱靶效应[120,121];(2)使用高保真的Cas9核酸酶。利用工程化的Cas9变体可降低碱基编辑的脱靶情况。David R. Liu实验室通过将HF1-Cas9与BE3融合,建立的HF-BE3可明显降低原始BE3对非靶标位点的编辑[123]。Jin-Soo Kim实验室通过将Sniper Cas9与ABE融合建立的Sniper ABE7.10对ABE系统的脱靶效应有明显的改善[120];(3)缩短脱氨酶与Cas9核酸酶融合蛋白在细胞中的作用时间。利用碱基编辑器与sgRNA形成的核糖核蛋白复合体(ribonucleoprotein, RNP)取代其DNA表达形式直接转化细胞,可以使碱基编辑器在细胞内快速降解,从而降低脱靶效应。研究表明,递送BE3 RNP和ABE RNP均可提高相应碱基编辑系统的特异性[120,123]。
第二、全基因组范围内不可预测的脱靶。该类脱靶位点往往随机或有一定规律的分布在全基因组水平上,但通过目前的软件无法预测。体外优化的Digenome-seq、EndoⅧ或Endo V-seq的脱靶评估手段并不能真实有效的反应体内环境下碱基编辑系统的特异性[120,121,124]。由于脱氨酶需要结合在ssDNA处发挥作用,因此细胞内自身存在的ssDNA、转录区域、复制区域等成为了碱基编辑系统在全基因组水平的潜在脱靶对象。Lee等[125]发现在小鼠胚胎中BE3在sgRNA前后200个碱基处的脱靶效应高于ABE系统。而在小鼠中,杨辉实验室利用建立的GOTI技术结合全基因组测序分析,发现BE3在小鼠体内能够引发比自发突变高20倍的C-T的单核苷酸变异(single nucleotide variations, SNVs)[126];同一时间,本实验室在植物中发现了类似的现象,即BE3和HF1-BE3能够引起水稻全基因组水平的C-T SNVs的增加[127],且这两项研究均表明这些C-T SNVs在染色体间均匀分布,但呈现出在转录活跃区富集的趋势[126,127]。而对于融合其他类型胞嘧啶脱氨酶的CBE系统是否也存在该现象还需要进一步的评估。更早的研究发现,过表达不同胞嘧啶脱氨酶或UGI在酵母、大肠杆菌及人类细胞系中会导致基因组内C-T SNVs的增加[128~130]。这些问题的发现一方面暗示着现阶段将碱基编辑系统于疾病治疗等精准改造方面有一定的风险,另一方面促使科研人员更深层次的探索究竟是胞嘧啶脱氨酶、UGI还是两个组分共同作用的结果造成了全基因组水平的脱靶,从而改进开发出更加精准安全的CBE系统。而相比之下,ABE系统在全基因水平具有很高的特异性[126,127],这可能与ABE系统融合的腺嘌呤脱氨酶是经过人工定向进化以及ABE系统中并没有UGI组分有关。
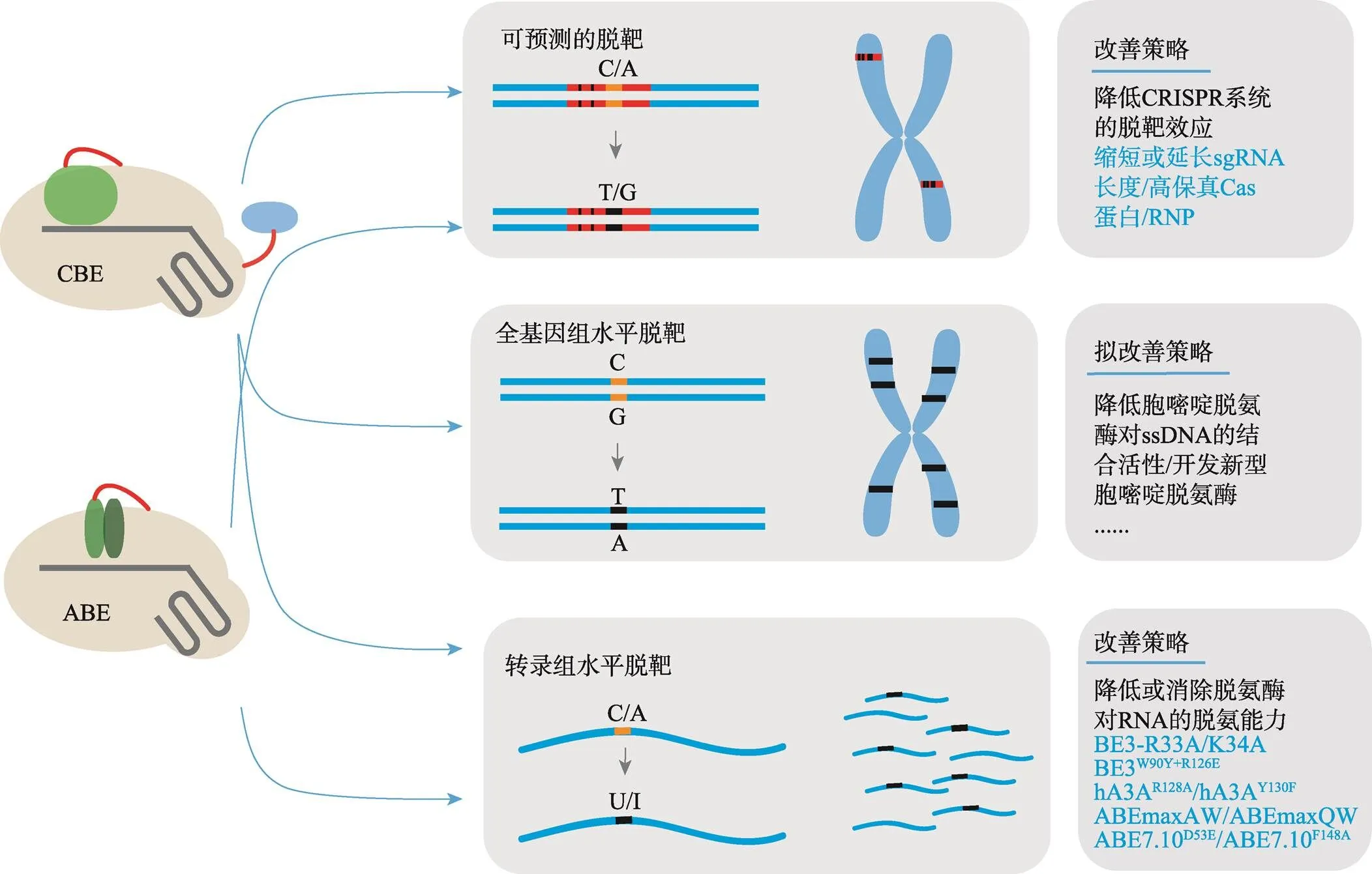
图3 碱基编辑系统的脱靶效应及改善策略
碱基编辑系统的脱靶效应分3个层次:传统的可预测的脱靶、全基因组水平的脱靶及转录组水平的脱靶。目前针对可预测的脱靶及转录组水平的脱靶已有较好的改善策略,而对于CBE系统在全基因组范围的脱靶只有初步的猜想。
第三、转录组水平的脱靶。无论是胞嘧啶脱氨酶还是腺嘌呤脱氨酶在之前的研究报道中都是能够作用于RNA或自身完全作用于RNA的脱氨酶,因此这就有可能导致碱基编辑系统在RNA水平产生脱靶。2019年,Keith Joung实验室通过转录组测序,率先证明了BE3和ABE系统在人类细胞系转录组水平存在严重的脱靶效应[131]。其中,BE3可在38%~58%的表达基因中以0.07%~100%的频率诱导成千上万的C-U的编辑;而ABEmax在RNA水平对碱基A的脱靶编辑也可高达100%[131]。随后,David R. Liu实验室也证明ABE在哺乳动物细胞系中存在很低但能够检测到的RNA脱靶[132]。杨辉实验室定量评价了CBEs和ABEs诱导的RNA单核苷酸变异,结果发现胞嘧啶碱基编辑器BE3和腺嘌呤碱基编辑器ABE7.10都能产生成千上万的非靶向RNA SNVs[133]。对于碱基编辑系统在RNA水平的严重脱靶现象,目前主要的解决方案是利用工程化的脱氨酶变体,即一般通过改造脱氨酶与RNA的活性结合位点从而来降低对RNA的编辑效率。依据此思路,Keith Joung实验室开发的BE3-R33A和BE3-R33A/K34A的变体可将BE3对RNA的编辑分别降低390倍和3800倍。杨辉实验室开发的BE3W90Y+R126E、hA3AR128A和hA3AY130F可直接将BE脱靶降低到本底水平[133]。对于ABE系统的改造,David R. Liu实验室开发的ABEmaxAW (TadA E59A, TadA* V106W)和ABEmaxQW (TadA E59Q, TadA* V106W)可在保证一定DNA水平的编辑效率的同时,降低其在RNA水平的编辑效率[132]。而杨辉实验室也开发了两种ABE变体,ABE7.10D53E和ABE7.10F148A,其中ABE7.10F148A可直接消除ABE系统在转录组水平的脱靶效应[133]。
总之,ABE系统因其在DNA水平具有较高的特异性,而在RNA水平的脱靶也有了很好的解决方案,因此就目前研究结果来看ABE系统具有较高的安全性。而CBE系统在DNA水平的脱靶效应需要进一步的改善,开发出更安全的CBE工具。
5 碱基编辑系统的发展前景
碱基编辑技术的开发为定向修正碱基突变和创制基因组中的关键核苷酸变异提供了重要工具,展现了其在遗传疾病治疗与动植物新品种培育等方面的重大应用价值。然而碱基编辑系统仍然存在一些不足之处。最为明显的是,CBE及ABE系统只能实现嘧啶对嘧啶、嘌呤对嘌呤的改变,即C-T、T-C、G-A和A-G的改变,而不能实现碱基之间的颠换,如C-G、C-A、A-T和A-C的转变。虽然目前有研究报道CBE系统可产生C至G或A的突变,但其发生有很大的随机性并且效率很低。另外,碱基编辑系统相比HR在全基因组及RNA水平的脱靶效应更高,安全性更低。受自身的编辑活性窗口及PAM的限制,使碱基编辑系统在实际应用中并不能对所有的靶点都能有效编辑。因此,碱基编辑系统在技术及应用层面等仍具有更深层次的开发潜力,如新型碱基编辑系统的开发、全基因组水平的特异性的提高、碱基编辑材料递送形式的优化等。
(1)开发新型碱基编辑系统。主要表现在碱基之间可进行颠换的碱基编辑系统的挖掘。以C变G为例,目前已有关于其设计思路及相关专利的报道[134]。当C被胞嘧啶脱氨酶脱氨之后会在UDG的作用下形成AP位点,进而招募TLS聚合酶如Rev1等结合在此处进行修复,该酶会在AP位点的对侧链偏向性的插入碱基C,留下一个可连接的缺口,最终在DNA连接酶及DNA修复过程中实现C-G的改变。David R. Liu实验室公开相关专利,采用上述思路先后尝试了多种构建策略:rAPOBEC1-d/nCas9-UDG、rAPOBEC1-d/nCas9-UDG变体(UdgX、UdgX*和UdgX_On等)以及rAPOBEC1-d/nCas9-UDG/(UDG变体)+TLS聚合酶(Rev1、Kappa、Lota和Eta)等,经测试后发现只有胞嘧啶脱氨酶同时融合UDG (或UDG变体)和TLS聚合酶时编辑效果最佳,但编辑效率并不理想。可能原因在于UDG只有在细胞处于S期才会将U切除形成AP位点,并且TLS聚合酶必须在ssDNA缺口处才能复制。美国宾夕法尼亚大学科学家Kiran S. Gajula针对此问题提出了可能的改善策略,即一方面可人工定向进化Rev1,理想的Rev1应具有5ʹ至3ʹ外切酶的活性,同时具有自主打开缺口并牢牢结合在AP位点处的功能,这样可大大增加其接触ssDNA的几率;另一方面可融合EvolvR系统中的enCas9变体,从而有可能提高C-G的效率[134]。因而,对于C-G及其他碱基之间相互转变的新型单碱基技术体系的开发需要不断的探索及优化。此外,如何简单高效的实现C-T及A-G的同时编辑也值得探索。虽然目前在动植物中已有相关报道,但均是采用直接融合不同Cas9变体的策略,通过不同Cas9识别不同PAM的特点而实现。但该方案使用的表达载体过大,编辑效率低且能靶向的位点局限性大。因此可尝试使用不同的适配体如MS2、PP7和boxB等招募不同的脱氨酶等方法,快速而高效的实现不同碱基的同时突变。
(2)提高碱基编辑系统的特异性。主要表现为需要开发在全基因组范围具有更高特异性和更高安全性的CBE系统。具体的解决方案可尝试YEE-BE3等其他新近开发的CBE碱基编辑器,由于YEE-BE3与DNA的结合活性较弱,对降低全基因组水平的脱靶效应也许会有一定的帮助[29];或者可人工进化脱氨酶找寻新的变体,在保证其正常编辑效率的同时能从本质上解决CBE在全基因组范围的脱靶。另外,可尝试优化胞嘧啶脱氨酶或UGI组分,如Keith Joung实验室在相关国际专利提出初步设想,即将胞嘧啶脱氨酶分割到两个载体中表达,一部分与ZF或TALE效应因子融合,另一部分与nCas9融合,只有当两部分同时靶向靶位点时,才能通过内含肽二聚化形成具有活性的胞嘧啶脱氨酶,从而有可能降低其在全基因组水平的脱靶风险。除了可改进脱氨酶组分之外,也可将nCas9组分替换为在全基因组水平更加特异的其他Cas蛋白,如dSaCas9或者dCas12a。另外,RNP相比DNA在细胞内更容易快速降,因此猜测使用CBE RNP形式对降低CBE在全基因组脱靶会有一定的改善效果。
(3)碱基编辑材料的形式及递送方式方面。如何将碱基编辑材料有效且安全的递送到细胞内是目前碱基编辑系统需要继续改进及发展的重要方面。目前,碱基编辑系统介导的编辑材料包括质粒(DNA)、体外转录物(transcripts, IVTs)以及RNP 3种形式发挥作用。目前在动物相关研究中,这3种形式的碱基编辑均已成功报道,主要通过注射、电转、腺相关病毒(AAV)及阳离子脂质体介导的转化方法实现,其中以mRNA的形式居多[12]。但如何缩小BE载体大小使其更容易装载入腺病毒载体、甚至开发出用于基因治疗的新型递送系统及可直接进行组织特异性的递送仍需进一步的探索。相比在哺乳动物细胞中的研究,碱基编辑材料的递送在植物中发展相对缓慢。对于质粒DNA介导的碱基编辑是目前最常用的形式,在植物中可通过基因枪轰击、农杆菌浸染及PEG介导的转化方法实现。但是当DNA整合到基因组中会带来因碱基编辑DNA元件持续表达导致的潜在脱靶效应的增加,且编辑的产品会引起公众的担忧。尽管本研究组采用瞬时表达碱基编辑系统的DNA方式可在一定程度上降低外源DNA的整合[28,82,108],但不是根本的解决办法。以碱基编辑IVTs或RNP的形式作为基因组编辑材料可以更加有效避免外源DNA整合事件的发生,整个过程中不涉及任何外源DNA,因而更有助于避免转基因的监管。本实验室率先利用BE3 mRNA形式在小麦中实现了基因的编辑[83]。对于RNPs形式的编辑,同样是本实验室初步报道了PEG介导的转化方法在小麦原生质体中初步实现了A3A-PBE-ΔUGI RNP的编辑[28],而对于RNP在植物个体中的碱基编辑并没有成功案例。因此,在植物中如何高效且安全地将碱基编辑RNP形式递送到植物细胞中从而获得DNA-free的突变植株,成为目前植物碱基编辑系统亟待解决的技术瓶颈。由于在植物遗传转化过程中对RNP递送数量及质量均有损失,因此需要摸索RNP各种遗传转化条件,如测试不同的纳米金颗粒包被RNP等。另外,可尝试一些新型基因组编辑材料递送方式,如磁纳米颗粒转化法、碳纳米管转化法、病毒载体转化法等。
总之,碱基编辑技术在人类疾病研究、动植物科学基础研究和育种等方面应用前景广阔,该技术的进一步优化及改造将推动农业生产,生物医药及生命科学等领域的快速发展。
致谢
感谢本实验室王延鹏青年研究员在图片制作和文章写作方面给予的帮助,感谢李超和靳帅博士研究生在文章写作方面给予的建议。
[1] Li J, Zhang Y, Chen KL, Shan QW, Wang YP, Liang Z, Gao CX. CRISPR/Cas: a novel way of RNA-guided genome editing., 2013, 35(11): 1265–1273.李君, 张毅, 陈坤玲, 单奇伟, 王延鹏, 梁振, 高彩霞. CRISPR/Cas系统: RNA靶向的基因组定向编辑新技术. 遗传, 2013, 35(11): 1265–1273.
[2] Shan QW, Gao CX. Research progress of genome editing and derivative technologies in plants., 2015, 37(10): 953–973.单奇伟,高彩霞. 植物基因组编辑及衍生技术最新研究进展. 遗传, 2015, 37(10): 953–973.
[3] Li DQ, Yang YP. Analysis on the international development trends of crop genome editing technology., 2017, 49(2): 179–190.李东巧, 杨艳萍. 作物基因组编辑技术国际发展态势分析. 中国科学: 生命科学, 2017, 49(2): 179–190.
[4] Zhang Y, Massel K, Godwin ID, Gao C. Applications and potential of genome editing in crop improvement., 2018, 19(1): 210.
[5] Zhang F. Development of CRISPR-Cas systems for genome editing and beyond., 2019, 52, e6, 1-31, doi: 10.1017/S0033583519000052.
[6] Jinek M, Chylinski K, Fonfara I, Hauer M, Doudna JA, Charpentier E. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity., 2012, 337(6096): 816–821.
[7] Jiao RJ, Gao CX. CRISPR/Cas9 can do anything?, 2017, 47(11): 1127–1129.焦仁杰, 高彩霞. CRISPR/Cas9无所不能? 中国科学: 生命科学, 2017, 47(11): 1127–1129.
[8] Symington LS, Gautier J. Double-strand break end resection and repair pathway choice., 2011, 45: 247–271.
[9] Landrum MJ, Lee JM, Benson M, Brown G, Chao C, Chitipiralla S, Gu B, Hart J, Hoffman D, Hoover J, Jang W, Katz K, Ovetsky M, Riley G, Sethi A, Tully R, Villamarin-Salomon R, Rubinstein W, Maglott DR. ClinVar: public archive of interpretations of clinically relevant variants., 2016, 44(D1): D862–868.
[10] Zhao K, Tung CW, Eizenga GC, Wright MH, Ali ML, Price AH, Norton GJ, Islam MR, Reynolds A, Mezey J, McClung AM, Bustamante CD, McCouch SR. Genome- wide association mapping reveals a rich genetic architecture of complex traits in., 2011, 2: 467.
[11] Chen K, Wang Y, Zhang R, Zhang H, Gao, C. CRISPR/Cas genome editing and precision plant breeding in agriculture., 2019, 70: 667–697.
[12] Rees HA, Liu DR. Base editing: precision chemistry on the genome and transcriptome of living cells., 2018, 19(12): 770–788.
[13] Molla KA, Yang Y. CRISPR/Cas-mediated base editing: technical considerations and practical applications., 2019, S0167-7799(19): 30053– 30058.
[14] Wei Y, Zhang XH, Li DL. The "new favorite" of gene editing technology-single base editors., 2017, 39(12): 1115–1121.魏瑜, 张晓辉, 李大力.基因编辑之“新宠”—单碱基基因组编辑系统.遗传, 2017, 39(12): 1115–1121.
[15] Yang B, Yang L, Chen J. Development and application of base editors., 2019, 2: 91–104.
[16] Hess GT, Tycko J, Yao D, Bassik MC. Methods and applications of CRISPR-mediated base editing in eukaryotic genomes., 2017, 68(1): 26–43.
[17] Liu JH, Liang PP, Shi G, Huang JJ.Research progress of base editing system., 2017, 39(6): 457–462.刘佳慧, 梁普平, 时光, 黄军就.单碱基基因编辑系统的研究进展. 世界科技研究与发展, 2017, 39(6): 457–462.
[18] Komor AC, Kim YB, Packer MS, Zuris JA, Liu DR. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage., 2016, 533(7603): 420–424.
[19] Nishida K, Arazoe T, Yachie N, Banno S, Kakimoto M, Tabata M, Mochizuki M, Miyabe A, Araki M, Hara KY, Shimatani Z, Kondo A. Targeted nucleotide editing using hybrid prokaryotic and vertebrate adaptive immune systems., 2016, 353(6305): aaf8729.
[20] Hess GT, Frésard L, Han K, Lee CH, Li A, Cimprich KA, Montgomery SB, Bassik MC. Directed evolution using dCas9-targeted somatic hypermutation in mammalian cells., 2016, 13(12): 1036–1042.
[21] Ma Y, Zhang J, Yin W, Zhang Z, Song Y, Chang X. Targeted AID-mediated mutagenesis (TAM) enables efficient genomic diversification in mammalian cells., 2016, 13(12): 1029–1035.
[22] Yang L, Briggs AW, Chew WL, Mali P, Guell M, Aach J, Goodman DB, Cox D, Kan Y, Lesha E, Soundararajan V, Zhang F, Church G. Engineering and optimising deaminase fusions for genome editing., 2016, 7: 13330.
[23] Komor AC, Zhao KT, Packer MS, Gaudelli NM, Waterbury AL, Koblan LW, Kim YB, Badran AH, Liu DR. Improved base excision repair inhibition and bacteriophage Mu Gam protein yields C:G-to-T:a base editors with higher efficiency and product purity., 2017, 3(8): eaao4774.
[24] Wang L, Xue W, Yan L, Li X, Wei J, Chen M, Wu J, Yang B, Yang L, Chen J. Enhanced base editing by co-expression of free uracil DNA glycosylase inhibitor., 2017, 27(10): 1289–1292.
[25] Huang TP, Zhao KT, Miller SM, Gaudelli NM, Oakes BL, Fellmann C, Savage DF, Liu DR. Circularly permuted and PAM-modified Cas9 variants broaden the targeting scope of base editors., 2019, 37(6): 626–631.
[26] Koblan LW, Doman JL, Wilson C, Levy JM, Tay T, Newby GA, Maianti JP, Raguram A, Liu DR. Improving cytidine and adenine base editors by expression optimization and ancestral reconstruction., 2018, 36(9): 843–846.
[27] Zafra MP, Schatoff EM, Katti A, Foronda M, Breinig M, Schweitzer AY, Simon A, Han T, Goswami S, Montgomery E, Thibado J, Kastenhuber ER, Sánchez-Rivera FJ, Shi J, Vakoc CR, Lowe SW, Tschaharganeh DF, Dow Le. Optimized base editors enable efficient editing in cells, organoids and mice., 2018, 36(9): 888– 893.
[28] Zong Y, Song Q, Li C, Jin S, Zhang D, Wang Y, Qiu JL, Gao C. Efficient C-to-T base editing in plants using a fusion of nCas9 and human APOBEC3A., 2018, 36(9): 950–953.
[29] Kim YB, Komor AC, Levy JM, Packer MS, Zhao KT, Liu DR. Increasing the genome-targeting scope and precision of base editing with engineered Cas9-cytidine deaminase fusions., 2017, 35(4): 371– 376.
[30] Hua K, Tao X, Zhu JK. Expanding the base editing scope in rice by using Cas9 variants., 2018, 17(2): 499–504.
[31] Jakimo N, Chatterjee P, Nip L, Jacobson JM. A Cas9 with complete PAM recognition for adenine dinucleotides., 2018, http://dx.doi.org/10.1101/429654.
[32] Li X, Wang Y, Liu Y, Yang B, Wang X, Wei J, Lu Z, Zhang Y, Wu J, Huang X, Yang L, Chen J. Base editing with a Cpf1-cytidine deaminase fusion., 2018, 36(4): 324–327.
[33] Kleinstiver BP, Sousa AA, Walton RT, Tak YE, Hsu JY, Clement K, Welch MM, Horng JE, Malagon-Lopez J, Scarfò I, Maus MV, Pinello L, Aryee MJ, Joung JK. Engineered CRISPR-Cas12a variants with increased activities and improved targeting ranges for gene, epigenetic and base editing., 2019, 37(3): 276–282.
[34] Hu JH, Miller SM, Geurts MH, Tang W, Chen L, Sun N, Zeina CM, Gao X, Rees HA, Lin Z, Liu DR. Evolved Cas9 variants with broad PAM compatibility and high DNA specificity., 2018, 556(7699): 57–63.
[35] Nishimasu H, Shi X, Ishiguro S, Gao L, Hirano S, Okazaki S, Noda T, Abudayyeh OO, Gootenberg JS, Mori H, Oura S, Holmes B, Tanaka M, Seki M, Hirano H, Aburatani H, Ishitani R, Ikawa M, Yachie N, Zhang F, Nureki O. Engineered CRISPR-Cas9 nuclease with expanded targeting space., 2018, 361(6408): 1259–1262.
[36] Tan J, Zhang F, Karcher D, Bock R. Engineering of high-precision base editors for site-specific single nucleotide replacement., 2019, 10(1): 439.
[37] Jiang W, Feng S, Huang S, Yu W, Li G, Yang G, Liu Y, Zhang Y, Zhang L, Hou Y, Chen J, Huang X. BE-PLUS: a new base editing tool with broadened editing window and enhanced fidelity., 2018, 28(8): 855–861.
[38] Wang X, Li J, Wang Y, Yang B, Wei J, Wu J, Wang R, Huang X, Chen J, Yang L. Efficient base editing in methylated regions with a human APOBEC3A-Cas9 fusion., 2018, 36(10): 946–949.
[39] Liu LD, Huang M, Dai P, Liu T, Fan S, Cheng X, Zhao Y, Yeap LS, Meng FL. Intrinsic nucleotide preference of diversifying base editors guides antibodyaffinity maturation., 2018, 25(4): 884–892.e3.
[40] Ren B, Yan F, Kuang Y, Li N, Zhang D, Zhou X, Lin H, Zhou H. Improved base editor for efficiently inducing genetic variations in rice with CRISPR/Cas9-guided hyperactive hAID mutant., 2018, 11(4): 623–626.
[41] Gehrke JM, Cervantes O, Clement MK, Wu Y, Zeng J, Bauer DE, Pinello L, Joung JK. An APOBEC3A-Cas9 base editor with minimized bystander and off-target activities., 2018, 36(10): 977–982.
[42] Liu Z, Chen S, Shan H, Chen M, Song Y, Lai L, Li Z. Highly precise base editing with CC context-specificity using engineered human APOBEC3G-nCas9 fusions., 2019, http://dx.doi.org/10.1101/658351.
[43] Li Y, Ma S, Sun L, Zhang T, Chang J, Lu W, Chen X, Liu Y, Wang X, Shi R, Zhao P, Xia Q. Programmable single and multiplex base-editing inusing RNA-guided cytidine deaminases., 2018, 8(5): 1701–1709.
[44] Li G, Liu Y, Zeng Y, Li J, Wang L, Yang G, Chen D, Shang X, Chen J, Huang X, Liu J. Highly efficient and precise base editing in discarded human tripronuclear embryos., 2017, 8(10): 776–779.
[45] Shevidi S, Uchida A, Schudrowitz N, Wessel GM, Yajima M. Single nucleotide editing without DNA cleavage using CRISPR/Cas9-deaminase in theembryo., 2017, 246(12): 1036–1046.
[46] Chang YJ, Xu CL, Cui X, Bassuk AG, Mahajan VB, Tsai YT, Tsang SH. CRISPR base editing in induced pluripotent stem cells., 2019, doi: 10.1007/7651_2019_243.
[47] Huang L, Dong H, Zheng J, Wang B, Pan L. Highly efficient single base editing inwith CRISPR/Cas9 cytidine deaminase fusion., 2019, 223–225: 44–50.
[48] Yang G, Zhu TY, Lu ZY, Li GL, Zhang H, Feng SJ, Liu YJ, Li JN, Zhang Y, Chen J, Guo XJ, Huang XX. Generation of isogenic single and multiplex gene knockout mice by base editing-induced STOP., 2018, 63(17): 1101–1107.
[49] Liu Z, Lu Z, Yang G, Huang S, Li G, Feng S, Liu Y, Li J, Yu W, Zhang Y, Chen J, Sun Q, Huang X. Efficient generation of mouse models of human diseases via ABE- and BE-mediated base editing., 2018, 9(1): 2338.
[50] Tang J, Lee T, Sun T. Single-nucleotide editing: from principle, optimization to application., 2019, doi: 10.1002/humu.23819.
[51] Chadwick AC, Wang X, Musunuru K.base editing of PCSK9 (Proprotein Convertase Subtilisin/ Kexin Type 9) as a therapeutic alternative to genome editing., 2017, 37(9): 1741–1747.
[52] Yang G, Zhou C, Wang R, Huang S, Wei Y, Yang X, Liu Y, Li J, Lu Z, Ying W, Li X, Jing N, Huang X, Yang H, Qiao Y. Base-editing-mediated R17H substitution in histone H3 reveals methylation-dependent regulation of Yap signaling and early mouse embryo development., 2019, 26(2): 302–312.e4.
[53] Zeng Y, Li J, Li G, Huang S, Yu W, Zhang Y, Chen D, Chen J, Liu J, Huang X. Correction of the marfan syndrome pathogenicmutation by base editing in human cells and heterozygous embryos., 2018, 26(11): 2631–2637.
[54] Liang P, Ding C, Sun H, Xie X, Xu Y, Zhang X, Sun Y, Xiong Y, Ma W, Liu Y, Wang Y, Fang J, Liu D, Songyang Z, Zhou C, Huang J. Correction of βmutant by base editor in human embryos., 2017, 8(11): 811–822.
[55] Zhang M, Zhou C, Wei Y, Xu C, Pan H, Ying W, Sun Y, Sun Y, Xiao Q, Yao N, Zhong W, Li Y, Wu K, Yuan G, Mitalipov S, Chen ZJ, Yang H. Human cleaving embryos enable robust homozygotic nucleotide substitutions by base editors., 2019, 20(1): 101.
[56] Zhang H, Pan H, Zhou C, Wei Y, Ying W, Li S, Wang G, Li C, Ren Y, Li G, Ding X, Sun Y, Li GL, Song L, Li Y, Yang H, Liu Z. Simultaneous zygotic inactivation of multiple genes in mouse through CRISPR/Cas9-mediated base editing., 2018, 145(20): dev168906.
[57] Kim K, Ryu SM, Kim ST, Baek G, Kim D, Lim K, Chung E, Kim S, Kim JS. Highly efficient RNA-guided base editing in mouse embryos., 2017, 35(5): 435–437.
[58] Liang P, Sun H, Sun Y, Zhang X, Xie X, Zhang J, Zhang Z, Chen Y, Ding C, Xiong Y, Ma W, Liu D, Huang J, Songyang Z. Effective gene editing by high-fidelity base editor 2 in mouse zygotes., 2017, 8(8): 601–611.
[59] Sasaguri H, Nagata K, Sekiguchi M, Fujioka R, Matsuba Y, Hashimoto S, Sato K, Kurup D, Yokota T, Saido TC. Introduction of pathogenic mutations into the mousegene by base editor and Target-AID., 2018, 9(1): 2892.
[60] Villiger L, Grisch-Chan HM, Lindsay H, Ringnalda F, Pogliano CB, Allegri G, Fingerhut R, Häberle J, Matos J, Robinson MD, Thöny B, Schwank G. Treatment of a metabolic liver disease bygenome base editing in adult mice., 2018, 24(10): 1519–1525.
[61] Li J, Liu Z, Huang S, Wang X, Li G, Xu Y, Yu W, Chen S, Zhang Y, Ma H, Ke Z, Chen J, Sun Q, Huang X. Efficient base editing in G/C-rich regions to model androgen insensitivity syndrome., 2019, 29(2): 174–176.
[62] Liu Z, Chen M, Chen S, Deng J, Song Y, Lai L, Li Z. Highly efficient RNA-guided base editing in rabbit., 2018, 9(1): 2717.
[63] Liu Z, Chen M, Shan H, Chen S, Xu Y, Song Y, Zhang Q, Yuan H, Ouyang H, Li Z, Lai L. Expanded targeting scope and enhanced base editing efficiency in rabbit using optimized xCas9(3.7)., 2019, doi: 10.1007/s00018-019-03110-8.
[64] Liu Z, Chen S, Shan H, Zhang Q, Chen M, Lai L, Li Z. Efficient and precise base editing in rabbits using human APOBEC3A-nCas9 fusions., 2019, 5: 31.
[65] Liu Z, Shan H, Chen S, Chen M, Zhang Q, Lai L, Li Z. Improved base editor for efficient editing in GC contexts in rabbits with an optimized AID-Cas9 fusion., 2019, doi: 10.1096/fj.201900476RR.
[66] Li Z, Duan X, An X, Feng T, Li P, Li L, Liu J, Wu P, Pan D, Du X, Wu S. Efficient RNA-guided base editing for disease modeling in pigs., 2018, 4: 64.
[67] Xie J, Ge W, Li N, Liu Q, Chen F, Yang X, Huang X, Ouyang Z, Zhang Q, Zhao Y, Liu Z, Gou S, Wu H, Lai C, Fan N, Jin Q, Shi H, Liang Y, Lan T, Quan L, Li X, Wang K, Lai L. Efficient base editing for multiple genes and loci in pigs using base editors., 2019, 10(1): 2852.
[68] Zhou S, Cai B, He C, Wang Y, Ding Q, Liu J, Liu Y, Ding Y, Zhao X, Li G, Li C, Yu H, Kou Q, Niu W, Petersen B, Sonstegard T, Ma B, Chen Y, Wang X. Programmable base editing of the sheep genome revealed no genome-wide off-target mutations., 2019, 10: 215.
[69] Li G, Zhou S, Li C, Cai B, Yu H, Ma B, Huang Y, Ding Y, Liu Y, Ding Q, He C, Zhou J, Wang Y, Zhou G, Li Y, Yan Y, Hua J, Petersen B, Jiang Y, Sonstegard T, Huang X, Chen Y, Wang X. Base pair editing in goat: nonsense codon introgression intoresults in longer hair., 2019, doi: 10.1111/febs.14983.
[70] Zhang Y, Qin W, Lu X, Xu J, Huang H, Bai H, Li S, Lin S. Programmable base editing of zebrafish genome using a modified CRISPR-Cas9 system., 2017, 8(1): 118.
[71] Tanaka S, Yoshioka S, Nishida K, Hosokawa H, Kakizuka A, Maegawa S.targeted single- nucleotide editing in zebrafish., 2018, 8(1): 11423.
[72] Park DS, Yoon M, Kweon J, Jang AH, Kim Y, Choi SC. Targeted base editing via RNA-guided cytidine deaminases inembryos., 2017, 40(11): 823–827.
[73] Shi Z, Xin H, Tian D, Lian J, Wang J, Liu G, Ran R, Shi S, Zhang Z, Shi Y, Deng Y, Hou C, Chen Y. Modeling human point mutation diseases inwith a modified CRISPR/Cas9 system., 2019, 33(6): 6962–6968.
[74] Hua K, Tao X, Han P, Wang R, Zhu JK. Genome engineering in rice using Cas9 variants that recognize NG PAM sequences., 2019, 12(7): 1003– 1014.
[75] Li J, Sun Y, Du J, Zhao Y, Xia L. Generation of targeted point mutations in rice by a modified CRISPR/Cas9 system., 2017, 10(3): 526–529.
[76] Lu Y, Zhu JK. Precise editing of a target base in the rice genome using a modified CRISPR/Cas9 system., 2017, 10(3): 523–525.
[77] Ren B, Liu L, Li S, Kuang Y, Wang J, Zhang D, Zhou X, Lin H, Zhou H. Cas9-NG greatly expands the targeting scope of the genome-editing toolkit by recognizing NG and other atypical PAMs in rice., 2019, 12(7): 1015–1026.
[78] Zhong Z, Sretenovic S, Ren Q, Yang L, Bao Y, Qi C, Yuan M, He Y, Liu S, Liu X, Wang J, Huang L, Wang Y, Baby D, Wang D, Zhang T, Qi Y, Zhang Y. Improving plant genome editing with high-fidelity xCas9 and non-canonical PAM-targeting Cas9-NG., 2019, 12(7): 1027–1036.
[79] Qin R, Li J, Li H, Zhang Y, Liu X, Miao Y, Zhang X, Wei P. Developing a highly efficient and wildly adaptive CRISPR-SaCas9 toolset for plant genome editing., 2018, 17(4): 706–708.
[80] Tang X, Ren Q, Yang L, Bao Y, Zhong Z, He Y, Liu S, Qi C, Liu B, Wang Y, Sretenovic S, Zhang Y, Zheng X, Zhang T, Qi Y, Zhang Y. Single transcript unit CRISPR 2.0 systems for robust Cas9 and Cas12a mediated plant genome editing., 2019, 17(7): 1431– 1445.
[81] Ren B, Yan F, Kuang Y, Li N, Zhang D, Lin H, Zhou H. A CRISPR/Cas9 toolkit for efficient targeted base editing to induce genetic variations in rice., 2017, 60(5): 516–519.
[82] Zong Y, Wang Y, Li C, Zhang R, Chen K, Ran Y, Qiu JL, Wang D, Gao C. Precise base editing in rice, wheat and maize with a Cas9-cytidine deaminase fusion., 2017, 35(5): 438–440.
[83] Zhang R, Liu J, Chai Z, Chen S, Bai Y, Zong Y, Chen K, Li J, Jiang L, Gao C. Generation of herbicide tolerance traits and a new selectable marker in wheat using base editing., 2019, 5(5): 480–485.
[84] Chen Y, Wang Z, Ni H, Xu Y, Chen Q, Jiang L. CRISPR/Cas9-mediated base-editing system efficiently generates gain-of-function mutations in., 2017, 60(5): 520–523.
[85] Tian S, Jiang L, Guo S, Li M, Zhang H, Ren Y, Gong G, Zong M, Liu F, Chen Q, Xu Y. Engineering herbicide-resistant watermelon variety through CRISPR/ Cas9-mediated base-editing., 2018, 37(9): 1353–1356.
[86] Qin L, Li J, Wang Q, Xu Z, Sun L, Alariqi M, Manghwar H, Wang G, Li B, Ding X, Rui H, Huang H, Lu T, Lindsey K, Daniell H, Zhang X, Jin S. High-efficient and precise base editing of C•G to T•A in the allotetraploid cotton () genome using a modified CRISPR/Cas9 system., 2019, doi: 10.1111/pbi.13168.
[87] Shimatani Z, Kashojiya S, Takayama M, Terada R, Arazoe T, Ishii H, Teramura H, Yamamoto T, Komatsu H, Miura K, Ezura H, Nishida K, Ariizumi T, Kondo A. Targeted base editing in rice and tomato using a CRISPR-Cas9 cytidine deaminase fusion., 2017, 35(5): 441–443.
[88] Shimatani Z, Fujikura U, Ishii H, Terada R, Nishida K, Kondo A. Herbicide tolerance-assisted multiplex targeted nucleotide substitution in rice., 2018, 20: 1325–1331.
[89] Wu Y, Xu W, Wang F, Zhao S, Feng F, Song J, Zhang C, Yang J. Increasing cytosine base editing scope and efficiency with engineered Cas9-pmCDA1 fusions and the modified sgRNA in rice., 2019, 10: 379.
[90] Veillet F, Perrot L, Chauvin L, Kermarrec MP, Guyon-Debast A, Chauvin JE, Nogué F, Mazier M. Transgene-free genome editing in tomato and potato plants using-mediated delivery of a CRISPR/Cas9 cytidine base editor., 2019, 20(2): E402.
[91] Bastet A, Zafirov D, Giovinazzo N, Guyon-Debast A, Nogué F, Robaglia C, Gallois JL. Mimicking natural polymorphism inby CRISPR-Cas9 base editing is associated with resistance to potyviruses., 2019, 17(9): 1736–1750.
[92] Endo M, Mikami M, Endo A, Kaya H, Itoh T, Nishimasu H, Nureki O, Toki S. Genome editing in plants by engineered CRISPR-Cas9 recognizing NG PAM., 2019, 5(1): 14–17.
[93] Banno S, Nishida K, Arazoe T, Mitsunobu H, Kondo A. Deaminase-mediated multiplex genome editing in., 2018, 3(4): 423–429.
[94] Zheng K, Wang Y, Li N, Jiang FF, Wu CX, Liu F, Chen HC, Liu ZF. Highly efficient base editing in bacteria using a Cas9-cytidine deaminase fusion., 2018, 1: 32.
[95] Gu T, Zhao S, Pi Y, Chen W, Chen C, Liu Q, Li M, Han D, Ji Q. Highly efficient base editing inusing an engineered CRISPR RNA-guided cytidine deaminase., 2018, 9(12): 3248–3253.
[96] Chen W, Zhang Y, Zhang Y, Pi Y, Gu T, Song L, Wang Y, Ji Q. CRISPR/Cas9-based genome editing inand cytidine deaminase-mediated base editing inspecies., 2018, 6: 222–231.
[97] Wang Y, Wang S, Chen W, Song L, Zhang Y, Shen Z, Yu F, Li M, Ji Q. CRISPR-Cas9 and CRISPR-assisted cytidine deaminase enable precise and efficient genome editing in., 2018, 84(23): e01834–18.
[98] Kuscu C, Parlak M, Tufan T, Yang J, Szlachta K, Wei X, Mammadov R, Adli M. CRISPR-STOP: gene silencing through base-editing-induced nonsense mutations., 2017, 14(7): 710–712.
[99] Billon P, Bryant EE, Joseph SA, Nambiar TS, Hayward SB, Rothstein R, Ciccia A. CRISPR-mediated base editing enables efficient disruption of eukaryotic genes through induction of STOP codons., 2017, 67(6): 1068–1079.e4.
[100] Jia K, Lu Z, Zhou F, Xiong Z, Zhang R, Liu Z, Ma Y, He L, Li C, Zhu Z, Pan D, Lian Z. Multiple sgRNAs facilitate base editing-mediated i-stop to induce complete and precise gene disruption., 2019, doi: 10.1007/s13238-019-0611-6.
[101] Zhang S, Samocha KE, Rivas MA, Karczewski KJ, Daly E, Schmandt B, Neale BM, MacArthur DG, Daly MJ. Base-specific mutational intolerance near splice sites clarifies the role of nonessential splice nucleotides., 2018, 28(7): 968–974.
[102] Gapinske M, Luu A, Winter J, Woods WS, Kostan KA, Shiva N, Song JS, Perez-Pinera P. CRISPR-SKIP: programmable gene splicing with single base editors., 2018, 19(1): 107.
[103] Yuan J, Ma Y, Huang T, Chen Y, Peng Y, Li B, Li J, Zhang Y, Song B, Sun X, Ding Q, Song Y, Chang X. Genetic modulation of RNA splicing with a CRISPR- guided cytidine deaminase., 2018, 72(2): 380–394.e7.
[104] Xue C, Zhang H, Lin Q, Fan R, Gao C. Manipulating mRNA splicing by base editing in plants., 2018, 61(11): 1293–1300.
[105] Li Z, Xiong X, Wang F, Liang J, Li JF. Gene disruption through base editing-induced messenger mRNA missplicing in plants., 2018, 222(2): 1139–1148.
[106] Li Q, Li Y, Yang S, Huang S, Yan M, Ding Y, Tang W, Lou X, Yin Q, Sun Z, Lu L, Shi H, Wang H, Chen Y, Li J. CRISPR-Cas9-mediated base-editing screening in mice identifies DND1 amino acids that are critical for primordial germ cell development., 2018, 20(11): 1315–1325.
[107] Gaudelli NM, Komor AC, Rees HA, Packer MS, Badran AH, Bryson DI, Liu DR. Programmable base editing of A•T to G•C in genomic DNA without DNA cleavage., 2017, 551(7681): 464–471.
[108] Li C, Zong Y, Wang Y, Jin S, Zhang D, Song Q, Zhang R, Gao C. Expanded base editing in rice and wheat using a Cas9-adenosine deaminase fusion., 2018, 19(1): 59.
[109] Chatterjee P, Jakimo N, Jacobson JM. Minimal PAM specificity of a highly similar SpCas9 ortholog., 2018, 4(10): eaau0766.
[110] Li G, Liu X, Huang S, Zeng Y, Yang G, Lu Z, Zhang Y, Ma X, Wang L, Huang X, Liu J. Efficient generation of pathogenic A-to-G mutations in human tripronuclear embryos via ABE-mediated base editing., 2019, 17: 289–296.
[111] Ryu SM, Koo T, Kim K, Lim K, Baek G, Kim ST, Kim HS, Kim DE, Lee H, Chung E, Kim JS. Adenine base editing in mouse embryos and an adult mouse model of Duchenne muscular dystrophy., 2018, 36(6): 536–539.
[112] Huang S, Liao Z, Li X, Liu Z, Li G, Li J, Lu Z, Zhang Y, Li X, Ma X, Sun Q, Huang X. Developing ABEmax-NG with precise targeting and expanded editing scope to model pathogenic splice site mutations., 2019, 15: 640–648.
[113] Liang P, Sun H, Zhang X, Xie X, Zhang J, Bai Y, Ouyang X, Zhi S, Xiong Y, Ma W, Liu D, Huang J, Songyang Z. Effective and precise adenine base editing in mouse zygotes., 2018, 9(9): 808–813.
[114] Yang L, Zhang X, Wang L, Yin S, Zhu B, Xie L, Duan Q, Hu H, Zheng R, Wei Y, Peng L, Han H, Zhang J, Qiu W, Geng H, Siwko S, Zhang X, Liu M, Li D. Increasing targeting scope of adenosine base editors in mouse and rat embryos through fusion of TadA deaminase with Cas9 variants., 2018, 9(9): 814–819.
[115] Ma Y, Yu L, Zhang X, Xin C, Huang S, Bai L, Chen W, Gao R, Li J, Pan S, Qi X, Huang X, Zhang L. Highly efficient and precise base editing by engineered dCas9-guide tRNA adenosine deaminase in rats., 2018, 4: 39.
[116] Qin W, Lu X, Liu Y, Bai H, Li S, Lin S. Precise A•T to G•C base editing in the zebrafish genome., 2018, 16(1): 139.
[117] Hua K, Tao X, Yuan F, Wang D, Zhu JK. Precise A•T to G•C base editing in the rice genome., 2018, 11(4): 627–630.
[118] Yan F, Kuang Y, Ren B, Wang J, Zhang D, Lin H, Yang B, Zhou X, Zhou H. Highly efficient A•T to G•C base editing by Cas9n-guided tRNA adenosine deaminase in rice., 2018, 11(4): 631–634.
[119] Kang BC, Yun JY, Kim ST, Shin Y, Ryu J, Choi M, Woo JW, Kim JS. Precision genome engineering through adenine base editing in plants., 2018, 4(7): 427–431.
[120] Kim D, Kim DE, Lee G, Cho SI, Kim JS. Genome-wide target specificity of CRISPR RNA-guided adenine base editors., 2019, 37(4): 430–435.
[121] Kim D, Lim K, Kim ST, Yoon SH, Kim K, Ryu SM, Kim JS. Genome-wide target specificities of CRISPR RNA-guided programmable deaminases., 2017, 35(5): 475–480.
[122] Kim D, Luk K, Wolfe SA, Kim JS. Evaluating and enhancing target specificity of gene-editing nucleases and deaminases., 2019, 88: 191–220.
[123] Rees HA, Komor AC, Yeh WH, Caetano-Lopes J, Warman M, Edge ASB, Liu DR. Improving the DNA specificity and applicability of base editing through protein engineering and protein delivery., 2017, 8: 15790.
[124] Liang P, Xie X, Zhi S, Sun H, Zhang X, Chen Y, Chen Y, Xiong Y, Ma W, Liu D, Huang J, Songyang Z. Genome- wide profiling of adenine base editor specificity by EndoV-seq., 2019, 10(1): 67.
[125] Lee HK, Willi M, Miller SM, Kim S, Liu C, Liu DR, Hennighausen L. Targeting fidelity of adenine and cytosine base editors in mouse embryos., 2018, 9(1): 4804.
[126] Zuo E, Sun Y, Wei W, Yuan T, Ying W, Sun H, Yuan L, Steinmetz LM, Li Y, Yang H. Cytosine base editor generates substantial off-target single-nucleotide variants in mouse embryos., 2019, 364(6437): 289–292.
[127] Jin S, Zong Y, Gao Q, Zhu Z, Wang Y, Qin P, Liang C, Wang D, Qiu JL, Zhang F, Gao C. Cytosine, but not adenine, base editors induce genome-wide off-target mutations in rice., 2019, 364(6437): 292–295.
[128] Petersen-Mahrt SK, Harris RS, Neuberger MS. AID mutatessuggesting a DNA deamination mechanism for antibody diversification., 2002, 418(6893): 99–103.
[129] Rogozin IB, Iyer LM, Liang L, Glazko GV, Liston VG, Pavlov YI, Aravind L, Pancer Z. Evolution and diversification of lamprey antigen receptors: evidence for involvement of an AID-APOBEC family cytosine deaminase., 2007, 8(6): 647–656.
[130] Burns MB, Lackey L, Carpenter MA, Rathore A, Land AM, Leonard B, Refsland EW, Kotandeniya D, Tretyakova N, Nikas JB, Yee D, Temiz NA, Donohue DE, McDougle RM, Brown WL, Law EK, Harris RS. APOBEC3B is an enzymatic source of mutation in breast cancer., 2013, 494(7437): 366–370.
[131] Grünewald J, Zhou R, Garcia SP, Iyer S, Lareau CA, Aryee MJ, Joung JK. Transcriptome-wide off-target RNA editing induced by CRISPR-guided DNA base editors., 2019, 569(7756): 433–437.
[132] Rees HA, Wilson C, Doman JL, Liu DR. Analysis and minimization of cellular RNA editing by DNA adenine base editors., 2019, 5(5): eaax5717.
[133] Zhou C, Sun Y, Yan R, Liu Y, Zuo E, Gu C, Han L, Wei Y, Hu X, Zeng R, Li Y, Zhou H, Guo F, Yang H. Off-target RNA mutation induced by DNA base editing and its elimination by mutagenesis., 2019, 571(7764): 275–278.
[134] Gajula KS. Designing an elusive C•G→G•C CRISPR base editor., 2018, 44(2): 91–94.
Progress on base editing systems
Yuan Zong, Caixia Gao
Base editing is a newly developed precise genome editing technique based on the CRISPR/Cas system. According to different base modification enzymes, the current base editing systems can be divided into cytosine base editors (CBE) and adenine base editors (ABE). They use cytosine deaminases or artificially evolved adenine deaminases to perform single-base editing, and achieve C to T (G to A) or A to G (T to C) substitutions, respectively. Due to high efficiency, independence of DNA double-strand breaks, and no need for donor DNA, base editing systems have been successfully applied in diverse species including animals, plants and other organisms since the first report in 2016. Therefore, base editing systems will have a high prospect of providing important support for gene therapy and crop genetic improvement in the future.In this review, we describe the development and current applications of base editing systems for basic research and biotechnology, highlight the challenges, and discuss the directions for future research in this important field. The information presented may facilitate interested researchers to grasp the principles of base editing, to use relevant base editing tools in their own studies, or to innovate new versions of base editing in the future.
CRISPR/Cas; base editing;cytosine base editor (CBE); adenine base editor (ABE)
2019-07-19;
2019-08-07
国家自然科学基金项目(编号:31788103)资助[Supported by the National Natural Science Foundation of China (No.31788103)]
宗媛,博士,研究方向:遗传学。E-mail: zongyuan@genetics.ac.cn
高彩霞,博士,研究员,博士生导师,研究方向:植物基因组编辑。E-mail: cxgao@genetics.ac.cn
10.16288/j.yczz.19-205
2019/8/21 10:05:03
URI: http://kns.cnki.net/kcms/detail/11.1913.R.20190821.1004.004.html
(责任编委: 张博)
