重新设计与快速驯化创造新型作物
2019-09-24杨新萍于媛许操
杨新萍,于媛,许操
重新设计与快速驯化创造新型作物
杨新萍1,3,于媛1,2,3,许操1,3
1. 中国科学院遗传与发育生物学研究所,植物基因组学国家重点实验室,种子创新研究院,北京 100101 2. 中国科学院大学,北京 100049 3. 中国科学院–英国约翰英纳斯中心植物和微生物科学联合研究中心,北京 100101
作物驯化和遗传改良对于产量性状的过分追求常导致其抗逆性和遗传多样性的降低,在全球气候变化加剧和自然灾害频发的大背景下,该效应使得世界粮食稳产和食物安全面临威胁,亟需创新育种策略。作物重新设计与快速驯化是指选用耐逆、品质营养等性状或其他目标性状优异的野生或者半野生植物,综合运用基因组学、基因编辑和合成生物学等方法,对其农艺性状进行重新设计,在保持其原有优异性状的前提下,快速驯化获得新型作物的全新育种策略。本文回顾了作物驯化的发展历程及其对农业发展和人类文明的贡献,着重阐述了育种策略创新的紧迫性,并对作物重新设计与快速驯化创造新型作物的可行性、最新进展和发展前景进行探讨和展望。
作物重新设计;快速驯化;野生资源;基因编辑
农作物的栽培与驯化是农业文明起源的重要标志。《周书》记载:“神农之时,天雨粟,神农遂耕而种之。作陶冶斤斧,为耒耜鉏蓐,以垦草莽。然后五谷兴助,百果藏食”。在漫长的驯化选择历程中,作物逐渐失去了其野生祖先种的遗传和生理特征,最终形成高产优质的现代栽培种。驯化是一个复杂的进化历程,是一种生物的繁殖逐渐受到另一种生物的利用与掌控的过程。驯化与人类生存与社会发展息息相关,通过对野生动植物进行有目的的改变,人类可以稳定地获取食物以果腹维生,制作衣服以御寒蔽体,制造饲料以饲养家禽家畜[1]。驯化大约开始于13 000~10 000年前的新石器时代,是人类文明发展史上的里程碑事件。自此,人类逐渐由采集狩猎为主的生活方式,转变为聚群而居、栽培养殖的农耕生活[2]。安定的农耕生活加之充足的食物,使得人口密度大大提高进而催生了阶级结构,大量的劳动力从农业生产活动中解脱出来,从事其他工作,由此产生社会分工及人类社会的雏形。因此,驯化是人类智慧的体现,也是农业发展的源动力,在一定程度上促生了农业文明。
将野生植物驯化成农作物是人类驯化活动的主要目标之一。目前农业生产中种植的主要农作物,如水稻(L.)、玉米(L.)、小麦()、棉花()、高粱()和番茄()等,都是由早期人类从自然界的野生植物中选择和驯化而来[2,3]。目前约有160个科的2500种植物被人类驯化,其中250种植物已被完全驯化[4]。人们发现,不同作物在从野生种到栽培种的驯化过程中,尽管作物本身形态和起源地千差万别,但它们的生长势、生物量、繁殖能力、产量和品质等重要农艺性状在被驯化时往往具有很多相似之处或具有类似的选择趋势,这种重要农艺形状控制位点/基因在驯化时受到平行选择的现象称为“驯化综合征”(domestication syndrome)[5,6]。当完成最初的驯化后,作物被人为地扩散到不同的地域。部分农艺性状受到持续一致的选择,而有些性状却因当地的环境条件和文化习惯不同,受到进一步的适应性选择,逐渐形成不同的地方品种。因此,作物品种的多样性往往与地域自然环境和人文环境密切相关,人类对作物的需求也伴随着社会发展和文明进步而持续更新。
1 社会发展和人类进步对作物提出更高要求
农业是人类文明的基石,农作物的更新换代往往与人类文明发展和社会生产方式的变革同步。20世纪40年代起,随着农业机械化和化学合成技术等工业文明成果的日臻成熟和推广应用,作物品种也随着第一次绿色革命的兴起而实现更新换代,这使得以大规模单一品种的机械化种植,化肥及杀虫剂、除草剂等在农业生产中大量施用为特征的农业模式替代了传统的小范围农耕模式。这一变化一方面大大提高了全球粮食产量,在一定程度上缓解了全球饥饿问题;另一方面深刻地改变了传统的农业生产模式。高产作物品种对现代农业设施及现代农业管理方式的高度依赖,使得农作物能够在拥有现代化农业设施的、贫瘠的地区进行种植,这大大拓宽了农业生产范围;然而,高产品种的单一化、同质化种植使得不同地区的区域性品种逐渐消失,削弱了农业生态系统和物种遗传的多样性,降低了对病虫害的耐受力。如印度在绿色革命前约有3000个水稻品种,而现在广泛种植的则仅约10个[7]。更为严峻的是,集中化的种植方式加剧了病虫害的传播,而杀虫剂等农药的过度施用则又提高了病虫害的耐药性,同时也对土壤、水系造成了极大的污染[8]。
随着现代科学技术的迅猛发展,人类社会逐步进入以信息革命、人工智能为特征的智能文明时代。技术的革新与进步,对农作物自身的特质也提出了新的要求,例如,当前作物是否能适应现代社会及未来智能社会的数字化、智能化、信息化的生产方式?是否具备更高的环境适应性以应对全球气候变化?更为重要的是,随着物质文明的极大丰富和生活水平大幅度提高,人们开始对食物安全、食物的外观、口味及营养多样性提出更多维度和个性化要求。但是,越来越多的研究结果表明,现代农业中广泛种植的栽培种所含的微量营养物质较低,有可能是因长期驯化及育种过程中单方面追求高产导致了相关基因的丢失所造成的[9],而有限的种质资源和遗传多样性进一步限制了对现有品种的遗传改良。同时,随着人们的食品安全意识日渐提高,人们对天然、绿色、安全食品的需求也与日俱增,这也对高度依赖化肥农药的现代化农业生产模式及该模式下的农作物品种提出了更高要求。大数据和人工智能时代的来临对农业生产方式及作物品种提出了新的要求,将引导农业生产从机械化向智能化转变。当前人工智能在农业生产中的应用已经取得了初步成效,它通过贯穿于农业生产的各个阶段的智能控制对农作物生长进行实时监测和管理,解放了劳动力,降低了农业劳动强度,提高了农业生产效率;而通过对农业生产过程中的各项信息指标进行收集、分析与处理,可以建立农业生产大数据信息库,对农业生产提供更深层次的反馈指导调节[10]。因此,农业生产方式的变革亟需与之匹配的新型作物,培育和创造能够兼容智能农业生产模式的、满足人们生活多样化需求的、环境友好型的“智能作物”,这已经成为未来农业发展的必然趋势。
2 全球气候变化呼唤新型作物
自18世纪中叶工业革命以来,人类活动特别是发达国家工业化进程中的经济活动,如化石燃料燃烧、森林大面积砍伐等,致使全球气候发生急剧变化。全球气候变化问题已然成为21世纪人类面临的最大的环境问题,也是人类面临的最复杂的挑战之一[11]。据美国气象学会发布的2017年气候变化报告称,自1901年起,全球气温平均每世纪升高0.7℃,从1975年起开始,该数据升高至1.5~1.8℃,持续的全球变暖趋势将导致气候带迁移,新病虫害出现,自然灾害频发[12],这些均对现有的作物和农业生产产生了严重影响,导致粮食减产,威胁农业生产和人类食物安全。
在全球气候变化的背景下,我国农业气象灾害、水资源短缺、农业病虫害的发生程度都呈加剧趋势。气候变化造成的一系列环境问题严重威胁着我国的农业生产及粮食安全。据统计,严重干旱造成粮食平均减产15%~25%,大面积洪涝减产要在30%~50%,低温冷害造成减产20%左右,其中水稻可达45%。据预测,到2030年,我国种植业产量在总体上因全球气候变暖将会减少5%~10%,其中小麦、水稻和玉米三大作物均以减产为主(中国政府网,http:// www.gov.cn/govweb/jrzg/2006-05/18/content_284434.htm)。中国农业生产总值因病虫害每年损失可达20%~25%,而农业病虫害的发生、发展以及流行都与气候条件有着密切的关系,甚至部分病虫害的发生主要取决于气象条件。另外,气候变化还可能引起新病虫害种类的出现,或导致某些次要病虫害上升为主要病虫害,使得农业生产因病虫害造成的损失变得更为严重[13]。受全球气候变化的影响,在保持人民消费水平持续增长的前提下,中国未来粮食生产有可能不能满足粮食需求增长,存在7%~8%的粮食缺口[14]。全球气候变化对现有作物的耐逆能力提出了严峻挑战,对主要粮食和蔬菜作物高产稳产造成了严重威胁。因此,培育具有更好的耐逆性和稳健性、可以智能响应环境变化的新型作物是人类应对气候变化的必然选择。
3 培育新型作物亟需创新育种策略
在长期的驯化选择过程中,只有与“驯化综合征”相关的少数基因受到强烈选择被保存下来,那些基因组范围内没有受到选择的基因位点则由于“选择牵连”(hitchhiking effect)或“驯化瓶颈”(domestication bottleneck)等效应严重丢失,致使基因组水平的遗传多样性显著降低[15,16]。野生种则主要依赖于群体内部的多样性和个体适应性在自然环境中进行生存繁衍。与其野生种祖先相比,栽培作物的群体遗传结构多样性降低。同时,因为人类对作物特定性状的过分或者片面追求,使得大量被“忽视的性状”如风味营养性状在驯化选择中逐渐丢失。据统计,20世纪人类丢失了75%的作物多样性[11];预测到2055年,现有的主要栽培作物品种中将有20%的野生种质因全球气候变化而消失[11,16]。作物遗传多样性的下降,降低了作物基因资源库的丰度,使得可以用于遗传改良的基因池越来越小,限制了优异性状基因资源的发掘和利用[9]。野生种因具有丰富的基因资源为抗逆育种做出了重要贡献。以耐逆育种为例:传统耐逆育种通过挖掘自然遗传变异,寻找耐逆的数量性状位点,通过大规模杂交将野生种的优良性状通过基因渐渗的方式转移到栽培种中,运用分子标记辅助选择方式筛选出高抗、高产的新品种[17,18]。但这种方法耗时长,一般需要5~10代才能得到理想株系,而且由于连锁效应,非目标基因也会转移至回交后代中造成连锁累赘,损害了作物的品质或者产量性状,常使得耐逆育种“顾此失彼”,难以兼顾产量或品质与抗逆性。因此,尽管近年来人们对于植物响应生物与非生物胁迫的分子机制的认识不断加深,但耐逆育种工作一直存在瓶颈,进展较为缓慢[19~21]。
转基因育种通过基因工程手段,将目的基因片段导入生物活细胞DNA中,培养获得新的种质资源。这种技术可以实现跨物种的基因交流,对目标性状改良的针对性强,极大地提高了育种效率,同时还不涉及到生殖隔离、连锁累赘等问题[22]。近年来通过转基因的方式过表达或者沉默一些耐逆相关基因可以一定程度上提高不同作物的耐逆性[23],但植物响应逆境是一个非常复杂的生物学过程,它不仅涉及细胞渗透压、离子胁迫以及由这些胁迫引发的次生伤害如氧化损伤等,还牵涉植物发育与胁迫响应的整体协调,相当多的耐逆性状是多基因、多维时空协作调控的结果,很难通过转入单基因实现作物耐逆性的显著提升[19,24]。例如,番茄耐盐性是由多基因控制的,耐盐基因已被定位在至少7条染色体上,如第1、2、3、7、8、9和12号染色体[25,26],这在很大程度上限制了番茄耐盐性状的转基因改良。目前已知耐逆基因及其多维调控网络,有的需要过量表达,有的则需要下调表达才能发挥功效,而同时转入多基因以及多种表达调控模式对于载体构建、组织培养、基因转化等转基因技术是一个巨大的挑战,转基因后代的遗传稳定性也存在不可预知性。
综上所述,传统育种和现有的转基因育种技术为作物改良和品种创制做出了重要贡献,但仍难以满足人类社会发展和全球气候变化对新型作物的迫切需求,亟需创新育种策略以培育智能响应环境变化的新型作物。
4 重新设计与快速驯化是全新育种策略
野生植物相较于现有栽培作物普遍具有较强的耐逆性和环境适应力,而且它们往往因旺盛的次生代谢而产生更多的风味和营养物质。基因组学、转录组学、代谢组学等多重组学技术的发展使得人们对作物驯化的基因组和遗传学基础的认识越来越清晰,而基因编辑和合成生物学等技术的迅猛发展使得精准和快速的基因操作以及代谢流重编程成为可能。根据人类生产方式和生活方式的需求,以优异的野生种质为底盘材料,对作物的目标性状进行重新设计,在保持野生种天然优异性状的前提下,通过基因编辑、合成生物学等方法导入目标性状,实现野生植物的快速驯化是创造新型作物的全新策略 (图1)。作物的重新设计与快速驯化应具备优异的野生种质资源、明确的驯化基因、高效的基因操控技术等3个必要条件。
世界范围内存在大量营养风味独特、能够在极度贫瘠、恶劣气候和高度盐碱化的土地中生长良好的野生种质,它们被世界各国的种子资源库整理收集,为重新设计与快速驯化提供了丰富的种质资源[27,28]。大量的驯化基因相关研究表明,野生植物的性状丢失和栽培作物性状的获得往往是由于基因突变所造成的基因功能的丧失、歧化或是新功能获得的结果。随着人们对驯化机理研究的不断深入,多种作物驯化的遗传和分子机理被解析,一些重要的驯化基因不断被鉴定出来[29,30]。驯化基因一般具备3个特点:(1)与驯化性状之间存在明确的相关性;(2)在驯化过程中受到强烈的正向选择;(3)在由一个群体演化而成的不同种群中几乎完全丧失了多样性,具有单一性[4]。如大麦种子产量相关基因[31],玉米籽粒裸露相关基因[32],水稻籽粒芒结构相关基因[33],高粱、水稻、玉米中籽粒脱落相关基因[34],以及影响番茄生殖方式基因[35]等。与驯化密切相关的基因大多为转录因子和激酶调控过程中的关键基因,这类基因在植物的生长发育、结实和成熟过程中都起着重要的调控作用[6,36,37]。作物重要的产量与品质性状大多由少数几个基因控制,这使得利用基因编辑技术对野生植物进行重新设计与驯化成为可能。而驯化基因的鉴定为快速导入产量和品质性状、保持耐逆境性状、实现野生植物的重新设计与快速驯化提供了丰富的基因资源。
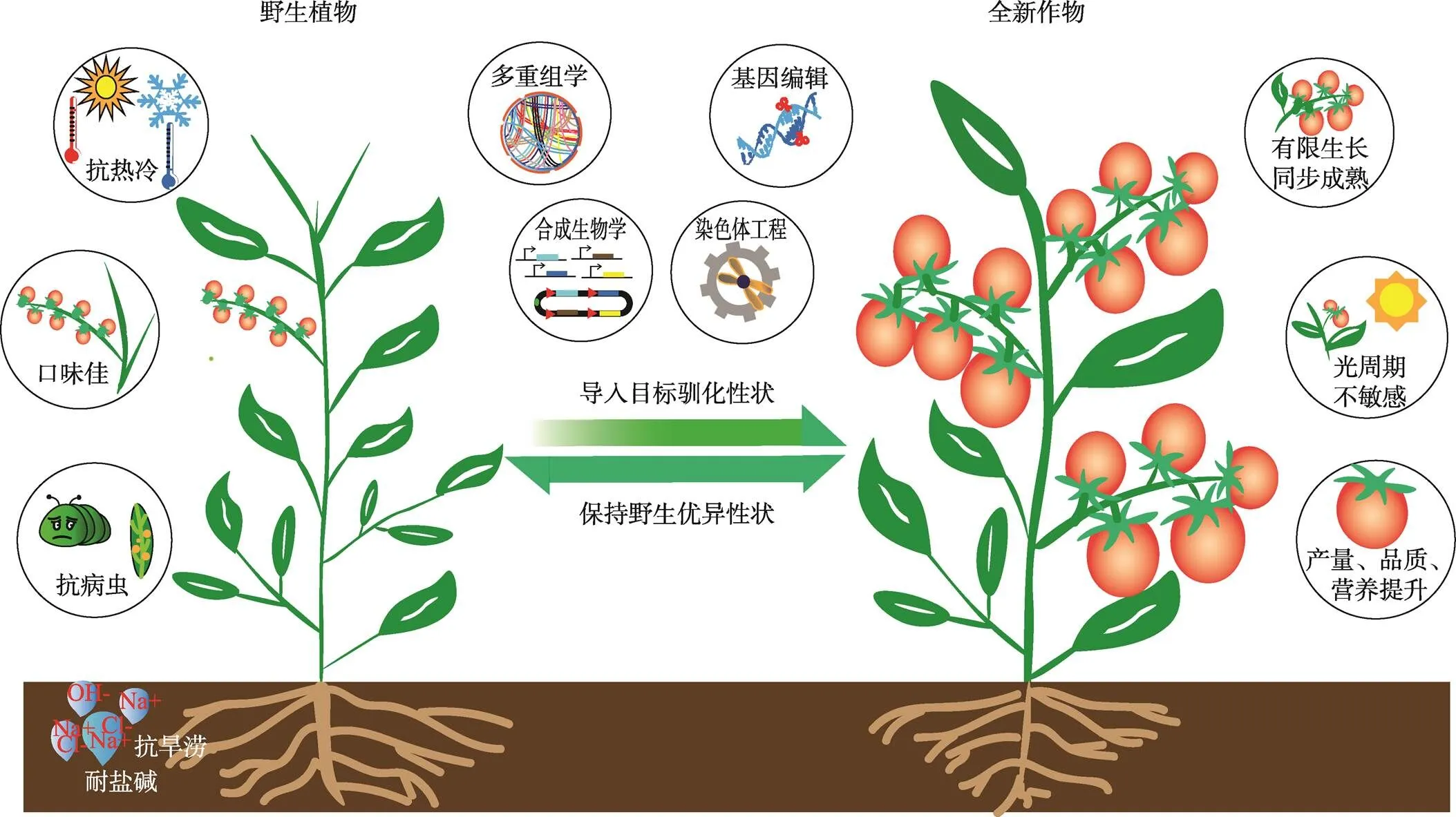
图1 野生植物的快速驯化与新型作物的重新设计模式图
作物驯化的基因组学和遗传学基础不断被解析的同时,基因编辑技术也在不断发展。基因编辑技术是以特异性改变遗传物质靶向序列为目标的技术,可以对靶基因基因及其调控选件进行定点突变、插入或敲除[38,39]。近年来,锌指核酸酶(zinc finger nuclease, ZFN)、类转录激活因子效应核酸酶(transcription activator-like effector nuclease, TALEN)、规律成簇的间隔短回文重复(clustered regularly interspaced short palindromic repeats, CRISPR)和单碱基编辑(base editing, BE)技术相继出现,从第一代DNA核酸酶编辑系统ZFNs、第二代TALENs到第三代CRISPR/Cas9系统,基因编辑效率不断提高,成本逐渐降低,应用范围不断扩大。ZFNs、TALENs和CRISPR/Cas9等3种基因编辑技术都是在基因组靶标位点引起DNA双链断裂(double-strand breaks, DSBs),进而激活细胞内部修复机制的基础上建立的。细胞内DNA双链断裂的修复机制包括易引起随机插入、缺失的异源末端连接(non-homologous end joining, NHEJ)和需要同源模板存在才可以激活的同源重组修复(homology directed repair, HDR)。值得注意的是,CRISPR/Cas9编辑系统与ZFNs和TALENs编辑系统最大的不同之处在于,CRISPR/Cas9系统可以通过较小的sgRNA靶向特定DNA序列,而不是通过蛋白酶本身与DNA序列结合。这一特点使得CRISPR/Cas9编辑系统可以同时实现多个基因多个靶位点的编辑。目前在烟草、拟南芥、水稻和番茄等作物中同时实现多基因编辑的CRISPR/Cas9系统已研究较为成熟[40,41]。2016年,单碱基编辑技术的开发实现了在不引起DNA双链断裂和无需同源模板情况下的单个碱基(A-G、T-C)转换,有效地规避了基于双链DNA断裂后NHEJ和HDR修复的基因编辑技术的不足[42,43]。除了对内源基因进行定向敲除,CRISPR/Cas9还可以调控(激活/抑制)靶基因的表达。失去核酸酶活性的dCas9(dead Cas9)可以作为DNA 转录调控因子,通过融合具有转录调控功能的转录激活因子或抑制因子来调控基因表达水平,从而形成一个研究基因表达调控的有效工具[44]。基因编辑技术的进步为实现作物的重新设计和野生植物的快速驯化提供了有力的技术保障。
5 作物重新设计与快速驯化进展
2018年10月1日,中国、巴西、美国和德国等国科学家分别在、等杂志同时发布了世界首例快速驯化作物及相关研究成果,引起国际社会和科技界的广泛关注[45~47]。其中杂志以“Super-tomato shows what plant scientists can do”为题,杂志以“CRISPR can make old tomatoes, new tomatoes”为题,对番茄重新设计与快速驯化研究进行了评价和推介,认为这些研究首次通过基因编辑实现野生植物的快速驯化,为精准设计和创造耐盐碱作物提供了全新策略[48]。植物综述杂志也刊文“A CRISPR way for fast-forward crop domestication”,指出野生植物的重新设计与快速驯化是精准育种的革命性进展[49]。
5.1 快速驯化野生植物创造新型番茄
番茄有16个野生或近缘野生种,其中野生种12个,它们在耐逆境、抗病虫、果实品质、风味和营养等方面各具特色[27,28]。为了创新耐逆和营养育种策略,中国科学院遗传与发育生物学研究所许操研究组和高彩霞研究组合作,选用天然耐盐碱和抗细菌疮痂病的野生醋栗番茄()为基础材料,运用基因编辑技术精准靶向多个产量和品质性状控制基因的编码区及调控区,在不牺牲其对盐碱和疮痂病天然抗性的前提下,将产量和品质性状精准地导入了野生番茄,消除了野生番茄开花的光周期敏感性,突破了栽种的地理范围限制,实现了野生植物驯化的第一步。同时将野生醋栗番茄开花晚、坐果稀的无限生长型(indeterminate)的株型变成了“双有限”生长型(double determinate)的紧凑株型,提高了坐果率、果实成熟的同步性和收获指数。而对小肽基因及其信号途径下游基因的顺式调控元件和上游开放阅读框的编辑使野生番茄果实变大,维生素C含量升高。盐处理和疮痂病菌接种实验表明,上述重要农艺性状的精准导入并没有影响野生番茄的天然抗性。上述研究直接选用天然耐逆性优异的野生番茄做底盘材料,避开了复杂的耐逆性状,首次通过重新设计与快速驯化创制了耐盐碱、高抗疮痂病、高营养品质的新型番茄[45]。与此同时,巴西、美国、德国等国家的研究人员合作,运用基因编辑技术对野生醋栗番茄株型、果实形状、大小和番茄红素含量相关基因进行编辑,可以将果实大小提升3倍,使番茄红素含量较比传统樱桃番茄提高5倍以上,为重新设计和快速驯化获得外观佳和营养高的番茄提供了范例[46]。
5.2 快速驯化小宗作物创造新型作物
小宗作物(orphan crop)是指分布在较为局限的地区,并被当地人食用的作物。在世界农业现代化迅猛发展,全球化快速推进的背景下,随着小麦、玉米、水稻和番茄等大宗作物的广泛种植,小宗作物生存空间逐渐缩小甚至消失。然而,这些小宗作物往往具有优良的品质或营养性状,并可以在土壤贫瘠、高海拔、高寒旱的恶劣环境中生长。随着全球人口增长和环境恶化,保障食物供应一直是很多经济不发达地区的首要问题,增加人类食谱的丰富性也是提高生活品质的重要目标,而这些小宗作物能在与其原产地相似的环境恶劣地区依然保持优质高产地生长,可见其具有十分广阔的应用潜力。对于我国而言,小宗作物往往极具地域特色和特殊食用或者药用价值并因此而衍生出可观的经济价值,对推动乡村振兴和精准扶贫具有重要意义。很多小宗作物在生物学上与其他大宗作物亲缘关系较远,它们可能蕴藏着其他大宗作物不具有的特殊营养物质或者药用成分,未来可能为人类提供更为丰富的营养和药用物质来源。与大宗粮食和果蔬作物相比,小宗作物相对来说属于驯化的“半成品”,借助比较基因组学和基因编辑技术实现小宗作物的快速驯化,将丰富现有栽培作物种类,改善人类的食谱,对保障人类食物安全也具有重要意义。
美国冷泉港实验室的Lippman教授团队使用基因编辑对小宗作物灯笼果()进行了快速驯化。灯笼果具有优异的逆境耐受能力,丰富的营养成分和特别的风味物质,它与番茄、土豆、辣椒、茄子等同属茄科。通过比较基因组学分析,研究人员使用CRISPR/Cas9基因编辑技术对番茄、和基因的同源基因、和的编码区进行编辑,对后代进行基因型及表型鉴定发现,和基因不同的编辑形式使得灯笼果花期提前、株型紧凑、产量提高,而编辑基因增加了灯笼果果实大小。通过上述基因的编辑,改良了灯笼果的食用品质和园艺性状,实现了灯笼果的快速驯化,为小宗作物的快速驯化提供了范例[47]。
5.3 快速驯化创新育种策略
四倍体马铃薯()作为马铃薯生产及研究中的主要对象,存在遗传背景复杂、基因组高度杂合、育种周期长以及繁殖系数低、储运成本高、易携带病虫害等缺陷。自然界中70%的马铃薯种质资源为二倍体,然而却存在自交不亲和的问题。中国农业科学院黄三文研究组利用CRISPR/Cas9基因组编辑技术对马铃薯控制自交不亲和的基因进行了定点突变,获得了自交亲和的二倍体马铃薯,并通过自交获得了不含有外源片段的马铃薯新材料,成功地用二倍体替代四倍体,用杂交种子替代薯块,拓展了自交亲和马铃薯资源,为马铃薯的快速驯化和遗传改良提供了新策略[50]。
6 结语与展望
番茄、灯笼果、马铃薯等作物重新设计与快速驯化的实现证实了这一全新育种策略的可行性和广阔前景。近期,华中农业大学严建兵教授和德国马普分子植物生理研究所Alisdair Fernie教授在刊文,重申了“再驯化”(re-domestication)和“重新驯化”(domestication)的概念,对作物重新设计与快速驯化的技术路线和育种策略进行了阐述[30]。作者认为,快速发展的现代生物技术和基因组学研究驱动的育种技术变革正在叩开“设计育种”的大门,而将大数据和精准育种与基因编辑技术相结合,则有望引发“第三次绿色革命”。因此,收集和发掘更多性状优异的野生种质资源,进一步加强作物基因组学、代谢组学等多重组学研究,鉴定更多驯化基因,将为作物重新设计和快速驯化提供更多底盘材料和靶基因;加强植物器官再生和基因转化方面的研究,提高基因编辑技术的靶向效 率,突破植物基因替换的技术瓶颈,优化合成生物学及代谢流重编程技术,将为作物快速驯化提供技术支撑。
[1] Hancock JF. Contributions of domesticated plant studies to our understanding of plant evolution., 2005, 96(6): 953–963.
[2] Diamond J. Evolution, consequences and future of plant and animal domestication., 2002, 418(6898): 700– 707.
[3] Crawley MJ, Brown SL, Hails RS, Kohn DD, Rees M. Transgenic crops in natural habitats., 2001, 409 (6821): 682–683.
[4] Meyer RS, Purugganan MD. Evolution of crop species: genetics of domestication and diversification., 2013, 14(12): 840–852.
[5] Gepts P. A comparison between crop domestication, classical plant breeding, and genetic engineering., 2002, 42(6): 1780–1790.
[6] Doebley JF, Gaut BS, Smith BD. The molecular genetics of crop domestication., 2006, 127(7): 1309–1321.
[7] Sumberg J, Keeney D, Dempsey B. Public agronomy: Norman Borlaug as ‘Brand Hero’ for the green revolution., 2012, 48(11): 1587–1600.
[8] Hardin LS. Bellagio 1969: the green revolution., 2008, 455(7212): 470–471.
[9] Smýkal P, Nelson M, Berger J, von Wettberg E. The impact of genetic changes during crop domestication., 2018, 8(7): 119.
[10] Liakos K, Busato P, Moshou D, Pearson S, Bochtis D. Machine learning in agriculture: a review., 2018, 18(8): 2674.
[11] Wheeler T, von Braun J. Climate change impacts on global food security., 2013, 341(6145), 508–513.
[12] Hartfield G, Blunden, J, Arndt DS. State of the climate in 2017., 2018, 99(8): Si–S310.
[13] Wang CY, Zhao JB, Li X. Research on agriculture adaptation strategy for warming and drying climate in North China., 2006, 29(5): 646–652.王长燕, 赵景波, 李小燕. 华北地区气候暖干化的农业适应性对策研究. 干旱区地理, 2006, 29(5): 646–652.
[14] Liu LT, Liu XJ, Lun F, Wu L, Lu CX, Guo JH, Qu TT, Liu G, Shen L, Cheng SK. Research on China’s food security under global climate change background., 2018, 33 (6): 927–939.刘立涛, 刘晓洁, 伦飞, 吴良, 鲁春霞, 郭金花, 曲婷婷, 刘刚, 沈镭, 成升魁.全球气候变化下的中国粮食安全问题研究. 自然资源学报, 2018, 33(6): 927–939.
[15] Tanksley SD, McCouch SR. Seed banks and molecular maps: unlocking genetic potential from the wild., 1997, 277(5329): 1063–1066.
[16] Massawe F, Mayes S, Cheng A. Crop diversity: an unexploited treasure trove for food security., 2016, 21(5): 365–368.
[17] Minhas PS, Rane J, Pasala RK. Abiotic stress management for resilient agriculture. Abiotic Stress Management for Resilient Agriculture, Springer, 2017.
[18] Yu SB, Xu WJ, Vijayakumar CHM, Ali J, Fu BY, Xu JL, Jiang Y. Z, Marghirang R, Domingo J, Aquino C, Virmani S S, Li ZK. Molecular diversity and multilocus organizationof the parental lines used in the international rice molecular breeding program., 2003, 108(1): 131– 140.
[19] Luo LJ. Breeding for water-saving and drought-resistance rice (WDR) in China., 2010, 61(13): 3509–3517.
[20] Vinocur B, Altman A. Recent advances in engineering plant tolerance to abiotic stress: achievements and limitations., 2005, 16(2): 123–132.
[21] Witcombe JR, Hollington PA, Howarth CJ, Reader S, Steele KA. Breeding for abiotic stresses for sustainable agriculture., 2008, 363(1492): 703–716.
[22] Sánchez-Martín J, Keller B. Contribution of recent technological advances to future resistance breeding., 2019, 132(3): 713–732.
[23] Shi H, Lee B, Wu SJ, Zhu JK. Overexpression of a plasma membrane Na+/H+ antiporter gene improves salt tolerance in., 2003, 21(1): 81–85.
[24] Wang Z, Yang C, Chen H, Wang P, Wang P, Song C, Zhang X, Wang D. Multi-gene co-expression can improve comprehensive resistance to multiple abiotic stresses inL., 2018, 274: 410–419.
[25] Villalta I, Bernet GP, Carbonell EA, Asins MJ. Comparative QTL analysis of salinity tolerance in terms of fruit yield using two Solanum populations of F7 lines., 2007, 114(6): 1001–1017.
[26] Li J, Liu L, Bai Y, Zhang P, Finkers R, Du Y, Visser RGF, van Heusden AW. Seedling salt tolerance in tomato., 2011, 178(3): 403–414.
[27] Rick C. M. Potential genetic resources in tomato species: clues from observations in native habitats., 1973, 255–269.
[28] Rick CM. Genetic variability in tomato species., 1983, 1(2): 81–87.
[29] Østerberg JT, Xiang W, Olsen LI, Edenbrandt AK, Vedel SE, Christiansen A, Landes X, Andersen MM, Pagh P, Sandøe P, Nielsen J, Christensen SB, Thorsen BJ, Kappel K, Gamborg C, Palmgren M. Accelerating the domestication of new crops: feasibility and approaches., 2017, 22(5): 373–384.
[30] Fernie AR, Yan J.domestication: an alternative route toward new crops for the future., 2019, 12(5): 615–631.
[31] Komatsuda T, Pourkheirandish M, He C, Azhaguvel P, Kanamori H, Perovic D, Stein N, Graner A, Wicker T, Tagiri A, Lundqvist U, Fujimura T, Matsuoka M, Matsumoto T, Yano M. Six-rowed barley originated from a mutation in a homeodomain-leucine zipper I-class homeobox gene., 2007, 104(4): 1424–1429.
[32] Wang H, Nussbaum-Wagler T, Li B, Zhao Q, Vigouroux Y, Faller M, Bomblies K, Lukens L, Doebley JF. The origin of the naked grains of maize., 2005, 436(7051): 714–719.
[33] Hua L, Wang DR, Tan L, Fu Y, Liu F, Xiao L, Zhu Z, Fu Q, Sun X, Gu P, Cai H, McCouch SR, Sun C., a domestication gene associated with long, barbed awns in wild rice., 2015, 27(7): 1875–1888.
[34] Lin Z, Li X, Shannon LM, Yeh CT, Wang ML, Bai G,Peng Z, Li J, Trick HN, Clemente TE, Doebley J, Schnable PS, Tuinstra MR, Tesso TT, White F, Yu J. Parallel domestication of thegenes in cereals., 2012, 44(6): 720–724.
[35] Chen KY, Cong B, Wing R, Vrebalov J, Tanksley SD. Changes in regulation of a transcription factor lead to autogamy in cultivated tomatoes., 2007, 318(5850): 643–645.
[36] Meyer RS, Purugganan MD. Evolution of crop species: genetics of domestication and diversification., 2013, 14(12): 840–852.
[37] Shang Y, Ma Y, Zhou Y, Zhang H, Duan L, Chen H, Zeng J, Zhou Q, Wang S, Gu W, Liu M, Ren J, Gu X, Zhang S, Wang Y, Yasukawa K, Bouwmeester HJ, Qi X, Zhang Z, Lucas WJ, Huang S. Plant science. Biosynthesis, regulation, and domestication of bitterness in cucumber., 2014, 346(6213): 1084–1088.
[38] Doudna JA, Charpentier E. Genome editing. The new frontier of genome engineering with CRISPR-Cas9., 2014, 346(6213): 1258096.
[39] Shalem O, Sanjana NE, Zhang F. High-throughput functional genomics using CRISPR–Cas9., 2015, 16(5): 299–311.
[40] Xing HL, Dong L, Wang ZP, Zhang HY, Han CY, Liu B, Wang XC, Chen QJ. A CRISPR/Cas9 toolkit for multiplex genome editing in plants., 2014, 14: 327.
[41] Lowder LG, Zhang D, Baltes NJ, Paul JW, Tang X, Zheng X, Voytas DF, Hsieh TF, Zhang Y, Qi Y. A CRISPR/Cas9 toolbox for multiplexed plant genome editing and transcriptional regulation., 2015, 169(2): 971–985.
[42] Gaudelli NM, Komor AC, Rees HA, Packer MS, Badran AH, Bryson DI, Liu DR. Programmable base editing of T to G C in genomic DNA without DNA cleavage., 2017, 551(7681): 464–471.
[43] Chen K, Wang Y, Zhang R, Zhang H, Gao C. CRISPR/Cas genome editing and precision plant breeding in agriculture., 2019, 70: 667–697.
[44] Piatek A, Ali Z, Baazim H, Li L, Abulfaraj A, Al-Shareef S, Aouida M, Mahfouz MM. RNA-guided transcriptional regulation in planta via synthetic dCas9-based transcription factors., 2015, 13(4): 578–589.
[45] Li T, Yang X, Yu Y, Si X, Zhai X, Zhang H, Dong W, Gao C, Xu C. Domestication of wild tomato is accelerated by genome editing., 2018, 36(12): 1160– 1163.
[46] Zsögön A, Čermák T, Naves ER, Notini MM, Edel KH, Weinl S, Freschi L, Voytas DF, Kudla J, Peres LEP.domestication of wild tomato using genome editing., 2018, 36(12): 1211–1216.
[47] Lemmon ZH, Reem NT, Dalrymple J, Soyk S, Swartwood KE, Rodriguez-Leal D, Van Eck J, Lippman ZB. Rapid improvement of domestication traits in an orphan crop by genome editing., 2018, 4(10): 766–770.
[48] Super-tomato shows what plant scientists can do., 2018, 562(7725): 8–8.
[49] Khan MZ, Zaidi SS, Amin I, Mansoor S. A CRISPR way for fast-forward crop domestication., 2019, 24(4): 293–296.
[50] Ye M, Peng Z, Tang D, Yang Z, Li D, Xu Y, Zhang C, Huang S. Generation of self-compatible diploid potato by knockout of., 2018, 4(9): 651–654.
domestication to create new crops
Xinping Yang1,3, Yuan Yu1,2,3, Cao Xu1,3
Crop improvement by domestication and traditional breeding often results in fitness penalties and loss of genetic diversity, which greatly threatens crop production and food security under the challenging global climate.domestication has been proposed as a novel strategy for crop breeding. By combining multi-omics, genome editing and synthetic biology approaches, domestication of wild or semi-wild plant species can be accelerated by rapidly introducing desirable traits without causing an associated drag on their inherent traits. In this review, we summarize the history of crop domestication, emphasize the urgency for breeding strategy innovation, and discuss recent progress ofcrop domestication.
crop design; fast domestication; wild species; genome editing
2019-05-28;
2019-07-30
中组部青年千人计划项目资助[Supported by the Thousand Younth Talents Program]
杨新萍,博士后,研究方向:基因编辑及作物定向驯化。E-mail: xinping.yang@genetics.ac.cn
于媛,博士研究生,研究方向:基因编辑及作物定向驯化。E-mail: yuyuan@genetics.ac.cn
杨新萍和于媛并列第一作者。
许操,研究员,博士生导师,研究方向:小肽信号与植物细胞通讯,基因编辑及作物定向驯化。E-mail: caoxu@genetics.ac.cn
10.16288/j.yczz.19-151
2019/8/21 10:04:59
URI: http://kns.cnki.net/kcms/detail/11.1913.R.20190821.1004.002.html
(责任编委: 严建兵)
