小麦A基因组测序与进化研究进展
2019-09-24史晓黎何伊琳凌宏清
史晓黎,何伊琳,2,凌宏清,2
小麦A基因组测序与进化研究进展
史晓黎1,何伊琳1,2,凌宏清1,2
1. 中国科学院遗传与发育生物学研究所,植物细胞与染色体工程国家重点实验室,北京 100101 2. 中国科学院大学生命科学学院,北京 101408
小麦是世界上广泛种植的主要粮食作物,养活了全世界35%以上的人口。获取高质量的基因组图谱对于推动小麦基础理论和遗传育种研究至关重要。然而,庞大而复杂的基因组一度使小麦基因组测序被认为是“不可能完成的任务”。随着高通量测序和组装技术的成熟,近年来多个小麦基因组序列图谱陆续发布,序列组装质量日臻完善。仅最近两年就公布了5个不同倍性的小麦参考基因组序列,包括两个二倍体祖先种乌拉尔图小麦(, AA)和粗山羊草(, DD)、野生和栽培四倍体二粒小麦(ssp., BBAA)和六倍体普通小麦(, BBAADD)。其中,作为多倍体小麦A亚基因组供体的乌拉尔图小麦基因组测序和分析是由中国科学院遗传与发育生物学研究所牵头完成。本文主要对小麦A基因组的结构解析和进化分析等方面的研究进展进行了综述,以期为相关领域的科研人员提供参考信息,促进小麦的基础和应用研究。
小麦;乌拉尔图小麦;小麦A基因组;基因组测序;染色体演化
小麦(L.)是世界上适应性最广、栽培面积最大的主要粮食作物,年种植面积达2.2亿公顷,年产量超过7.4亿吨,养活了世界上35%的人口,分别提供了人类所需热能和蛋白质的19%和20% (http://www.fao.org/, http://www.wheatgenome.org)。我国是世界上小麦种植面积最大和总产量最高的国家,也是全球最大的小麦消耗国,常年种植面积在2400万公顷左右,年产量超过1.2亿吨(http:// www.xiaomai.cn/html/news/20150226/363917.html)。随着世界人口的不断增加,预计到2050年将达到96亿,对小麦的需求量将在现有产能基础上增加60%[1],再加之全球气温升高和极端天气频发,使小麦生产面临着严峻挑战。为了保证我国乃至全世界的粮食安全,维持小麦生产的增产和稳产,迫切需要加强小麦的基础理论和育种应用研究,培育出更多环境友好型的高产优质小麦新品种。
世界三大主要粮食作物中,水稻和玉米基因组的测序分别于2002年和2009年相继完成[2~4],并有力地推动了这两大作物的基础和分子育种研究。小麦基因组庞大(16 Gb)且含有大量重复序列,致使小麦基因组测序和组装变得极为困难。基因组测序的缓慢进展,严重制约了小麦的基础和应用研究。近年来,在科学家们的不懈努力下,随着高通量测序和生物信息分析技术的发展,小麦基因组测序研究取得了重大突破。自2003年普通小麦3B染色体物理图谱发表以来[5],许多不同倍性的小麦基因组测序研究结果相继发表。仅在2017和2018年就先后公布了A[6]、D[7]、AB[8]和ABD[9]4个小麦基因组的参考序列,其中乌拉尔图小麦A基因组的测序与分析是由中国科学院遗传与发育生物学研究所植物细胞与染色体工程国家重点实验室主导完成。本文总结了本实验室多年来在乌拉尔图小麦A基因组测序和进化研究领域的相关工作以及小麦A基因组的测序研究进展,以期为相关领域的科研人员提供参考信息,促进小麦的基础理论和分子育种研究。
1 小麦的起源及染色体组型
小麦是小麦属(L.)植物的统称,起源于中东的新月沃地(Fertile Crescent)。根据含有的染色体组型,可以将小麦属归纳为6个种,包括2个二倍体种、2个四倍体种和2个六倍体种,有A、B、D和G 4种染色体组。二倍体小麦皆为A染色体组,如乌拉尔图小麦(Thumanjan ex Gandilyan,AA, 2n=2x=14)和一粒小麦(L., AmAm, 2n=2x=14)。四倍体小麦中具有AB和AG两类染色体组型。具AB染色体组的四倍体小麦(L., BBAA, 2n=4x=28)是由A基因组供体乌拉尔图小麦与B基因组供体拟斯卑尔脱山羊草(未完全确定)(, SS, 2n=2x=14)在50~300万年前经天然杂交和染色体加倍而形成,其中包括野生二粒小麦(ssp.(Körn. Ex Asch. &Graebn.) Thell.)、栽培二粒小麦(ssp.(Schrank ex Schübl.) Thell.)、圆锥小麦(L.)和硬粒小麦(Desf.)等不同类型。而AG染色体组的四倍体小麦也是由含A基因组的乌拉尔图小麦与1种含有G基因组的拟斯卑尔脱山羊草(未知种)杂交和染色体加倍而来(Zhuk, AAGG, 2n=4x=28),包括野生阿拉拉特小麦(ssp.)和栽培的提莫菲维小麦(ssp.)两类。六倍体小麦在自然界中存在ABD和AmAG两个染色体组类型。具ABD染色体组的普通小麦(L., BBAADD, 2n=6x=42)是由栽培二粒小麦(ssp., BBAA, 2n=4x=28)与具有D染色体组二倍体粗山羊草((Coss.) Schmal, DD, 2n=2x=17)在大约9000年前经天然杂交和染色体加倍而形成。而具AmAG染色体组的茹科夫斯基小麦(Menabde&Ericzjan, AmAmAAGG, 2n=6x=42)是由四倍体提莫菲维小麦(AAGG)与一粒小麦(AmAm)杂交进化而来[10,11]。
在小麦属的6个物种中,六倍体普通小麦的种植面积最广,占全世界小麦总生产的95%,其次是四倍体硬粒小麦,约占5%[10]。其余的二倍体栽培一粒小麦、四倍体提莫菲维小麦和六倍体茹科夫斯基小麦只在局部地区或在植物园里有少量种植。
2 小麦A基因组测序与结构解析
如前所述,小麦属所有的6个物种中都含有A基因组,表明小麦A基因组是小麦进化的基础性基因组,在小麦进化过程中起着核心作用。含有A基因组的二倍体乌拉尔图小麦是小麦A基因组的原始供体种,主要分布在中东地区,如黎巴嫩、伊朗、伊拉克、土耳其和叙利亚等,其基因组大小是普通小麦基因组的1/3,约为5 Gb。因此,对二倍体乌拉尔图小麦基因组进行测序,将大大简化基因组测序研究的难点,同时可为普通小麦基因组分析提供参考,也将为多倍体小麦的进化与驯化研究提供重要信息。此外,乌拉尔图小麦基因组的测序还有助于鉴定和分离普通小麦A基因组上的重要农艺性状基因以及加快小麦的遗传改良。
鉴于小麦A基因组的重要性,中国科学院遗传与发育生物学研究所植物细胞与染色体工程国家重点实验室组织了小麦基因组测序攻关团队,以小麦A基因组原始供体种乌拉尔图小麦为研究对象,采取先草图后精细图的分步实施策略,开展乌拉尔图小麦基因组测序研究。
2.1 乌拉尔图小麦基因组草图绘制
2009年,小麦基因组测序攻关团队与华大基因研究院以及美国加州大学戴维斯分校的Jan Dvorak教授、罗明成博士等合作,利用当时刚出现的高通量二代测序技术,选取二倍体乌拉尔图小麦G1812系为材料,进行全基因组鸟枪法测序。利用Hiseq2000测序平台,对57种不同插入片段大小(200~20 000 bp)的测序文库进行测序,共获得450 Gb的有效序列,用华大基因研究院开发的SOAPdenovo (v.1.05)基因组组装软件进行了拼接,通过4年的艰辛研究,于2013年率先在国际上完成和公布了乌拉尔图小麦基因组序列草图[12]。该草图总长为4.66 Gb,Scaffold N50长度为63.6 kb,不含N的完整序列总长为3.92 Gb,Contig N50长度为3.42 kb,预测出34 879个蛋白编码基因。乌拉尔图小麦基因组序列草图的完成和公布,得到了国内外的广泛关注和新闻媒体的大量报道,研究成果入选了2013年度中国科学十大进展。
2.2 乌拉尔图小麦基因组精细图谱绘制
在完成乌拉尔图小麦基因组草图绘制的基础上,为了获得高质量的小麦A基因组的参考基因组序列,小麦基因组测序攻关团队与中国科学院遗传与发育生物学研究所基因组分析平台梁承志研究员合作,采用BAC-by-BAC测序策略,并与第三代测序技术PacBio (Pacific Biosciences sequencing)大片段单分子实时(single-molecular real-time, SMRT) 测序和新的基因组物理图谱构建方法(BioNano genome map 和10× Genomics linked reads)相结合,对乌拉尔图小麦G1812系基因组进行了测序和组装,并于2018年5月公布了乌拉尔图小麦基因组的高质量参考基因组序列[6]。首先构建了RⅠ、dⅢ和Ⅰ共3种不同酶切的BAC文库,对451 584个BAC克隆进行指纹分析,搭建出20 702个BAC重叠群,利用最小重叠法原则(minimal tiling path principle)筛选出47 223个BAC克隆,混合成984个BAC混池(48 BACs/池);提取BAC DNA,构建了300 bp的插入片段测序文库,利用高通量的Illumina HiSeq2500平台进行测序,然后对单个BAC序列进行组装,并用纠错后的全基因组SMRT序列进行补洞;进一步根据BAC重叠群信息、BioNano物理图谱和10× Genomics linked reads将获得的BAC序列进行拼接和组装,最终获得长度为4.86 Gb的组装基因组(Scaffold N50为3.67 Mb),其中无N序列总长为4.79 Gb (Contig N50为344 kb),为乌拉尔图小麦基因组(4.94 Gb)的97%。预测出41 507个蛋白编码基因,基因平均长度为1453 bp,编码蛋白的平均长度为332氨基酸。此外,还鉴定出了31 269个miRNAs、5810个lncRNAs、3620个tRNAs、80个核糖体rRNAs和2519个snRNAs。并利用高密度的单核甘酸多态性(single nucleotide polymorphism, SNP)遗传图谱将4.67 Gb序列铆钉到了染色体的相应位置,绘制出小麦A基因组7条染色体的假分子(pseudomolecule),其从第1至第7条染色体长度分别为583 994 449、755 818 500、747 043 632、619 526 116、662 474 153、576 970 508和719 848 703 bp。基因组各组分(如基因、重复序列、SSR等)在染色体上分布的偏好性分析显示,在每条染色体上的两个末端区的基因密度和重组交换率显著高于染色体的中间区域,而重复序列主要分布在染色体中间区域[6]。
3 比较基因组学及染色体进化分析
利用获得的高质量乌拉尔图小麦基因组数据,研究人员对基因组中重复序列、基因家族的组成特征、庞大基因组的形成原因、染色体演化、群体基因组学等方面进行深入分析。
3.1 乌拉尔图小麦基因组中的重复序列
基因组注释显示,乌拉尔图基因组中81.42% (3.9 Gb)的组装序列为重复序列,其中RNA反转座子重复序列占71.83% (3.44 Gb),DNA转座子重复序列为7.41% (355 Mb)。在反转座子重复序列中,主要是长末端重复反转座子(long-terminal repeat retrotransposons, LTR-RTs),由Gypsy和Copia两类LTR反转座子组成,它们分别占全基因组的42.71%和24.30%,在乌拉尔图小麦基因组的进化过程中起到了重要作用。通过计算完整Gypsy和Copia反转座子LTR区的突变率估计其插入时间,发现Gypsy类反转座子插入高峰大约在1.5百万年前,而Copia类反转座子的插入高峰时间约在0.8~0.9百万年,从而证明在乌拉尔图小麦基因组进化过程中先发生了Gypsy的爆发,然后再出现Copia反转座子的大量插入。并且这两个最大组分的反转座子在染色体上的分布偏好性也存在明显差异,Gypsy主要分布在染色体中间部位,而Copia更多分布在染色体末端。
3.2 基因家族比较分析
利用组装的乌拉尔图小麦基因组序列预测的基因家族与已报道的禾本科植物二穗短柄草()、水稻()、高粱()和玉米()的基因家族进行了比较分析,鉴定出1567个乌拉尔图小麦基因组特异基因家族,含4610个基因,功能注释显示多数基因参与逆境胁迫反应[6]。另外,还发现含NB-ARC功能域的抗病基因在乌拉尔图小麦基因组中发生了大量扩增[12],并推断这些基因的扩张可能赋予小麦抵御恶劣生存环境的广泛适应性。对转录因子家族的比较分析显示,B3转录因子家族中的REM (reproductive meristem)亚家族在普通小麦、乌拉尔图小麦和粗山羊草的基因组中均呈现显著扩增。基因主要参与春化及花发育,编码B3 REM转录因子的基因在麦类作物的扩增很可能和小麦的春化反应有关[6]。
3.3 乌拉尔图小麦基因组扩张机制及结构变异
乌拉尔图小麦基因组大小约为5000 Mb,是玉米基因组(2300 Mb[4])的2.3倍、高粱基因组(730 Mb[13])的6.8倍、水稻基因组(420 Mb[2])的12倍、二穗短柄草基因组(272 Mb[14])的18.4倍,这表明乌拉尔图小麦基因组在进化过程中发生了大量扩增。基因组测序为揭示基因组扩增的机制提供了机遇。
通过对二穗短柄草与乌拉尔图小麦的共线性分析,发现乌拉尔图小麦基因组中约21%的基因在基因密度上与对应的二穗短柄草共线性区段的基因密度相当,但79%的乌拉尔图小麦基因在进化过程中被大量的Gypsy和Copia反转座子插入隔离,使其基因间的平均距离与二穗短柄草基因组相比增大了近20倍[12],这是首次从全基因组水平上解释了小麦基因组庞大的原因。
在乌拉尔图基因组内识别了5个成对的共线性旁系同源基因区域,它们是7000万年前的一次全基因组复制事件的产物。对该复制事件在水稻基因组内遗留的成对共线性旁系同源基因在乌拉尔图小麦中的保守性分析显示,高达85%的水稻同源基因对在乌拉尔图小麦中已检测不到。其中,47%的基因对在乌拉尔图小麦中发生了单拷贝缺失,38%的水稻基因对的两个拷贝在对应的乌拉尔图小麦的同源共线区域内均缺失[6]。这些结果表明在与水稻分离后,乌拉尔图小麦的基因组发生过大规模的重组或变异。
比较乌拉尔图小麦基因组序列和已报道的中国春小麦基因组序列,鉴定出了3个大的染色体结构变异:(1)乌拉尔图小麦4和5号染色体间的长臂末端发生相互易位,该易位发生在A、B和D基因组分开后,四倍体小麦形成前;(2)中国春小麦基因组中7B向4A的单向染色体片段易位;(3)中国春4A染色体中的一个含着丝粒的大片段倒位。后面两个结构变异时间发生在四倍化过程中或四倍化后[6]。此外,对乌拉尔图小麦7号染色体和中国春7A染色体的DNA序列比较发现,两个染色体的DNA序列相似性为90%,但具各自独有的转座子插入[6]。
3.4 乌拉尔图小麦染色体进化分析
染色体是基因的载体,不同物种具有不同的染色体数目。从进化角度来看,禾本科的所有物种都是从一个共同祖先逐渐演化而来。在已测序的禾本科物种中,水稻1n = 12、高粱和玉米1n = 10、二穗短柄草1n = 5,而乌拉尔图小麦1n = 7。那么乌拉尔图小麦基因组的7条染色体是如何从禾本科植物共同祖先的染色体组演化而成?通过将乌拉尔图小麦基因组中的基因与水稻、高粱和二穗短柄草基因组内旁系同源基因的共线性分布的分析,结合Salse等[15]的染色体进化假设,研究人员推演出乌拉尔图小麦7条染色体的进化模型。便于理解本文对该模型进行了进一步总结归纳,绘制了乌拉尔图小麦7条染色体进化示意图(图1)。如图所示,禾本科植物鼻祖的染色体基数为7,在距今大约7000万年前经历了一次全基因组复制[16],变为14,其中4条染色体发生两两融合,形成了禾本科植物的12条祖先染色体。这12条禾本科祖先染色体在水稻中得以很好保存,而这12条祖先染色体再经断裂、融合和染色体片段重排等,演化出了乌拉尔图小麦的7条染色体。乌拉尔图小麦的3号和6号染色体分别来自禾本科祖先的1号和2号(也即水稻1号和2号)染色体。而乌拉尔图小麦的1号、2号、4号和7号染色体分别由祖先的两条染色体融合而成。其中,祖先的10号(水稻10号)染色体插入祖先的5号(水稻的5号)染色体的着丝粒区,形成乌拉尔图小麦的1号染色体;祖先的7号(水稻7号)染色体插入到祖先的4号(水稻4号)染色体的着丝粒区,形成乌拉尔图小麦的2号染色体;祖先的11号(水稻11号)染色体插入祖先的3号(水稻3号)染色体的着丝粒区,形成乌拉尔图小麦的4号染色体;祖先的8号(水稻8号)染色体插入到祖先的6号(水稻6号)染色体的着丝粒区,形成了乌拉尔图小麦的7号染色体[6]。这种由着丝粒区插入完成的染色体融合在二穗短柄草中也有报道,并且在二穗短柄草中还观察到更为复杂的多层嵌套插入[14]。区别于上述染色体融合方式,乌拉尔图小麦的5号染色体由祖先的12号(水稻12号)和9号(水稻9号)染色体头尾相接而成,此外其3′端粒区还融合了位于祖先3号(水稻3号)染色体的3′近端粒区和5′端粒区的两个染色体小片段[6]。水稻、高粱、玉米、乌拉尔图小麦和二穗短柄草的进化树显示[14],二穗短柄草与乌拉尔图小麦有更近的亲缘关系,但这两个物种的染色体演化模式完全不同,表明乌拉尔图小麦和二穗短柄草的染色体组进化是独立发生的。
3.5 乌拉尔图小麦群体基因组学分析
如前所述,乌拉尔图小麦主要分布在新月沃地。利用从亚美尼亚、伊朗、伊拉克、叙利亚、土耳其和黎巴嫩等6个国家不同生态环境收集到的147份乌拉尔图小麦系(accession)为材料,进行了群体基因组学分析;通过叶片转录组测序,鉴定出144 806个高质量SNP,根据获得的SNP进行系统发育分析,可将147份乌拉尔图小麦聚类成3个大类群[6]。类群1 (第Ⅰ组)包含来自多个国家的30个品种;类群2 (第Ⅱ组)包含64个品种,其中88%来自黎巴嫩;类群3 (第Ⅲ组)包含53个品种,其中92%来自土耳其。这3个类群材料的采集地点在海拔高度上存在明显差异,类群2大多数材料的采集海拔高度超过1000米,而绝大多数类群1和类群3材料的采集海拔高度低于1000米。通过对小麦白粉菌f. sp.(Bgt, race E09)接种鉴定发现,乌拉尔图小麦的这3个类群在抗白粉病菌侵染方面存在明显差异。来自黎巴嫩高海拔的类群2 (第Ⅱ组)中,92.2%的系表现出抗性,而来自于低海拔的类群1 (第Ⅰ组) 和3 (第Ⅲ组)的大部分系(分别为96.7%和90.6%)为易感型[6]。这些研究结果暗示不同海拔高度生态环境与乌拉尔图小麦适应性进化密切相关。
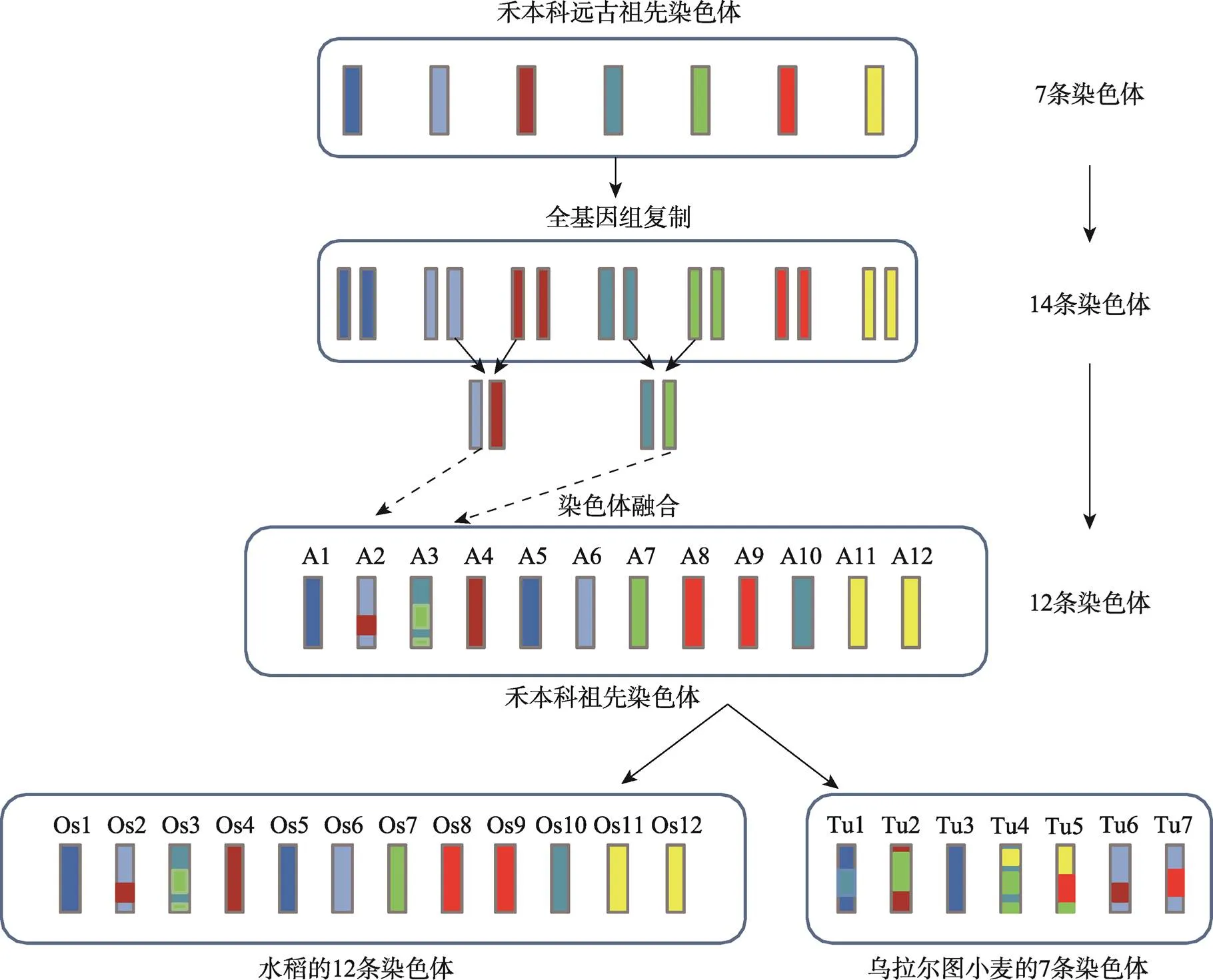
图1 乌拉尔图小麦7条染色体的进化模型
3.6 乌拉尔图小麦基因组的测序促进了小麦的基础研究
乌拉尔图小麦是普通小麦A亚基因组的供体,具有较六倍体小麦更为简单的基因组结构,对于研究小麦A基因组基因型和表型的相互关联能够提供更加直接和确定性的证据支持。受多倍化和长期驯化的影响,普通小麦A亚基因组与之相比已经有了相当的变化,为研究来自不同亚基因组同源基因在多倍化和驯化过程中发生的变化提供依据。乌拉尔图小麦是小麦抗白粉病基因的重要来源。利用乌拉尔图小麦基因组序列设计的分子标记,结合遗传作图和RNA测序,Zou等[17]在很短时间内从具有白粉病抗性的乌拉尔图小麦PI428309品系中定位并克隆了白粉病抗性基因,这也是第一个从乌拉尔图小麦中克隆的抗病基因。此外,研究人员利用水稻等作物的已知基因序列与乌拉尔图小麦序列进行比对,鉴定和克隆了多个小麦重要农艺性状基因,如控制小麦粒长的基因。Dong等[18]利用水稻基因序列与乌拉尔小麦图基因组序列比对,获得了水稻在小麦A基因组上的同源基因,利用乌拉尔图小麦基因组序列设计引物,扩增出了六倍体小麦基因组中A亚基因组的,并通过基因的单倍型分析,结合表型数据证明了该基因在普通小麦中参与对籽粒性状的调控。这些结果表明,乌拉尔图小麦基因组结构解析推动了小麦的基础和应用研究。
4 小麦A基因组研究的未解之谜
综上所述,小麦的A基因组在多倍体小麦进化过程中起到了核心作用。它的起源很明确,来自于二倍体乌拉尔图小麦。普通小麦的A亚基因组历经了从供体乌拉尔图小麦→野生四倍体小麦→栽培四倍体小麦→栽培六倍体小麦的进化和驯化。在这一系列进化和驯化过程中,A基因组如何改变其自身的结构、基因组成、基因表达等来适应多倍化环境和接纳B、D基因组这一基础科学问题目前仍是未解之谜。二倍体供体乌拉尔图小麦[6]、野生二粒小麦[8]、栽培二粒小麦[19]和六倍体普通小麦[9]基因组测序的完成和高质量参考基因组序列的公布,为研究这一基础科学问题提供了前所未有的新机遇。通过深入比较分析将揭示多倍体物种进化和人工驯化过程的分子机制及基础理论,同时也有助于小麦基因组的功能分析和遗传改良。
[1] Rosegrant MW, Cline SA. Global food security: challenges and policies., 2003, 302(5652): 1917–1919.
[2] Goff SA, Ricke D, Lan TH, Presting G, Wang R, Dunn M, Glazebrook J, Sessions A, Oeller P, Varma H, Hadley D, Hutchison D, Martin C, Katagiri F, Lange BM, Moughamer T, Xia Y, Budworth P, Zhong J, Miguel T, Paszkowski U, Zhang S, Colbert M, Sun WL, Chen L, Cooper B, Park S, Wood TC, Mao L, Quail P, Wing R, Dean R, Yu Y, Zharkikh A, Shen R, Sahasrabudhe S, Thomas A, Cannings R, Gutin A, Pruss D, Reid J, Tavtigian S, Mitchell J, Eldredge G, Scholl T, Miller RM, Bhatnagar S, Adey N, Rubano T, Tusneem N, Robinson R, Feldhaus J, Macalma T, Oliphant A, Briggs S. A draft sequence of the rice genome (L. ssp.)., 2002, 296(5565): 92–100.
[3] Yu J, Hu S, Wang J, Wong GK, Li S, Liu B, Deng Y, Dai L, Zhou Y, Zhang X, Cao M, Liu J, Sun J, Tang J, Chen Y, Huang X, Lin W, Ye C, Tong W, Cong L, Geng J, Han Y, Li L, Li W, Hu G, Huang X, Li W, Li J, Liu Z, Li L, Liu J, Qi Q, Liu J, Li L, Li T, Wang X, Lu H, Wu T, Zhu M, Ni P, Han H, Dong W, Ren X, Feng X, Cui P, Li X, Wang H, Xu X, Zhai W, Xu Z, Zhang J, He S, Zhang J, Xu J, Zhang K, Zheng X, Dong J, Zeng W, Tao L, Ye J, Tan J, Ren X, Chen X, He J, Liu D, Tian W, Tian C, Xia H, Bao Q, Li G, Gao H, Cao T, Wang J, Zhao W, Li P, Chen W, Wang X, Zhang Y, Hu J, Wang J, Liu S, Yang J, Zhang G, Xiong Y, Li Z, Mao L, Zhou C, Zhu Z, Chen R, Hao B, Zheng W, Chen S, Guo W, Li G, Liu S, Tao M, Wang J, Zhu L, Yuan L, Yang H. A draft sequence of the rice genome (L. ssp.)., 2002, 296(5565): 79–92.
[4] Schnable PS, Ware D, Fulton RS, Stein JC, Wei F, Pasternak S, Liang C, Zhang J, Fulton L, Graves TA, Minx P, Reily AD, Courtney L, Kruchowski SS, Tomlinson C, Strong C, Delehaunty K, Fronick C, Courtney B, Rock SM, Belter E, Du F, Kim K, Abbott RM, Cotton M, Levy A, Marchetto P, Ochoa K, Jackson SM, Gillam B, Chen W, Yan L, Higginbotham J, Cardenas M, Waligorski J, Applebaum E, Phelps L, Falcone J, Kanchi K, Thane T, Scimone A, Thane N, Henke J, Wang T, Ruppert J, Shah N, Rotter K, Hodges J, Ingenthron E, Cordes M, Kohlberg S, Sgro J, Delgado B, Mead K, Chinwalla A, Leonard S, Crouse K, Collura K, Kudrna D, Currie J, He R, Angelova A, Rajasekar S, Mueller T, Lomeli R, Scara G, Ko A, Delaney K, Wissotski M, Lopez G, Campos D, Braidotti M, Ashley E, Golser W, Kim H, Lee S, Lin J, Dujmic Z, Kim W, Talag J, Zuccolo A, Fan C, Sebastian A, Kramer M, Spiegel L, Nascimento L, Zutavern T, Miller B, Ambroise C, Muller S, Spooner W, Narechania A, Ren L, Wei S, Kumari S, Faga B, Levy MJ, McMahan L, Van Buren P, Vaughn MW, Ying K, Yeh CT, Emrich SJ, Jia Y, Kalyanaraman A, Hsia AP, Barbazuk WB, Baucom RS, Brutnell TP, Carpita NC, Chaparro C, Chia JM, Deragon JM, Estill JC, Fu Y, Jeddeloh JA, Han Y, Lee H, Li P, Lisch DR, Liu S, Liu Z, Nagel DH, McCann MC, SanMiguel P, Myers AM, Nettleton D, Nguyen J, Penning BW, Ponnala L, Schneider KL, Schwartz DC, Sharma A, Soderlund C, Springer NM, Sun Q, Wang H, Waterman M, Westerman R, Wolfgruber TK, Yang L, Yu Y, Zhang L, Zhou S, Zhu Q, Bennetzen JL, Dawe RK, Jiang J, Jiang N, Presting GG, Wessler SR, Aluru S, Martienssen RA, Clifton SW, McCombie WR, Wing RA, Wilson RK. The B73 maize genome: complexity, diversity, and dynamics., 2009, 326(5956): 1112–1115.
[5] Paux E, Sourdille P, Salse J, Saintenac C, Choulet F, Leroy P, Korol A, Michalak M, Kianian S, Spielmeyer W, Lagudah E, Somers D, Kilian A, Alaux M, Vautrin S, Bergès H, Eversole K, Appels R, Safar J, Simkova H, Dolezel J, Bernard M, Feuillet C. A physical map of the 1-gigabase bread wheat chromosome 3B., 2008, 322(5898): 101–104.
[6] Ling HQ, Ma B, Shi X, Liu H, Dong L, Sun H, Cao Y, Gao Q, Zheng S, Li Y, Yu Y, Du H, Qi M, Li Y, Lu H, Yu H, Cui Y, Wang N, Chen C, Wu H, Zhao Y, Zhang J, Li Y, Zhou W, Zhang B, Hu W, van Eijk MJT, Tang J, Witsenboer HMA, Zhao S, Li Z, Zhang A, Wang D, Liang C. Genome sequence of the progenitor of wheat A subgenome., 2018, 557(7705): 424–428.
[7] Luo MC, Gu YQ, Puiu D, Wang H, Twardziok SO, Deal KR, Huo N, Zhu T, Wang L, Wang Y, McGuire PE, Liu S, Long H, Ramasamy RK, Rodriguez JC, Van SL, Yuan L, Wang Z, Xia Z, Xiao L, Anderson OD, Ouyang S, Liang Y, Zimin AV, Pertea G, Qi P, Bennetzen JL, Dai X, Dawson MW, Müller HG, Kugler K, Rivarola-Duarte L, Spannagl M, Mayer KFX, Lu FH, Bevan MW, Leroy P, Li P, You FM, Sun Q, Liu Z, Lyons E, Wicker T, Salzberg SL, Devos KM, Dvořák J. Genome sequence of the progenitor of the wheat D genome., 2017, 551 (7681): 498–502.
[8] Avni R, Nave M, Barad O, Baruch K, Twardziok SO, Gundlach H, Hale I, Mascher M, Spannagl M, Wiebe K, Jordan KW, Golan G, Deek J, Ben-Zvi B, Ben-Zvi G, Himmelbach A, MacLachlan RP, Sharpe AG, Fritz A, Ben-David R, Budak H, Fahima T, Korol A, Faris JD, Hernandez A, Mikel MA, Levy AA, Steffenson B, Maccaferri M, Tuberosa R, Cattivelli L, Faccioli P, Ceriotti A, Kashkush K, Pourkheirandish M, Komatsuda T, Eilam T, Sela H, Sharon A, Ohad N, Chamovitz DA, Mayer KFX, Stein N, Ronen G, Peleg Z, Pozniak CJ, Akhunov ED, Distelfeld A. Wild emmer genome architecture and diversity elucidate wheat evolution and domestication., 2017, 357(6346): 93–97.
[9] International Wheat Genome Sequencing Consortium (IWGSC), Appels R, Eversole K, Feuillet C, Keller B, Rogers J, Stein N, Pozniak CJ, Stein N, Choulet F, Distelfeld A, Eversole K, Poland J, Rogers J, Ronen G, Sharpe AG, Pozniak C, Ronen G, Stein N, Barad O, Baruch K, Choulet F, Keeble-Gagnère G, Mascher M, Sharpe AG, Ben-Zvi G, Josselin AA, Stein N, Mascher M, Himmelbach A, Choulet F, Keeble-Gagnère G, Mascher M, Rogers J, Balfourier F, Gutierrez-Gonzalez J, Hayden M, Josselin AA, Koh C, Muehlbauer G, Pasam RK, Paux E, Pozniak CJ, Rigault P, Sharpe AG, Tibbits J, Tiwari V, Choulet F, Keeble-Gagnère G, Mascher M, Josselin AA, Rogers J, Spannagl M, Choulet F, Lang D, Gundlach H, Haberer G, Keeble-Gagnère G, Mayer KFX, Ormanbekova D, Paux E, Prade V, Šimková H, Wicker T, Choulet F, Spannagl M, Swarbreck D, Rimbert H, Felder M, Guilhot N, Gundlach H, Haberer G, Kaithakottil G, Keilwagen J, Lang D, Leroy P, Lux T, Mayer KFX, Twardziok S, Venturini L, Appels R, Rimbert H, Choulet F, Juhász A, Keeble-Gagnère G, Choulet F, Spannagl M, Lang D, Abrouk M, Haberer G, Keeble-Gagnère G, Mayer KFX, Wicker T, Choulet F, Wicker T, Gundlach H, Lang D, Spannagl M, Lang D, Spannagl M, Appels R, Fischer I, Uauy C, Borrill P, Ramirez-Gonzalez RH, Appels R, Arnaud D, Chalabi S, Chalhoub B, Choulet F, Cory A, Datla R, Davey MW, Hayden M, Jacobs J, Lang D, Robinson SJ, Spannagl M, Steuernagel B, Tibbits J, Tiwari V, van Ex F, Wulff BBH, Pozniak CJ, Robinson SJ, Sharpe AG, Cory A, Benhamed M, Paux E, Bendahmane A, Concia L, Latrasse D, Rogers J, Jacobs J, Alaux M, Appels R, Bartoš J, Bellec A, Berges H, Doležel J, Feuillet C, Frenkel Z, Gill B, Korol A, Letellier T, Olsen OA, Šimková H, Singh K, Valárik M, van der Vossen E, Vautrin S, Weining S, Korol A, Frenkel Z, Fahima T, Glikson V, Raats D, Rogers J, Tiwari V, Gill B, Paux E, Poland J, Doležel J, Číhalíková J, Šimková H, Toegelová H, Vrána J, Sourdille P, Darrier B, Appels R, Spannagl M, Lang D, Fischer I, Ormanbekova D, Prade V, Barabaschi D, Cattivelli L, Hernandez P, Galvez S, Budak H, Steuernagel B, Jones JDG, Witek K, Wulff BBH, Yu G, Small I, Melonek J, Zhou R, Juhász A, Belova T, Appels R, Olsen OA, Kanyuka K, King R, Nilsen K, Walkowiak S, Pozniak CJ, Cuthbert R, Datla R, Knox R, Wiebe K, Xiang D, Rohde A, Golds T, Doležel J, Čížková J, Tibbits J, Budak H, Akpinar BA, Biyiklioglu S, Muehlbauer G, Poland J, Gao L, Gutierrez-Gonzalez J, N'Daiye A, Doležel J, Šimková H, Číhalíková J, Kubaláková M, Šafář J, Vrána J, Berges H, Bellec A, Vautrin S, Alaux M, Alfama F, Adam-Blondon AF, Flores R, Guerche C, Letellier T, Loaec M, Quesneville H, Pozniak CJ, Sharpe AG, Walkowiak S, Budak H, Condie J, Ens J, Koh C, Maclachlan R, Tan Y, Wicker T, Choulet F, Paux E, Alberti A, Aury JM, Balfourier F, Barbe V, Couloux A, Cruaud C, Labadie K, Mangenot S, Wincker P, Gill B, Kaur G, Luo M, Sehgal S, Singh K, Chhuneja P, Gupta OP, Jindal S, Kaur P, Malik P, Sharma P, Yadav B, Singh NK, Khurana J, Chaudhary C, Khurana P, Kumar V, Mahato A, Mathur S, Sevanthi A, Sharma N, Tomar RS, Rogers J, Jacobs J, Alaux M, Bellec A, Berges H, Doležel J, Feuillet C, Frenkel Z, Gill B, Korol A, van der Vossen E, Vautrin S, Gill B, Kaur G, Luo M, Sehgal S, Bartoš J, Holušová K, Plíhal O, Clark MD, Heavens D, Kettleborough G, Wright J, Valárik M, Abrouk M, Balcárková B, Holušová K, Hu Y, Luo M, Salina E, Ravin N, Skryabin K, Beletsky A, Kadnikov V, Mardanov A, Nesterov M, Rakitin A, Sergeeva E, Handa H, Kanamori H, Katagiri S, Kobayashi F, Nasuda S, Tanaka T, Wu J, Appels R, Hayden M, Keeble-Gagnère G, Rigault P, Tibbits J, Olsen OA, Belova T, Cattonaro F, Jiumeng M, Kugler K, Mayer KFX, Pfeifer M, Sandve S, Xun X, Zhan B, Šimková H, Abrouk M, Batley J, Bayer PE, Edwards D, Hayashi S, Toegelová H, Tulpová Z, Visendi P, Weining S, Cui L, Du X, Feng K, Nie X, Tong W, Wang L, Borrill P, Gundlach H, Galvez S, Kaithakottil G, Lang D, Lux T, Mascher M, Ormanbekova D, Prade V, Ramirez-Gonzalez RH, Spannagl M, Stein N, Uauy C, Venturini L, Stein N, Appels R, Eversole K, Rogers J, Borrill P, Cattivelli L, Choulet F, Hernandez P, Kanyuka K, Lang D, Mascher M, Nilsen K, Paux E, Pozniak CJ, Ramirez-Gonzalez RH, Šimková H, Small I, Spannagl M, Swarbreck D, Uauy C. Shifting the limits in wheat research and breeding using a fully annotated reference genome., 2018, 361(6403): eaar7191
[10] Fuller DQ, Lucas L. Wheats: Origins and Development. In: Encyclopedia of Global ArchaeologyNew York, Springer, 2014.
[11] Peng JH, Sun DF, Nevo E. Domestication evolution, genetics and genomics in wheat., 2011, 28(3): 281–301.
[12] Ling HQ, Zhao S, Liu D, Wang J, Sun H, Zhang C, Fan H, Li D, Dong L, Tao Y, Gao C, Wu H, Li Y, Cui Y, Guo X, Zheng S, Wang B, Yu K, Liang Q, Yang W, Lou X, Chen J, Feng M, Jian J, Zhang X, Luo G, Jiang Y, Liu J, Wang Z, Sha Y, Zhang B, Wu H, Tang D, Shen Q, Xue P, Zou S, Wang X, Liu X, Wang F, Yang Y, An X, Dong Z, Zhang K, Zhang X, Luo MC, Dvorak J, Tong Y, Wang J, Yang H, Li Z, Wang D, Zhang A, Wang J. Draft genome of the wheat A-genome progenitor., 2013, 496 (7443): 87–90.
[13] Paterson AH, Bowers JE, Bruggmann R, Dubchak I, Grimwood J, Gundlach H, Haberer G, Hellsten U, Mitros T, Poliakov A, Schmutz J, Spannagl M, Tang HB, Wang XY, Wicker T, Bharti AK, Chapman J, Feltus FA, Gowik U,Grigoriev IV, Lyons E, Maher CA, Martis M, Narechania A, Otillar RP, Penning BW, Salamov AA, Wang Y, Zhang LF, Carpita NC, Freeling M, Gingle AR, Hash CT, Keller B, Klein P, Kresovich S, McCann MC, Ming R, Peterson DG, Mehboob-ur-Rahman, Ware D, Westhoff P, Mayer KF, Messing J, Rokhsar DS. Thegenome and the diversification of grasses., 2009, 457(7229): 551–556.
[14] International Brachypodium Initiative. Genome sequencing and analysis of the model grass., 2010, 463(7282): 763–768.
[15] Salse J, Bolot S, Throude M, Jouffe V, Piegu B, Quraishi UM, Calcagno T, Cooke R, Delseny M, Feuillet C. Identification and characterization of shared duplications between rice and wheat provide new insight into grass genome evolution., 2008, 20(1): 11–24.
[16] Wang X, Shi X, Hao B, Ge S, Luo J. Duplication and DNA segmental loss in the rice genome: implications for diploidization., 2005, 165(3): 937–946.
[17] Zou S, Wang H, Li Y, Kong Z, Tang D. The NB-LRR gene Pm60 confers powdery mildew resistance in wheat., 2018, 218(1): 298–309
[18] Dong LL, Wang FM, Liu T, Dong ZY, Li A, Jing RL, Mao L, Li YW, Liu X, Zhang KP, Wang DW. Natural variation of TaGASR7-A1 affects grain length in common wheat under multiple cultivation conditions., 2014, 34(3): 937–947
[19] Maccaferri M, Harris NS, Twardziok SO, Pasam RK, Gundlach H, Spannagl M, Ormanbekova D, Lux T, Prade VM, Milner SG, Himmelbach A, Mascher M, Bagnaresi P, Faccioli P, Cozzi P, Lauria M, Lazzari B, Stella A, Manconi A, Gnocchi M, Moscatelli M, Avni R, Deek J, Biyiklioglu S, Frascaroli E, Corneti S, Salvi S, Sonnante G, Desiderio F, Marè C, Crosatti C, Mica E, Özkan H, Kilian B, De Vita P, Marone D, Joukhadar R, Mazzucotelli E, Nigro D, Gadaleta A, Chao S, Faris JD, Melo ATO, Pumphrey M, Pecchioni N, Milanesi L, Wiebe K, Ens J, MacLachlan RP, Clarke JM, Sharpe AG, Koh CS, Liang KYH, Taylor GJ, Knox R, Budak H, Mastrangelo AM, Xu SS, Stein N Hale I, Distelfeld A, Hayden MJ, Tuberosa R, Walkowiak S, Mayer KFX, Ceriotti A, Pozniak CJ, Cattivelli L. Durum wheat genome highlights past domestication signatures and future improvement targets., 2019, 51(5): 885–895.
Progress on wheat A genome illustration and its evolutional analysis
Xiaoli Shi1, Yilin He1,2, Hongqing Ling1,2
Wheat is one of the main food crops and widely grown in the world. It feeds more than 35% of the world's population. Obtaining high-quality genome sequences of wheat is important for its basic and breeding researches. However, the large and complex genome of wheat once led to its genome sequencing as an "impossible task". Recently, with the development of high-throughput sequencing and assembly technology, many wheat genome sequences have been released, and their sequencing and assembly quality is being improved continuously. In the last two years, five wheat reference genomes with different ploidy levels have been published, including two diploid ancestors(AA) and(DD), wild and cultivated tetraploid wheatssp.(BBAA) and hexaploid wheat(BBAADD). Among them, the sequencing and analysis of thegenome, a donor of polyploid wheat A subgenome, was led by the Institute of Genetics and Developmental Biology of the Chinese Academy of Sciences. In this review, we summarize the research progress on structure and evolution analyses of thegenome to provide some valuable information for promoting the basic and applied researches of wheat.
wheat;; wheat A genome; genome sequencing; chromosome evolution
2019-08-10;
2019-09-10
国家自然科学基金项目(编号:31871273)资助[Supported by the National Natural Science Foundation of China (No.31871273)]
史晓黎,博士,助理研究员,研究方向:生物信息和植物比较基因组学。E-mail: xlshi@genetics.ac.cn
凌宏清,研究员,博士生导师,研究方向:小麦基因组学和植物营养分子生物学。E-mail: hqling@genetics.ac.cn
10.16288/j.yczz.19-233
2019/9/16 14:55:15
URI: http://kns.cnki.net/kcms/detail/11.1913.R.20190916.0912.001.html
(责任编委: 夏先春)
