现代代谢组学平台建设及相关技术应用
2019-09-24张凤霞王国栋
张凤霞,王国栋
现代代谢组学平台建设及相关技术应用
张凤霞,王国栋
中国科学院遗传与发育生物学研究所,植物基因组学国家重点实验室,种子创新研究院,北京 100101
代谢组作为生命科学研究的5个层面(基因组、转录组、蛋白质组、代谢组和表型组)之一越来越受到科研工作者的关注。色谱–质谱联用技术由于其高分离能力、高灵敏度等优点在代谢组学(定性和定量)领域发挥重要的作用,基于色谱–质谱联用技术的代谢组学已成功应用于代谢表型差异研究、基因功能鉴定和转基因生物安全性评价等多个研究方向。本文以中国科学院遗传与发育生物学研究所代谢组学平台为例,详细介绍了现代代谢组学平台色谱–质谱联用仪器的硬件组成,以及不同技术平台在现在系统生物学研究中的具体应用。
小分子化合物;代谢组学平台;色谱–质谱联用;系统生物学
小分子代谢物作为细胞调控过程的终产物,其种类和数量的变化被视为生命体对基因或环境变化的最终响应,对这些化合物在动态和静态上进行全面的定性定量分析,即为代谢组学研究。它是目前系统生物学研究的一个重要组成部分,主要通过鉴定动植物体内代谢物变化与特定基因表达变化之间的联系,研究代谢产物在动植物生长发育与环境之间应答中的作用,从而进一步揭示生物体在特定时间、特定环境下代谢网络的整体状态,深入解析生命活动规律的内在机制[1]。根据不同的研究目的,代谢组学可以笼统划分为靶标代谢组学与非靶标代谢组学;从应用技术层面上代谢组学研究又可以分为代谢靶标分析(target analysis)、代谢轮廓分析(metabolic profiling analysis)、代谢指纹分析(metabolic fingerprinting analysis)及代谢组学(metabolomics)等4个层次[2]。目前,色谱–质谱联用技术是代谢组学研究中的主流检测技术,常用仪器包括气相色谱–质谱联用(gas chromatography-mass spectrometry, GC-MS)、液相色谱–质谱联用(liquid chromatography- mass spectrometry, LC-MS)和毛细管电泳质谱联用(capillary electrophoresis-mass spectrometry, CE-MS)等。本文将以中国科学院遗传与发育生物学研究所代谢组学平台为例,从代谢组学平台的硬件组成和实际应用两个层面逐一介绍。
1 代谢组学平台常用硬件设施
中国科学院遗传与发育生物学研究所代谢组学平台是在研究所和植物基因组学国家重点实验室的大力支持下成立的。该平台是以色谱–质谱联用分析技术为基础的小分子代谢物定性和定量分析平台,建立针对不同化学性质代谢产物(如挥发性、高极性等化合物、低丰度的植物激素等)的分析方法,形成能够多层次、多角度地对各种生物组织进行代谢靶标、代谢轮廓以及代谢指纹分析能力。2009年研究所启动了代谢组学平台建设,2010年购置、安装第一台气相色谱–质谱联用仪,2011年购置高灵敏、高通量的超高效液相色谱串联三重四极杆质谱联用仪(ultra high performance liquid chromatography coupled to triple quadrupole mass spectrometer, UHPLC-MS- QQQ),2013年安装了高分辨的超高效液相色谱串联四极杆飞行时间质谱联用仪(ultra high performance liquid chromatography coupled to ion mobility quadrupole- time of flight mass spectrometer, UHPLC-MS-Q-TOF),2018年配备了高分辨、高灵敏且具有离子淌度功能的超高效液相色谱串联四级杆飞行时间质谱联用仪(UHPLC-MS-IM-Q-TOF),引入了第三维度的分离功能。这些大型质谱仪均配备了大功率UPS电源(uninterruptible power supply, UPS),避免了意外断电对质谱仪造成的损害,保障了大型质谱仪的正常运行。图1所示为代谢组学平台目前所拥有的大型质谱仪。同时还配备有氮气发生器、空气压缩机、氮气发生器及空压机一体机和大型精密空调,既保证了质谱仪的连续供气,又为质谱仪提供一个温度恒定的环境。另外,实验室还配备了2台光照培养箱、2台全自动调节植物温室等植物培养必需设备、3台 −80℃超低温冰箱、1台高速冷冻离心机、2台小型控温离心机、1台真空浓缩仪、1台样品研磨仪、1套氮吹仪、1套旋转蒸发仪、3台精密天平、1台冷冻干燥仪、多台涡旋仪和超声仪等样品保存与预处理必须设备,保障了代谢组学平台工作的顺利开展。
2 代谢组学平台技术与应用
色谱法(chromatography)是一种物理或化学分离的分析方法,分离原理主要是利用物质在流动相与固定相之间的分配系数差异而实现分离,是分析混合物最有力的手段,具有高灵敏度、高选择性、高效能、分析速度快和应用范围广等优点[3]。质谱分析法(mass spectrometry, MS)是将化合物形成离子和碎片离子,按其质荷比()的不同进行分离测定,以及成分和结构鉴定的一种分析方法,具有应用范围广、灵敏度高和分析速度快的特点[3]。色谱法进行定性的依据是保留值,很难对复杂混合物进行准确定性;而质谱法具有很强的结构鉴定能力,可以测定化合物的分子量、分子式及有关结构信息,但其不具备分离能力,很难直接对复杂混合物进行定性,因此色谱与质谱技术联用可同时实现复杂混合物的分离、定性与定量的目的[4]。在色谱与质谱联用系统中,色谱充当了质谱的分离和进样装置,而质谱则是色谱的检测器,既弥补了各自的不足,又发挥了各自的功能优势。

图1 基于色谱–质谱联用技术的代谢组学平台仪器构成
A:7980A-5975C GC-MS;B:1290 Infinity UHPLC-6495 QQQ MS;C:1290 Infinity UHPLC-G6550A Q-TOF MS;D:1290 Infinity II UHPLC-G6560B Ion Mobility Q-TOF MS。
代谢组研究所分析的化合物分子量大小、含量、极性、挥发性等物理化学性质差异较大,因此要求分析方法具有高灵敏、高通量和无偏向性的特点,至今为止,尚未有一种分析方法能够满足上述所有要求。而色谱–质谱联用技术既具有色谱的高分离度、高通量也具有质谱的普适性、高灵敏度和高专一性,因而成为代谢组学研究最主要的工具[5~11]。
代谢组学平台目前拥有先进且功能互补的色谱–质谱联用仪。该平台的技术特色主要是利用色谱–质谱联用技术从多层次、多角度对代谢小分子进行快速定性、定量分析。根据个性化的科研需求已建立了涵盖代谢组学、代谢轮廓和代谢靶标分析的20多种分析方法。下面将对不同代谢组学技术平台的应用进行详细介绍。
2.1 基于GC-MS技术的代谢组学平台
GC-MS具有仪器性能稳定、分析重复性好、灵敏度高等特点,有丰富的质谱库可用于化合物的定性,且可提供保留指数信息以辅助定性,适于挥发性、半挥发性化合物或者经衍生后具有挥发性的热稳定性好的化合物检测。目前,GC-MS技术已经比较成熟,在所有联用技术中发展最为完善,应用很非常广泛,已成为代谢组学研究的最有利的工具之一[12~16]。
近年来,代谢组学在农作物研究领域及动物的发育研究中被广泛应用[17~22],为满足科研人员的科研需求,代谢组学平台相继开发了基于GC-MS技术的代谢组学、代谢轮廓及代谢靶标的分析方法。首先,在前人研究的基础上本课题组改进了主生代谢产物的提取及衍生方法,避免了两步衍生法中易出现的衍生不完全的问题,实现了对生物体中三羧酸循环、糖酵解、氨基酸、脂肪酸等代谢途径相关代谢物的高灵敏检测。该方法已被成功应用于拟南芥()、水稻(L.)、棉花(spp)、油菜(L.)、大豆((Linn.) Merr.)、烟草(L.)、果蝇()和蓝藻()等生物组织的相关研究中[23~41]。其中,Tan等[23]利用该方法通过检测转基因拟南芥种子中葡萄糖、果糖和蔗糖的含量,以及检测转基因油菜籽中油脂的含量,证明了和是油菜遗传改良的可靠靶点,该方法在分子育种上具有一定的技术可行性,可推广应用于其他油料作物的改良中。Ding等[26]通过对突变体果蝇脂肪组织进行的代谢组分析,发现突变体在糖酵解通向线粒体三羧酸循环的代谢环节出现了瓶颈,导致合成脂肪的原料(柠檬酸)含量大幅度减少;该研究发现钙离子依赖的线粒体代谢维持脂肪细胞脂质稳态,揭示了-钙离子–线粒体代谢–脂肪储积的调控途径,为致病基因的分子功能和机体脂肪代谢调控提供了新的认知,也为脂肪营养不良疾病提供了多个潜在治疗手段。
其次,本平台建立了植物体中挥发性代谢物谱的检测方法。该方法采用固相微萃取(solid phase micro-extraction, SPME)装置对样品中的挥发性化合物进行富集,而后再进行GC-MS分析,可同时检测醛、酮类化合物、单萜及倍半萜类等挥发性小分子代谢物。该方法已成功应用于蔬菜作物黄瓜(L.)、番茄(Mill.),农作物类小麦(L.)、水稻以及模式植物拟南芥等植物样品中挥发性代谢物的检测[27,28]。Wei等[28]系统分析了黄瓜测序品种9930的不同发育阶段的共计23个样品组织的挥发性化合物,共检测到85个挥发性化合物,有多达36种挥发性萜类化合物,且挥发性化合物的含量呈现出组织特异性。他们通过基因–挥发物关联分析的方法构建了黄瓜挥发性化合物的代谢网络,系统性研究和阐明黄瓜挥发性化合物的生物合成通路和调控的分子机制。该研究也为进一步明确挥发性化合物在植物–环境互作中的生理功能,以及黄瓜风味品质改良育种奠定了坚实基础。
本平台还建立了植物叶片或花药组织中角质、蜡质和总脂肪酸代谢谱的分析方法,为农作物的育种研究提供了很好的技术支持。该方法可以实现只需提供1份样品即可得到3类代谢谱的数据,解决了珍贵样品取样难的问题。同时,要注意采样及与预处理过程中保持样品的完整性,否则植物组织内源性的小分子化合物就会泄露出来,尤其会影响表面蜡质的代谢谱,造成谱图中出现“杂质峰”。图2为植物体表面蜡质、角质以及脂肪酸代谢分析的总离子流色谱图。目前为止,利用该方法已成功检测了拟南芥和盐芥()叶片、玉米(L.)叶片与花药、小麦叶片以及水稻叶片和花药组织[31,32]。Chen等[31]采用该方法检测了玉米花药中的蜡质和角质组成,结果表明()参与了玉米C16/C18 ω-羟基脂肪酸的氧化途径,并与和共同调控花药角质层和花粉外分泌的发育。
2.2 基于UHPLC-MS技术的代谢组学平台
UHPLC-MS是由超高效液相色谱仪、离子源和质谱仪构成。其中较常用的离子化技术包括电喷雾电离(electrospray ionization, ESI)和大气压化学电离(atmospheric pressure chemical ionization, APCI)。ESI比较适合分析极性化合物,同时它还可以生成多电荷离子,因此也能够分析一些生物大分子(如蛋白质、多肽等)。APCI主要产生单电荷离子,适于分析非极性和弱极性小分子代谢物。作为UHPLC的检测器,三重四极杆质谱具备全扫描、子离子扫描、母离子扫描、中性丢失扫描以及多反应监测扫描等多种扫描模式,具有高灵敏和高选择性的特点。因此,UHPLC-MS-QQQ在化合物的快速筛选及复杂基质中微量靶标代谢物的定量分析中有着独特的优势,在代谢靶标分析中的应用最为广泛[42~44]。但是,由于其分辨率较低,故而在化合物鉴定中的表现稍微逊色。而UHPLC-MS-QTOF具有高灵敏、高通量和高分辨率的特点;它既可以在一级质谱图中可提供精确的分子质量,又可以从二级质谱图中得到丰富的离子碎片的精确质量数信息。它可用于化合物的定量,并在化合物的定性分析中具有绝对优势,因此,多被用于非靶标代谢组学或代谢轮廓分析。具有离子淌度功能的四级杆串联飞行时间质谱仪还可以为代谢物的鉴定提供“第三维”分离信息—碰撞截面积(collision cross section, CCS),大大提高了代谢物定性的准确性。CCS值具有一定专属性,可用于区分同分异构体以及带电荷数。离子淌度质谱在脂质组学、区分D和L型氨基酸异构体、聚糖异构体以及糖结合物的分离鉴定中发挥了很好的作用[45~48]。因此,具有离子淌度功能的UHPLC-MS- QTOF将在代谢物的结构鉴定中发挥其优异的作用。
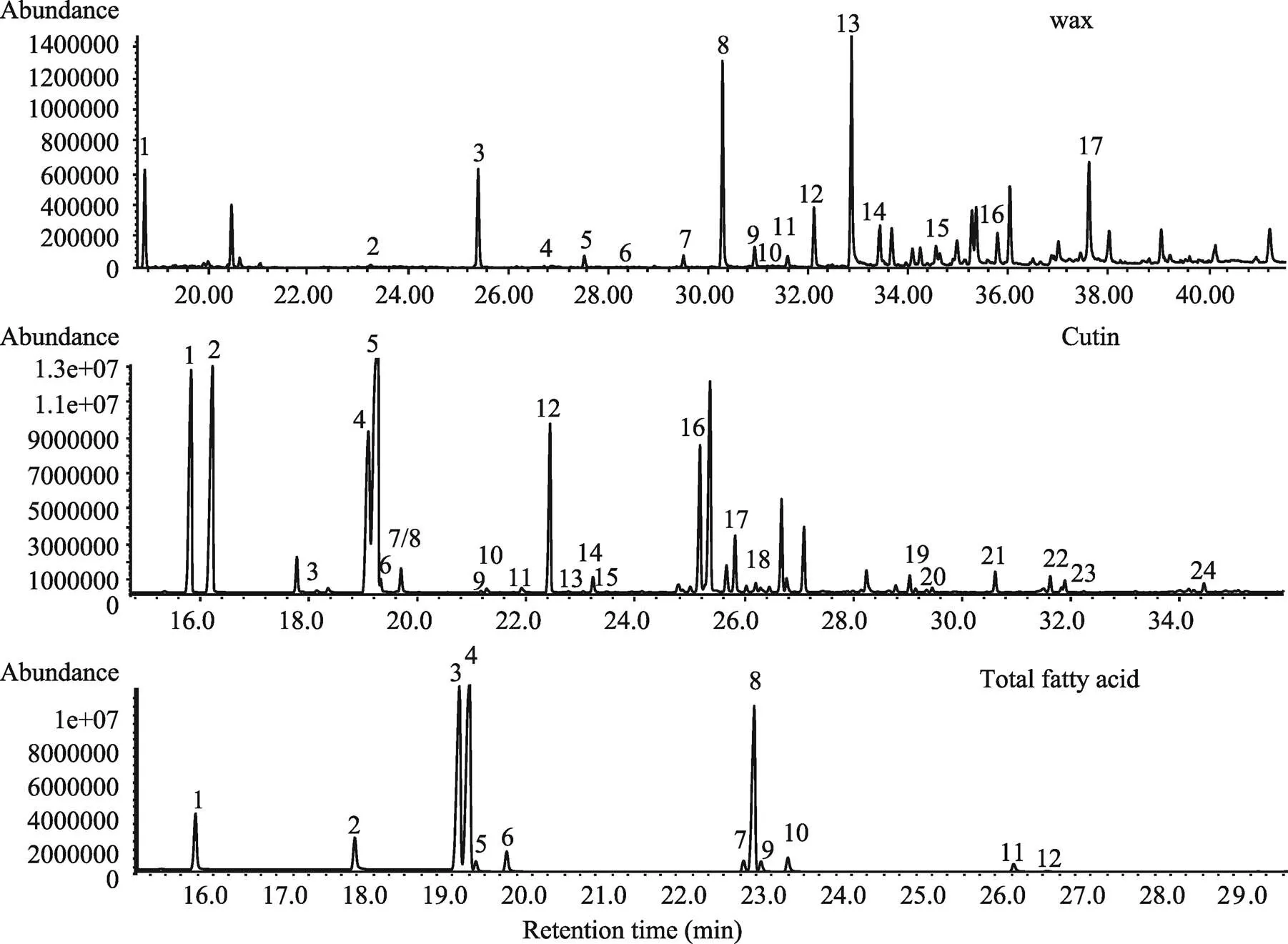
图2 基于气相色谱–质谱联用技术的植物体表面蜡质、角质及总脂肪酸代谢谱分析
根据个性化的研究需求,代谢组学平台建立了高灵敏、高通量的NAD代谢途径中相关代谢物的代谢靶标的定量分析方法,可同时检测18种相关代谢物。在此基础上,充分利用平台仪器的优势功能,提出了整合UHPLC-MS-QTOF与UHPLC-MS-QQQ技术的研究策略来研究NAD补救合成途径中相关基因的功能,并解析NA衍生物在植物代谢中的分子机制及其生理功能[49~51]。首先,利用高分辨的UHPLC-MS-QTOF对野生型(Col-0)和突变体的拟南芥样品进行代谢组学分析,结果在突变体中发现了两个差异比较显著的质谱信号,经推测应为NA的六碳糖糖基化产物(质谱测量误差分别为1.75 ppm和0.35 ppm)。通过对两个化合物的二级质谱分析及对应的酶活产物作为标准品进行验证,鉴定出两个化合物分别为NA的羧酸位和氮位的葡萄糖基化产物。其次,利用UHPLC-MS-QQQ技术分别对不同基因型的拟南芥干种子和萌发种子进行NAD相关代谢物的定量分析,结合其他生理生化的实验结果证明了NAGT活性是在十字花科植物进化过程中才逐渐获得,NAGT活性的获得为植物适应环境提供选择优势。该研究同时为进一步研究植物中其他NA衍生物的生物学功能奠定了基础[49]。
另外,平台还建立了高灵敏、高通量的基于UHPLC-MS-QQQ技术的氨基酸定量分析方法。图3为20种游离氨基酸分析的典型总离子流图,在9分钟内实现了20种氨基酸的快速分离检测,该方法可用于各种植物组织、动物组织及微生物等各生物体中的游离氨基酸的检测。Zhou等[52]利用该方法通过对秀丽线虫和小鼠模型的研究,证明了赖氨酸的正常代谢对于线粒体稳态的维持以及动物的发育是必须的。人类Ⅱ型高赖氨酸血症(酵母氨酸血症)是一种严重影响人类发育的氨基酸代谢缺陷症,本研究揭示了该病的发病机理,并为其治疗提供了重要依据。
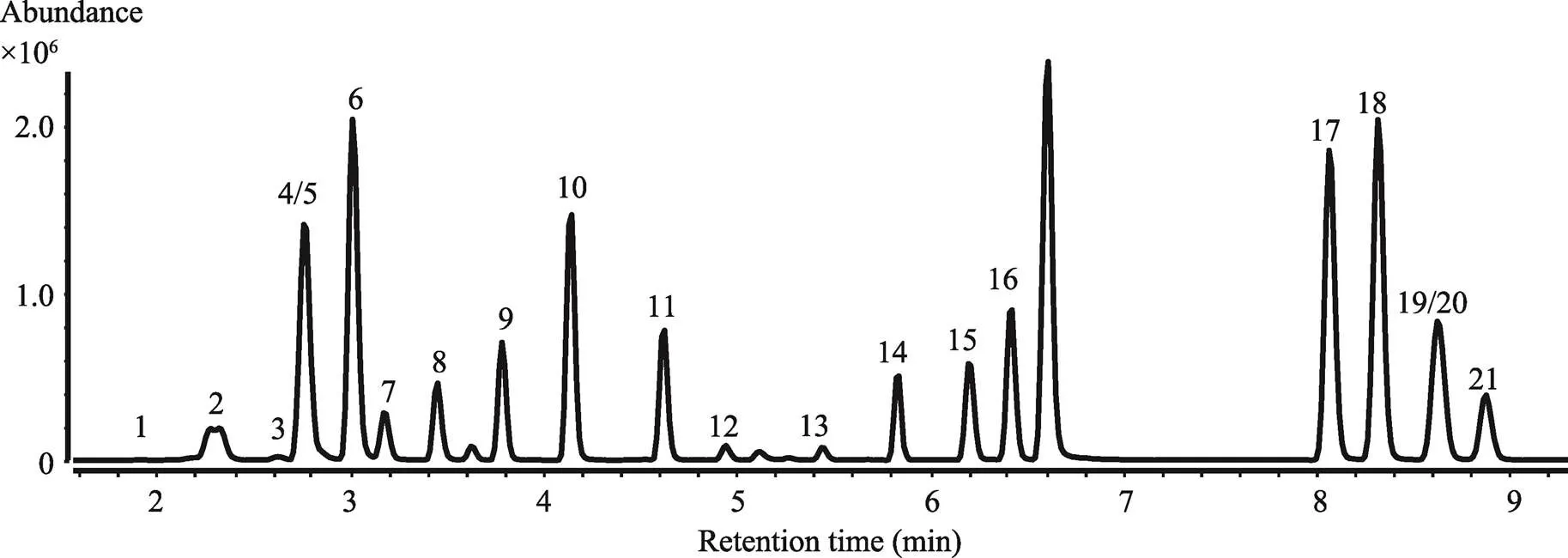
图3 基于液相色谱串联三重四级杆质谱技术的生物体内游离氨基酸代谢谱分析
1:L-His;2:L-Asn;3:L-Arg;4:L-Gln;5:L-Ser;6:L-Gly;7:L-Asp;8:L-Glu;9:L-Thr;10:L-Ala;11:L-Pro;12:L-Orn;13:L-Lys;14:L-Tyr;15:L-Met;16:L-Val;17:L-Ile;18:L-Leu;19:L-Phe;20:L-NorLeu(IS);21:L-Trp。
本平台建立了基于UHPLC-MS-QTOF技术的多个种类代谢物的代谢轮廓分析方法,包括黄酮和异黄酮类代谢谱分析、三萜皂苷类代谢谱分析及萜酚类代谢谱分析等。前期本课题组综合代谢组学、转录组学和生化等技术手段对啤酒花()苦味酸生物合成途径进行了系统研究, 功能鉴定短侧链脂肪酸CoA连接酶[53]。近年来,Li等[54]和Ban等[55]利用UHPLC-MS QTOF技术的代谢轮廓分析方法结合UHPLC-MS-QQQ技术的靶标化合物定量的方法相继深入解析了苦味酸和黄腐醇的的生物合成途径。Ban等[55]研究发现并鉴定两个不具备催化活性的查尔酮异构酶基因(和)参与并调控啤酒花中黄腐醇的生物合成。发现能够与黄腐醇合成通路中上下游蛋白查尔酮合成酶(CHS)和膜定位的异戊烯基转移酶()互作并提高它们的活性,形成的代谢复合体可以高效催化黄腐醇的生物合成;而则能够结合黄腐醇代谢途径中柚皮素查尔酮和脱甲基黄腐醇等中间化合物,稳定这些化合物的开环结构,进而维持其生理活性。同时还进一步发现结合并提高酶促活性的功能在所有的陆生植物中是保守,暗示其在植物从水生到陆生的演化过程中曾发挥着重要作用。
2.3 代谢组学平台应用服务范围
本平台自成立以来,紧跟研究所的发展需求,开发个性化的技术方法,及时满足科研人员的科研需求,既得到了研究所的肯定,也得到了科研人员的一致好评。截至2018年底,仪器总运行有效机时约为25 000 h,其中约5000 h为对外服务测试的有效机时,对外服务测试样本数约15 000个。与平台合作过课题组(研究所内及北京生命科学大型仪器区域中心各单位)约为50多个,合作发表文章30余篇,合作单位包括清华大学、北京大学、北京科技大学、中国水稻所、北京大北农科技集团股份有限公司生物技术中心、四川农业大学、中国农业大学、云南大学、山东农业大学、中国农业科学院蔬菜研究所和青岛烟草所等多个科研单位。代谢组学平台可提供各种生物体的代谢组学、代谢轮廓及代谢靶标分析的科研服务工作,已开发的方法详见表1。
3 结语与展望
色谱–质谱联用技术强大的分离能力和高灵敏度的检测能力为代谢组分析提供了强有力的技术保障。未来更快速的液相色谱技术及高端的质谱技术的出现将会为现代代谢组学平台的发展注入新的动力。目前,代谢组分析的瓶颈为代谢物的结构鉴定(即定性分析)。色谱–质谱联用技术是鉴定代谢物最直接有效的办法,即将代谢物的保留时间、精确质量数、多级质谱碎片同标准品进行比对确认,但是实际研究中往往无法获得商品化的代谢物的标准品。所以,在色谱–质谱联用技术平台的基础上引入核磁共振技术(nuclear magnetic resonance, NMR)将是未来代谢组分析的主要发展方向之一。另外一个值得关注的发展方向就是质谱成像技术(mass spectrometry imaging, MSI),其优点是直接分析生物样品的切片,相对于传统的色质分析技术对样品的破坏性,质谱成像技术可以获得与生物学问题更为直接的小分子化合物的分布和相对定量信息。目前质谱成像技术不但可以绘制出对应分子或离子在生物样本的二维分布图,也可以通过对连续切片的二维分布图进行进一步软件处理,获得待测物在样本中的三维空间分布。随着生物样品制备和质谱技术的飞速发展,质谱成像技术(特别是与转录组和蛋白组数据结合)将会有越来越广泛的应用[56]。

表1 代谢组学平台已开发的分析方法
[1] Fiehn O. Metabolomics – the link between genotypes and phenotypes., 2002, 48(1–2): 155–171.
[2] Feihn O, Kopka J, Dörmann P, Altmann T, Trethewey RN, Willmitzer L. Metabolite profiling for plant functional genomics., 2000, 18(11): 1157–1161.
[3] Sun YQ, Li FM, Li ZW, Wu YT, Zheng ST, Yu YQ, Hu YZ. Analycal Chemistry. Beijing: People's Medical Publishing House, 2001, 184, 206.孙毓庆, 李发美, 李章万, 吴玉田, 郑斯成, 郁韵秋, 胡育筑等编著. 分析化学(第四版,下册). 北京:人民卫生出版社, 2001, 184, 206.
[4] Xu GW, Lu X, Yang J, Wang Y, Zhao XJ, Shi XZ, Tian J, Kong HW, Wang C, Yuan KL, Yin PY, Wang JS, Ma CF, Gao P. Method and Application for Metabolomics. Beijing: Science Press, 2008, 29.许国旺, 路鑫, 杨军, 王媛, 赵欣捷, 石先哲, 田晶, 孔宏伟, 王畅, 袁凯龙, 尹沛源, 汪江山, 马晨菲, 高鹏等编著. 代谢组学—方法与应用. 北京: 科学出版社, 2008, 29.
[5] Fu J, Schoeman JC, Harms AC, van Wietmarschen HA, Vreeken RJ, Berger R, Cuppen BV, Lafeber FP, van der Greef J, Hankemeier T. Metabolomics profiling of the free and total oxidised lipids in urine by LC-MS/MS: application in patients with rheumatoid arthritis., 2016, 408(23): 6307–6319.
[6] Schoeman JC, Harms AC, van Weeghel M, Berger R, Vreeken RJ, Hankemeier T. Development and application of a UHPLC–MS/MS metabolomics based comprehensive systemic and tissue-specific screening method for inflammatory, oxidative and nitrosative stress., 2018, 410(10): 2551–2568.
[7] Ory P, Bonnet A, Mondeguer F, Breitwieser M, Dubillot E, Graber M. Metabolomics based on UHPLC-QToF- and APGC-QToF-MS reveals metabolic pathways reprogramming in response to tidal cycles in the sublittoral speciesexposed to aerial emergence., 2019, 29: 74–85.
[8] Trammell SA, Brenner C. Targeted, LCMS-based metabolomics for quantitative measurement of NAD+metabolites., 2013, 4(5): e201301012.
[9] Ichihashi Y, Kusano M, Kobayashi M, Suetsugu K, Yoshida S, Wakatake T, Kumaishi K, Shibata A, Saito K, Shirasu K. Transcriptomic and metabolomic reprogramming from roots to haustoria in the parasitic plant,., 2018, 59(4): 724–733.
[10] Fitian AI, Nelson DR, Liu C, Xu Y, Ararat M, Cabrera R. Integrated metabolomic profiling of hepatocellular carcinoma in hepatitis C cirrhosis through GC/MS and UPLC/MS- MS., 2014, 34(9): 1428–1444.
[11] Farag MA, Gad HA, Heiss AG, Wessjohann LA. Metabolomics driven analysis of six nigella species seeds via UPLC-qTOF-MS and GC–MS coupled to chemometrics., 2014, 151: 333–342.
[12] Nam KH, Shin HJ, Pack IS, Park JH, Kim HB, Kim CG. Metabolomic changes in grains of well-watered and drought-stressed transgenic rice., 2015, 96(3): 807–814.
[13] Han S, Micallef SA. Environmental metabolomics of the tomato plant surface provides insights oncolonization., 2016, 82(10): 3131–3142.
[14] Zhao LJ, Huang Y, Hu J, Zhou H, Adeleye A, Keller AA.1H NMR and GC-MS based metabolomics reveal defense and detoxification mechanism of cucumber plant under nano- Cu stress., 2016, 50(4): 2000–2010.
[15] Warth B, Parich A, Bueschl C, Schoefbeck D, Neumann NK, Kluger B, Schuster K, Krska R, Adam G, Lemmens M, Schuhmacher R. GC–MS based targeted metabolic profiling identifies changes in the wheat metabolome following deoxynivalenol treatment., 2015, 11(3): 722–738.
[16] Gao XX, Locke S, Zhang JZ, Joshi J, Wang-Pruski GF. Metabolomics profile of potato tubers after phosphite treatment., 2018, 9(4): 845–864.
[17] Valette M, Rey M, Gerin F, Comte G, Wisniewski-Dyé F. A common metabolomic signature is observed upon inoculation of rice roots with various rhizobacteria., 2019, DOI: 10.1111/jipb.12810.
[18] Xiong Q, Cao C, Shen T, Zhong L, He H, Chen X. Comprehensive metabolomic and proteomic analysis in biochemical metabolic pathways of rice spikes under drought and submergence stress., 2019, 1867(3): 237–247.
[19] Roldan MVG, Engel B, de Vos RCH, Vereijken P, Astola L, Groenenboom M, van de Geest H, Bovy A, Molenaar J, van Eeuwijk F, Hall RD. Metabolomics reveals organ- specific metabolic rearrangements during early tomato seedling development., 2014, 10(5): 958– 974.
[20] Feng YC, Fu TX, Zhang LY, Wang CY, Zhang DJ. Research on differential metabolites in distinction of rice (L.) origin based on GC-MS., 2019, https://doi.org/10.1155/2019/1614504.
[21] do Nascimento TP, Santos MCB, da Silva Lima LRS, Nascimento FR, Cameron LC, Ferreira MSL. Dataset on phenolic profile of seven wheat genotypes along maturation., 2018, 21: 284–288.
[22] Tzin V, Pozo NF, Richter A, Schmelz EA, Schoettner M, Schäfer M, Ahern KR, Meihls LN, Kaur H, Huffaker A, Mori N, Degenhardt J, Mueller LA, Jander G. Dynamic maize responses to aphid feeding are revealed by a time series of transcriptomic and metabolomic assays., 2015, 169(3): 1727–1743.
[23] Tan H, Yang X, Zhang X, Zheng X, Qu C, Mu J, Fu FY, Li J, Guan R, Zhang H, Wang G, Zuo J. Enhanced seed oil production in canola by conditional expression ofandin developing seeds., 2011, 156(3): 1577–1588.
[24] Yang X, Nian J, Xie Q, Feng J, Zhang F, Jing H, Zhang J, Dong G, Liang Y, Peng J, Wang G, Qian Q, Zuo J. Rice ferredoxin-dependent glutamate synthase regulates nitrogen– carbon metabolomes and is genetically differentiated betweenandsubspecies., 2016, 9(11): 1520–1534.
[25] Xu F, Fang J, Ou SJ, Gao S, Zhang F, Du L, Xiao Y, Wang H, Sun X, Chu J, Wang G, Chu C. Variations in CYP78A13 coding region influence grain size and yield in rice., 2015, 38(4): 800–811.
[26] Ding L, Yang X, Tian H, Liang J, Zhang F, Wang G, Wang Y, Ding M, Shui G, Huang X. Seipin regulates lipid homeostasis by ensuring calcium‐dependent mitochondrial metabolism., 2018, 37(17): e97572.
[27] Liu X Li F, Tang J, Wang W, Zhang F, Wang G, Chu J, Yan C, Wang T, Chu C, Li C. Activation of the jasmonic acid pathway by depletion of the hydroperoxide lyase OsHPL3 reveals crosstalk between the HPL and AOS branches of the oxylipin pathway in rice., 2012, 7: e50089.
[28] Wei G, Tian P, Zhang F, Qin H, Miao H, Chen Q, Hu Z, Cao L, Wang M, Gu X, Huang S, Chen MS, Wang G. Integrative analyses of nontargeted volatile profiling and transcriptome data provide molecular insight into VOC diversity in cucumber plants ()., 2016, 172(1): 603–618.
[29] Wang C, Chen Q, Fan D, Li J, Wang G, Zhang P. Structural analyses of short-chain prenyltransferases identify an evolutionarily conserved GFPPS clade inplants., 2015, 9(2): 195–204.
[30] Chen Q, Fan D, Wang G. Heteromeric geranyl(geranyl) diphosphate synthase is involved in monoterpene biosynthesis inflowers., 2015, 8(9): 1434–1437.
[31] Chen X, Zhang H, Sun H, Luo H, Zhao L, Dong Z, Yan S, Zhao C, Liu R, Xu C, Li S, Chen H, Jin W. IRREGULAR POLLEN EXINE1 is a novel factor in anther cuticle and pollen exine formation., 2017, 173(1): 307–325.
[32] An X, Dong Z, Tian Y, Xie K, Wu S, Zhu T, Zhang D, Niu C, Ma B, Hou Q, Bao J, Zhang S, Li Z, Wang Y, Yan T, Sun X, Zhang Y, Wan X. ZmMs30 encoding a novel GDSL lipase is essential for male fertility and valuable for hybrid breeding in maize, 2019, 12(3): 343–359.
[33] Fang C, Ma Y, Wu S, Liu Z, Wang Z, Yang R, Hu G, Zhou Z, Yu H, Zhang M, Pan Y, Zhou G, Ren H, Du W, Yan H, Wang Y, Han D, Shen Y, Liu S, Liu T, Zhang J, Qin H, Yuan J, Yuan X, Kong F, Liu B, Li J, Zhang Z, Wang G, Zhu B, Tian Z. Genome-wide association studies dissect the genetic networks underlying agronomical traits in soybean., 2017, 18(1): 161.
[34] Li Q, Fang C, Duan Z, Liu Y, Qin H, Zhang J, Sun P, Li W, Wang G, Tian Z. Functional conservation and divergence of GmCHLI genes in polyploid soybean., 2016, 88(4): 584–596.
[35] Wang B, Chu J, Yu T, Xu Q, Sun X, Yuan J, Xiong G, Wang G, Wang Y, Li J. Tryptophan-independent auxin biosynthesis contributes to early embryogenesis in., 2015, 112(15): 4821–4826.
[36] Song Q, Li Q, Liu Y, Zhang F, Ma B, Zhang W, Man W, Du W, Wang G, Chen S, Zhang J. Soybean GmbZIP123 gene enhances lipid content in the seeds of transgenicplants., 2013, 64(14): 4329–4341.
[37] Yu S, Cao L, Zhou C, Zhang T, Lian H, Sun Y, Wu J, Huang J, Wang G, Wang J. Sugar is an endogenous cue for juvenile-to-adult phase transition in plants., 2013, 2: e00269.
[38] Yu H, Zhang F, Wang G, Liu Y, Liu D. Partial deficiency of isoleucine impairs root development and alters transcript levels of the genes involved in branched-chain amino acid and glucosinolate metabolism in., 2013, 64(2): 599–612.
[39] Yu H, Du X, Zhang F, Hu Y, Liu S, Jiang X, Wang G, Liu D. A mutation in the E2 subunit of the mitochondrial pyruvate dehydrogenase complex inreduces plant organ size and enhances the accumulation of amino acids and intermediate products of the TCA cycle., 2012, 236(2): 387–399.
[40] Li C, Wang Y, Liu L, Hu Y, Zhang F, Mergen S, Wang G, Schläppi MR, Chu C. A rice plastidial nucleotide sugar epimerase is involved in galactolipid biosynthesis and improves photosynthetic efficiency., 2011, 7(7): e1002196.
[41] Li W, Zhang H, Li X, Zhang F, Liu C, Du Y, Gao X, Zhang Z, Zhang X, Hou Z, Zhou H, Sheng X, Wang G, Guo Y. Intergrative metabolomic and transcriptomic analyses unveil nutrient remobilization events in leaf senescence of tobacco., 2017, 7(1): 12126.
[42] McCloskey D, Utrilla J, Naviaux RK, Palsson BO, Feist AM. Fast Swinnex filtration (FSF): a fast and robust sampling and extraction method suitable for metabolomics analysis of cultures grown in complex media., 2015, 11(1): 198–209.
[43] Zimmermann M, Thormann V, Sauer U, Zamboni N. Nontargeted profiling of coenzyme a thioesters in biological samples by tandem mass spectrometry., 2013, 85(17): 8284−8290.
[44] Snyder NW, Basu SS, Zhou Z, Worth AJ, Blair IA. Stable isotope dilution liquid chromatography/mass spectrometry analysis of cellular and tissue medium- and long-chain acyl-coenzyme a thioesters., 2014, 28(16): 1840–1848.
[45] Zheng X, Deng L, Baker ES, Ibrahim YM, Petyuk VA, Smith RD. Distinguishing d- and l-aspartic and isoaspartic acids in amyloid β peptides with ultrahigh resolution ion mobility spectrometry., 2017, 53(56): 7913–7916.
[46] Zheng X, Zhang X, Schocker NS, Renslow RS, Orton DJ, Khamsi J, Ashmus RA, Almeida IC, Tang K, Costello CE, Smith RD, Michael K, Baker ES. Enhancing glycan isomer separations with metal ions and positive and negative polarity ion mobility spectrometry-mass spectrometry analyses., 2017, 409(2): 467–476.
[47] Barroso A, Giménez E, Konijnenberg A, Sancho J, Sanz- Nebot V, Sobott F. Evaluation of ion mobility for the separation of glycoconjugate isomers due to different types of sialic acid linkage, at the intact glycoprotein, glycopeptide and glycan level., 2017, 173: 22–31.
[48] Hinz C, Liggi S, Griffin JL. The potential of ion mobility mass spectrometry for high-throughput and high-resolution lipidomics., 2017, 42: 42–50.
[49] Li W, Zhang F, Chang Y, Zhao T, Schranz ME, Wang G. Nicotinate-Glucosylation is an evolutionarily metabolic trait important for seed germination under stress conditionsin., 2015, 27(7): 1907–1924.
[50] Li W, Zhang F, Wu R, Jia L, Li G, Guo Y, Liu C, Wang G. A novel-methyltransferase inappears to feed a conserved pathway for nicotinate detoxification among land plants and is associated with lignin biosynthesis., 2017, 174(3): 1492–1504.
[51] Wu R, Zhang F, Liu L, Li W, Pichersky E, Wang G. MeNA, controlled by reversible methylation of nicotinate, is a NAD precursor that undergoes long-distance transport in., 2018, 11(10): 1264–1277.
[52] Zhou J, Wang X, Wang M, Chang Y, Zhang F, Ban Z, Tang R, Gan Q, Wu S, Guo Y, Zhang Q, Wang F, Zhao L, Jing Y, Qian W, Wang G, Guo W, Yang C. The lysine catabolite saccharopine impairs development by disrupting mitochondrial homeostasis., 2019, 218(2): 580–597.
[53] Xu H, Zhang F, Liu B, Huhman D, Sumner LW, Dixon R, Wang G. Characterization of the formation of branched short-chain fatty acid: CoAs for bitter acid biosynthesis in hop glandular trichomes., 2013, 6(4): 1301– 1317.
[54] Li H, Ban Z, Qin H, Ma L, King AJ, Wang G. A heteromeric membrane-bound prenyltransferase complex from hop catalyzes three sequential aromatic prenylations in the bitter acid pathway., 2015, 167(3): 650–659.
[55] Ban Z, Qin H, Mitchell AJ, Liu B, Zhang F, Weng JK, Dixon RA, Wang G. Noncatalytic chalcone isomerase-fold proteins inare auxiliary components in prenylated flavonoid biosynthesis., 2018, 115(22): E5223–E5232.
[56] He H, Chen L, Qin L, Liu Y, Wang X. Mass spectrometry imaging for in situ analysis of endogenous molecules in plants., 2017, 47(10): 1043–1064.何慧欣, 陈路路, 秦亮, 刘亚琴, 王晓东. 质谱成像及其在植物内源性分子原位表征中的应用进展. 中国科学: 生命科学, 2017, 47(10), 1043–1064.
Current metabolomics platforms: technical composition and applications
Fengxia Zhang, Guodong Wang
Metabolomics (defined as comprehensive small molecule chemical analysis), together with genomics, transcriptomics, proteomics and phenomics, now plays a fundamental role in system biological studies. Chromatography- mass spectrometry machines, which have the characteristics of high resolution and high sensitivity, are widely used for metabolomics analysis, both qualitatively and quantitatively. With the fast development of the chromatography-mass spectrometry technology, metabolomics analysis has been successfully applied in various biological research fields. Here, we introduce the different chromatography-mass spectrum machines used for metabolomics analysis and their applications to various biological issues by mainly using the metabolomics platform in Institute of Genetics and Developmental Biology as a casestudy.
small molecule chemicals; metabolomics platform; chromatography-mass spectrometry; system biology
2019-04-30;
2019-06-30
植物基因组学国家重点实验室(编号:SKLPG2016A-13,SKLPG2016B-13)资助[Supported by the State Key Laboratory of Plant Genomics of China (Nos. SKLPG2016A-13, SKLPG2016B-13)]
张凤霞,博士,工程师,研究方向:代谢组学分析方法及应用。E-mail: zhangfx@genetics.ac.cn
王国栋,博士,研究员,研究方向:植物功能代谢组学。E-mail: gdwang@genetics.ac.cn
10.16288/j.yczz.19-122
2019/8/12 14:49:56
URI: http://kns.cnki.net/kcms/detail/11.1913.R.20190812.1449.003.html
(责任编委: 杨永华)
