链霉菌沉默生物合成基因簇激活策略的研究进展
2019-08-28吴旭日陈依军
戴 岩,吴旭日,陈依军
(中国药科大学生命科学与技术学院化学生物学研究室,南京211198)
链霉菌的次级代谢产物或其衍生物一直都是药物发现和开发的重要来源,特别是抗感染药物和抗肿瘤药物[1-3]。然而,自20世纪50年代至60年代次级代谢产物发现的热潮后,基于菌株发酵和产物分离鉴定的传统方法难以再挖掘出具有新骨架或新活性单元的次级代谢产物。近年来。随着DNA测序技术和生物信息学的蓬勃发展,研究人员意识到链霉菌等微生物中存在大量的新型生物合成基因,次级代谢产物药物的发现也随之进入了第二个黄金时代[4]。
链霉菌次级代谢产物是由成簇存在的基因,即所谓的生物合成基因簇(biosynthetic gene cluster,BGC)编码一系列功能相关性酶或肽链依照一定次序发挥作用后产生的天然化合物。大量已发表的链霉菌基因组信息表明,次级代谢产物BGCs的数量远超已发现的次级代谢物总量,意味着链霉菌存在巨大的生物合成潜力。以antiSMASH为代表的生物信息学软件可以批量预测链霉菌基因组中的BGCs[5],但迄今依然只有极少数的 BGCs及其相应的代谢产物被识别鉴定,这是因为次级代谢产物的合成受到严格调控,通常需要在特定的环境下才能激活相关的BGCs。由于常规实验条件的限制,这些表达量极低甚至完全不表达的BGCs被定义为沉默BGCs。因此通过激活链霉菌沉默BGCs发掘新次级代谢物对突破新药研发的瓶颈具有重要意义。
如图1所示,目前链霉菌沉默生物合成基因簇的研究流程为:(1)链霉菌基因组的提取及测序;(2)生物信息学工具预测和分析BGCs;(3)利用合成生物学等手段激活沉默BGCs;(4)新次级代谢产物的检测;(5)新产物的分离鉴定。本文首先概述了最新的生物信息学工具,根据预测原理分为规则依赖性和规则无关性两类[6];之后重点讨论了在天然宿主和异源宿主中激活链霉菌沉默BGCs的有效策略,为基于链霉菌次级代谢产物的药物开发提供有效的方法学参考。
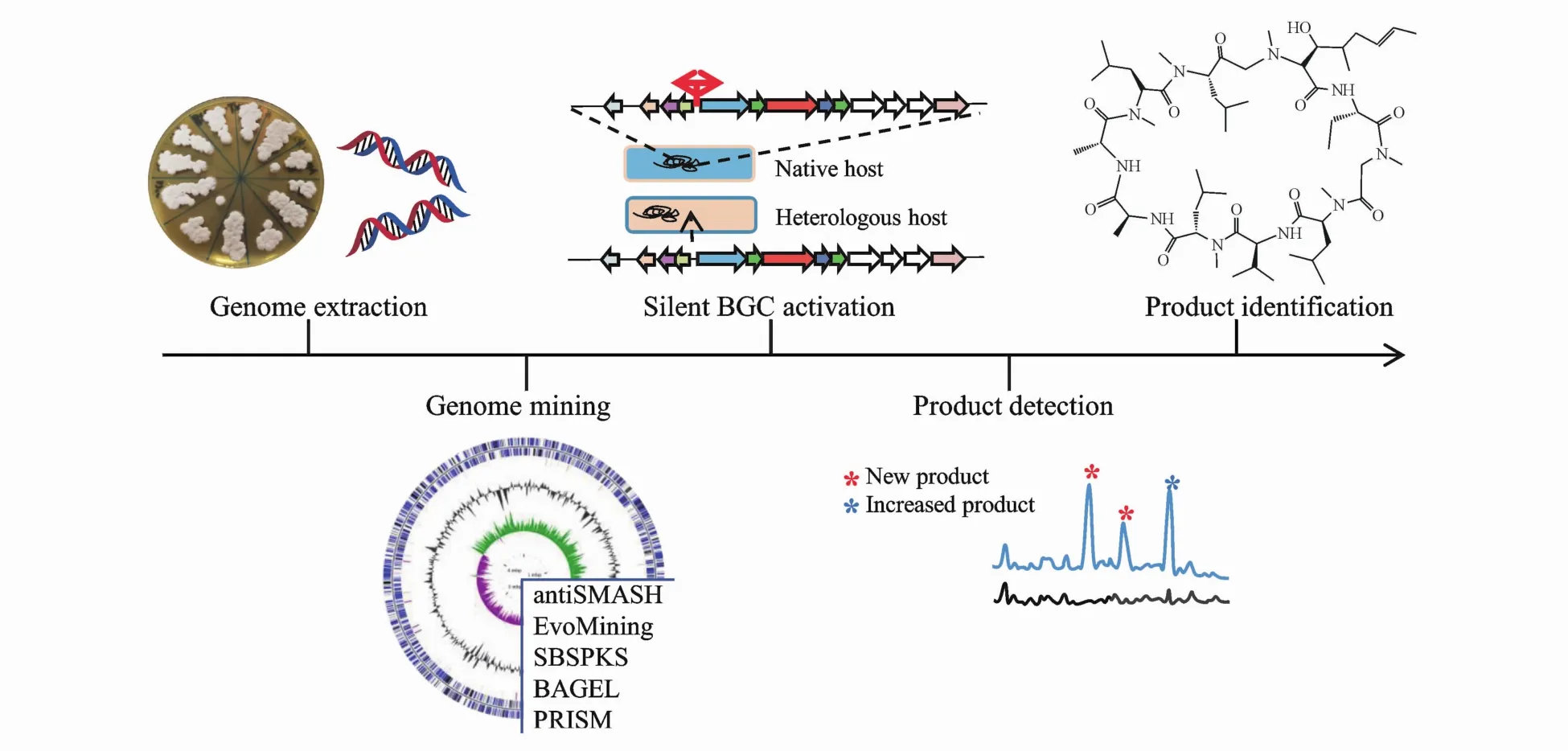
Figure 1 Workflow for a secondarymetabolite discovery strategyBGC:Biosynthetic gene cluster
1 生物合成基因簇的分析工具
挖掘链霉菌基因组的生物信息学工具根据运行原则分为两类:规则依赖性工具和规则无关性工具(表1)。前者利用预定义的规则集通过已知BGCs特征基因的识别精确预测BGCs的类型,规则无关性工具则是运用机器学习或系统进化树分析等实现BGCs的预测。
1.1 规则依赖性工具
根据BGCs编码的蛋白种类以及合成的终产物类型,用于挖掘链霉菌基因组的生物信息学工具主要分为两组。第1组是针对非核糖体肽合酶/聚酮合酶(NRPS/PKS)的工具,作为非核糖体肽和聚酮的合成机器,多模块酶复合体NRPS/PKS利用不同模块激活不同的氨基酸或丙二酰,顺序缩合后进一步修饰实现终产物的结构多样性(图2)。这些工具使用预定义的规则集建立算法识别与生物合成途径相关的特征基因,例如SBSPKS,PKS/NRPS利用腺苷化结构域的保守性比较分析预测新的NRPS基因簇[7],NRPSpredictor[8]进一步列出了该结构域所有活性位点可能结合的底物。SBSPKS第2版[9]还添加了NRPS的缩合,差向异构化,环化结构域预测;在线工具 NaPDoS[10]通过识别NRPS的缩合域,PKS的聚酮结构域及其亚型来预测NRPS/PKS基因簇。antiSMASH是目前链霉菌基因组挖掘中使用率最高的工具,不仅包括了不同特征基因的比较算法,更是建立了基于同源性的代谢模型管路以及综合的次级代谢基因簇数据库[11-12]。但是,基于预测的 BGCs推测天然产物的结构仍然是一大难点,PRISM[13]利用生物合成逻辑编写算法实现了PKS结构的初步预测。
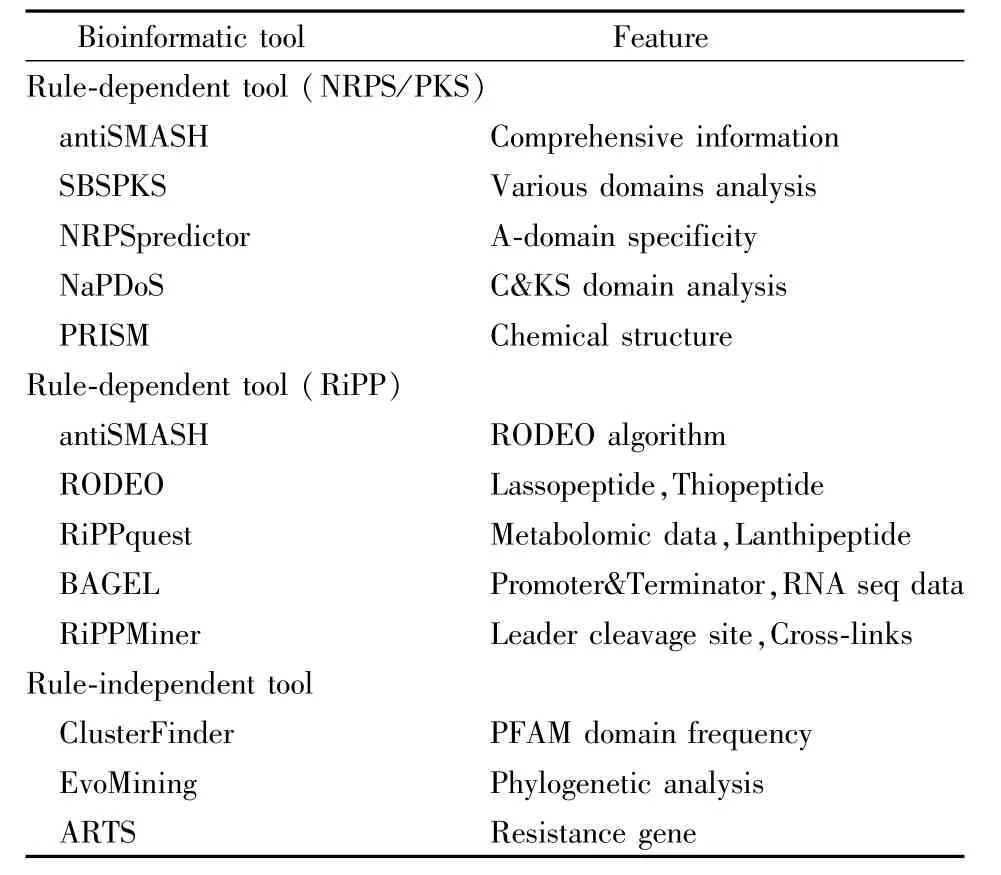
Table 1 Bioinformatic tools for BGCs prediction
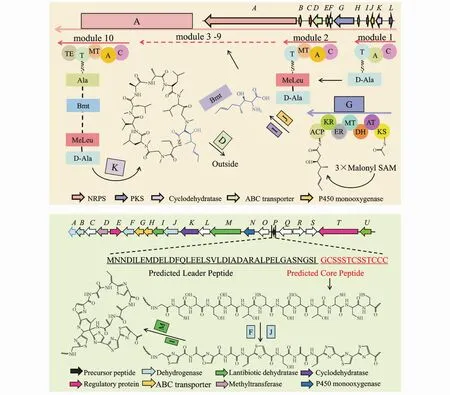
Figure 2 Schematic diagrams for NRPS/PKS(above)and RiPP(below)pathways
第2组是挖掘核糖体合成翻译后修饰肽(RiPP)(图 2)的工具,antiSMASH 4.0[11]利用启发式算法计算得分,联合SVM以及基序分析可以预测15种不同的RiPP BGCs,相比于剖面隐马尔科夫模型,这些从 RODEO[14]借鉴的算法更适用于RiPPs挖掘。RODEO根据现已公开的基因组信息先后预测了1 300多种环肽类,508种硫肽类BGCs[14-15];RiPPquest[16]将 RiPP BGCs的基因型与代谢组学数据集对应的化学表型联系起来,成功地在Streptomyces viridochromogenes中发现了新的羊毛硫肽类化合物。作为首个挖掘RiPPs的网页工具,BAGEL第4版[17]还整合了 RNA表达数据以及启动子和终止子的预测;RiPPMiner[18]也因为前导肽分割位点及翻译后修饰交联的预测功能广受关注。
1.2 规则无关性工具
大多数基因挖掘平台都根据已知BGCs中参与关键生物合成步骤的酶的基因作为锚或探针来筛选预测出已知类型的BGCs,虽然这种规则依赖性工具预测敏感度,阳性率和置信度高,但难以识别使用不同酶机制的BGCs。
为了扩大基因挖掘的范围,ClusterFinder[19]使用隐马尔科夫模型在全基因组范围内计算已知BGCs中出现的PFAM结构域的频率来预测BGCs,即使在完全没有特征基因的情况下富含这些PFAM结构域的基因组区域极有可能是新的BGCs。但根源上PFAM结构域频率的计算依然基于已知的 BGCs,而 EvoMining[20]的开发给非典型BGCs的预测提供了全新的视角,它以基础代谢酶为起点通过建立系统发育树检测已知BGCs中关键酶的旁系同源,假想它们有潜力作用于次级代谢物合成,基于这一理论Cruz-Morales等[20]发现了两种以前从未报道过的酶,其中一个参与一种新的砷有机化合物的代谢合成。此外,基于抗性基因开发的ARTS[21]也是挖掘新型BGCs的有效工具。但这些规则无关性的生物信息学手段通常假阳性率高,最好能与规则依赖性的工具联合使用,从而实现高质量的基因组挖掘。次级代谢产物生物信息门户(SMBP)的网站[6]为目前流行的大多数生物信息学工具提供了一站式目录和链接,可以快速访问这些挖掘工具和数据库。
2 沉默生物合成基因簇在天然宿主中的激活
沉默BGCs激活的策略主要分为两种:其一,在天然宿主菌中解除沉默BGCs的抑制因素;其二,避开现有调控体系,直接将BGCs异源表达或重构BGCs的控制元件后转移到异源宿主中。
2.1 OSMAC及其衍生策略
通过调整链霉菌的培养参数,譬如培养基组成、温度、pH、通气甚至容器类型等,诱导沉默BGCs的表达是在单菌株水平下改变整个次级代谢产物谱最常见的策略,Bode等[22]将其定义为OSMAC(one strain many compounds)。这种方法相对经济简单但结果多样性,特别适用于基因组信息不全面或存在遗传隔离的链霉菌菌种。Rateb等[23]从干旱沙漠土壤中分离一株链霉菌后利用OSMAC摸索出8种培养基得到了一系列多样的次级代谢产物。
低浓度抗生素、信号分子和组蛋白去乙酰化酶抑制剂等诱导子的添加[24]和共培养策略也是从OSMAC衍生出来的,后者利用链霉菌与其他一种或多种微生物共同培养,基于竞争原则模拟动态自然环境来诱导新活性物质的产生,Streptomyces endus与Tsukamurella pulmoni共培养后产生了新的次级代谢物 alchivemycin A[25]。
上述方法引起的代谢特征变化是全局性的,因此后续比较分析代谢谱时难度大大增加,而且该策略相较于基因工程手段盲目性强,目的性差。基因组指导OSMAC能适当缩减培养参数范围,通过基因组分析能初步解读出典型的生物合成组装线及它们的产物类型,根据产物类型提供足够的前体,关键元素等可大大提高沉默基因簇表达概率。
2.2 调控体系的编辑
2.2.1 过表达正调控因子 链霉菌中有35~40个调控蛋白家族,按功能划分为正调控因子和负调控因子两类。其中,过表达正调控基因如AraC,LuxR,SARP等是激活沉默基因簇的有效方法,例如Du等[26]构建了7株重组链霉菌菌株,重组载体上分别含有一种SARP基因及组成型启动子,成功挖掘出一种新的Ⅱ型聚酮ishigamide;属于LuxR家族的转录激活基因vemR组成型表达时,Streptomyces venezuelae产生了一个新的二芳基聚酮化合物venemycin[27];连续过表达AraC家族的3个调控基因sgcR1,sgcR2和sgcR3产生了 enediyne C-1027[28]。由于过表达质粒构建简单,转入目的链霉菌的效率较高,这种方法可能会进一步发展用于单菌株水平高通量激活沉默基因簇。
2.2.2 解除或下调负调控因素 敲除负调控基因是挖掘新次级代谢产物的经典手段之一,在模式链霉菌中,传统的基因敲除策略是自杀型或温敏型质粒介导的同源双交换重组,但往往耗时耗力。为了改善基因编辑可行性,CRISPR-Cas9技术被高效精确地运用于链霉菌靶基因的敲除,当没有同源臂指导同源定向修复(HDR)(图 3-A)时,Cas9通过sgRNA介导的DNA双链断裂(DSB)可以进行非同源末端连接(NHEJ)修复,这会导致靶序列出现不同大小的片段敲除。通过共表达LigD连接酶优化链霉菌NHEJ途径(图3-B),靶基因的缺失,替换或插入降至3个碱基以内,因此靶基因发生功能变化或丧失[29]。虽然此法基因编辑位点比较准确,也无需构建同源臂,但结果序列过于随机,无法保证靶基因功能的完全丧失。而同时提供用于HDR的同源臂时,则能实现靶基因的精准敲除[30],基于这一原理Tong等[31]还开发了一个高效编辑放线菌基因组的CRISPR-Cas9工具盒。
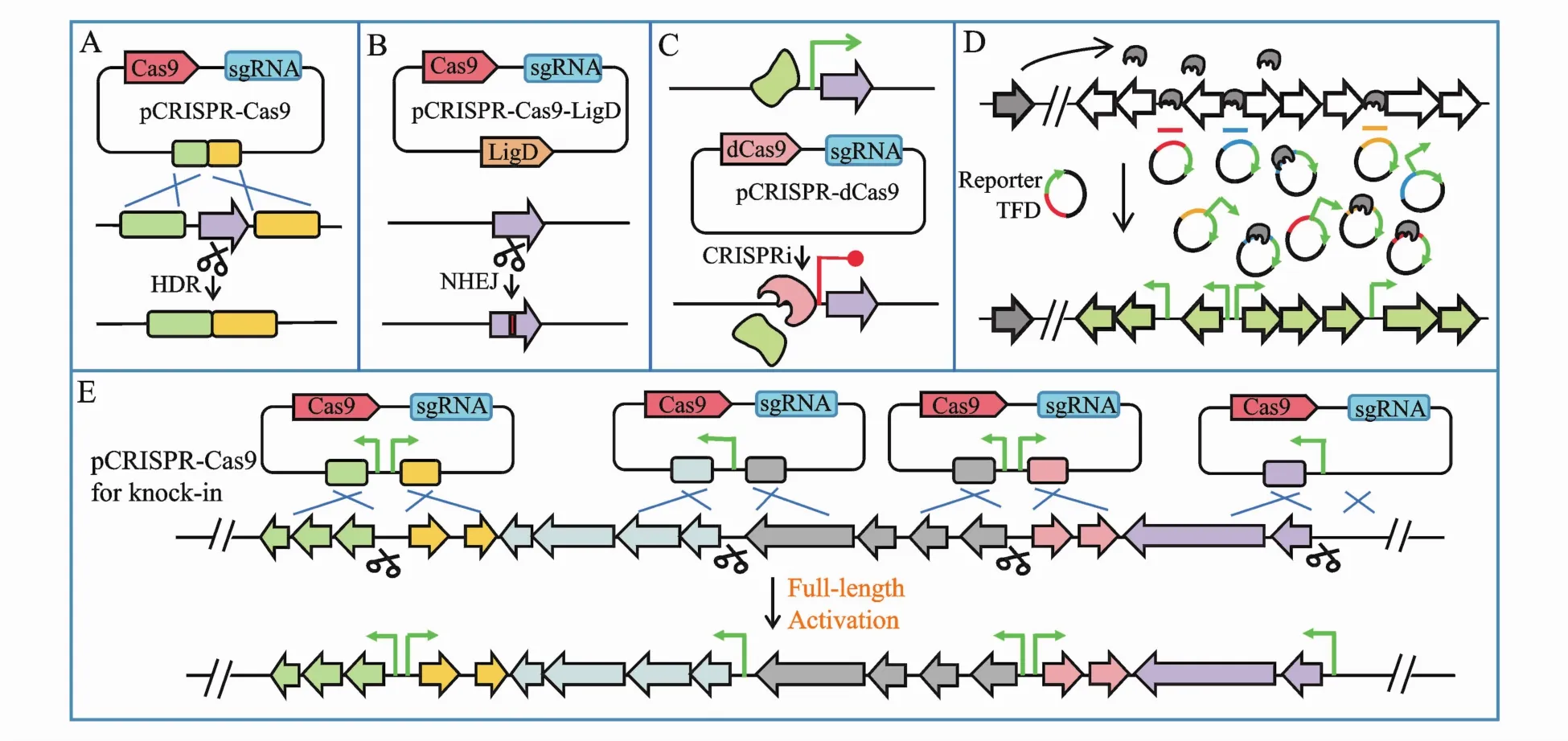
Figure3 Approaches to activate silent BGCs in native hostsA:CRISPR-Cas9-HDR;B:Reconstituted CRISPR-Cas9-NHEJ;C:CRISPRi;D:TFD;E:CRIPR-Cas9-guided knock in
此外,Cas9催化活性失活的突变体(dCas9)介导的序列特异性干扰基因表达(CRISPRi)很有希望用于高通量抑制链霉菌中的负调控基因表达从而激活一系列沉默BGCs[32](图3-C)。针对负调控因子基因的启动子区域设计sgRNAs,CRISPR-dCas9复合体因此能与转录机器竞争性结合这段非编码区,使负调控因子的转录大大降低甚至无法转录。但这种方法是可逆的,当撤去温度压力时,CRISPR-dCas9会渐渐丢失,改造菌株回复到野生型。
转录因子诱捕(transcription factor decoy,TFD)这一概念最初由Mcarthur等[33]提出,他们构建了一系列哑铃状寡核苷酸模拟基因组调控元件,竞争性结合抑制型转录因子从而实现基因簇的正常转录并产生相应的代谢物。Wang等[34]开发的TFD则是将基因组调控元件序列与报告基因一同构建在高拷贝载体pKC1139上,接合转移后提供温度和抗性双重压力使得重组pKC1139稳定游离在链霉菌体内(图3-D)。该方法利用TFD序列竞争性结合抑制型转录因子成功激活了8个沉默的NRPS/PKS基因簇并挖掘出一个新的■唑类化合物。
2.3 强启动子的引入
除了编辑调控体系,另一种激活沉默BGCs的方法是在靶BGC上游敲入强启动子,从而起始生物合成基因的转录并合成相应的代谢产物。相比于传统的基因同源重组双交换,CRISPR-Cas9的介入显著提高了启动子的敲入效率。基于HDR原理,Zhang等[35]利用 CRISPR-Cas9成功在5种链霉菌中引入kasO*p替换天然启动子,成功激活了多个沉默的BGCs,并诱导了相应次级代谢物的产生,包括在Streptomyces viridochromogene中产生的一个新型Ⅱ型聚酮。
BGCs中常见多操纵子结构,因此激活完整长度的BGC并产生相应次级代谢产物物需构建一系列敲入质粒替换靶BGC每个操纵子上游的天然启动子(图3-E)。不同的操纵子组合产生不同的中间产物,还可以为生物合成机制的研究提供参考[36]。但如果一个新次级代谢物的合成涉及全局性的转录变化,这种途径特异性敲入启动子的策略则无法适用。
2.4 报告子导向的筛选
上述TFD策略中利用报告子大大简化了后续筛选[34],同样高通量诱导子筛选(high-throughput elicitor screening,HiTES)[37](图 4-A)利用报告子的特性表征靶BGC的表达,可以快速鉴定沉默BGCs是否被激活。针对白色链霉菌中NRPS沉默基因簇sur构建2种报告子质粒,一种使3个串联的eGFP(eGFP×3)定点插入到sur的天然启动子(Psur)下游,另一种将Psur与eGFP×3融合后异位整合到基因组上,利用HiTES发现 ivermectin和etoposide是激活sur基因簇的强诱导子,并成功分离鉴定了14种新次级代谢物。
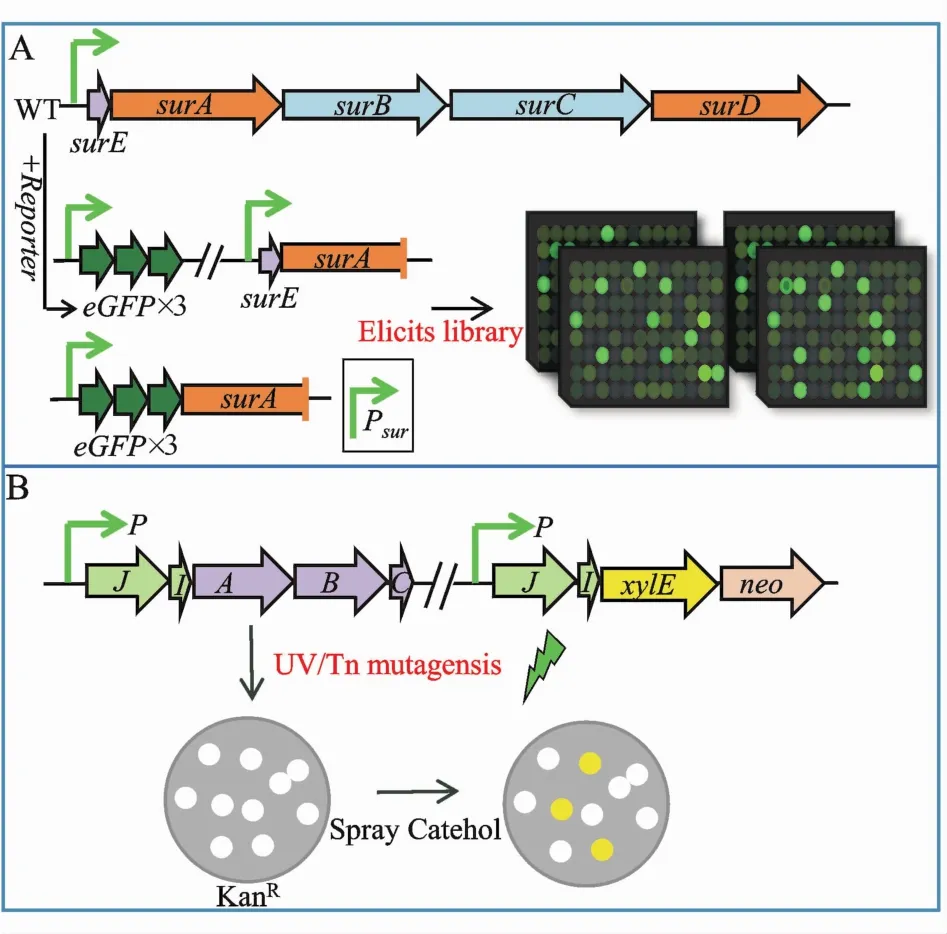
Figure 4 Approaches to activate silent BGCs in native hostsA:HiTES(high-throughput elicitor screening);B:RGMS(reporterguided mutant selection)
报告子导向的突变体筛选(reporter-guided mutant selection,RGMS)(图 4-B)也是一个有效且适用范围广的新策略,采用基因组规模的随机突变建立遗传多样性,然后通过双报告子筛选直接识别靶激活的突变体。Guo等[38]将沉默pga基因簇启动子融合报告基因后异位整合到基因组中,利用RGMS成功在Streptomyces sp.PGA64中激活了pga基因簇并挖掘了两个新蒽醌氨基糖苷类代谢物。此方法中双报告子的特征需区别于链霉菌本身的性质,xylE-neo报告子只适用于原菌株卡那霉素敏感,单克隆显示为非黄色[38]。
3 沉默生物合成基因簇的异源表达
异源宿主相比于天然宿主存在众多优势:培养条件简单,调控体系研究透彻,转录元件容易控制等,直接克隆或者重构BGCs后再异源表达可以避开原宿主中的调控体系,实现沉默BGCs的激活并产生相应的次级代谢产物。
3.1 直接克隆
异源表达最直接的方法是直接克隆靶BGC转入异源宿主中。传统克隆技术依赖文库的构建和筛选不仅耗时耗力,而且单个cosmid或细菌人工染色体无法完整地克隆链霉菌中长达几十甚至几百kb的BGCs。基于DNA重组开发了一些直接克隆大片段BGCs的策略,例如酵母转化偶联重组(transformation-associated recombination,TAR)系统,Red/ET介导的同源重组,Cas9相关染色体片段的捕获(Cas9-assisted targeting of chromosome segments,CATCH),点特异性重组 (site-specific recombination,SSR)等。
酿酒酵母中发现的TAR系统被Kim等[39]用于特异性捕捉微生物的BGC后,渐渐发展成链霉菌大片段BGCs的克隆工具。基因组DNA与过量TAR克隆载体共转化到酵母细胞中,由于克隆载体上提前构建了靶BGC序列两端的同源臂,重组会高频发生,从而形成环状酵母人工染色体分子。这种方法可以成功地从复杂基因组中克隆出长达250 kb的DNA片段,但在酵母转化子中靶BGC的捕捉效率只有0.5%~2%[40],因此阳性克隆极少,筛选过程繁琐耗时。
在不断改造噬菌体重组体系Red和RecET的过程中,Fu等[41]发现全长的 RecE联合 RecT和Redγ能在大肠埃希菌中实现高效的线线同源重组(linear-linear homologous recombination,LLHR)。此法与TAR克隆类似,效率略高于TAR,但克隆的BGC长度一般不超过50 kb。
TAR和Red/ET介导的同源重组因为基因组片段存在非特异性结合,阳性克隆产率极低。而sgRNA指导的Cas9能精确切割靶BGC边界处的DNA序列,对基因组DNA用CRISPR-Cas9预处理可大幅提高同源重组效率(图 5),TAR联合CRISPR-Cas9后阳性克隆产率甚至提高到32%[40];Red/ET介导的同源重组也利用 CRISPRCas9极大改善了靶BGC的捕获效率[42]。相比于TAR和Red/ET介导的体内直接克隆,CATCH[43]结合CRISPR-Cas9和Gibson组装,可在体外捕捉长达100 kb的基因组片段,耗时短且阳性克隆率高,靶BGC长50 kb时阳性克隆率为60%。但CATCH对基因组DNA的纯度和完整性要求较高,需要十分精细的体外操作。
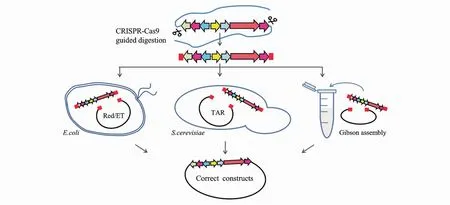
Figure 5 CRISPR-Cas9 assisted direct cloning
基于点特异性重组的直接克隆法也不断被开发(图6),例如丝氨酸整合酶介导的 SSR[44],链霉菌噬菌体φBT1成对的整合位点attB/attP分别通过同源重组单交换到靶BGC边界,然后表达φBT1重组酶切割成对的整合位点导致靶BGC环化至pKC1139上,利用此方法从Streptomyces roseosporus基因组中直接克隆了长分别为45 kb,157 kb的napsamycin和 daptomycin基因簇,克隆效率达80%[44]。Cre/loxP[45]是 酪 氨 酸 整 合 酶 介 导 的SSR,与φBT1相似,loxP位点首先被整合到靶BGC边界,Cre重组酶表达识别 loxP位点进而环化靶BGC。
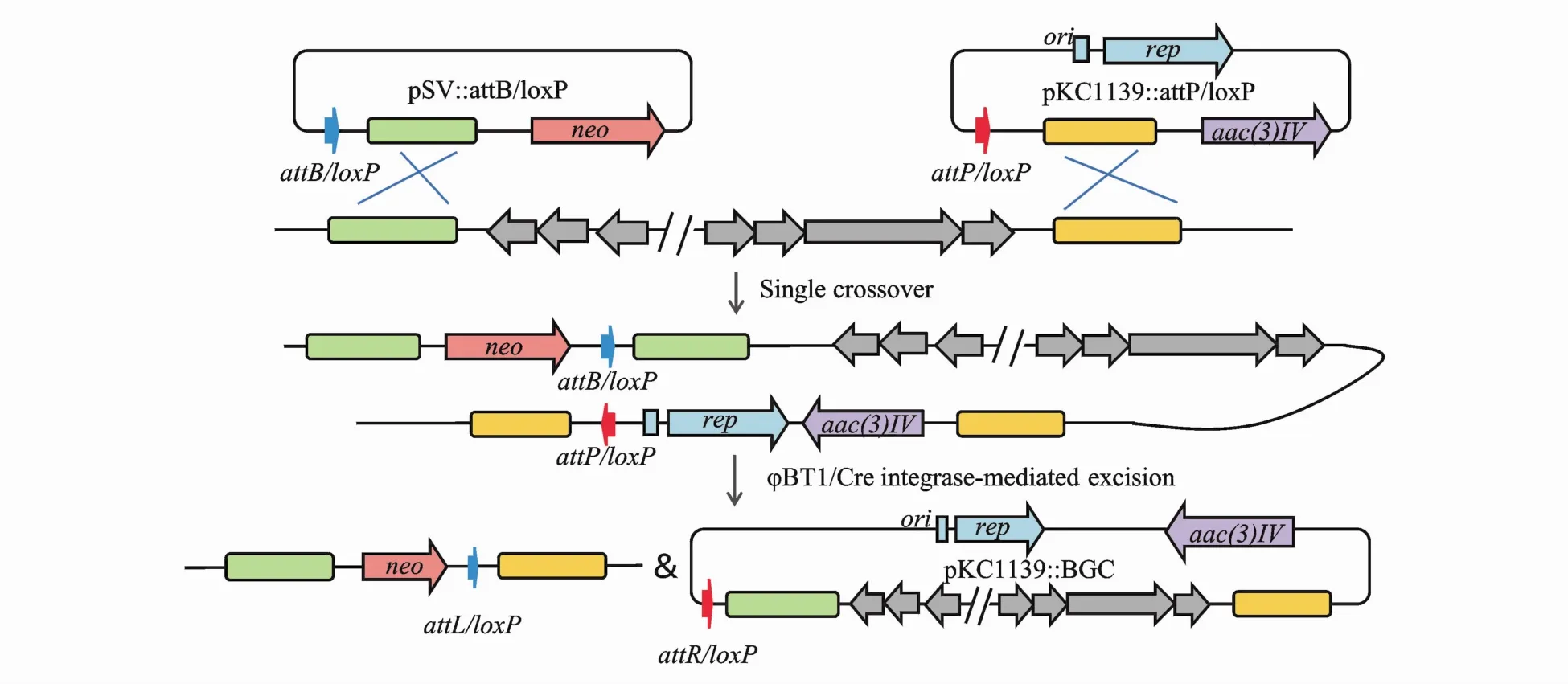
Figure 6 Site-specific recombination for direct cloning
3.2 重构生物合成基因簇
沉默BGCs直接克隆后接合转移到异源宿主中依然不能激活时,往往通过改变原有的调控元件重构BGCs后实现异源表达。例如,taromycin基因簇直接克隆到异源菌中不表达,但敲除负调控基因后可被激活[46]。最常见的重构策略是将天然启动子替换成组成型启动子,mCRISTAR(multiplexed-CRISPR-TAR)[47]是目前最有效的手段(图 7),CRISPR-Cas9在BGC各启动子处介导双链断裂形成线状DNA片段,而组成型启动子含有BGC特异性的同源臂,在酵母细胞中利用TAR与这些片段重组。这套多启动子插入体系为避免启动子间的同源重组,需对启动子进行正交设计。基于这一方法,tetarimycin和lazarimide和芳香聚酮AB1210基因簇重构后在异源宿主白色链霉菌中被激活[47]。另一个重构BGCs的方法是通过DNA组装完成BGC的全部构建[48],包括生物合成基因,组成型启动子等各种元素都从线性DNA片段出发。
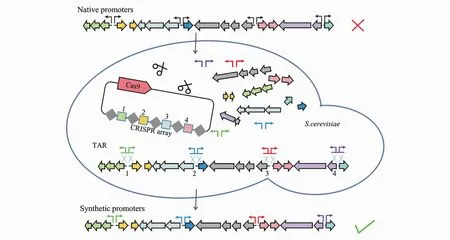
Figure 7 Multiplexed-CRISPR-TAR for BGCs refactoring
4 展 望
本文阐述的链霉菌沉默BGCs的激活策略虽已成功应用于多种次级代谢产物的发掘,但仍存在较大的完善空间:(1)现有的生物信息学工具对全新模式的生物合成途径以及次级代谢产物结构的预测置信度低,有待算法的进一步突破;(2)在天然宿主中利用基因操作激活沉默BGCs的策略不适用于培养困难或遗传隔离的菌种,所以传统OSMAC策略仍需进一步创新发展;(3)基于异源表达的沉默BGCs激活策略虽避开了天然宿主的限制因素,给基因编辑提供了较高的自由度,但简单的异源宿主环境通常无法提供次级代谢产物生物合成的调控网络和代谢需求,因此改造调控系统和代谢途径构建兼容性好的通用底盘菌株迫在眉睫;(4)TFD,HiETS及RGSGs策略实现了单菌株水平批量挖掘新次级代谢产物的目的,但链霉菌的种间差异性与基因编辑的可行性限制了通量的进一步提高。然而,随着合成生物学技术和生物信息学算法的不断升级改进,链霉菌沉默BGCs的激活将逐渐实现高通量挖掘次级代谢产物的目标,从而为药物先导化合物的开发提供丰富的结构基础。
