2017-2018年吉林部分地区规模化猪场猪细环病毒的遗传变异分析
2019-08-22刘东旭李国江苗丽娟沙万里尹柏双
刘东旭,李国江,苗丽娟,沙万里,尹柏双∗
(1.吉林农业科技学院,吉林吉林132101;2.预防兽医学吉林省重点实验室,吉林吉林132101)
猪细环病毒(Torque teno sus virus,TTSuV)是一种无囊膜、环状、单股负链DNA病毒,病毒粒子直径约 30 ~32 nm[1]。 人细环病毒(Torque teno virus,TTV)是由日本学者Nishizawa等在1997年使用代表性差异分析技术发现的一种新病毒。1999年,美国学者首次从猪血清中分离到该病毒后,已在中国、加拿大、日本等国家发现TTSuVs的流行[2]。 根据核苷酸序列差异,TTSuVs可分为TTSuVl和 TTSuV2两种基因型。其中 TTSuVl有4 种亚型,即 TTSuVla、TTSuVlb、TTSuVlc和 TSuVld。TTSuV2 有 四 种 亚 型,即 TTSuV2a、TTSuV2b、TTSuV2c和TTSuV2d[3]。该病毒编码区中包含3个开放阅读框(ORF1-ORF3),1个 TATA盒和1个PolyA。其中ORF1基因是编码衣壳蛋白和复制相关结构域的基因,不同毒株之间差异较大,可用于TTSuVs的分型鉴定。
吉林省虽是养猪大省,但到目前为止仍没有关于猪群TTSuVs遗传变异分析的报道。为此,本研究对2017-2018年吉林省猪生态养殖及疫病防控科技创新中心收集的574份血清样品进行检测,并对其部分毒株进行ORF1序列分析,了解吉林部分地区规模化猪场TTSuVs的基因特性,为进一步研究TTSuVs的遗传进化提供依据。
1 材料与方法
1.1 病料的收集 收集2017-2018年来自吉林地区10家规模化猪场的血清样品574份。猪场所在地分别为:长春市、吉林市、白城市、四平市、通话市、白山市、延吉市7个地区。同时记录每份样品的基本信息,包括猪的身体状况、年龄及品种等。
1.2 材料与试剂 UNIQ-10柱式病毒基因组抽提试剂盒购自上海生物工程技术服务有限公司;PCR克隆载体pMD18-T、感受态细胞JM109、蛋白酶K、DNA分子量标准DL2000、Taq酶、dNTP购自TaKaRa公司;DNA凝胶回收试剂盒、溴化乙锭(EB)、琼脂糖、琼脂粉购自维特杰公司。
1.3 引物的设计与合成 在对比分析GenBank上公布的国内外不同TTSuVs毒株ORF1基因序列的基础上,选取保守区,并在引物设计软件primer 6.0的辅助下设计引物。各PCR反应体系的引物序列如表1所示。

表1 分段扩增所用的引物序列Tab 1 Primer sequences for segmented amplification
1.4 病毒DNA的提取及TTSuVs阳性率的检测应用UNIQ-10柱式病毒基因组抽提试剂盒,按照说明书,提取574份检测样品中的病毒DNA并进行PCR鉴定,分析吉林部分地区规模化养猪场TTSuVs的阳性率。
1.5 TTSuVs ORF1全基因扩增及测序 为研究TTSuVs在同一感染体内不同亚型间的基因特点,本研究根据所收集样品的地点,对每家规模化猪场选取同时感染TTSuV1和TTSuV2的样品1份,应用所设计的引物,扩增各代表株的ORF1全基因序列。将扩增产物纯化并与pMD18-T载体连接后,42℃热击转化到JM109感受态细胞中,进行蓝白斑筛选。筛选后挑取白色菌落,经鉴定后进行测序。
1.6 TTSuVs ORF1基因的序列分析 采用DNAMAN、DNAStar、MEUA等生物信息学软件对TTSuVs测序株ORF1基因进行分析,使用参考株资料见表2。
2 结果与分析
2.1 吉林部分地区规模化猪场TTSuVs流行情况调查 对从10家规模化猪场收集到的574份血清样品进行PCR鉴定,其中共检出201份TTSuV1阳性样品,其感染率为36.75%;363份TTSuV2阳性样品,感染率为63.2%;TTSuV1与TTSuV2的混合感染样品有174份,混合感染率为30.3%。在吉林部分地区规模化猪场中,TTSuV1感染率最高的城市是长春市,感染率为48.97%;TTSuV2感染率最高的城市是延吉市,感染率为76.28%;而混合感染率最高的城市同样也是长春市,感染率为42.82%。结果表明,吉林省部分地区规模化猪场中,TTSuV2的感染率普遍高于TTSuV1,且各个年龄段的猪均可感染TTSuVs(表3-表5)。
2.2 TTSuVs ORF1基因的扩增 根据所收集样品的地点,每家规模化猪场选取同时感染TTSuV1和TTSuV2的样品1份,并进行TTSuV1、TTSuV2 ORF1全基因序列扩增,扩增结果见图1和图2。将PCR结果测序后,通过软件拼接为完整的ORF1全基因序列,大小为2000 bp左右,与预期结果相符,完成了TTSuVs ORF1基因的扩增与测序。

表2 本研究使用参考株资料表Tab 2 The reference strain list
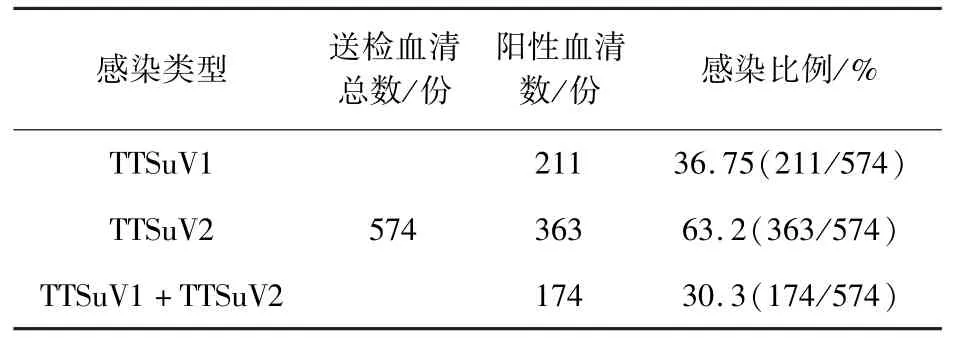
表3 2017-2018年吉林部分地区TTSuVs感染情况Tab 3 The infection type of TTSuVs in the part of Jilin during 2017-2018
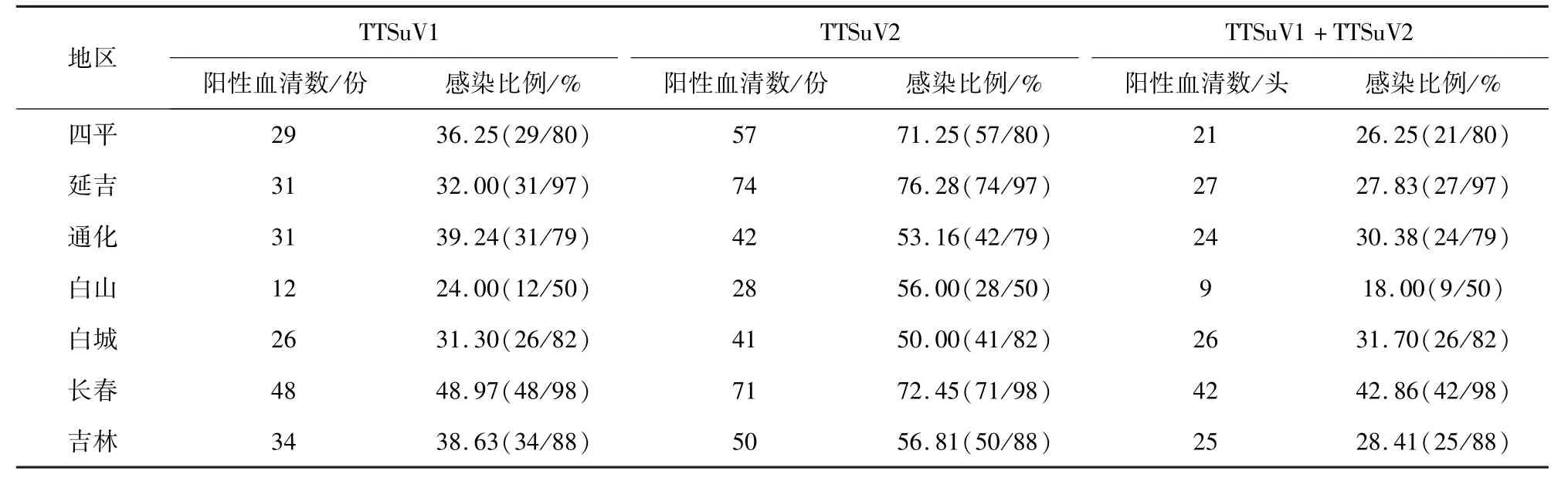
表4 吉林部分地区2017-2018年TTSuVs分布情况Tab 4 Distribution of TTSuVs in parts of Jilin during 2017-2018
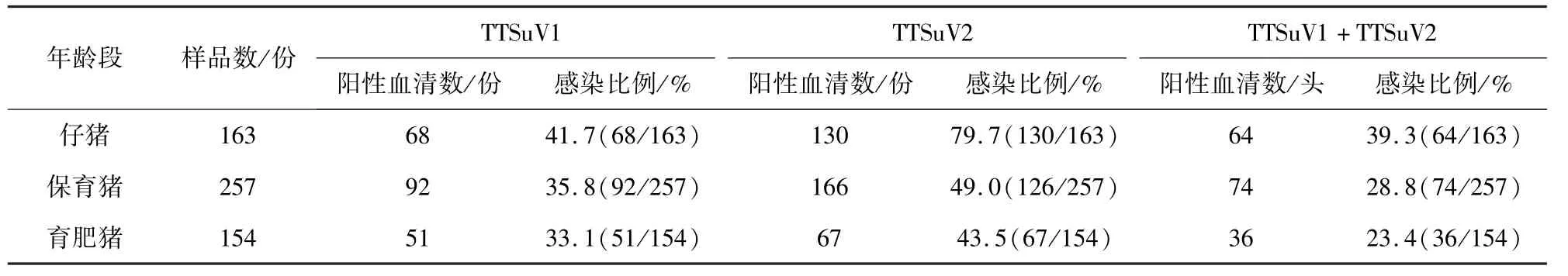
表5 吉林地区2017-2018年TTSuVs不同年龄段分布情况Tab 5 The pig of different age groups of TTSuV in Jilin during 2017-2018

图1 TTSuV1 ORF1全基因分段扩增结果Fig 1 Results of TTSuV1 ORF1 gene amplification

图2 TTSuV2 ORF1全基因分段扩增结果Fig 2 Results of TTSuV2 ORF1 gene amplification
2.3 TTSuVs ORF1基因核苷酸同源性分析 将所测得的TTSuVs ORF1全基因序列与国内外参考株的ORF1序列进行对比分析,结果见图3、图4。结果显示,10株TTSuV1测序株之间的核苷酸同源性为75.7% ~94.7%,其与 2004年巴西参考株AY823990之间同源性最高,为98.0%;与2018年巴西参考株MH170071的同源性最低,为61.2%。10株TTSuV2测序株之间的核苷酸同源性为77.5% ~94.0%,其与 2010年西班牙参考株GU570205及2010年中国参考株JF694118之间同源性最高,为 97.1%;与 2015年日本参考株KR054750同源性最低,为77.5%。

图3 10株TTSuV1测序株与国内外参考株ORF1核苷酸序列的同源性对比结果Fig 3 Comparison of homology between 10 sequencing strains of TTSuV1 and ORF1 sequences from domestic and foreign strains

图4 10株TTSuV2测序株与国内外参考株ORF1核苷酸序列的同源性对比结果Fig 4 Comparison of homology between 10 sequencing strains of TTSuV2 and ORF1 sequences from domestic and foreign strains
2.4 TTSuVs ORF1基因编码的蛋白氨基酸同源性分析 将测序结果推导出的氨基酸序列与国内外参考株的ORF1氨基酸序列进行对比分析,结果见图5、图6。结果显示,10株TTSuV1测序株之间的氨基酸同源性为48.1% ~88.2%,其与2010年中国参考株HM633253之间同源性最高,为96.2%;与2010年西班牙参考株GU570198、GU570200同源性最低,为34.2%。10株TTSuV2测序株之间的氨基酸同源性为70.6% ~87.4%,其与2010年西班牙参考株 GU570205之间同源性最高,为93.6%;与2015年日本参考株KR054750同源性最低,为 72.4%。
2.5 TTSuVs ORF1基因编码的蛋白氨基酸序列突变位点分析 TTSuVs测序株ORF1基因推导的氨基酸序列与国内外参考株的ORF1氨基酸序对比存在多处氨基酸的差异,结果见图7、图8。结果显示,在296~464aa处是TTSuV1 ORF1的超变区,10株TTSuV1测序株在392aa和397aa均有与1b型相同的5个氨基酸的缺失;TTSuV1-JL在10aa处有一个氨基酸的突变(R变K),与其他毒株均不相同。与2017年中国参考株 KY937993相比,TTSuV1-JL、TTSuV1-CC1和 TTSuV1-CC2在64~65aa处有两个氨基酸的缺失;TTSuV1-TH1在620~622aa处有三个氨基酸的缺失。200~280aa是TTSuV2 ORF1的超变区,10株TTSuV2测序株在39aa和47aa处均有两个氨基酸的缺失,与2012年的中国参考株 JQ782385相同;TTSuV2-BC和TTSuV2-YB在292~294aa处存在与2010年中国参考株HM633220相同的三个氨基酸的缺失;TTSuV2-JL、TTSuV2-BC、TTSuV2-YB 和TTSuV2-SP2在377aa处有与2012年中国参考株JQ782385相同的一个氨基酸的缺失。

图5 10株TTSuV1测序株与国内外参考株ORF1氨基酸序列的同源性对比结果Fig 5 Comparison of amino acid sequences between 10 sequencing strains of TTSuV1 and ORF1 sequences from domestic and foreign strains

图6 10株TTSuV2测序株与国内外参考株ORF1氨基酸序列的同源性对比结果Fig 6 Comparison of amino acid sequences between 10 sequencing strains of TTSuV2 and ORF1 equences from domestic and foreign strains

图7 部分TTSuV1 ORF1基因编码氨基酸序列对比结果Fig 7 Comparison of TTSuV1 ORF1 gene coding amino acid sequences

图8 部分TTSuV2 ORF1基因编码氨基酸序列对比结果Fig 8 Comparison of TTSuV2 ORF1 gene coding amino acid sequences
2.6 TTSuVs ORF1基因遗传进化树分析 为进一步分析吉林部分地区TTSuVs毒株与其他参考株在进化上的关系,将20株测序株与国内外发表的参考株共同构建遗传进化树,结果见图9、图10。结果显示,在吉林部分地区规模化猪场中,测序的10株TTSuV1均属于 TTSuV1-lb型,其中测序株TTSuV1-JL、TTSuV1-CC1和 TTSuV1-CC2与巴西参考株亲缘关系较近;测序株 TTSuV1-BC、TTSuV1-YB、TTSuV1-SP1和 TTSuV1-SP2与2010年中国参考株HM633253亲缘关系较近。10株 TTSuV2测序株中,TTSuV2-JL、TTSuV2-SP2、TTSuV2-YB和TTSuV2-BC属于TTSuV2-2a型;TTSuV2-CC1、TTSuV2-CC2、TTSuV2-TH2 和TTSuV2-SP1属于 TTSuV2-2c型;TTSuV2-BS与TTSuV2-TH1属于TTSuV2-2b型。测序结果表明,在吉林部分地区规模化猪场中流行的TTSuV1均属于TTSuV1-lb型,而TTSuV2流行情况较为复杂,各种亚型均有涉及。
3 讨论与结论
TTSuVs的感染不会立刻引发疾病,然而TTSuVs却可以影响一些疾病的发展,甚至影响它们的结果。目前,人类的TTV已证实与自身免疫风湿性疾病、肝脏病变和呼吸性疾病有关[4],这也使得近年来对TTSuVs的研究成为热点。
本研究对2017-2018年间吉林部分地区规模化猪场的574份样品进行检测,结果显示,TTSuV1感染率为 36.75%;TTSuV2感染率为 63.2%;TTSuV1与TTSuV2的混合感染率为30.3%,与王礞礞2009年的调查结果相近,但与徐茜2013年对江西省猪TTSuVs感染率的调查结果相反(TTSuV1的感染率为72.9%,TTSuV2的感染率为69.8%,总混合感染率为53.0%),说明TTSuVs已在我国多省份流行,但流行情况各个地域间差异较大[5]。
细环病毒的基因组具有高度变异性和多样性,其中约2000 bp的ORF1全基因序列分型法是公认的经典分型方法之一[6]。ORF1全序列分型法是根据其核苷酸的差异来确定不同基因型,即差异程度在30%~40%之间的为不同基因型,大于40%为不同基因群[7]。ORF1全基因序列占TTSuVs基因总长度的60%,所以ORF1全基因序列分型法可以代表TTSuVs基因组多变性特点,并且该方法相对来说更加经济有效[8-9]。对20株TTSuVs测序株ORF1基因同源性分析发现,在吉林部分地区规模化猪场中流行的TTSuV1均属于TTSuV1-lb型,而TTSuV2流行情况较为复杂,各种亚型均有涉及。

图9 TTSuV1测序株与参考株ORF1基因进化树分析Fig 9 Phylogenetic tree analysis of ORF1 gene of TTSuV1 sequencing and reference strains

图10 TTSuV2测序株与参考株ORF1基因进化树分析Fig 10 Phylogenetic tree analysis of ORF1 gene of TTSuV2 sequencing and reference strains
现阶段,国内外对于TTSuVs的研究大部分停留在流行情况调查及检测方法的建立,而对于病毒的基因与其致病性及如何逃避宿主免疫的机制研究较少[10]。学者对于人TTV基因组研究发现其普遍存在G和A之间的转换[11],而在本次的调查中G和A也出现了较高的突变率,这可能与7 APOBEC3(Apolipoprotein B editing component 3)蛋白有关,但具体相关性仍需进一步的探查。
本研究对多株TTSuVs基因研究发现,其核苷酸同源性大于60%,而氨基酸同源性不及60%。对于一般的病毒,氨基酸同源性会大于其核苷酸同源性,但TTSuVs却是核苷酸同源性大于氨基酸同源性,这和其他病毒显著不同[12]。ORF1可能是TTSuVs编码的最大蛋白,可能在宿主体内诱发宿主抗体的形成,推断ORF1的多样性可以让其逃避宿主的免疫攻击。
通过序列比较分析,本研究发现了一些保守区域:TTGGCCGCCGCCG,ACGAGTGCCGAAAGCTCT,ATGGATCGTCTGA,这与2011年国内学者发现的保守区并不完全相同[13],但是均属于高CG区,与人源TTV保守区相似[14]。对其研究显示,突变的原因可能是在几个不同的个体内进行序列的改变和进化,推测TTSuVs与近来发现单股DNA病毒相似,其高频率的突变可能和病毒基因组的单链或和参与复制的蛋白的编码能力有关。
近几年来国内学者对于TTSuVs的基因研究显示,TTSuV2 ORF1核苷酸序列较为保守,其测序的TTSuV2 ORF1核苷酸序列可与2010年中国参考株HM633231的同源性高达97%[15]。虽然在本研究中TTSuV1测序株与2010年中国参考株HM633253之间同源性最高可达96.2%,与2017年中国参考株KY937993之间同源性最高也达94.2%,但其突变频率也最为频繁,其序列之间的多样性大于30%,而 TTSuV2序列之间的多样性大于15%。TTSuV1和人源TTV在这点上具有相似性[16],人源TTV基因组序列之间的多样性在30%以上,这一高百分比对于DNA病毒来说是极不常见的。
有学者推测,只有一小部分人源TTV拥有可以成功感染宿主的所有组分,与HIV病毒一样,需要不同的TTV共感染才可满足持续性感染发生的条件[17],即感染人类的 TTV不是一个基因型而是TTV的一个族群。为此,本研究选取同一感染体来源的TTSuVs进行分析,结果显示,同一感染体中的两种TTSuVs亚型在氨基酸及核苷酸分析上与其他单独感染猪的TTSuVs亚型没有明显的共同差异或突变,TTSuVs不同亚型的共感染是否可以引起持续性感染以及其感染机制是什么,将是下一步的主要研究方向。
总之,本次流行病学调查更好地了解了TTSuVs在吉林部分地区规模化猪场中的流行情况,掌握了TTSuVs发生、发展的动态规律,这将有助于揭示TTSuVs遗传变异规律,丰富其分子生物学特性内容,并为今后预防和控制TTSuVs提供理论依据,也为TTSuVs的机理研究奠定基础。
