基于知识图谱的原发性肝癌知识问答系统
2019-08-05曹明宇李青青杨志豪林鸿飞
曹明宇,李青青,杨志豪,王 磊,张 音,林鸿飞,王 健
(1. 大连理工大学 计算机科学与技术学院,辽宁 大连 116024;2.军事医学科学院,北京 100850)
0 背景
随着大数据时代的到来,知识工程受到了广泛关注,如何从海量的数据中提取有用的知识,是大数据分析的关键。知识图谱技术提供了一种从海量文本和图像中抽取结构化知识的手段,并且已被广泛应用于智能搜索、智能问答、个性化推荐等领域,因而受到了广泛的关注。
知识图谱于2012年被Google正式提出[1],其初衷是为了提高搜索引擎的能力,增强用户的搜索质量及搜索体验。目前,已经存在的大规模知识库如Freebase、Wikidata、DBpedia、YAGO中,不仅包含大量的半结构化、非结构化数据,是知识图谱数据的重要来源,而且具有较高的领域覆盖面,与领域知识库存在大量的链接关系。除此之外,一些行业知识库(也称为垂直型知识库),如MusicBrainz、IMDB、豆瓣等也已经构建起用来描述特定行业领域的知识。值得注意的是,在中文知识图谱构建方面,中文开放知识图谱联盟OpenKG搭建了OpenKG.CN技术平台,吸引了国内最著名知识图谱资源的加入,如Zhishi.me、CN-DBPedia、PKUBase,并已经包含了来自于常识、医疗、金融、 城市、出行等15个类目的开放知识图谱[2]。同时,由于与人类健康密切相关,生物医学领域的知识受到密切关注。我们建立了一个生物医学领域的与肝细胞癌(Hepatocellular carcinoma,HCC)相关的知识图谱。肝细胞癌是成人中最常见的原发性肝癌,并且是肝硬化患者最常见的死亡原因[3]。构建肝细胞癌相关的知识图谱,结构化地表示肝细胞癌与其相关的蛋白质、药物、疾病、病症等之间的关系,对于医学研究者来说具有重要的意义。
问答系统是自然语言处理领域的一个重要方向。它接受自然语言问题的输入,从知识库中查询到相应的答案,并以自然语言文本的形式返回给用户。传统上人们获取知识的途径主要依赖于搜索引擎,然而搜索引擎只是单纯的关键字查询,缺乏对用户意图的理解,需要用户从返回的网页中筛选自己想获取的信息。与传统的搜索引擎相比,问答系统极大地增强了用户获取知识的便捷性,不但节省了筛选信息的时间,还能精确地获得更符合需求的答案。
传统的问答系统大多基于文档检索,使用爬虫从网络上爬取百科数据、问答对等知识,再使用关键词检索或模板匹配的方式查询答案。这种方式的知识来源并非结构化,包含大量的冗余信息,需要时间来进行检索。而知识图谱作为一种结构化、关联化的数据来源,可以为问答系统提供更加高质量的数据信息,面向领域的问答系统也层出不穷。杜泽宇等[4]利用哈工大LTP 语义依存分析SDP及基于Word2Vec(https://code.google.com/archive/p/word2vec/)的语义相似度计算,开发了面向电商领域的问答系统,极大地增强了电商网站用户沟通的便捷性与时效性。
近年来,随着“看病难”“挂号难”等社会问题出现,很多在线求医问药的网站兴起,用户可以更便捷地在网络上描述自己的病情并得到医生的指导。但是这种方式仍然需要人力的维护,并且医生通常推荐面诊来确定病情。同时,由于大众对疾病的知识了解过少,常常不能对医生给予的治疗方案的合理性进行判断。针对以上问题,本文提出了一个面向肝细胞癌的基于知识图谱的自动问答系统,可以回答与肝癌相关的药物、疾病、表征等问题,帮助用户更充分地了解肝癌相关知识,缓解医疗机构的压力。
本文提出的基于肝细胞癌知识图谱的问答系统有一套流水线式的结构。首先,基于当前主流的BiLSTM-CRF神经网络模型,对问题中的药物、疾病等实体进行识别; 然后将结合TFIDF与预训练的词向量,得到问题向量,将其与预先定义 的问题模板进行相似度匹配,得到最相似的问题模板;再根据模板对应的语义信息,使用Cypher查询语句到知识图谱中查询答案;最后生成自然语言回答并返回给用户。
1 肝细胞癌知识图谱的构建
1.1 肝细胞癌知识的获取
海量生物医学文本中蕴含着大量医学实体及关系,新实体和新关系的更新速度比专业的数据库系统更快,而专业数据库中的知识更加精准可靠。基于上述两种知识来源的特点,本文同时使用以下两种方式获取肝细胞癌相关知识:①应用深度学习技术。对医学指南和PubMed摘要文本进行命名实体识别,再对实体对进行关系分类,从中抽取出与HCC相关的三元组; ②从SemMedDB[5]中抽取所有与HCC相关的三元组。
具体的知识获取步骤描述如下。
首先获取了UpToDate临床顾问(http://www.uptodate.com)中与肝细胞癌相关的医学指南,以及在PubMed中下载与肝细胞癌相关的1 000篇MEDLINE摘要,然后使用基于深度学习的方法,对文本进行命名实体识别和关系抽取,得到与肝细胞癌相关的关系三元组。对得到的三元组进行去重,将实体和关系映射到生物医学本体中,形成了肝细胞癌与其相关的基因、蛋白质、单个药物、药物组合、疾病、病症以及治疗方法之间的关系三元组。
SemMedDB是使用SemRep工具(https://semrep.nlm.nih.gov)从MEDLINE摘要中进行关系抽取得到的,包含9 100万个关系预测的数据库[5]。该数据库支持Semantic MEDLINE Web应用程序,它集成了PubMed搜索、SemRep预测、自动汇总和数据可视化。我们使用SQL语句从该数据库中检索与肝细胞癌直接相关的实体以及关系三元组,共得到46 172个三元组。然后对三元组进行去重,去重后得到4 547个三元组。在这些三元组中,每个实体的类型定义为概念唯一标识符、实体标准名、实体类型以及实体在文本中的名字。关系属性定义为关系类型和关系来源。
结合以上两种方式,我们得到了与肝细胞癌相关的实体和关系三元组。其中,使用深度学习的方法从医学指南和PubMed文摘中获得416个实体和500条关系;从SemMedDB中抽取了共2 723个实体和4 547条关系。对于实体和关系的详细统计,如表1所示。

表1 肝细胞癌相关的实体和关系数据统计
1.2 知识表示
三元组是知识图谱的一种通用表示方式,即g=(e,r,s),其中e={e1,e2,…,e|E|} 是知识库中的实体集合,共包含|E|种不同实体;R={r1,r2,…,r|E|}是知识库中的关系集合,共包含|R|种不同关系;S∈E×R×E代表知识库中的三元组集合。三元组的基本形式主要包括实体1、关系、实体2和概念、属性、属性值等,实体是知识图谱中的最基本元素,不同的实体间存在不同的关系。概念主要指集合、类别、对象类型、事物的种类,例如,药物、疾病等;属性主要指对象可能具有的属性、特征、特性、特点以及参数,例如实体名、实体概念标识符等;属性值主要指对象指定属性的值,例如“肝细胞癌”“Q1148337”等。每个实体(概念的外延)可用一个全局唯一确定的ID来标识,每个属性—属性值对(attribute-value pair,AVP)可用来刻画实体的内在特性,而关系可用来连接两个实体,刻画它们之间的关联[6]。
1.3 知识存储
目前,图结构有两种通用的存储方案:RDF存储和图数据库(Graph Database)。图数据库的结构定义相比RDF数据库更为通用,实现了用图结构中的节点、边以及属性来进行图数据的存储。我们使用当前流行的开源图数据库Neo4j(https://neo4j.com/)进行知识图谱的存储,优点是数据库本身提供完善的图查询语言,支持各种图挖掘算法。
Neo4j提供Cypher语句来导入数据和查询图形数据,Cypher是描述性的图形查询语言,语法简单,功能强大。除此之外,对于大规模的数据,Neo4j还提供了neo4j-import 工具,可以快速地将大量的节点(实体)和边(关系)导入图数据库。我们将医学指南、PubMed摘要文本和SemMedDB中抽取的肝细胞癌相关的三元组通过Cypher CREATE 语句、Cypher LOAD CSV 语句以及neo4j-import 工具导入Neo4j数据库。图1展示了肝细胞癌知识图谱的部分关系三元组。
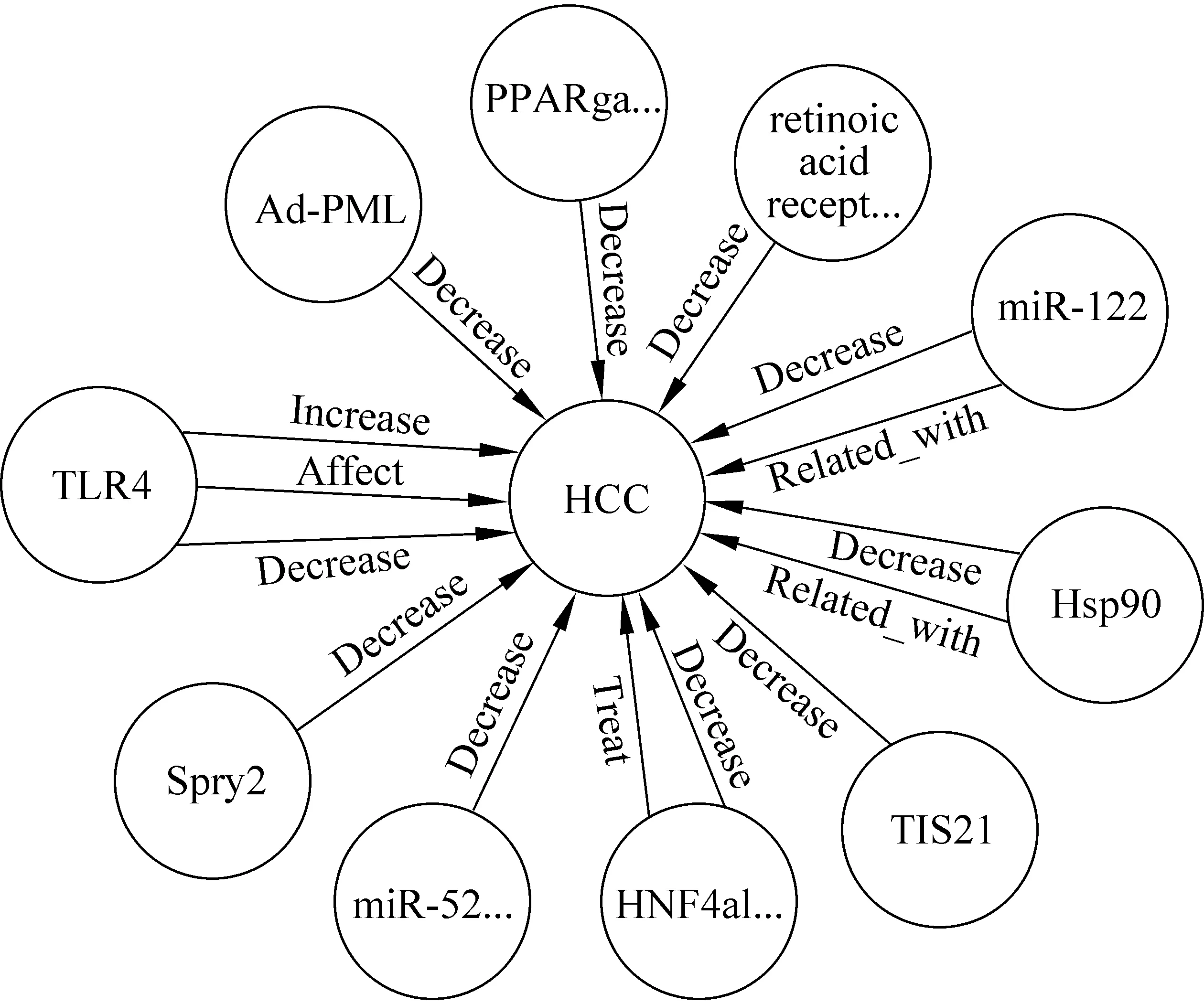
图1 肝细胞癌知识图谱中的部分关系三元组
2 基于肝细胞癌知识图谱的问答系统
本文设计并实现了基于肝细胞癌知识图谱的英文问答系统,为用户在线求医提供了更加智能化的方式,提升了在线求医的便捷性及实时性。该系统主要包括以下几个模块:①疾病和药物的实体识别; ②问题的模板匹配; ③基于Neo4j数据库的查询; ④基于Web的图形化展示。具体的流程如下所述:
(1) 输入问题:在对话框内输入与疾病、药物、表征相关的英文医学问题,如:“Whichmedicinecantreataids?”“WhatarethemanifestationsofHCC”。
(2) 问题的预处理及分词:由于医学领域的命名实体识别是以单个词为单位,而很多实体常与标点符号直接相连,如果问题的分词不准确,则会直接影响实体识别的准确性,也会对之后的模板匹配造成影响。比如上述问题中的“aids?”被分解为“aids”和“?”两个单词。
(3) 医学实体识别:由于本文实现的问答系统主要是面向普通用户的,所以在这一环节识别出问题中最常涉及的问题,包括疾病名、药物名及表征名。识别结果是一个形如[entity name1,label1]的列表,例如,问题“Whichmedicinecantreataids?”,识别结果为[‘aids’ ,‘disease’]。
(4) 问题模板匹配:根据识别出的实体信息,将问题与相应的问题模板集进行匹配。本文分别从Literal、Synonym两个层面,利用TFIDF及基于Word2Vec词向量的匹配方式,为原问题匹配到最相似的问题模板。这一环节实现了问题理解的功能,可以得到问题中蕴含的关系,如:疾病—药物、疾病—表征等。
(5) 基于图形化数据库的查询:根据环节(3)、(4) 识别出的实体名及关系类型,理解问题的语义,使用Python语言中的Neo4j-driver模块,在本文构建的肝细胞癌知识图谱中查询对应的实体或属性。
(6) 答案生成:根据问题的意图及查询到的结果,生成符合对话逻辑并且语法通顺的自然语言答案,返回给用户。
针对医学实体识别、问题模板匹配和基于知识图谱的查询的详细介绍如下。
2.1 生物医学实体识别
本文实体识别模型的训练使用biocreative评测提供的 CDR语料集。传统的命名实体识别使用统计学习的机器学习方法,如条件随机场(conditional random fields,CRF),对文本中的人名、地名、机构名等实体进行识别。而医学领域的实体通常命名规则复杂,实体名中可能包含数字或符号,并且实体的边界更容易被误识别。针对这些问题,本文使用当前流行的BiLSTM(Bi-directional LSTM)+CRF模型[7],在疾病、药物和表征实体上分别达到了0.887、0.905和0.866的综合分类率(F1值)。
长短时记忆模型(long short-term memory,LSTM)[8]是循环神经网络(recurrent neural network,RNN)的一种。RNN的最大特点是可以接受序列的输入,产生对应的序列输出,不同时刻的输入之间存在着依赖关系,当前时刻的输出不仅取决于当前时刻的输入,还和上一时刻的输出有关。由于这样的特点,RNN很适合用于学习单个词的上下文信息。但是RNN仍然存在着长距离依赖的问题,即当前时刻的输入受距离当前更近的时刻影响更大,而受距离较远的之前时刻输出的影响相对较小,这就导致RNN不能充足地学习到远距离的上下文信息。LSTM在RNN的基础上增加了门机制,可以控制信息在时刻之间传递的程度,更好地学习当前词的上下文信息。双向长短时记忆循环模型由两个不同方向的LSTM组成,两个LSTM分别从前向和后向学习单词的上下文信息,再将二者拼接起来,作为当前时刻的输出。
本文的命名实体工作,首先使用预先训练好的词向量,将词映射为低维空间中稠密的50维词向量,随后将句子的词向量序列输入到BiLSTM中,用神经网络自动学习前向及后向的上下文特征,最后在输出层使用softmax来预测每个单词的标签。这种方法的缺陷是对每个词的标签都进行独立的预测,不能参考上下文中已经预测出的标签,导致预测出的标签序列可能是不合逻辑的。例如,标签I后面是不可能紧跟着标签B的,但神经网络无法利用到这个信息。为了实现标签级别的全局优化,本文在神经网络的输出后增加一个条件随机场层进行句子级的序列标注。CRF层的参数是一个 (k+2)×(k+2)的矩阵A(之所以要加2是因为要为句子首部添加一个起始转移状态,在句子尾部添加一个终止转移状态),Aij表示的是从第i个标签到第j个标签的转移得分,进而在为一个位置进行标注的时候可以利用此前已经标注过的标签。结合了BiLSTM和CRF的命名实体识别,可以充分学习每个单词的上下文信息及上下文标签信息,从局部和全局两个层面,对词标签的分类进行更好的优化,达到良好的实体识别效果。
2.2 问题模板匹配
常见的用于问题理解的技术有基于模板匹配[9]、基于检索模型及基于深度学习的模型方法[10]。本文实现的问答系统使用了模板匹配的方式。与其他两种问题理解的方式相比,模板匹配只需要根据常见的问题设计问题模板,并实现匹配模板(即计算问题与模板间的相似度)的算法,无须对大量人工标注的语料进行深度学习,也不需要从大量的QA文本中检索相似的问题。本文模板匹配的具体流程如下。
(1) 根据问题中可能包含的实体数量及实体类别,针对每一种情况,本文设计了6种情况共107个问题模板,问题模板集的信息见表2。

表2 不同实体情况的问题模板集
(2) 根据实体识别环节中识别出的实体类别及数量,在与实体情况对应的问题模板集中进行相似度匹配,选择相似度最高的问题模板。本文的相似度匹配结合TFIDF算法[11]与Word2Vec词向量[12],对于输入的问题中的每个词,首先计算该词的TF(term frequency,词频),即在该问题中出现的频率,词的TF越高就越表明它能代表这个问题。然后计算该词的IDF(inverse document frequency,逆向文件频率),由总模板问题数目除以包含该词的模板问题数目得到,IDF可以衡量一个词的区分能力。TF和IDF的乘积便代表这个词在当前问题中的权重,将问题中所有词的词向量加权求和,得到问题的向量。我们分别计算模板问题及用户提出问题的问题向量,再分别计算句子向量之间的Cosine和 Euclidean距离,最后取平均作为提出问题与模板问题的相似度。
2.3 基于知识图谱的查询
本文使用Cypher语言在图形数据库中查询答案。该语言是neo4j图形数据库的查询语言,遵循SQL(structured query language)语法。问题模板中包含着问题的语义,根据预先定义的模板问题到数据库中关系的映射可以得到关系名,结合识别出的医学实体名,根据规则生成Cypher语句。
用于查询与已知实体具有特定关系的相关实体名的Cypher语句模板如下:Match (a)-[: RelationName]-(b) where b.name=‘EntityName’ return a.name。其中,EntityName和RelationName用之前得到的实体和关系名替换。例如,对于问题“Which medicine can treat HCC?”,首先识别出实体[HCC, drug],匹配问题模板可以得到该问题在数据库中的对应关系为“Treats”,然后根据实体名、实体类别和关系名,按照规则生成Cypher语句:Match (a)-[: Treats]-(b) where b.name=‘HCC’ return a.name,根据返回的结果,生成自然语言回答:“HCC can be treated by acrylamide, transaminase, Bortezomib, etc.”
3 实验与结果分析
由于目前没有肝细胞癌相关的标准问答语料,我们人工设计了50个与模板问题语义相近的肝细胞癌相关问题,对其答案进行评测,以验证本文提出的问答系统的性能。除此之外,也从英文医疗问答网站(https://www.drugs.com)中爬取了100个与肝细胞癌相关的真实问题进行了实验。
(1) 由于本文提出的问答系统知识来源是结构化的知识图谱,因此,对于一个问题,当其命名实体识别结果准确、问题模板匹配符合语义且返回有效自然语言回答时,就认为该问题得到了正确回答。
从实验结果可以看出,76%人工设计的问题可以得到正确回答。尽管有些问题使用了与模板问题不同的表示方式来表示语义,基于问题向量进行相似度匹配的方式仍然可以为大多数问题匹配到语义相同的模板。对于医疗问答网站爬取的问题,28%可以得到正确回答,例如“Should I take Cipro for HCC?”,这类包含药物-疾病关系语义信息的问题大多可以被正确地理解。对于网络爬取的问题,实体识别的准确性良好,但语义理解的准确度偏低。一些不能准确回答的问题,例如,“Does Xgeva need to be refrigerated?”,问题的语义是咨询药物的保存事宜。由于本文搭建的知识图谱侧重于药物、疾病、表征等实体关系,而药物的保存事宜、服用方法等应该被存储于实体属性中,由于信息抽取的并不完备,未能返回满意的答案,这也是未来知识图谱需要完善的部分。
(2) 现有实验说明本文提出的问答系统可以有效地基于肝细胞癌知识图谱,对肝细胞癌相关的药物—疾病、疾病—表征、药物—药物及药物—表征等问题进行回答。同时由于本文使用流水线式的结构,每个子模块都具备进一步优化的可能性。结合回答失败问题的原因,本文的未来工作包括:①将实体的属性信息补充加入知识图谱; ②扩充知识图谱的疾病覆盖率; ③使用深度学习技术对问题理解进行更深入的研究。
4 结论
本文针对成人中常见的原发性肝细胞癌,从医学指南和相关医学文摘及SemMedDB知识库中抽取其知识三元组,构建了原发性肝细胞癌的知识图谱。在此基础上,实现了流水线式的问答系统。实验表明,该问答系统可以回答药物—疾病、药物—表征、药物-药物等语义信息的问题。下一步的工作包括使用深度学习方法来提高问题理解的准确度、扩展该问答系统可回答问题的种类以及丰富知识图谱中实体的属性信息。
