肝细胞癌相关差异基因的生物信息学及预后分析
2019-05-16朱亚玲赵洪波蒋保三张越美
朱亚玲,赵洪波,蒋保三,王 刚,张越美,刁 勇*
(1.华侨大学医学院,中国福建泉州362021;2.昆明医科大学分子临床医学研究院暨云南省干细胞和再生医学重点实验室,中国云南昆明650500)
肝细胞癌(hepatocellular carcinoma,HCC)是常见的恶性肿瘤之一,占原发性肝癌的70%~90%,是全球范围内致死率排名第二的恶性肿瘤[1]。遗传因素[2]和环境因素(酒精、黄曲霉毒素[3]、慢性乙型肝炎病毒[4]等)被认为是导致肝细胞癌发生和发展的主要原因[5~6]。虽然诊断技术、外科治疗、化疗和分子靶向治疗技术有所改善,但由于肝细胞癌患者被诊断时多为中晚期,因此患者的5年生存率仍不尽如人意[7~8]。
肝细胞癌的发生是一个多基因参与、多因子协调的复杂的生物学过程。研究发现,血管生成功能失调、慢性炎症、内分泌激素、脂肪因子和细胞代谢的改变均可能参与肿瘤的形成过程[9]。尽管已经发现了一些肝细胞癌相关的重要驱动基因,但其发生、发展和转移的机制尚不清楚,临床上仍然缺乏与患者预后相关的生物标志物[10~11]。因此,深入研究肝细胞癌发病及进展过程潜在的生物学机制,对于开发新的诊断标志物和治疗策略具有重要的意义。
近年来,高通量基因芯片和测序技术作为基因表达分析的工具,已被广泛用于识别肿瘤发生过程中遗传信息的改变,为研究肝细胞癌基因表达情况及发现关键基因的变化规律提供了基础[12]。本研究利用基因表达数据库(Gene Expression O-mnibus,GEO)中肝细胞癌组织和非癌组织的基因表达阵列数据筛选肝细胞癌相关差异表达基因(differentially expressed genes,DEGs),并对差异表达基因进行生物信息学分析,同时结合KM plotter(Kaplan Meier plotter)数据库进行预后分析,以期为揭示肝细胞癌发生、发展的分子机制和探索肝细胞癌诊断、治疗和预后相关的潜在候选生物标志物提供理论依据。
1 材料与方法
1.1 数据获取
数据GSE84402[13]从NCBI基因表达数据库GEO(https://www.ncbi.nlm.nih.gov/geo)中下载得到。该数据采用的芯片平台是GPL570(Affymetrix Human Genome U133 Plus 2.0 Array),包括14例肝细胞癌组织和14例非癌组织。
1.2 HCC差异表达基因筛选
采用GEO数据库中基于R语言的网络分析工具GEO2R[14](https://www.ncbi.nlm.nih.gov/geo/info/geo2r)分析GSE84402中肝细胞癌组织和非癌组织的差异表达基因。该工具利用Bioconductor项目中的R语言软件包GEOquery和Limma,根据Benjamini和Hochberg提出的假阳性率控制法[15],进行多重检验校正。差异表达基因同时满足以下条件:FDR(false discovery rate)<0.05,|log2FC|>1。
1.3 差异表达基因的生物信息学分析
生物学信息注释及可视化数据库(Database for Annotation,Visualization and Integrated Discovery,DAVID)是基因功能分析的在线生物信息学工具,可对差异表达基因进行GO(Gene Ontology)富集分析及KEGG(Kyoto Encyclopedia of Genes and Genomes)通路分析。将差异表达基因数据导入DAVID 6.8(https://david.ncifcrf.gov/)[16],以 FDR<0.05为显著性基因富集的临界值,对差异显著的基因进行功能注释,并分析其参与的生物学过程及通路。
1.4 蛋白质互作网络分析及hub genes的筛选
蛋白质相互作用数据库STRING(https://string-db.org/)可利用已知或预测蛋白质之间的相互作用构建互作网络[17]。将差异基因导入STRING 10.5分析工具,将最低互作分值(minimum required interaction score)设置成最高可信(highest confidence 0.9),删除与其他蛋白质无相互作用的节点后,分析差异基因所编码的蛋白质之间的相互作用。将互作网络数据导入Cytoscape 3.6.1软件[18],计算节点的度(degree),列出degree排名前20位的中心节点蛋白质,并与GO和KEGG富集分析得到的显著性基因取交集,得到的基因即为核心基因(hub genes)。
1.5 Hub genes与预后的相关性分析
KM plotter数据库(http://kmplot.com/)从GEO、EGA及TCGA数据库下载基因表达数据、无复发和总生存信息,利用集成基因表达和临床数据的PostgreSQL服务器对数据进行处理,依据所提出的生物标志物的不同分位数表达式将患者样本分成两组[19]。本研究根据hub genes的表达水平,以中位数值将KM plotter数据库中纳入的364例肝癌患者分成高表达组和低表达组,分析两组患者预后的相关性。通过Kaplan Meier生存图比较两组病人队列,计算危险比(hazard radio,HR)及95%置信区间(confidence interval,CI),差异比较用logrank P检验,logrank P<0.05表示差异具有统计学意义。
2 结果
2.1 差异表达基因筛选结果
通过对肝细胞癌组织和非癌组织的基因表达数据进行分析,共筛选出1 307个表达差异明显的基因,其中上调基因741个,下调基因566个。排名前10的差异表达基因见表1。
2.2 差异表达基因的GO富集分析结果
采用DAVID 6.8对741个上调基因和566个下调基因进行GO生物学过程富集分析。上调基因主要富集到细胞分裂、姐妹染色单体黏着和分离、有丝分裂核分裂、DNA复制、细胞周期等25个生物学过程;下调基因主要涉及氧化还原、急性期反应、环氧酶P450通路、异源代谢过程、药物代谢过程等23个生物学过程。上、下调基因前5位的生物学过程富集见表2。
2.3 差异表达基因的KEGG通路分析结果
采用DAVID 6.8对741个上调基因和566个下调基因进行KEGG通路富集分析。上调基因中有2条富集通路呈显著性(FDR<0.05),下调基因中有10条富集通路呈显著性(FDR<0.05)。上调基因主要涉及细胞周期和DNA复制信号通路,下调基因主要涉及代谢途径、补体途径、视黄醇代谢、化学物致癌作用、胆汁分泌、脂肪酸降解等。上调及下调基因前5位的显著富集通路见表3。
2.4 差异表达基因的蛋白质相互作用网络分析及hub genes的筛选
通过STRING 10.5及Cytoscape 3.6.1软件分析,得到包含567个差异表达基因及3 095条互作关系的蛋白质相互作用网络(图1)。结果显示最大degree值为107。Degree值排名前20位的中心节点蛋白质见表4。将编码前20位中心节点蛋白质的差异基因与GO和KEGG富集得到的显著性基因取交集,得到BUB1 (mitotic checkpoint serine/threonine kinase,丝裂原检查点丝氨酸/苏氨酸激酶)、BUB1B(mitotic checkpoint serine/threonine kinase B,丝裂原检查点丝氨酸/苏氨酸激酶B)、CCNA2(cyclin A2,细胞周期蛋白A2)、CCNB1(cyclin B1,细胞周期蛋白 B1)、CCNB2(cyclin B2,细胞周期蛋白 B2)、CDC20(cell division cycle 20,细胞分裂周期蛋白20)、CDK1(cyclin-dependent kinase 1,周期蛋白依赖激酶1)、MAD2L1(mitotic arrest deficient 2 like 1)、PLK1(Polo-like kinase 1,Polo样激酶1)等9个hub genes,其相互作用网络包括36条互作关系(图2A)。KEGG分析发现hub genes主要富集在细胞周期等通路(图2B)。

表2 前5位差异表达基因的生物学过程富集Table 2 Biological process enrichment results of DEGs(Top 5)
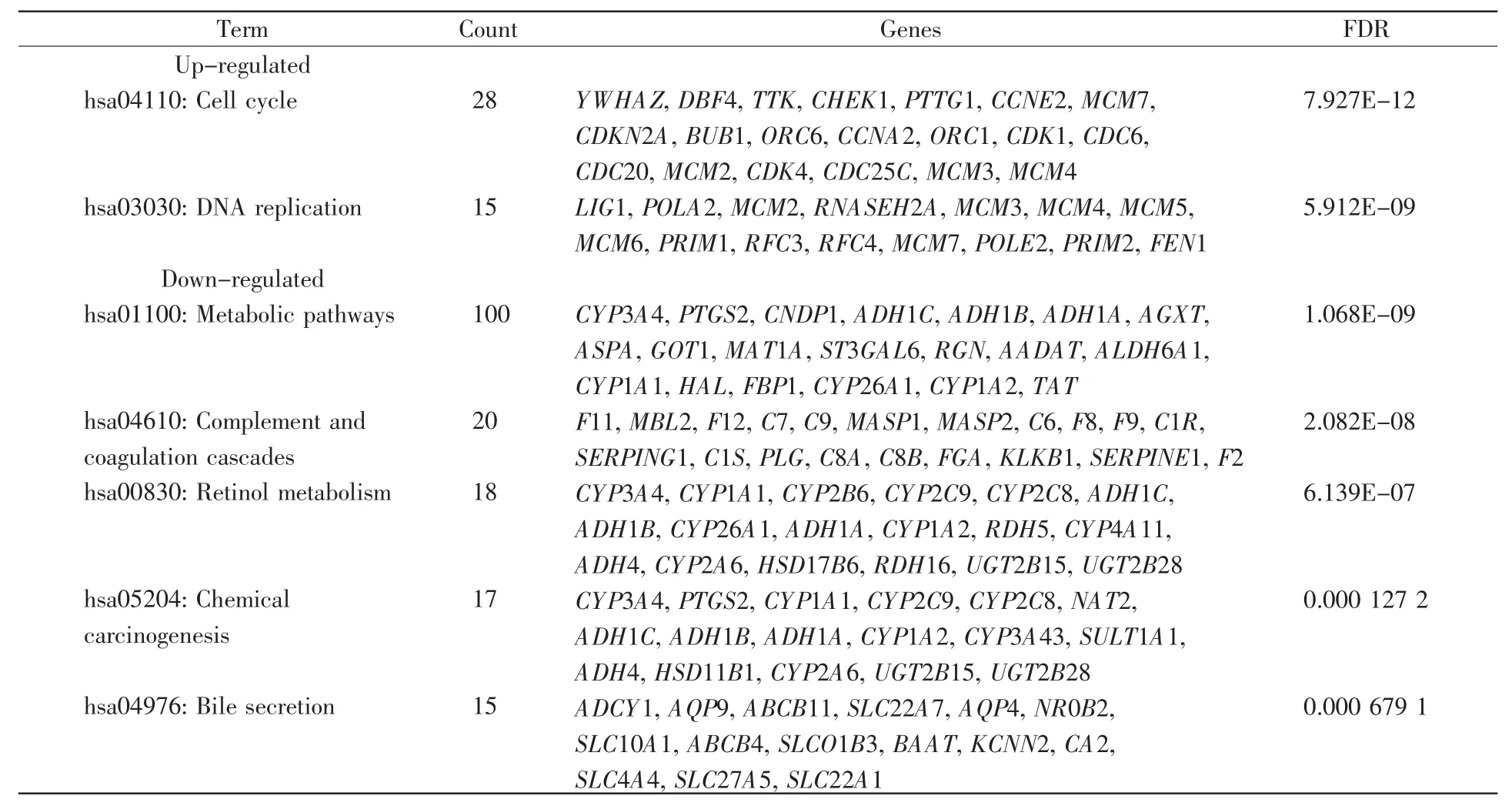
表3 前5位差异表达基因的KEGG通路富集Table 3 KEGG pathway analysis of DEGs(Top 5)
2.5 Hub genes与预后的相关性
利用KM plotter数据库中纳入的364例肝癌患者的基因表达及临床数据,对hub genes进行总生存期的分析,相关数据如下:BUB1(HR=1.85;95%CI=1.3~2.64;logrank P=0.000 52),BUB1B(HR=1.82;95%CI=1.28~2.59;logrank P=0.000 71),CC-NA2(HR=1.69;95%CI=1.19~2.4;logrank P=0.002 9),CCNB1(HR=1.92;95%CI=1.34~2.74;log-rank P=0.000 26),CCNB2(HR=1.62;95%CI=1.14~2.29;logrank P=0.006 7),CDC20(HR=2.3;95%CI=1.6~3.3;logrank P=3.4E-06),CDK1(HR=1.69;95%CI=1.19~2.4;logrank P=0.002 9),MAD2L1(HR=1.88;95%CI=1.33~2.68;logrank P=0.000 33),PLK1(HR=1.89;95%CI=1.33~2.71;logrank P=0.000 36),以上结果显示9个hub genes在肝癌患者中的表达均上调,各基因logrank P<0.05,说明hub genes高表达组与低表达组肝癌患者生存期差异具有统计学意义,提示hub genes的高表达与肝癌患者预后不良相关(图 3)。
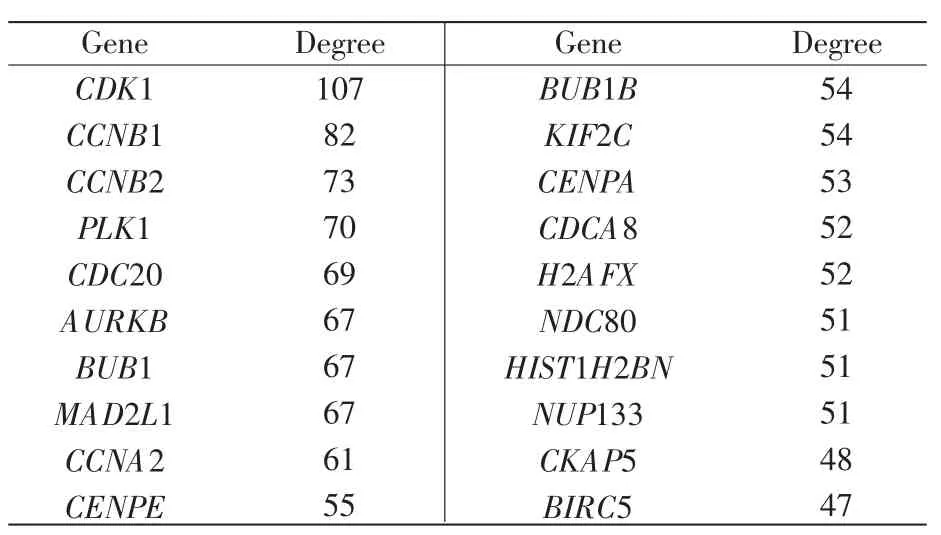
表4 蛋白质互作网络中排名前20的中心节点蛋白质Table 4 The central node proteins in the PPI network(Top 20)

图1 差异表达基因的蛋白质相互作用网络红色节点为表达上调的基因,绿色节点为表达下调的基因。Fig.1 PPI network of DEGsRed nodes denote up-regulated genes,while green nodes denote down-regulated genes.
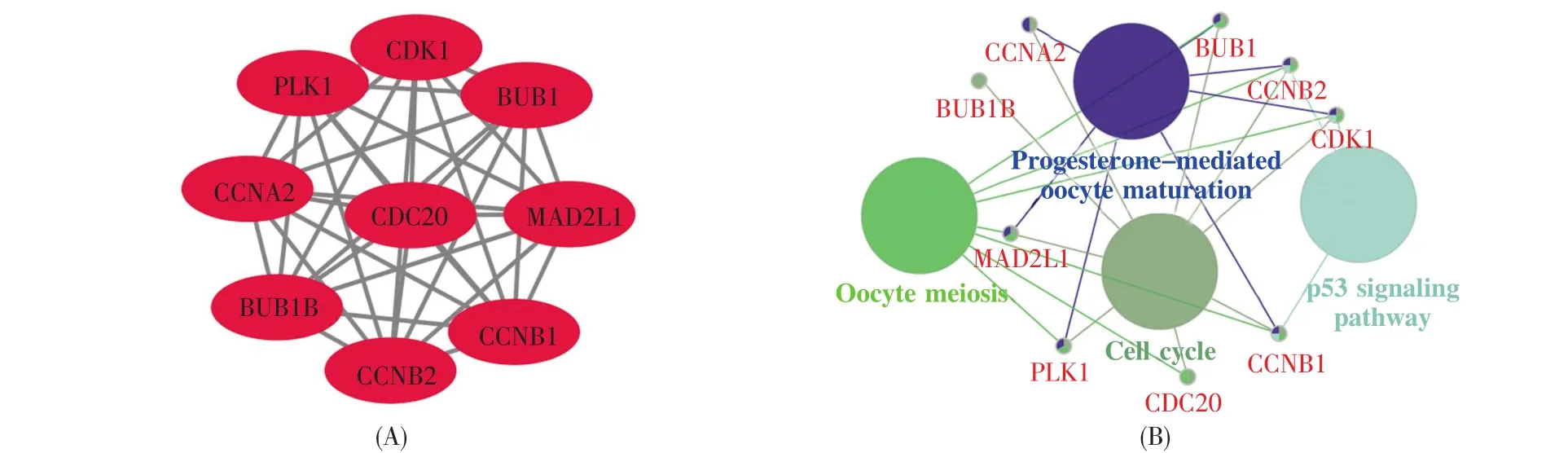
图2 Hub genes分析(A)Hub genes的蛋白质相互作用网络;(B)Hub genes的KEGG通路分析。Fig.2 Result analysis of hub genes(A)PPI network of hub genes;(B)KEGG pathway analysis of hub genes.
3 讨论
肝细胞癌是世界范围内常见恶性肿瘤之一,每年全世界约有778 000例新发病例和745 000例死亡病例,其中仅中国就占一半[1]。虽然肝细胞癌的诊断和治疗技术取得了显著进展,但因其具有浸润、转移和复发的特性,最终导致肝细胞癌患者的预后仍然较差。
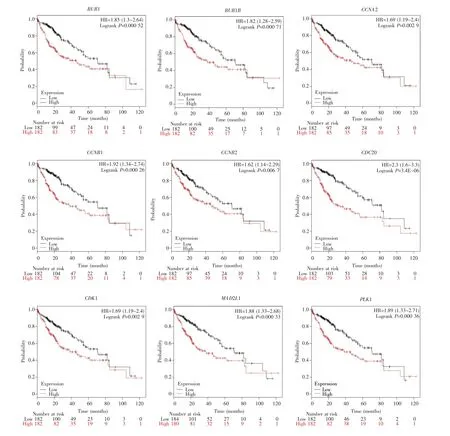
图3 Hub genes的总生存期分析HR:危险比;Logrank P<0.05表明差异具有统计学意义。红色曲线表示高表达组,黑色曲线代表低表达组。Fig.3 Analysis of effects of hub genes on overall survivalHR:Hazard radio;Logrank P<0.05 stands for significant difference.Black and red lines represent low and high expression groups,respectively.
本研究从GEO数据库检索包含肝细胞癌组织和非癌组织的基因表达谱数据GSE84402,利用生物信息学工具进行深入挖掘分析,共筛选出1 307个差异表达基因。通过GO分析发现差异表达基因主要参与了有丝分裂、细胞周期、DNA复制及物质代谢等生物学过程。KEGG通路分析显示差异表达基因主要涉及细胞周期、DNA复制及物质代谢等通路。通路分析结果与GO富集分析结果一致,表明细胞周期、DNA复制及物质代谢等功能的异常,在肝癌发生发展的过程中发挥着重要作用。进一步筛选出BUB1、BUB1B、CCNA2、CCNB1、CCNB2、CDC20、CDK1、MAD2L1、PLK1 等9个hub genes,分析发现9个hub genes均与细胞周期调控相关。有研究表明细胞周期相关蛋白质的异常表达与多种肿瘤的发生和发展密切相关,且多是由于编码蛋白质的上游信号通路的突变或基因损伤所致[20]。采用KM plotter数据库对hub genes进行总生存期的分析,结果显示9个hub genes在肝细胞癌患者中均为高表达基因,且与肝细胞癌的预后显著相关。
BUB1基因编码的丝氨酸/苏氨酸激酶参与细胞有丝分裂过程[21],其表达增加可改变有丝分裂纺锤体组装检查点通路,在细胞增殖和肿瘤进展中发挥重要的作用[22]。Xu等[23]研究发现,miR-490-5p可通过调节肝癌细胞TGFβ/Smad通路,抑制BUB1表达,从而抑制肝癌细胞的增殖、浸润和迁移能力,进一步降低细胞存活率并促进细胞凋亡。BUB1B基因定位于15号染色体的15q15.1区,BUB1B蛋白是一种具有纺锤体分离调控功能的蛋白激酶[24]。大量研究表明BUB1B参与了多种肿瘤形成过程[25~28],但其在肝癌中的作用机制鲜有报道,仍需进一步实验研究。CCNA2在细胞有丝分裂的S/G2期高水平表达,CCNA2异常表达会导致染色体结构不稳定,进而诱导肿瘤的发生[29]。Hung等[30]发现肝癌细胞中精氨琥珀酸裂解酶(argininosuccinate lyase,ASL)与CCNA2相互作用,并通过非酶途径促进肝癌的形成。CCNB1可促进细胞从G2期向M期转变,但在肿瘤细胞中CCNB1过度表达,并与CDK1结合导致细胞无限增殖[31]。Chai等[32]发现沉默肝癌细胞的FOXM1基因可显著降低CCNB1的表达水平,表明CCNB1在FOXM1诱导的肝癌细胞增殖中有重要作用,证明FOXM1-CCNB1与肝癌患者预后不良相关。CCNB2在多种肿瘤组织中过表达,且与肿瘤的侵袭和临床治疗效果差相关,肝癌细胞中KPNA2可通过促进CCNB2/CDK1的表达,诱导肝癌细胞增殖,且与肝癌患者总生存时间和无病生存时间显著降低有关[33]。CDC20编码与细胞周期的APC/C相互作用的调节蛋白,肝细胞中CDC20高表达可加速细胞增殖,促进肝癌的发生和发展,但其分子机制及预后意义有待深入研究[34]。CDK1是细胞周期的重要调节因子[35~36]。研究表明:肝细胞癌组织的CDK1表达水平明显高于非癌组织,CDK1在凋亡蛋白诱导的肝癌细胞凋亡中起着重要的调控作用[37];CDK1的高表达与肝癌患者整体存活率低有关,利用CDK1抑制剂阻断CDK1/pdk1/β-Cat信号转导,可提高索拉菲尼治疗肝癌临床前模型的疗效,为临床提出个性化治疗方案提供依据[38]。MAD2L1被认为是染色体控制通路的重要介质,MDA2L1表达水平异常可导致染色体不稳定,并促进肿瘤的进展,其在肝癌中的表达水平与肿瘤的大小、分期和分级密切相关[39]。Li等[40]报道MDA2L1在肝癌组织和细胞中高表达,miR-200c-5p可通过下调MAD2L1的表达,抑制肝癌细胞增殖、迁移和浸润,并促进细胞凋亡。PLK1被认为是肝癌中的一个重要致癌因子[41],其在调节细胞有丝分裂、胞质分裂、DNA损伤应答等细胞周期过程中发挥重要作用[42~43]。研究发现抑制PLK1表达,可抑制肝癌细胞增殖[44];PLK1可作为肝癌的预后标志物与治疗靶点,但其作用机制仍需进一步阐明[45~46]。
综上所述,本研究利用生物信息学的方法对肝细胞癌的基因表达谱数据进行挖掘和分析,得到了1 307个差异表达基因,其中BUB1、BUB1B、CCNA2、CCNB1、CCNB2、CDC20、CDK1、MAD2L1和PLK1等9个hub genes经总生存期分析发现与肝细胞癌患者预后不良有关,该结果为揭示肝细胞癌发生和发展的分子机制,筛选有效的诊断和预后相关的生物标志物及药物治疗靶点提供了参考。
