不同遗传背景猪TNF-α基因启动子区多态性与其血清含量的关联分析
2019-04-02李梦姝孙福亮柳俭强李兆华金海国张立春
李梦姝,孙福亮,柳俭强,曹 阳,李兆华,金海国,张立春*
(1.延边大学农学院,吉林延边130021;2.吉林省农业科学院,吉林公主岭136100)
肿瘤坏死因子(Tumor necrosis factor,TNF)是由活化的单核- 巨噬细胞分泌的一种细胞因子。TNF-α 在机体天然免疫系统中起重要的作用,除了能够杀伤肿瘤细胞,刺激单核- 巨噬细胞分泌细胞因子外,还能够诱导促炎症反应及多种免疫级联反应,是维持机体稳定、抵御各种致病因子必不可少的免疫调节因子,在炎性反应、细胞免疫、肿瘤免疫等多种生理和病理过程中发挥重要作用[1]。与此同时TNF-α 基因异常同样可以对机体造成严重危害[2]。研究报道表明TNF-α 的启动子区多态性与多种疾病的易感性密切相关。人及其它物种TNF-α 基因均含有丰富的多态性[3-5]。研究显示猪TNF-α 基因启动子区的G-791 位点多态性影响TNF-α 基因mRNA 的表达,提示TNF-α 基因多态性可以作为抗逆性相关的遗传标记[6]。本研究室前期的实验结果同样表明在相同条件下,不同遗传背景猪群血清TNF-α 含量存在极显著差异,表现为野杂猪和民猪极显著高于大白猪,推测野杂猪和民猪TNF-α 基因启动子区可能存在促进其转录翻译的调控元件并可能在高抗逆性状构成中发挥一定的作用[7]。对3 个猪群体TNF-α编码区的序列分析并未显示其存在单核苷酸多态位点(SNP)。基于此,本实验采用高分辨率熔解曲线方法(HRM)分析了不同遗传背景猪的TNF-α 基因启动子区多态性,及该多态性与血清中TNF-α 含量的相关性,为深入研究TNF-α 基因抗病功能、不同遗传背景猪群体抗病抗逆性状的遗传机制奠定基础。
1 材料与方法
1.1 实验动物及实验样品采集 野猪(含75 %长白山野猪血统)与当地民猪的杂交猪(简称野杂猪)(30头)、民猪(25 头)及纯种大白猪健康后备母猪(29 头)分别来自吉林省白山市隆兴牧业有限公司兴隆猪场、吉林省农业科学院畜牧分院民猪保种场和四平红嘴集团梨树种猪场,均为5 月龄~6 月龄。实验样品为3 个猪群体外周血,部分用于分离血清,采用ELISA 法检测血清TNF-α 含量,部分血液样品经ACD 抗凝,按照DNA 提取试剂盒说明书进行外周血DNA 的提取。
1.2 主要试剂 血液DNA 提取试剂盒购自Axygen公司;TaqMasterMix、DNA Marker 均购自康为世纪生物科技有限公司;基因分型试剂盒Light cycler 480 High Resolution Melting Master 购自罗氏公司。
1.3 引物的设计与合成 参照GenBank 数据库中猪TNF-α 基因启动子区序列(GQ339057.1)设计引物TNF-α-P1 及多态性检测引物TNF-α-P2 (表1)。引物均由新海基因检测有限公司合成。
1.4 不同遗传背景猪群体TNF-α 基因扩增及其SNP 鉴定 以野杂猪、民猪及大白猪外周血的DNA 为模板,以TNF-α-P1 为引物扩增其TNF-α 基因启动子区。PCR 产物经琼脂糖凝胶电泳检测、回收纯化后由金唯智生物公司测序,测序结果经DNAStar 软件分析,寻找TNF-α 基因启动子SNP 位点并对其中的G-1256、G-1239 进行基因多态性分析。
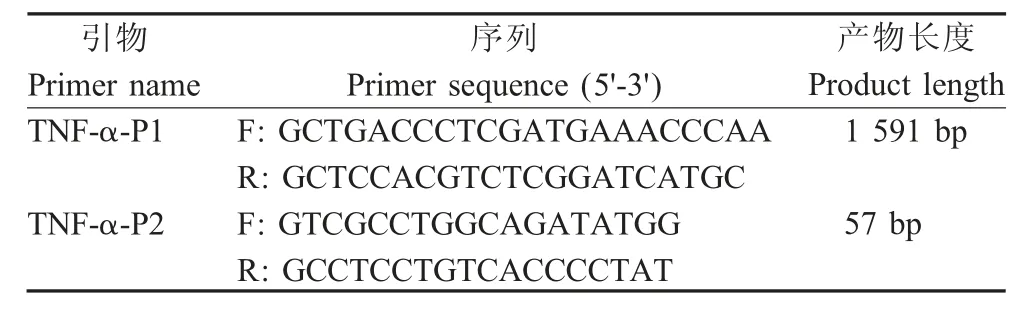
表1 引物序列信息Table 1 Information of the primers
1.5 TNF-α 基因启动子区多态性检测及分析 采用HRM 法利用TNF-α-P2 的上下游引物PCR 扩增TNF-α 基因启动子区,扩增结束后进行HRM 分型,分型条件为:95 ℃1 min,40 ℃1 min,65 ℃1 s;在65 ℃升温至95 ℃的过程中以25 次/℃的速度收集荧光,最后降温至40 ℃。检测结果经Gene scanning 软件进行分析,根据得到的基因型,挑选部分样品由金唯智生物公司测序分析。
1.6 数据统计 采用SPSS23 软件,经单因素方差分析方法分别对3 个不同群体猪个体TNF-α 基因启动子区的基因型与其血清TNF-α 含量进行差异显著性分析。
1.7 TNF-α 基因启动子转录因子结合位点预测利用转录因子数据库JASPAR (http://jaspar.genereg.net),分析本实验获得的TNF-α 基因启动子区的单倍型个体核苷酸序列,遴选出因碱基突变导致的潜在转录因子结合位点的变化。
2 结 果
2.1 不同遗传背景猪群体TNF-α 基因启动子区克隆与其SNP 鉴定 以野杂猪、民猪及大白猪外周血基因组DNA 为模板,PCR 扩增出该3 个群体TNF-α 基因启动子区,片段约为1.6 kb (图1)。对PCR 产物测序并进行多态位点筛查,结果显示3 个群体TNF-α 基因启动子区存在7 个SNP (表2)。表明3 个不同群体猪TNF-α 基因启动子区存在碱基突变,因此对其进行后续研究。

图1 TNF-α 基因启动子区的PCR 扩增Fig.1 Amplification of TNF-α gene promoter region by PCR
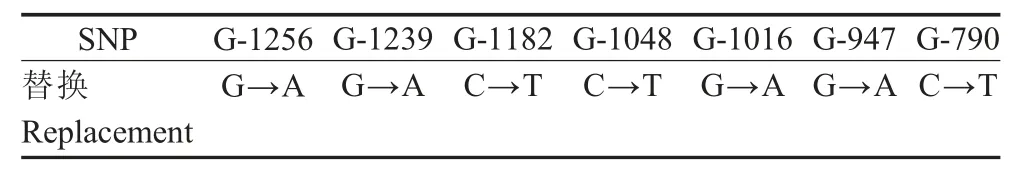
表2 TNF-α 基因启动子区的SNPTable 2 The SNP of TNF-α
2.2 不同遗传背景猪群体TNF-α 基因启动子多态性分析 选取TNF-α 基因启动子G-1256、G-1239位点区作为研究对象,通过HRM 方法对3 个不同遗传背景猪群体进行基因型检测。Gene scaning 分析结果显示3 个群体猪中该区域基因型主要分为4种类型,依次命名为AA、BB、CC 和DD (图2),且3 个群体猪的该区域基因型频率存在明显差异。其中CC 基因型在3 个猪群体中均表现为优势基因型,尤以野杂猪群体CC 基因型频率最高,达73 %(22/30),民猪群体次之,达52 % (13/25),大白猪最低,仅31 % (9/29)(表3)。 将PCR 产物测序分析,结果显示BB 和CC 基因型个体为纯合型,G-1256位点均为G,G-1239 位点分别是G 和A,DD 基因型G-1239 位点为A/G 双峰,表明DD 基因型为BB、CC 基因型的杂合型。AA 基因型则相对复杂,其在G-1256 和G-1239 位点均表现为A/G 双峰(图2)。由于该区域基因型由2 个SNP 组成,结合测序结果显示3 个群体中该区域实际上存在3 种单倍型(Haplotype),即:GG、GA 和AG。但鉴于AA 基因型个体数量稀少且仅在大白猪群体中存在,推测G-1256 A 可能在大白猪品种培育过程中通过突变产生了新的多态位点。由于不同遗传背景猪TNF-α 基因启动子区基因型种类与频率的差异性,提示自然状态下该区域可能承受较大的环境选择压力。
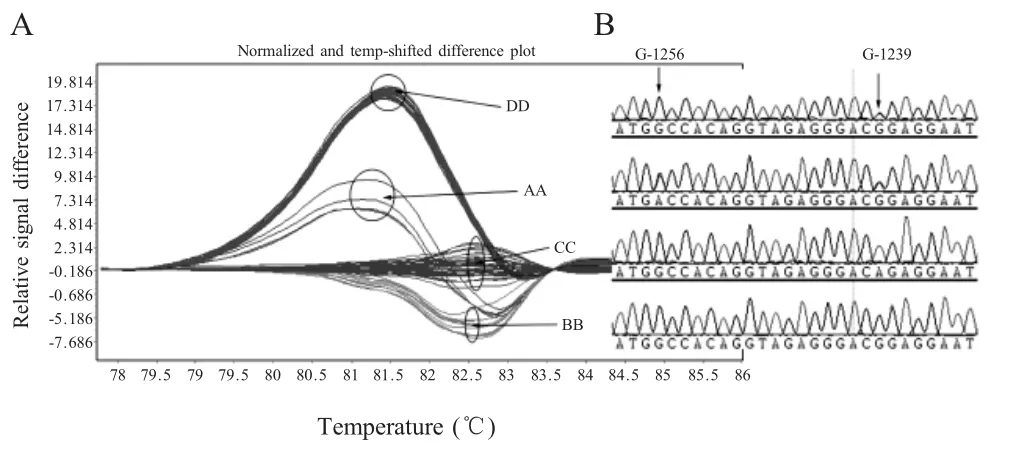
图2 3 个猪群体TNF-α 基因启动子区HRM 分型(A)与测序结果(B)Fig.2 The genotyping (A)and sequencing (B)of TNF-α gene promoter region from three pig populations by HRM
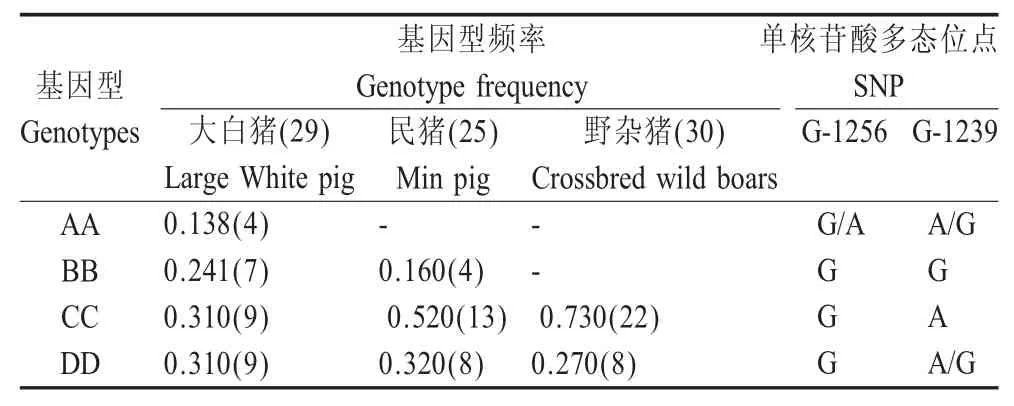
表3 3 个群体猪TNF-α 基因启动子区基因型频率及SNP 分析Table 3 The genotype frequences and the polymorphisms on TNF-α gene promoter region in three pig population
2.3 TNF-α 基因启动子多态性与血清TNF-α 含量相关性分析 为探究该基因座位多态性是否对TNF-α 基因转录及分泌产生影响,本实验进一步以3 个群体不同个体的TNF-α 基因启动子区基因型与血清中该基因含量进行相关性分析。3 个群体分别进行独立单因素方差分析结果显示,野杂猪和大白猪TNF-α 基因启动子区CC 基因型个体血清TNF-α含量显著高于其它基因型个体(p<0.05)。尽管民猪CC 基因型个体血清中该基因含量与其它品种猪相比并未表现出显著性差异,但其血清TNF-α 含量却表现出高于其它基因型个体的趋势(图3)。将3 个群体作为一个整体进行单因素方差分析同样显示CC基因型个体血清TNF-α 含量显著高于BB 基因型及DD 基因型(p<0.05)。提示CC 基因型在提高TNF-α基因转录表达方面可能具有普遍性。
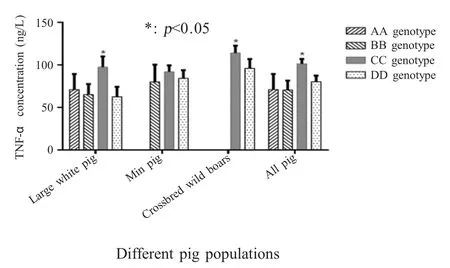
图3 3 个群体不同基因型猪血清TNF-α 的含量差异显著性分析Fig.3 The contrast of TNF-α in the peripheral blood of different genotype in three pig populations
2.4 TNF-α 基因启动子区潜在转录因子结合位点预测 截取一定长度GG、GA 和AG 单倍型序列,利用JASPAR 数据库在线分析工具进行潜在转录因子结合位点预测。结果显示不同单倍型序列潜在转录因子结合位点存在较大差异。其中GA 单倍型预测潜在转录因子27 个;GG 单倍型30 个;AG 单倍型16 个。其中3 种类型单倍型共同拥有1 个转录因子,GA 和GG 单倍型共有20 个转录因子潜在识别位点;GG 和AG 共有9 个;GA 和AG 单倍型则没有(图4)。进一步分析显示仅GA 和AG 单倍型存在自身特异性转录因子。其中GA 单倍型存在与免疫相关的潜在NF-κB 亚基RelB 因子结合位点,而AG单倍型潜在转录因子识别位点与免疫因子转录调控无关,表明RelB 因子在TNF-α 基因的转录中起重要调控作用。
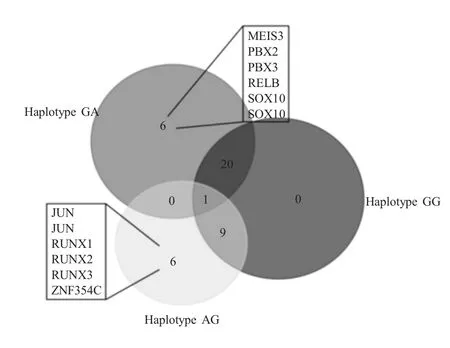
图4 不同单倍型序列转录因子预测与分析Fig.4 Prediction and analysis of transcription factors of different haplotypes sequences
3 讨 论
我国地方猪种类资源丰富、种质特性鲜明、抗病性能高,猪的抗病抗逆性状是由微效多基因控制的数量性状[8],目前尚未对地方猪抗病性状进行有效解析。本研究室在前期实验中发现在相同条件下,野杂猪、民猪及大白猪健康群体血清TNF-α 含量存在显著差异,提示TNF-α 可能参与高抗病性状的构建[7]。本实验对该3 种猪的TNF-α 基因启动子区多态性进行检验,并经统计学方法分析了血清TNF-α 含量。结果显示3 个群体TNF-α 启动子区不同基因型与血清TNF-α 含量存在一定的相关性,为解析不同遗传背景猪血清TNF-α 含量差异的遗传基础提供了参考依据。
目前猪TNF-α 基因编码区尚未发现存在基因多态性,基因多态性主要存在于其启动子区。Szydlowsk 等发现大白猪TNF-α 基因启动子区G-791 位点发生的C→T 突变,该位点可能与大白猪背膘厚等经济性状相关[6]。而孙丽等利用PCR-SSCP 方法分析了大白猪TNF-α 基因启动子区G-791 位点的多态性,结果表明G-791 位点可能影响TNF-α 基因mRNA 的表达且TT 基因型个体TNF-α 基因mRNA 在十二指肠和空肠显著上调表达[9]。除大白猪外,其它品种猪TNF-α 基因启动子区同样存在多态位点。牛步月等利用直接测序法证实民猪、长白猪TNF-α基因5' 侧翼区存在G-1048 多态位点,并进一步证实不同基因型对仔猪腹泻和生长性状有一定影响[10]。本实验利用测序对野杂猪、民猪和大白猪TNF-α 基因启动子区进行了多态性筛查,结果表明3 个群体猪该区段的确存在SNP。本实验通过测序方法在受试猪群体TNF-α 基因启动子区发现共计7 个SNP,但对上述3 个群体猪TNF-α 基因编码区进行克隆,测序结果显示编码区不存在包括无义突变在内的任何碱基突变,表明不同遗传背景猪TNF-α 基因氨基酸序列的保守性(数据未显示)。提示猪TNF-α 基因启动子区受到的选择压力较小,且其TNF-α 基因可能主要通过调节基因表达而非功能改变来调节其在免疫反应中的功能。
不同于传统的PCR-SSCP、PCR-RFLP 等SNP 研究方法,本实验采用低成本、高通量、快速的HRM法分析了TNF-α 基因启动子G-1256 和G-1239 区的基因型,显示3 个猪群基因型及基因型频率存在较大差别:大白猪群体存在4 种基因型,民猪3 种,野杂猪则只有2 种。本实验中AA、BB 新基因型在民猪和大白猪群体内的出现提示随着选育进程的增加,该区域SNP 可能经历从开始产生并逐渐增加的过程。目前普遍认为猪驯化或选育过程中一般伴随着抗病性能及抗性相关基因遗传多样性能丢失,本研究室对上述3 个猪群体Mx1基因的研究也证明了此观点[11]。但本实验结果似乎提示TNF-α 基因启动子区随着选育进程的增加其遗传多样性也呈现增加的趋势。综合考虑不同基因型与血清TNF-α 含量相关性分析结果即可推测出新的突变位点G-1256 A 的出现很可能抑制了TNF-α 基因转录表达,从而间接降低了血清TNF-α 含量,表明新的突变位点极有可能是有害突变。也从侧面提示在现代猪品种选育,规模化饲养模式及环境控制水平不断提升的客观条件下,可能降低了包括TNF-α 基因在内的相关免疫基因的环境选择压力,从而导致猪种环境适应能力的降低。TNF-α 基因转录受到NF-κB 信号通路的调控,转录因子结合位点分析显示G-1239 A 构成NF-κB 信号通路转录因子RelB 潜在结合位点,而该位点所构成的CC 基因型在野杂猪中频率最高,结合CC 基因型个体在猪群体中血清TNF-α 含量显著上升的结果,提示RelB 在TNF-α 基因转录调控过程发挥着重要作用,也间接表明TNF-α 基因启动子区NF-κB 转录信号识别区的增减对基因的转录调控的重要作用。考虑到高抗逆表型猪群(野杂猪)具有更高的血清TNF-α 含量,且该表型猪群优势基因型(CC 基因型)构成RelB 潜在结合位点,表明NF-κB信号通路在抗逆表型构成中可能具有重要作用,但其具体功能尚需进一步的实验验证。
