不同来源中国李(Prunus salicina L.)的多样性与近缘种关系
2019-02-20魏潇章秋平刘宁张玉萍徐铭刘硕张玉君马小雪刘威生
魏潇,章秋平,刘宁,张玉萍,徐铭,刘硕,张玉君,马小雪,刘威生
不同来源中国李(L.)的多样性与近缘种关系
魏潇,章秋平,刘宁,张玉萍,徐铭,刘硕,张玉君,马小雪,刘威生
(辽宁省果树科学研究所,辽宁营口 115009)
【目的】中国李资源丰富、分布广泛。更好地明晰不同来源中国李栽培品种的多样性、遗传结构差异以及与同域近缘种的关系,将有利于明确中国李驯化扩散历程以及近缘种在栽培驯化过程中的作用,促进中国李地方品种资源的深入挖掘和新品种的选育。【方法】利用均匀分布于基因组的22对SSR分子标记,采用荧光毛细管电泳检测技术对48份种质进行基因分型,其中包括38份不同来源的中国李种质、10份变异类型或近缘种。通过GenAlEx 6.41软件评估22对SSR引物的多态性,对参试种质按不同来源分析遗传多样性;利用NTSYS-pc 2.1软件构建48份材料的树状聚类分析图;并根据贝叶斯模型的Structure 2.2软件分析不同居群间的遗传结构差异。【结果】基于48份供试材料的数据,22对SSR引物等位变异范围为3—21个,平均每个位点检测到13.54个;总共检测到298个等位变异,其中有51.8%的等位变异属于稀有等位变异。在不同居群间进行比较,根据平均有效等位变异()、平均Shannon’s多样性指数()、观察杂合度()和期望杂合度()可以看出,南方品种群的多样性最高,其次为东北品种群;而杏李的多样性最低,且明显低于华北品种群。通过分子方差分析,认为中国李的多样性有69%的遗传变异来源于居群内,仅有31%的遗传变异来源于居群间。基于遗传分化系数和s遗传距离的数据比较,认为不同居群间存在显著的遗传分化,同时不同地理来源种质间存在适当的基因交流。树状聚类分析暗示国外育成品种与我国南方品种群具有较近的亲缘关系;而华北品种群与杏李关系密切;东北品种群与乌苏里李关系紧密。群体结构分析可以将栽培中国李种质资源划分为南方小果脆肉品种群、南方大果品种群(包括国外育成品种)、华北品种群和东北品种群。【结论】我国南方地区中国李的多样性最为丰富,按东北品种群、国外品种群、华北品种群顺序依次降低。东北品种群为了提高适应性融入了乌苏里李基因;杏李是从华北品种群中高度驯化后的特化类型,且该类型通过无性繁殖保存了其高度杂合性状态。我国南方江浙地区的大果型种质对国外育成品种起着重要作用。
中国李;遗传多样性;群体结构;基因流
0 引言
【研究意义】中国李(L.)起源于我国,是世界上栽培最广泛的李亚属(Subgenus)李组(Section)植物,均为二倍体(2n=16)[1]。在我国,南自广东、广西和云贵高原,北至黑龙江省牡丹江等地均有中国李栽培,地区分布广泛[2]。与中国李相比,近缘种杏李()和乌苏里李()却属于局域种(或类型),分布范围非常有限。研究中国李不同居群间的遗传结构以及近缘种在中国李扩散过程中的作用,对地方品种资源的深入挖掘和新品种选育具有重要意义。【前人研究进展】前人已经利用RAPD标记[3-4]和ISSR标记[5]对李亚属不同物种间的亲缘关系进行了研究。杏李()是原产于我国华北地区的栽培种,常被推测为普通杏和中国李种间杂交形成[6],也有人认为是中国李的变种[5]。乌苏里李()是生长在我国黑龙江等地的一种野生种,其具有极强的抗寒能力[2],但FAUST等[7]认为其是中国李的一个变种,而非独立种。然而,杏李、乌苏里李与同域分布的中国李之间的基因交流情况还有待深入研究。有关中国李不同品种群间演化关系的报道较少。郭忠仁[8]通过孢粉学观察认为中国李的传播是从南向北进行的,南方中国李最原始。乔玉山[9]将中国李品种分为南方品种群和北方品种群两大类。基于分子标记的多样性将中国李划分为3种不同类群,即东北品种群、华北品种群和南方品种群[10-11]。陈圣林等[12]对三峡库区的14份中国李品种进行分析后,发现该区域地方品种与国外引进品种存在明显差异。事实上,国外栽培品种是我国某地区栽培李经由日本传到美国后与近缘种美洲李()或樱桃李()杂交改良所形成[13],但是从中国何地引入并没有确切记录。【本研究切入点】以往对中国李的研究仅局限于中国李内部多样性比较和简单的类群划分,有关中国李不同品种群间的多样性差异、群体结构以及近缘种在中国李驯化中的作用还不清楚。【拟解决的关键问题】本研究以近缘种为对照,分析中国李驯化扩散过程中不同品种群间的多样性差异,明确近缘种在栽培驯化中的作用。
1 材料与方法
试验于2015年5月至2016年7月在辽宁省果树科学研究所国家果树种质熊岳李杏圃与辽宁省北方果树资源与育种重点实验室进行。
1.1 试材及取样
本试验材料共48份,其中美洲李(,2n=16)、樱桃李(,2n=16)和普通杏(,2n=16)各取1份材料为组外对照。依据中国李初级核心种质筛选结果[14],从不同来源的中国李材料中选取各地生产中主要栽培品种,其中华北品种群5份、东北品种群11份、南方品种群17份、国外育成品种5份,共计38份。另外,近缘种包括5份杏李(,2n=16)和2份乌苏里李(,2n=16)(表1)。

表1 供试材料信息
括号内为居群缩写。下同 The abbreviation of population is in brackets. The same as below
1.2 DNA提取及SSR基因型检测
于2015年春季,采集不同样品幼嫩叶片,并用硅胶干燥保存。采用DOYLE等[15]的改良CTAB法提取总基因组DNA;利用紫外分光光度计和1%浓度的琼脂糖凝胶电泳检测DNA质量和浓度,并稀释至20 ng·μL-1。
本研究中,参照SCHUELKE等[16]设计带有M13接头的SSR引物(Tailed primer M13 microsatellite markers,TP-M13-SSR):即正向引物的3′末端增加一段序列为TGTAAAACGACGGCCAGT的接头引物;反向引物序列不变。22对SSR引物均来自属公共图谱不同染色体上,具体信息见表2。PCR反应采用20 μL反应体系,其中DNA模板5 μL、2×Taq Master Mix(北京康为世纪生物公司)10 μL、带M13接头的正向引物0.4 μL、通用M13荧光引物1.6 μL、反向引物2 μL。PCR扩增反应程序如下:在95℃下解链5 min;整个PCR反应共40个循环,前15个循环为94℃ 45 s,55℃ 45 s,72℃ 45 s,后25个循环为94℃ 45 s,52℃ 45 s,72℃ 45 s;最后,在72℃下延伸10 min,保存于4℃。

表2 SSR位点的多样性指数
SSR位点、连锁群和位置均来自于属参照图谱(T×E图谱)的信息,具体参见Dirlewanger等[17],位点后文献序号是该位点引物信息来源。o:总等位变异数;:有效等位变异数;:Shannon’s指数;:观察杂合度;:期望杂合度;:Wright’s固定系数;:基因流。下同
The locus, linkage groups and locations in the table were derived from thereference map (T×E map), as shown in Dirlewanger.[17], and the literature number was the information sources of primers in locus.: Total number of alleles;: Effective number of alleles;: Shannon’s information index;: Observed heterozygosity;: Expected heterozygosity;: Wright’s Fixation index;: Gene flow. The same as below
上机前对PCR产物进行变性和纯化[25]。将纯化的PCR产物按比例稀释30倍后,取1 μL纯化后的PCR产物、0.12 μL Liz-500 Size-Standard内标和7 μL的甲酰胺进行混合,在95℃下变性5 min,并立即冷却10 min。利用ABI 3730 DNA遗传分析仪进行荧光电泳检测,并利用ABI GeneMapper V3.0软件与Peak Scanner Software V1.0软件对电泳检测结果进行基因型分型整理。
1.3 数据整理及分析
利用GenAlEx 6.41软件整理基因型数据,计算不同位点或不同居群间的多样性指数、AMOVA(analysis of molecular variance)分析以及不同居群间的Nei’s遗传距离和遗传分化系数等[26]。
采用NTSYS-pc 2.1软件对48份材料构建聚类分析图;利用Structure 2.2软件计算不同居群间的遗传结构差异[27],选择混合祖先模型(ancestry admixture models)和相关等位变异频率模型(allele frequencies correlated model)运算。设定种群分类亚群数(K值)为1到10变化,每次种群划分重复运行10次,每次运行迭代次数(markov chain monte carlo,MCMC)为100 000,运行步长(length of burnin peroid)为100 000。依据Evanno等[28]的方法,利用每个K值所对应的LnPD(D)值计算不同分类亚群的ΔK,并绘制ΔK趋势图。按照Earl等[29]的方法,利用CLUMPP软件进行重复抽样,使用DISTRUCT软件绘制柱形图。
2 结果
2.1 SSR位点的多态性比较
在本研究中,利用22对SSR标记检测了48份不同种质的基因型,共获得298个等位变异,每位点平均检测到13.54个。不同位点上检测到的等位变异数量明显不同,其中PceGA025、CPDCT016和CPDCT028位点检测到的等位变异数最多,均为21个;而CPDCT027位点仅检测到3个等位变异(表2)。有效等位变异()变异范围为1.889(CPDCT027位点)—12.03(PceGA025位点),整个群体有143.50个,平均有效等位变异数为6.52。与检测到的总等位变异数相比,有51.8%的等位变异属于稀有等位变异。Shannon’s多样性指数()变异范围从PceGA025位点的2.969到CPDCT027位点的0.703,整个群体不同位点上平均Shannon’s多样性指数为2.002,但大多数位点上的Shannon’s多样性指数均较小(表2)。
观察杂合度()变异范围从CPSCT039位点的0.958到CPDCT027位点的0.333,整个群体不同位点上的平均值为0.686;而期望杂合度()变异范围从PceGA025位点的0.917到CPDCT027位点的0.471,整个群体不同位点上的平均值为0.798(表2)。大多数SSR位点上的值小于值,表明供试材料在大多数位点上表现出杂合度不足。从Wright’s固定系数值可以看出,多数位点符合Hardy-Weinberg平衡(接近于0)。除了CPSCT004位点的基因流值大于1外,其余SSR位点上的基因流值均小于1,表明不同SSR位点上的等位变异在供试材料间存在着遗传分化。
2.2 不同品种群间的多样性比较
从表3可以看出,不同品种群间的多样性存在明显差异。通过平均有效等位变异()和平均Shannon’s多样性指数()可以看出,南方品种群的多样性最高,其次为东北品种群;而杏李的多样性最低,且明显低于华北品种群。通过观察杂合度和期望杂合度可以看出,南方品种群和东北品种群具有较高的遗传多样性,而华北品种群与国外品种群相对较低。杏李多样性最低,且观察杂合度远远高于期望杂合度,这种杂合度过量可能与居群间的基因交流相关。该居群固定系数=-1,表明该居群内繁衰退明显,且与外居群间无任何基因交流。

表3 不同品种群体间的多样性比较
2.3 不同品种群间的遗传分化
为了分析不同品种群间的多样性差异来源以及近缘种对栽培品种群的影响,通过AMOVA分析,不同品种群间的遗传差异达到极显著水平(F=3.79,<0.01),但是大部分遗传多样性仍主要来源于居群内变异(69%),居群间的遗传变异仅有31%。
遗传分化系数是衡量不同居群间的遗传分化程度,一般认为,当大于0.05时,居群间存在着中等遗传分化;当大于0.15时居群间存在较大的遗传分化[30]。通过表4可以看出,不同栽培中国李居群间均存在着适当的遗传分化,其中国外品种群与南方品种群的遗传分化程度最小;而乌苏里李与所有中国李品种群存在较大的遗传分化;杏李则与北方品种群间有较小的遗传分化,与其余品种群间存在较大的遗传分化程度。同时,s遗传距离显示乌苏里李相对东北品种群、杏李相对北方品种群均存在较小的距离。
2.4 基于核SSR的聚类分析
基于SSR基因分型结果对所有中国李及其近缘种进行Jaccard遗传相似系数检测。以普通杏、美洲李和樱桃李为外组对照,对45份中国李及近缘种进行聚类分析,构建树状亲缘关系图。在UPGMA聚类图(图1)中,当阈值为0.83时,将供试材料划分为5个类群,第I类群包括杏李和所有华北品种群材料;第II类主要为以布尔班克为基础的国外育成品种,同时也包括浙江一带的天目蜜李、银醉李等大果型品种;第III类全部为我国南方地方品种,包括西南的青冬李等脆肉型品种、广东三华李、福建㮈李和芙蓉李等类型;第IV类群则包含东北品种和2份乌苏里李;第V类群仅有1份庐山李,其与其他品种聚类距离最远;自聚类树根部依次为组外对照材料普通杏、美洲李和樱桃李。树状聚类图表明,国外育成的中国李品种与我国南方地方品种具有较近的亲缘关系;而北方品种群与杏李关系密切;东北品种群与乌苏里李关系紧密。

图1 基于SSR标记和UPGMA聚类分析48份材料的树形图

表4 不同居群间的Nei’s遗传距离(右上角)与遗传分化系数Fst值(左下角)比较
2.5 不同来源种质间的群体结构
利用混合祖先模型和等位变异发生频率模型推测中国李不同种质间的群体结构。根据Evanno等[28]的方法,绘制∆K变化趋势图(图2)推测最佳分类群数。当群体数K=4时,∆K值最大,认为本研究的中国李种质资源划分为4类较为合理,即华北品种群、东北品种群、南方小果脆肉品种群、南方大果品种群。为了更清楚地阐明不同种质材料间的遗传关系,比较了在不同假设分类群条件下,供试材料的分类划分情况,如图3所示。当K=2时,杏李和来自华北的中国李品种(绿色)与其他供试材料分离为两个类群(红色);当K=3时,乌苏里李和来自东北地区的中国李品种(蓝色)从其余材料中分离出来,独立为新类群,这表明华北品种群和东北品种群分别与杏李、乌苏里李具有相似的血统或相近的血缘关系;当K=4时,来自南方的中国李品种被划分为2个类群,第一组为小果脆肉型品种群(橙色),另一组为大果型品种群(红色)。大多数国外育成品种均属于南方大果型品种群;而南方种质中大部分红肉型种质属于以上2种血缘的混合类型。当K=5时,4种不同来源的中国李中均分离出新的血缘类型(黄色),而杏李和乌苏里李并无此类血统。

图2 利用作图法推断参试种质的合理组群划分
3 讨论
3.1 不同来源中国李的多样性比较
在本研究中,22对SSR引物共检测到298个等位变异,平均每个SSR位点检测到13.54个,平均有效等位变异数()为6.52,平均多数性指数为2.0,平均观察杂合度()为0.686,平均期望杂合度()为0.798。本研究中检测到中国李的多样性略高于孙萍等[31]在47份中国李检测的结果(=10.85,=4.81,=0.746,=0.774);也高于Carrasco等[32]检测到的(=12.1,=5.2)。但是,Carrasco等[32]的研究中的和值略高于本研究检测结果,且大于,这可能与该研究中选用的中国李多为育成品种,含有较多的近缘种有关。
陈红等[33]通过ISSR标记认为贵州地区栽培的中国李多样性极为丰富(=0.508),本研究中SSR数据显示南方品种群的Shannon’s多样性指数为1.709。本研究通过不同地理来源居群间的多样性比较(表3),发现我国南方品种群的多样性(=5.141,=0.736)远高于其他居群,而东北品种群、国外品种群、华北品种群的多样性依次降低。郁香荷等[34]分析了405份来自全国各地的中国李资源果实大小变异后,发现单果重变异系数为47.09%,其中南方种质的单果重变异最大,而国外品种群和华北品种群的变异系数最小。结合树状聚类图,发现大部分南方种质资源被聚在第III类群中,包括西南的青冬李等脆肉李、广东三华李、福建㮈李和芙蓉李等类型,但是第II类群中仍有南方品种天目蜜李、银醉李,第I类群有巴塘李,第V类群仅由庐山李组成(图2)。这进一步表明,我国南方种质资源多样性较为丰富,但是这可能受南方品种群取样量大的影响。一般认为,中国李()起源于我国南方长江流域[13]。因此,南方品种群多样性丰富也可能与中国李起源中心有关。在李种质资源考察过程中,笔者在云南、四川一带发现了野生李林的生长,推测我国西南地区可能是中国李的原始驯化中心。
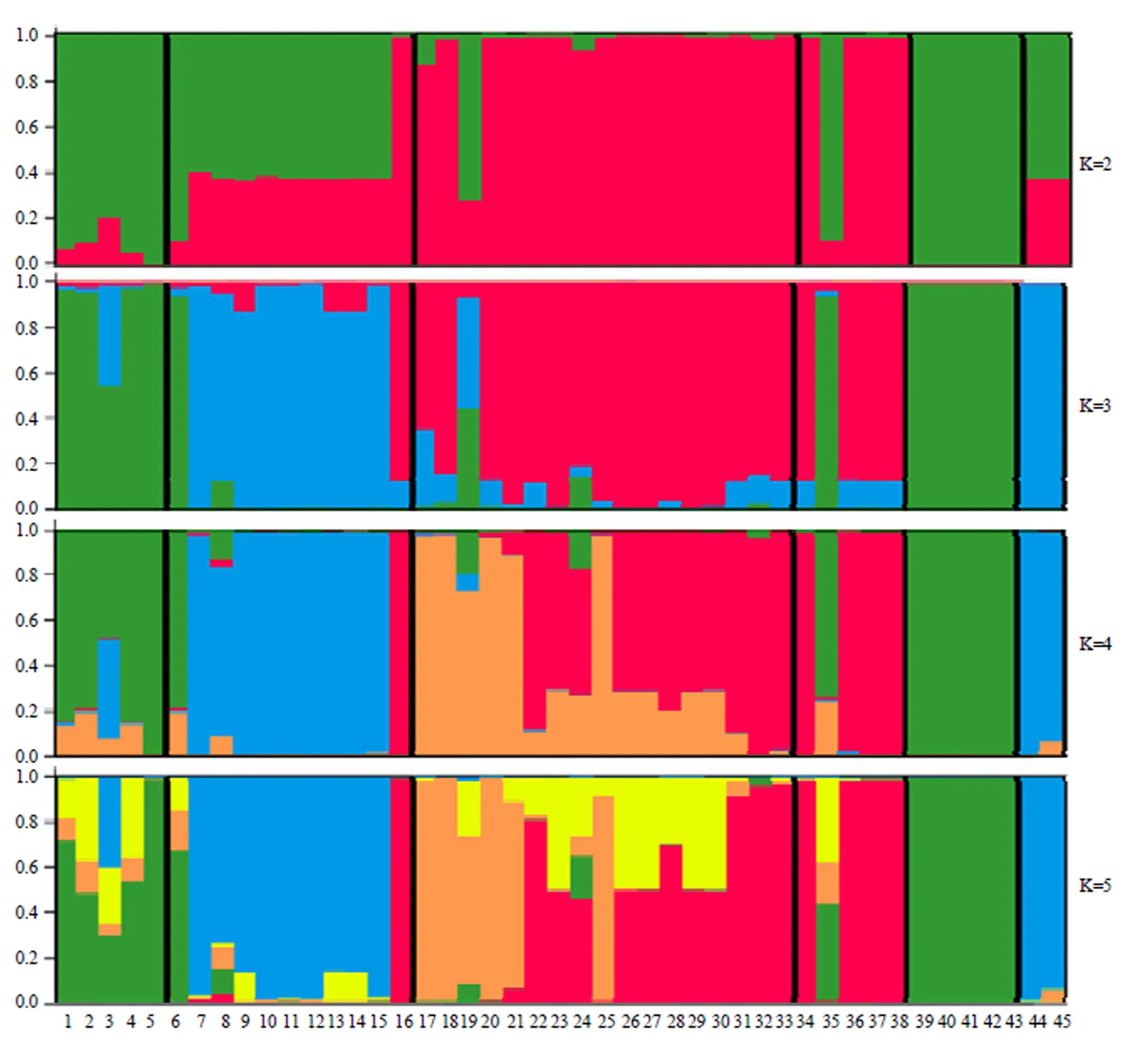
图3 中国李种质资源的群体结构
3.2 中国李不同品种群的形成与近缘种关系
在我国,中国李具有悠久的栽培历史,形成了许多优良地方品种群或类型。乔玉山[9]根据RAPD和ISSR标记将中国李品种分为南方品种群和北方品种群两大类。刘威生[10]则依据SSR标记划分为南方品种群、华北品种群和东北品种群3种不同类型,并且将国外育成品种划归到南方品种群。本研究认为中国李可以划分为华北品种群、东北品种群、南方小果脆肉品种群和南方大果品种群(包括国外引进品种)。郭忠仁[8]在孢粉学观察结果上认为中国李的传播是由南向北进行的,且南方李更原始。在本研究中,不同居群间的多样性是从南方品种群、东北品种群、国外品种群、华北品种群的多样性依次降低。东北品种群的多样性(=1.509)仅次于南方品种群,明显高于华北品种群(=0.977),这可能与生长在东北地区的乌苏里李()有关。在本研究中,乌苏里李与东北品种群中国李的遗传距离和遗传分化系数明显小于其他品种群,这也进一步表明乌苏里李对东北品种群形成具有重要作用。郁香荷等[34]发现东北品种群果实大小显著低于华北品种群,这可能是该品种群为了适应高纬度的严寒气候降低了果实大小的选择压力。因此,推测东北品种群在提高适应性方面融入了野生近缘种乌苏里李的基因。
在本研究中,杏李()的固定系数为-1,表现出极端的完全自交群体,且杏李的基因型高度杂合(=0.773)(表3)。但是,杏李的多样性极低,且与华北品种群遗传分化系数最小。因此,认为杏李是华北品种群中国李高度驯化后的特化类型,且该类型是通过无性繁殖进行传播的[35]。
3.3 南方中国李对现代育成品种形成的作用
SSR标记的亲缘关系分析能够提高育种过程中的亲本选配效率。为了适应果品市场质量需求,从上世纪末至本世纪初以来,我国先后从美国引进大果硬肉型品种,如幸运(Fortune)、安哥诺(Angeleno)和黑宝石(Black Diamond)等具有较强的耐贮运特性,并且这些品种在我国部分地区逐渐成为主栽品种。本研究SSR数据显示国外品种群多样性较低,且与南方品种群的遗传距离最小,仅为0.068。在左力辉等[11]和孙萍等[31]的研究中,也认为国外育成品种与我国南方品种亲缘关系较近。事实上,中国李被世界各国广泛栽培是从我国某地部分李品种通过日本传播到美国开始的[13]。为了提高栽培品种的本地适应性,国外育种者利用本土美洲李()、樱桃李()与仅有的少量中国李进行种间杂交,先后培育出百班克(Baibanke)、美丽李(Beauty)和福摩萨(Formosa)等系列种间杂交品种,这些材料成为国内外现代李育种的主要亲本来源[13]。在本研究中,国外育成品种与南方江浙一带的大果型品种聚类在一起,而与美洲李和樱桃李的关系较远,这可能是由于多代多次杂交后国外育成品种保留了较少的美洲李或樱桃李血缘。另外,以上结果也暗示国外育成品种的间接亲本可能来源于我国南方江浙一带,而我国育种亲本则多选择华北地方品种进行相互杂交,这为我国李育种提供了重要借鉴。本研究选用的标记种类和样本量有限,为此在后续研究中将通过大样本取样和全基因组变异检测(如GBS测序)等途径对以上结论进行进一步验证。
4 结论
南方品种群位于中国李栽培驯化中心,其多样性最为丰富;江浙一带的南方大果型种质对国外品种群的形成具有重要影响。在华北品种群中,通过高度驯化和人工选择形成一类以无性繁殖为基础的、果实香气浓郁的杏李()类型。东北品种群形成则与乌苏里李()基因渗透有关。
[1] REALES A, SARGENT D J, TOBUT K R, RIVERA D. Phylogenetics of Eurasian plums,L. section(Rosaceae),according to coding and non-coding chloroplast DNA sequences., 2010, 6: 37-45.
[2] 张加延, 周恩. 中国果树志·李卷. 北京: 中国林业出版社, 1998.
ZHANG J Y, ZHOU E.. Beijing: China Forestry Press, 1998. (in Chinese)
[3] 阮颖, 周朴华, 刘春林. 九种李属植物的RAPD亲缘关系分析. 园艺学报, 2002, 29(3): 218-223.
RUAN Y, ZHOU P H, LIU C L. Phylogenetic relationship among ninespecies based on random amplified polymorphic DNA., 2002, 29(3): 218-223. (in Chinese)
[4] LIU W S, LIU D C, FENG C J, ZHANG A M, LI S H. Genetic diversity and phylogenetic relationships in plum germplasm resources revealed by RAPD markers., 2006, 81(2): 242-250.
[5] LIU W S, LIU D C, ZHANG A M, FENG C J, YANG J M, YOON J, LI S H. Genetic diversity and phylogenetic relationships among plum germplasm resources in China assessed with inter-simple sequence repeat markers., 2007, 132(5): 619-628.
[6] BRETSCHNEIDER E.. Sampson Low, London, 1898: 827-833.
[7] FAUST M, Suranyi D. Origin and dissemination of plums., 1999, 23: 179-231.
[8] 郭忠仁. 我国南方中国李种质资源收集和利用研究[D]. 南京: 南京农业大学, 2006.
GUO Z R. Collection and utilization ofgermplasm resource in South China [D]. Nanjing: Nanjing Agricultural University, 2006. (in Chinese)
[9] 乔玉山. 中国李RAPD、ISSR和SSR反应体系的建立及其品种资源遗传多样性分析[D]. 南京: 南京农业大学, 2003.
QIAO Y S. Establishment of RAPD, ISSR and SSR reactions system and analysis of genetic diversity of Japanese plum cultivars [D]. Nanjing: Nanjing Agricultural University, 2003. (in Chinese)
[10] 刘威生. 李种质资源遗传多样性及主要种间亲缘关系的研究[D]. 北京: 中国农业大学, 2005.
LIU W S. Studies on the genetic diversity among plum germplasm resources and the phylogenetic relationships of main plum species [D]. Beijing: China Agricultural University, 2005. (in Chinese)
[11] 左力辉, 韩志校, 梁海永, 杨敏生. 不同产地中国李资源遗传多样性SSR分析. 园艺学报, 2015, 42(1): 111-118.
ZUO L H, HAN Z X, LIANG H Y, YANG M S. Analysis of genetic diversity offrom different producing areas by SSR markers., 2015, 42(1): 111-118. (in Chinese)
[12] 陈圣林, 王会娜, 孙颖, 陈显, 李建安. 三峡库区14个李品种遗传多样性的ISSR分析. 中南林业科技大学学报, 2012, 32(9): 130-134.
CHEN SL, WANG HN, SUN Y, CHEN X, LI JA. ISSR analysis on genetic diversity of fourteenvarieties in threegorges reservoir area., 2012, 32(9): 130-134. (in Chinese)
[13] TOPP B L, RUSSELL D M, NEUMULLER M, DALBO M, LIU W S. Plum//Badenes M L, Byrne D H.. Media: Springer Science Business, 2012: 571-621.
[14] 章秋平, 刘威生, 郁香荷, 刘宁, 张玉萍, 孙猛, 徐铭. 基于优化LDSS法的中国李()初级核心种质构建. 果树学报, 2011, 28(4): 617-623.
ZHANG Q P, LIU W S, YU X H, LIU N, ZHANG Y P, SUN M, XU M. Establishment and evaluation of primary core collection of Chinese plum (L.) germplasm., 2011, 28(4): 617-623. (in Chinese)
[15] DOYLE J J, DOYLE J L. Isolation of plant DNA from fresh tissue., 1990, 12: 13-15.
[16] SCHUELKE M. An economic method for the fluorescent labeling of PCR fragments., 2000, 18: 233-234.
[17] SOSINSKI B, GANNAVARAPU M, HAGER L D, BECK L E, KING G J, RYDER C D, RAJAPAKSE S, BAIRD W V, BALLARD R E, ABBOTT A G. Characterization of microsatellite markers in peach [(L.) Batsch]., 2000, 101(3): 421-428.
[18] DIRLEWANGER E, GRAZIANO E, JOOBEUR T, GARRIGA- CALDERÉ F, COSSON P, HOWAD W, ARÚS P. Comparative mapping and marker-assisted selection infruit crops., 2004, 101(26): 9891-9896.
[19] SORIANO J M, DOMINGO M L, ZURIAGA E, ROMERO C, BADENES M L, ZHEBENTYAYEVA T, ABBOTT A G. Identification of simple sequence repeat markers tightly linked toresistance in apricot., 2012, 30(2): 1017-1026.
[20] LOPES M S, SEFC K M, LAIMER M, DA CÂMARA M A. Identification of microsatellite loci in apricot., 2002, 2(1): 24-26.
[21] DIRLEWANGER E, COSSON P, TAVAUD M, ARANZANA M J, POIZAT C, ZANETTO A. Development of microsatellite markers in peach [(L.) Batsch] and their use in genetic diversity analysis in peach and sweet cherry (L.)., 2002, 105(1): 127-138.
[22] MNEJJA M, GARCIA-MAS J, HOWAD W, BADENES M L, ARÚS P. Simple-sequence repeat (SSR) markers of Japanese plum (Lindl.) are highly polymorphic and transferable to peach and almond., 2004, 4(2): 163-166.
[23] MNEJJA M, GARCIA-MAS J, HOWAD W, ARÚS P. Development and transportability acrossspecies of 42 polymorphic almond microsatellites., 2005, 5(3): 531-535.
[24] ARANZANA M J, GARCIA-MAS J, CARBÓ J, ARÚS P. Development and variability analysis of microsatellite markers in peach., 2002, 121: 87-92.
[25] 王凤格, 田红丽, 赵久然, 王璐, 易红梅, 宋伟, 高玉倩, 杨国航. 中国328个玉米品种(组合)SSR标记遗传多样性分析. 中国农业科学, 2014, 47(5): 856-864.
WANG F G, TIAN H L, ZHAO J R, WANG L, YI H M, SONG W, GAO Y Q, YANG G H. Genetic diversity analysis of 328 maize varieties (hybridized combinations) using SSR markers., 2014, 47(5): 856-864. (in Chinese)
[26] PEAKALL R, SMOUSE P E. GENALEX 6: Genetic analysis in Excel. Population genetic software for teaching and research., 2006, 6(1): 288-295.
[27] PRITCHARD J K, STEPHENS M, DONNELLY P J. Inference of population structure using multilocus genotype data., 2000, 155: 945-959.
[28] EVANNO G, REGNAUT S, GOUDET J. Detecting the number of clusters of individuals using the software STRUCTURE: A simulation study., 2005, 14: 2611-2620.
[29] EARL D A, VONHOLDT B M. Structure Harvester: a website and program for visualizing STRUCTURE output and implementing the Evanno method., 2012, 4: 359-361.
[30] 徐刚标. 植物群体遗传学. 北京: 科学出版社, 2009.
XU G B.. Beijing: Science Press, 2009. (in Chinese)
[31] 孙萍, 林贤锐, 沈建生. 基于SSR标记的李种质资源遗传多样性研究. 江西农业学报, 2017, 29(2): 33-39.
SUN P, LIN X R, SHEN J S. Study on genetic diversity of plum germplasm resources based on SSR markers., 2017, 29(2): 33-39. (in Chinese)
[32] CARRASCO B, DÍAZ C, MOYA M, GEBAUER M, GARCÍA- GONZÁLEZ R. Genetic characterization of Japanese plum cultivars () using SSR and ISSR molecular markers., 2012, 39(3): 533-543.
[33] 陈红, 杨迤然. 贵州李资源遗传多样性及亲缘关系的ISSR分析. 果树学报, 2014, 31(2): 175-180.
CHEN H, YANG Y R. Genetic diversity and relationship of plum resources in Guizhou analyzed by ISSR markers., 2014, 31(2): 175-180. (in Chinese)
[34] 郁香荷, 章秋平, 刘威生, 孙猛, 刘宁, 张玉萍, 徐铭. 中国李种质资源形态和农艺性状的遗传多样性分析. 植物遗传资源学报, 2011, 12(3): 402-407.
YU X H, ZHANG Q P, LIU W S, SUN M, LIU N, ZHANG Y P, XU M. Genetic diversity analysis of morphological and agronomic characters of Chinese plum (Lindl.) germplasm., 2011, 12(3): 402-407. (in Chinese)
[35] ZHANG Q P, WEI X, LIU W S, LIU N, ZHANG Y P, XU M, LIU S, ZHANG Y J, MA X X, DONG W X. The genetic relationship and structure of some natural interspecific hybrids insubgenus, based on nuclear and chloroplast simple sequence repeats., 2018, 65: 625-636.
Genetic Diversity of theL. from Different Sources and Their Related Species
WEI Xiao, ZHANG QiuPing, LIU Ning, ZHANG YuPing, XU Ming, LIU Shuo, ZHANG YuJun, MA XiaoXue, LIU WeiSheng
(Liaoning Institute of Pomology, Yingkou 115009, Liaoning)
【Objective】 There are abundant Japanese plums (L.) germplasm resources in China, which are distributed widely in geography. The better understanding of the diversity, the genetic structure and the relationship between sympatric related species, can be helpful to clarify the process of domestication of the cultivated groups of plum and the role of the related species, also further contribute to the in-depth exploration of local resources and enhance the fruit quality in breeding. 【Method】 The 22 pairs of SSR markers covering the entire genome were used to scan 48 samples, including 38 accessions offrom different sources and 10 accessions of the variation types or related species, by high-throughput fluorescence capillary electrophoresis platform. The polymorphism of 22 SSR loci and genetic diversity of 48 samples were evaluated via the software GenAlEx 6.41, and the dendrogram of these accessions was constructed by using the NTSYS-pc Version 2.1 program. The STRUCTURE 2.2 software based on Bayesian clustering method was used to analyze the genetic mixture of samples and to perform an assignment test on the studied individuals. 【Result】 The detected alleles of 22 SSR primers ranged from 3 to 21, with an average of 13.54 alleles for each locus. A total of 298 alleles were detected in these accessions, 51.8% of which were rare alleles. The values of average effective allele (), average Shannon's diversity index (), observation heterozygosity () and expectation heterozygosity () indicated that the diversity was the highest in the southern population, followed by the northeast population, which also indicated that the diversity of thewas lower than that of northern China. The analysis of molecular variance (AMOVA) showed that 69% of the diversity inwas within the population, and only 31% was among populations. On the basis of the data comparisons of the genetic differentiation coefficient ands genetic distance, the results showed that there were extremely significant genetic differentiation and appropriate genes mixture among different geographical populations. The results of cluster analysis implied that the breeding cultivars abroad were closely related to the local accessions in southern China, and the northern cultivars population was similar with, the northeast cultivars population had close relationship with. The genetic structure analysis indicated that the accessions inwere divided into four types: northern cultivars population, northeast cultivars population, southern cultivars of small fruit and crisp meat population, and southern cultivars of large fruit population (including foreign breeding cultivars). 【Conclusion】The diversity of the southern varieties were the most abundant in the, followed by that of Northeast China varieties, foreign varieties and Northern varieties.In order to enhance the adaptability, the northeast population might be introgressed with.might be a special type which was highly domesticated from Chinese plum in Northern China, and it had high heterozygosis owing to asexual reproduction by grafting. The large fruit accessions in Jiangsu and Zhejiang regions played an important role in modern breeding varieties abroad.
L.; genetic diversity; population structure; gene flow
10.3864/j.issn.0578-1752.2019.03.017
2018-09-28;
2018-12-10
国家自然科学基金(31401826)、物种品种资源保护项目(111821301354052003)、辽宁省北方果树资源与育种重点实验室项目(2018103002)
魏潇,Tel:15902483962;E-mail:weixiao6769@163.com。通信作者章秋平,Tel:13941786260;E-mail:lbzhangqiuping2@163.com。通信作者刘威生,Tel:13941718335;E-mail:wsliulaas@163.com
(责任编辑 赵伶俐)
