干酪乳杆菌CRISPR基因座分析
2019-02-20杨兰杨洋李伟勋ObaroakpoJOY逄晓阳吕加平
杨兰,杨洋,李伟勋,Obaroakpo JOY,逄晓阳,吕加平
干酪乳杆菌CRISPR基因座分析
杨兰,杨洋,李伟勋,Obaroakpo JOY,逄晓阳,吕加平
(中国农业科学院农产品加工研究所,北京 100193)
【目的】目前基于酿脓链球菌()spCas9为核心的CRISPR/Cas9基因编辑系统在乳酸菌上的应用受到很多限制,亟待开发适合于乳酸菌的基因编辑系统。对6株干酪乳杆菌()的CRISPR系统进行深入分析,并预测激活干酪乳杆菌自身Cas9蛋白所识别的PAM序列,为开发适用于乳酸菌的CRISPR/lcCas9基因编辑系统奠定基础。【方法】以已完成全基因组测序的6株干酪乳杆菌为研究对象,利用生物信息学方法对其CRISPR系统进行深入分析,重点对不同菌株的CRISPR系统结构进行解析,并且对Cas蛋白以及spacer的同源性进行分析,最后对CRISPR区重复序列的二级结构以及Cas9蛋白识别的PAM序列进行预测。【结果】6株干酪乳杆菌CRISPR系统具有相似的结构,均具有特征性的Cas9蛋白,并且Cas基因序列保守。预测到tracrRNA位于Cas9和Cas1之间,重复序列可以形成茎部长达7个碱基的二级结构。根据CRISPR的间隔区序列,6株干酪乳杆菌可被分为3个基因型,将间隔区逐一进行blast比对,结果表明6个间隔区比对上14个来源不同的原间隔序列,这些间隔序列均来源于不同质粒。干酪乳杆菌lcCas9蛋白识别PAM序列的1、3位碱基偏好T/C、A/C,2、4位碱基对G、A的偏好性比较大。【结论】6株干酪乳杆菌CRISPR系统均为type-ⅡA型,序列和重复序列高度保守。DR序列可以形成稳定的二级结构,TGMA为干酪乳杆菌Cas9蛋白高效识别的PAM序列。
干酪乳杆菌;CRISPR系统;spacer;;PAM
0 引言
【研究意义】随着分子生物学和高通量测序技术的飞速发展,人类已经完成了数以千计乳酸菌的全基因组测序,但是目前对于乳酸菌全基因组的研究仍然面临两大艰巨任务,一是如何更精确解读基因组测序得到的海量数据;二是如何对乳酸菌基因组进行遗传修饰,更加突出乳酸菌的益生功能。近年来,研究火热的CRISPR/Cas9基因编辑技术为高效完成上述两大任务提供了强有力的工具。目前应用最多的CRISPR/Cas9系统是以酿脓链球菌()的spCas9为核心构建的,已经广泛应用于真核生物的基因组编辑[1-3]。然而它在乳酸菌中的应用仍然受到很大限制,主要原因是spCas9在多数乳酸菌内有较高的细胞毒性;另外,spCas9蛋白较大、异源蛋白密码子偏好性等也是限制该系统在乳酸菌成功应用的重要原因。对乳酸菌自身CRISPR系统进行深入研究,解析乳酸菌抵御噬菌体等外源遗传基因侵染的机制,对于后续开发适合于乳酸菌的CRISPR/Cas9基因编辑系统具有重要意义。另一方面,干酪乳杆菌作为具有益生功能的微生物发酵剂被广泛的用于食品发酵中,具有降血压[4]、调节肠道菌群[5]和提高机体免疫力[6]等促进健康的作用。深入研究该菌CRISPR系统行使免疫功能的机制,将为解决乳酸菌工业发酵过程中易受到噬菌体的侵染导致发酵失败提供重要的理论依据。【前人研究进展】CRISPR(clustered regularly interspaced short palindromic repeat)/Cas(CRISPR associated)系统是细菌的一种获得性免疫系统,用来抵御噬菌体、质粒等外源DNA的侵害[7]。CRISPR基因座包括CRISPR序列和Cas蛋白,CRISPR序列由重复序列(repeat)和间隔序列(spacer)交替组成[8]。在前导区的调控下,CRISPR序列转录为pre-crRNA[9-10],与一种反式编码的小RNA(tracrRNA)通过碱基配对形成双链RNA(dsRNA)区域,在Cas蛋白存在下被RNase III切割加工成熟,然后与细菌自身的Cas核酸酶形成核酸蛋白复合体[11]。在type-II型系统中,当入侵的外源DNA和crRNA序列配对结合,同时Cas9蛋白识别对应的特异性PAM序列,就能够对入侵的DNA进行靶向切割,从而得到破坏外源DNA、实现自我防御的目的[12]。CRISPR/Cas系统的机制和功能日益清晰,科学家逐渐意识到可以将其应用于基因编辑。目前,CRISPR/Cas编辑系统飞速发展,已经被迅速应用于小鼠[13-15]、大鼠[16]、斑马鱼[17-18]、秀丽隐杆线虫[19]、拟南芥[20-21]及大肠杆菌[22-23]等多个物种,成为基因组精准编辑的有力工具[24-26],但是目前在乳酸菌中利用此系统进行成功编辑的研究还较少。OH等[27]将CRISPR/Cas9系统引入罗伊氏乳杆菌中,结合RED重组技术实现了对乳酸菌基因组的成功编辑,这是CRISPR/Cas9系统在乳酸菌上应用的首次报道。但是该研究也存在一些问题,其Cas9蛋白载体的构建是一种组成型表达,而且构建敲除系统所用质粒较多,电转化效率不是很高,影响了基因编辑的效率。目前在乳酸菌领域使用的基因编辑方法仍然是传统的基于基因同源重组的策略,至少需要24 d才能获得一个基因敲除,这是相当繁琐且耗时的[28]。【本研究切入点】目前的CRISPR/Cas9系统应用于乳酸菌还存在很多限制因素,亟待对该系统进行改进优化。本研究以干酪乳杆菌()的CRISPR系统为切入点,深入研究该菌的CRISPR系统行使免疫功能的机制,解析lcCas9识别的PAM序列。【拟解决的关键问题】本研究对干酪乳杆菌不同菌株CRISPR系统进行深入分析,重点解析lcCas9蛋白识别的PAM序列,以期对现有CRISPR/Cas9系统进行改良,为开发适用于乳酸菌的CRISPR/Cas9基因编辑系统奠定理论基础。
1 材料与方法
试验于2017年10月—2018年7月在中国农业科学院农产加工研究所乳品研究实验室进行。
1.1 材料
NCBI Genbank(https://www.ncbi.nlm.nih.gov/)数据库中已公布全基因组序列的6株干酪乳杆菌。这些菌株包括:干酪乳杆菌BD-Ⅱ(NC_017474.1)、干酪乳杆菌BL23(NC_010999.1)、干酪乳杆菌LC2W (NC_017473.1)、干酪乳杆菌W56(NC_018641.1)、干酪乳杆菌ZHANG(NZ_CP001084.1/NC_014334.2)、干酪乳杆菌LOCK919(NC_021721.1)。
1.2 方法
1.2.1 CRISPR系统结构分析 使用CRISPR-CAS++(https://crisprcas.i2bc.paris-saclay.fr/CrisprCasFinder/Index)以及CRISPRdisco软件对CRISPR系统进行查找,包括CRISPR序列及位置,确定CRISPR/Cas系统的类型。使用MEGA7对进行序列同源性分析。使用IBS 1.0.2进行基因结构作图。
1.2.2 TracrRNA位置预测 基于之前对大量tracrRNA同源物的研究[29],tracrRNA在CRISPR基因座内的位置并不是保守的,在4个典型的位置中发现了反重复序列,分别为:Cas9的上游,Cas9和Cas1之间以及CRISPR序列的上下游。为了预测新的tracrRNA,通过一系列序列比对筛选了CRISPR/Cas基因座非repeat-spacer序列,包括Cas9上游1 kb、Cas9-Cas1之间以及CRISPR序列上下游1 kb两条链的序列;验证CRISPR重复:反重复配对情况。将筛选出的反重复序列使用Promoter 2.0 prediction server(http://www.cbs.dtu.dk/services/Promoter/)预测启动子,使用ARNold(http://rna.igmors.u-psud.fr/)预测终止子。
1.2.3 DR序列的二级结构预测 在前导区的调控下,CRISPR基因座转录为单链RNA分子(pre- crRNA)[9-10]。由于CRISPR重复序列部分回文性质,其可能形成稳定的发夹二级结构[8,30]。使用RNAalifold web server(http://nibiru.tbi.univie.ac.at/cgi-bin/RNAWebSuite/ RNAalifold.cgi)进行二级结构预测,计算方法采用最小自由能法(minimum free energy,MFE),其他参数选择默认。
1.2.4 间隔区多样性及同源性分析 通过祖先间隔区和保守间隔区的分布可以将CRISPR序列分型将间隔区序列进行,使用Clustal X进行序列相似性分析。将6株菌的spacer逐一进行blast,查找预期匹配的原间隔序列,最多允许3个不匹配,并对原间隔序列进行溯源。筛选去除来源于CRISPR序列的同源序列,选择来源于噬菌体或质粒的序列作为潜在的外源遗传元件[31-33]。
1.2.5 PAM序列预测及其可视化 使用CRISPR target(http://bioanalysis.otago.ac.nz/CRISPRTarget/crispr_analysis.html)分析来源不同的原间隔序列非靶标链下游10个碱基序列。使用weblogo(http://weblogo.berkeley. edu/logo.cgi)对每个位点的碱基偏好性进行可视化。
2 结果
2.1 干酪乳杆菌CRISPR基因座结构
本研究的6株干酪乳杆菌中均只有一个已证实的CRISPR系统,同时在干酪乳杆菌ZHANG基因组上还存在5个疑似CRISPR区域,在干酪乳杆菌ZHANG质粒上存在1个疑似CRISPR区域,在干酪乳杆菌LOCK919质粒上存在一个疑似CRISPR区域,这些区域虽然存在简单的重复间隔区,但是均只有1—2个间隔区,且不存在,本研究对此不进行深入分析(表1)。分析6株干酪乳杆菌确认的CRISPR系统,发现4个(、、、)位于CRISPR序列侧翼。6株菌的基因组上均含有标志性基因,可以确定其CRISPR系统为type-ⅡA型。CRISPR序列最长的为1 424 bp,含有21个间隔序列,最短的为762 bp,含有11个间隔序列,重复序列均为36 bp,间隔序列为28—31 bp。对Cas蛋白序列保守型的比较分析表明,这些菌株的Cas蛋白之间具有高度的相似性。包括干酪乳杆菌BD-Ⅱ、干酪乳杆菌BL23、干酪乳杆菌LC2W、干酪乳杆菌W56、干酪乳杆菌ZHANG在内的菌株,他们的Cas蛋白序列相似性均达到了100%,仅LOCK919与他们有微小差别,但相似性仍然达到90%以上(图1)。
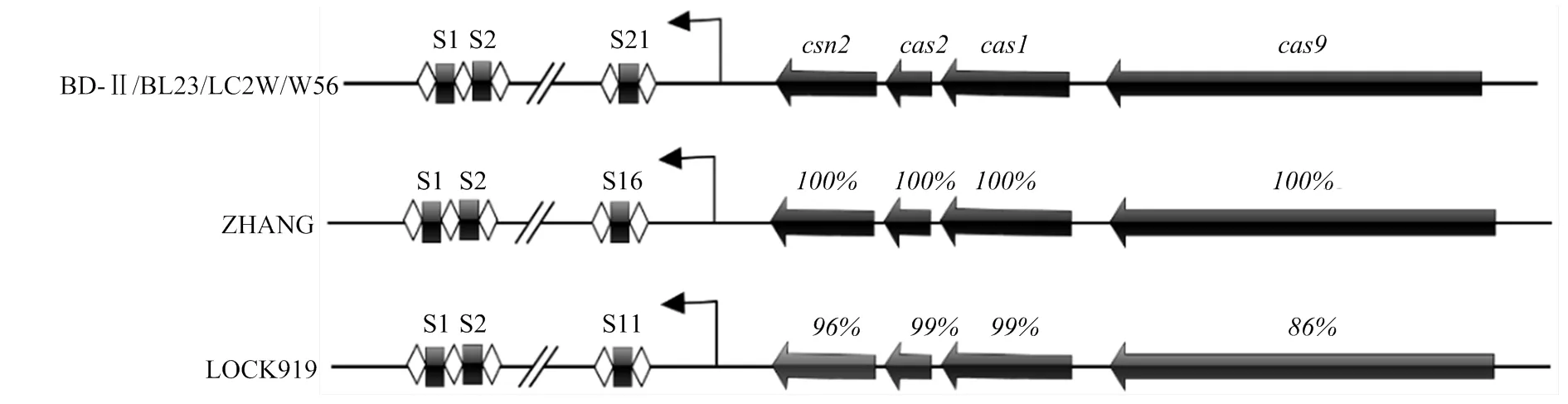
图1 干酪乳杆菌CRISPR基因座结构

表1 6株干酪乳杆菌CRISPR序列情况
2.2 TracrRNA位置预测
对6株菌株tracrRNA的预测表明,其tracrRNA均位于与之间,与转录方向相反(图2)。tracrRNA与repeat区有高达30 bp的配对,并成预测tracrRNA的启动子和终止子。启动子和转录终止子预测作为非必要的步骤,计算机模拟预测算法可以提供参考,若要获得准确的序列可以进行RNA深度测序验证。

图2 干酪乳杆菌tracrRNA在CRISPR基因座中的位置
2.3 DR序列的二级结构分析
DR序列转录成非信使RNA,以特殊的结构发挥功能。随着转录的进行,连续的单链重复序列形成二级结构,茎可长达4—8个碱基,从而产生更稳定的RNA结构。为了评估干酪乳杆菌CRISPR重复序列形成稳定的RNA二级结构的可能性,使用RNAalifold来预测重复序列的分子内RNA结构。预测到的DR序列二级结构自由能为-2.50 kcal/mol,茎长度达7个碱基,结构稳定(图3)。
2.4 间隔区多样性分析
本研究发现40个独特的间隔区类型(图4),根据祖先间隔区及保守间隔区的位置,6株菌被分为3种CRISPR类型。其中BD-Ⅱ、BL23、LC2W分享完全相同的间隔区,W56与他们共享祖先间隔区,但是有3个间隔区不同,这3个不同的间隔区又与之前的序列高度相似。ZHANG与GENOTYPE 1型共享一些保守的间隔区,但是具有不完全相同的祖先间隔区及部分独特间隔区。LOCK919享有所有的独特间隔区。

图3 重复序列的RNA二级结构
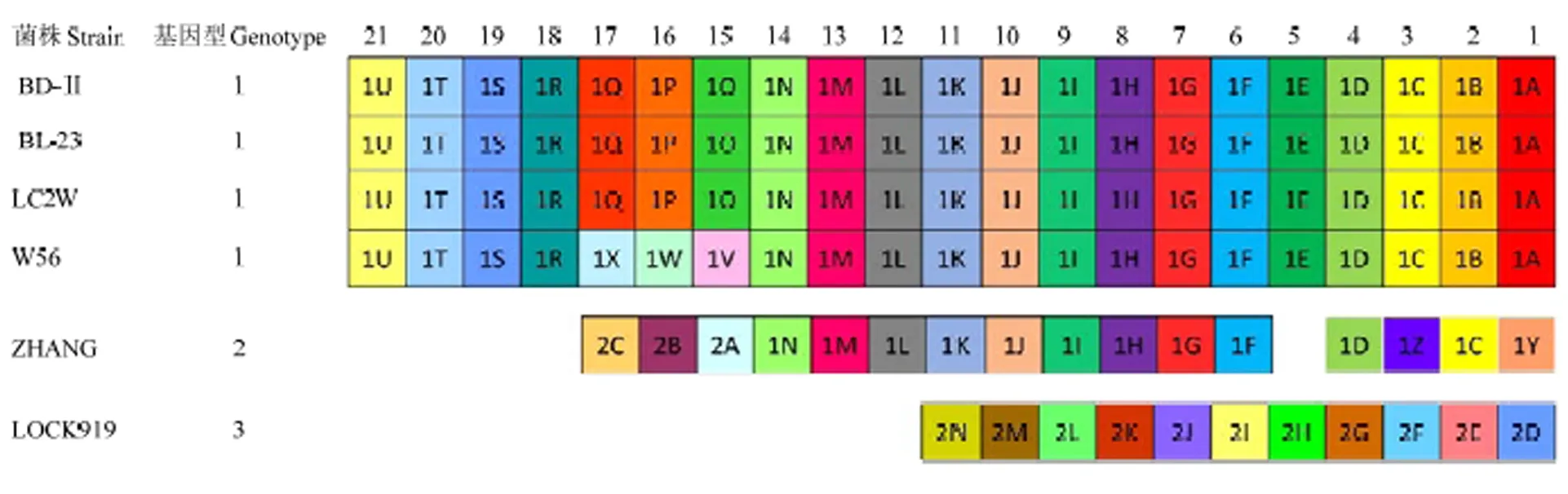
图4 6株干酪乳杆菌CRISPR间隔区多样性分析
2.5 间隔序列同源性分析
为了确定CRISPR间隔区的可能起源,研究其与已知序列的同源性。得到与干酪乳杆菌的部分间隔区高度匹配的噬菌体或质粒序列,其中包括某些完全匹配(表2)。BD-Ⅱ、BL23、LC2W、W56的spacer21序列均与乳杆菌的质粒pREN 100%匹配。ZHANG的spacer14序列与乳酸乳球菌乳酸亚种和乳酸乳球菌乳脂亚种的质粒仅有一个碱基错配。Spacer15序列与巴克乳杆菌、副干酪乳杆菌、卡氏双球菌、植物乳杆菌、短乳酸杆菌、乳明串珠球菌、戊糖片球菌中的质粒有1—3个碱基错配。
Blast的结果中40个间隔区只有6个间隔区比对上了原间隔序列,这其中还包括一些非完全匹配序列。另外匹配到原间隔序列的spacer均为新获取的间隔区,祖先间隔区均未匹配到有效序列,这可能是由于数据库的不完整以及噬菌体质粒进化过程中发生突变所致。ZHANG的spacer14和spacer15匹配到了多株菌的质粒,这表明一个间隔序列可以抵抗多种外源元件的侵入。仅有几个间隔区能够匹配上编码蛋白的基因,ZHANG的spacer15匹配到了来源于植物乳杆菌pXY3质粒的ORF4,BD-Ⅱ、BL23、LC2W、W56的spacer21以及ZHANG的spacer来源于副干酪乳杆菌的质粒均匹配到的是假定的蛋白,其余间隔区均未匹配到编码蛋白的序列。结果表明,所有间隔序列的来源均为质粒,但是这并不能说明抗噬菌体的能力较弱,仅有较少的新间隔区在数据库中得到匹配,绝大多数的间隔区没有得到匹配,噬菌体数据库不完善成为了主要限制因素,blast结果只能成为细菌抗噬菌体能力的一种参考。
2.6 干酪乳杆菌Cas9蛋白识别PAM序列的预测
分析结果表明,干酪乳杆菌PAM序列的1、3位碱基偏好T/C、A/C,2、4位碱基对G、A的偏好性比较大,推测干酪乳杆菌CRISPR系统识别效率最高的PAM序列为TGMA(图5)。但是通过生物信息学分析的方法预测到的原间隔序列有限,所以这一方法可以对CRISPR系统识别的PAM进行初步预测,对于将这一系统应用于基因编辑,后续将通过分子试验进一步验证。
3 讨论
CRISPR/Cas系统是细菌的获得性免疫系统,存在于46%细菌和84%古菌基因组中[31,34]。免疫功能由细菌基因组上的CRISPR序列和Cas蛋白执行,Cas1-Cas2复合物将新捕获的外源间隔序列整合入细菌的CRISPR区[35],间隔序列的插入代表着入侵者的特征遗传信息存储到宿主基因组。当入侵者再次侵染时,菌体自身的CRISPR系统会快速识别入侵者并引导相关Cas蛋白对入侵者DNA序列进行特异性切割破坏,从而发挥免疫作用。值得注意的是,间隔区在CRISPR基因座上的排列是按照进化时间顺序的,新获取的间隔区序列总是被整合到CRISPR位点的前导序列和第一个重复间隔单元之间[31]。因此,间隔区的位置可以代表他们被捕获时间的先后,这也为细菌提供了一种基于独特高变的基因座基因分型方法[36]。本研究通过分析确定其均为type-ⅡA型系统,并且基于CRISPR序列,将6株菌分为3个基因型。通过对其Cas蛋白序列对比分析,发现6株菌的Cas蛋白相似性较高,ZHANG与BD-Ⅱ等菌株Cas蛋白相似度均为100%,但是其间隔区却不完全相同,仅共享一部分保守间隔区。LOCK919与其他菌株Cas蛋白相似度达到90%左右,但是他们的间隔区完全不同,保守的祖先间隔区意味着菌株之间具有较高的亲缘关系,而后来获得的间隔区由于相关菌株暴露于不同的外来侵入性DNA而不同。虽然间隔区的差异巨大,但是repeat以及tracrRNA和Cas蛋白比对结果相似,是高度保守的。

表2 干酪乳杆菌spacer对应的原间隔区序列特点
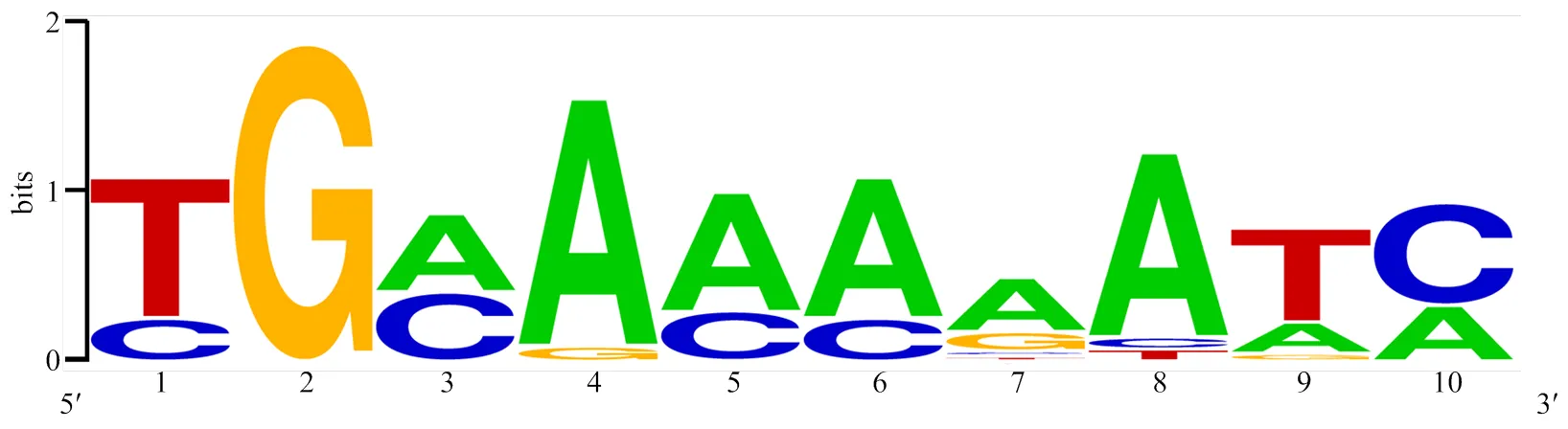
图5 干酪乳杆菌CRISPR系统识别PAM预测
通过对入侵者的靶标序列分析发现,原间隔序列侧翼位置上有一段2—7个碱基的前间隔序列邻近基序(Protospacer adjacent motif,PAM)[37]。现有研究表明,细菌CRISPR系统在获取新的间隔序列过程[38]和Cas9介导的靶序列特异性切割过程[10,39]中,PAM序列发挥着至关重要的作用。PAM存在于入侵者靶标序列侧翼但是在自身CRISPR序列中并不存在,这是宿主区分自我和非我的重要依据[40]。在不存在PAM的情况下,即使gRNA将Cas9蛋白引导到靶标基因的位置,Cas9蛋白也不会被激活,从而无切割活性。随着不同宿主来源的CRISPR系统被研究的逐渐深入,越来越多的PAM序列被解析出来。目前广泛使用的CRISPR/Cas9基因编辑系统是基于酿脓链球菌Cas9(SpCas9)为核心构建的,研究已证实该Cas9识别的经典PAM序列是5-NGG-3,进一步的研究表明5-NAG/NGA-3也是其识别的非经典PAM[40-42]。通过体外筛选试验,研究人员发现金黄色葡萄球菌Cas9(SaCas9)识别更长的PAM(5-NNGRR(T)-3),SaCas9体积小,有利于体内基因组编辑[43-44]。不同来源的Cas9基因序列相似性不同使他们识别不同的PAM序列,这使得可以不断探索不同物种Cas9蛋白所识别的PAM序列,丰富CRISPR工具库,提高基因编辑效率。本研究使用CRISPRtarget对干酪乳杆菌lcCas9所识别的PAM进行了初步预测。40个间隔序列在GenBank-Phage、RefSeq-Plasmid、RefSeq-Viral数据库中进行检索,仅有3个序列得到了有效匹配,但是其中2个匹配到了多达13个不同质粒。虽然间隔区是相同的,但是其匹配到的间隔区来源及错配数都是不同的,而且得到有效匹配的间隔序列均位于CRISPR的新获取间隔区,可靠性较强,表明TGMA非常有潜力成为高切割效率的PAM序列。
目前CRISPR/Cas9基因编辑方法在乳酸菌的应用并不广泛,受到了很多限制。对乳酸菌自身的CRISPR系统进行解析并基于此开发适合于乳酸菌的CRISPR/ cas9基因编辑系统,在乳酸菌遗传改良方面具有重要应用前景。本研究对干酪乳杆菌CRISPR基因座进行了全面的分析,预测了trcarRNA位置,并对PAM进行了初步预测,为解析干酪乳杆菌CRISPR系统提供了理论基础。同时也为解决乳酸菌工业发酵中噬菌体侵染这一产业难题提供了十分重要的理论依据,具有较高的应用价值和实际意义。
4 结论
6株干酪乳杆菌CRISPR系统均为type-ⅡA型,Cas基因序列和重复区序列保守,tracrRNA位于Cas9和Cas1蛋白之间。DR可以形成稳定的二级结构,根据间隔区序列可以将6株菌株分为3种基因型,TGMA为干酪乳杆菌CRISPR系统高效识别的PAM序列。
[1] COBB R E, WANG Y, ZHAO H. High-efficiency multiplex genome editing of Streptomyces species using an engineered CRISPR/Cas system., 2015, 4(6): 723-728.
[2] JIANG Y, CHEN B, DUAN C, SUN B, YANG J, YANG S. Multigene editing in thegenome via the CRISPR-Cas9 system., 2015, 81(7): 2506-2514.
[3] PYNE M E, MOO-YOUNG M, CHUNG D A, CHOU C P. Coupling the CRISPR/Cas9 system with lambda red recombineering enables simplified chromosomal gene replacement in., 2015, 81(15): 5103-5114.
[4] 陈冲, 宋丽菊, 齐盼盼, 王敏, 冯盨, 张杰, 赵建. 干酪乳杆菌在乳制品中的应用研究进展. 食品与发酵科技, 2015, 51(4): 88-91.
CHEN C, SONG L J, QI P P, WANG M, FENG W, ZHANG J, ZHAO J. Progress in the application of Lactobacillus casei in dairy products.2015, 51(4): 88-91. (in Chinese)
[5] KOEBNICK C, WAGNER I, LEITZMANN P, STERN U, ZUNFT H J. Probiotic beverage containingShirota improves gastrointestinal symptoms in patients with chronic constipation., 2016, 17(11): 655-659.
[6] AKOGLU B, LOYTVED A, NUIDING H, ZEUZEM S, FAUST D. Probiotic Lactobacillus casei Shirota improves kidney function, inflammation and bowel movements in hospitalized patients with acute gastroenteritis-A prospective study., 2015, 17: 305-313.
[7] MAKAROVA K S, WOLF Y I, KOONIN E V. Comparative genomics of defense systems in archaea and bacteria., 2013, 41(8): 4360-4377.
[8] BOLOTIN A,QUINQUIS B, SOROKIN A, EHRLICH S D. Clustered regularly interspaced short palindrome repeats (CRISPRs) have spacers of extrachromosomal origin., 2005, 151(Pt 8): 2551-2561.
[9] VISWANATHAN P, MURPHY K, JULIEN B, GARZA A G, KROOS L. Regulation of dev, an operon that includes genes essential fordevelopment and CRISPR-associated genes and repeats., 2007, 189(10): 3738-3750.
[10] KARVELIS T, GASIUNAS G, MIKSYS A, BARRANGOU R, HORVATH P, SIKSNYS V. CrRNA and tracrRNA guide Cas9- mediated DNA interference in., 2013, 10(5): 841-851.
[11] DELTCHEVA E, CHYLINSKI K, SHARMA C M, GONZALES K, CHAO Y, PIRZADA Z A, ECKERT M R, VOGEL J, CHARPENTIER E. CRISPR RNA maturation by trans-encoded small RNA and host factor RNase III., 2011, 471(7340): 602-607.
[12] VAN DER OOST J, JORE M M, WESTRA E R, LUNDGREN M, BROUNS S J. CRISPR-based adaptive and heritable immunity in prokaryotes., 2009, 34(8): 401-407.
[13] FUJII W, ONUMA A, SUGIURA K, NAITO K. Efficient generation of genome-modified mice via offset-nicking by CRISPR/Cas system., 2014, 445(4): 791-794.
[14] AIDA T, CHIYO K, USAMI T, ISHIKUBO H, IMAHASHI R, WADA Y, TANAKA K F, SAKUMA T, YAMAMOTO T, TANAKA K. Cloning-free CRISPR/Cas system facilitates functional cassette knock-in in mice., 2015, 16(1): 1-11.
[15] NAKAGAWA Y, SAKUMA T, SAKAMOTO T, OHMURAYA M, NAKAGATA N, YAMAMOTO T. Production of knockout mice by DNA microinjection of various CRISPR/Cas9 vectors into freeze- thawed fertilized oocytes., 2015, 15(1): 33.
[16] MA Y W, ZHANG X, SHEN B, LU Y D, CHEN W, MA J, BAI L, HUANG X X, ZHANG L F. Generating rats with conditional alleles using CRISPR/Cas9., 2014, 24(1): 122-125.
[17] VARSHNEY G K, PEI W, LAFAVE M C, IDOL J, XU L, GALLARDO V, CARRINGTON B, BISHOP K, JONES M, LI M. High-throughput gene targeting and phenotyping in zebrafish using CRISPR/Cas9., 2015, 25(7): 1030-1042.
[18] AUER T O, DUROURE K, DE CIAN A, CONCORDET J P, DEL BENE F. Highly efficient CRISPR/Cas9 mediated knock-in in zebrafish by homology-independent DNA repair., 2014, 24(1): 142.
[19] SHEN Z F, ZHANG X L, CHAI Y P, ZHU Z W, YI P S, FENG G X, LI W, OU G S. Conditional knockouts generated by engineered CRISPR-Cas9 endonuclease reveal the roles of coronin inneural development., 2014, 30(5): 625-636.
[20] MIAO J, GUO D S, ZHANG J Z, HUANG Q P, QIN G J, ZHANG X, WAN J M, GU H Y, QU L J. Targeted mutagenesis in rice using CRISPR-Cas system., 2013, 23(10): 1233-1236.
[21] FENG Z Y, MAO Y F, XU N F, ZHANG B T, WEI P L, YANG D L, WANG Z, ZHANG Z J, ZHENG R, YANG L, ZENG L, LIU X D, ZHU J K. Multigeneration analysis reveals the inheritance, specificity, and patterns of CRISPR/Cas-induced gene modifications in., 2014, 111(12): 4632-4637.
[22] LUN C, BIKARD D. Consequences of Cas9 cleavage in the chromosome of., 2016, 44(9): 4243-4251.
[23] JI W Y, LEE D, WONG E, DADLANI P, DINH D, HUANG V, KEARNS K, TENG S, CHEN S, HALIBURTON J, HALIBURTON J, HEIMBERG G, HEINEIKE B, RAMASUBRAMANIAN A, STEVENS T, HELMKE K J, ZEPEDA V, QI L S, LIM W A. Specific gene repression by CRISPRi system transferred through bacterial conjugation., 2014, 3(12): 929-931.
[24] JINEK M, CHYLINSKI K, FONFARA I, HAUER M, DOUDNA J A, CHARPENTIER E. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity., 2012, 337(6096): 816-821.
[25] JIANG W, BIKARD D, COX D, ZHANG F, MARRAFFINI L A. RNA-guided editing of bacterial genomes using CRISPR-Cas systems., 2013, 31(3): 233-239.
[26] BURGESS D J. Technology: A CRISPR genome-editing tool., 2013, 14(2): 80.
[27] OH J H, VAN PIJKEREN J P. CRISPR-Cas9-assisted recombineering in., 2014, 42(17): e131.
[28] BIDART G N, RODRIGUEZ-DIAZ J, MONEDERO V, YEBRA M J. A unique gene cluster for the utilization of the mucosal and human milk-associated glycans galacto-N-biose and lacto-N-biose in., 2014, 93(3): 521-538.
[29] CHYLINSKI K, LE RHUN A, CHARPENTIER E. The tracrRNA and Cas9 families of type II CRISPR-Cas immunity systems., 2013, 10(5): 726-737.
[30] HORVATH P, BARRANGOU R. CRISPR/Cas, the immune system of bacteria and archaea., 2010, 327(5962): 167-170.
[31] BARRANGOU R, FREMAUX C, DEVEAU H, RICHARDS M, BOYAVAL P, MOINEAU S, ROMERO D A, HORVATH P. CRISPR provides acquired resistance against viruses in prokaryotes.,2007, 315(5819): 1709-1712.
[32] GARNEAU J E, DUPUIS M E, VILLION M, ROMERO D A, BARRANGOU R, BOYAVAL P, FREMAUX C, HORVATH P, MAGADAN A H, MOINEAU S. The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA., 2010, 468(7320): 67-71.
[33] SAPRANAUSKAS R, GASIUNAS G, FREMAUX C, BARRANGOU R, HORVATH P, SIKSNYS V. TheCRISPR/ Cas system provides immunity in., 2011, 39(21): 9275-9282.
[34] MARRAFFINI L A, SONTHEIMER E J. CRISPR interference limits horizontal gene transfer in staphylococci by targeting DNA., 2008, 322(5909): 1843.
[35] BARRANGOU R, HORVATH P. A decade of discovery: CRISPR functions and applications., 2017, 2: 17092.
[36] BRINER A E, LUGLI G A, MILANI C, DURANTI S, TURRONI F, GUEIMONDE M, MARGOLLES A, VAN SINDEREN D, VENTURA M, BARRANGOU R. Occurrence and diversity of CRISPR-Cas systems in the genus bifidobacterium., 2015, 10(7): e0133661.
[37] DEVEAU H, BARRANGOU R, GARNEAU J E, LABONTE J, FREMAUX C, BOYAVAL P, ROMERO D A, HORVATH P, MOINEAU S. Phage response to CRISPR-encoded resistance in., 2008, 190(4): 1390-1400.
[38] PAEZ-ESPINO D, MOROVIC W, SUN C L, THOMAS B C, UEDA K, STAHL B, BARRANGOU R, BANFIELD J F. Strong bias in the bacterial CRISPR elements that confer immunity to phage., 2013, 4: 1430.
[39] GASIUNAS G, BARRANGOU R, HORVATH P, SIKSNYS V. Cas9- crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria., 2012, 109(39): E2579-E2586.
[40] MOJICA F J, DIEZ-VILLASENOR C, GARCIA-MARTINEZ J, ALMENDROS C. Short motif sequences determine the targets of the prokaryotic CRISPR defence system., 2009, 155(Pt 3): 733-740.
[41] HSU P D, SCOTT D A, WEINSTEIN J A, RAN F A, KONERMANN S, AGARWALA V, LI Y, FINE E J, WU X, SHALEM O, CRADICK T J, MARRAFFINI L A, BAO G, ZHANG F. DNA targeting specificity of RNA-guided Cas9 nucleases., 2013, 31(9): 827-832.
[42] ZHANG Y, GE X, YANG F, ZHANG L, ZHENG J, TAN X, JIN Z B, QU J, GU F. Comparison of non-canonical PAMs for CRISPR/ Cas9-mediated DNA cleavage in human cells., 2014, 4: 5405.
[43] RAN F A, CONG L, YAN W X, SCOTT D A, GOOTENBERG J S, KRIZ A J, ZETSCHE B, SHALEM O, WU X, MAKAROVA K S, KOONIN E V, SHARP P A, ZHANG F.genome editing usingCas9., 2015, 520(7546): 186-191.
[44] KLEINSTIVER B P, PREW M S, TSAI S Q, NGUYEN N T, TOPKAR V V, ZHENG Z, JOUNG J K. Broadening the targeting range ofCRISPR-Cas9 by modifying PAM recognition., 2015, 33(12): 1293-1298.
CRISPR Locus Analysisof
YANG Lan, YANG Yang, LI WeiXun, Obaroakpo JOY, PANG XiaoYang, LÜ JiaPing
(Institute of Food Science and Technology,Chinese Academy of Agricultural Sciences, Beijing 100193)
【Objective】The application of the CRISPR/Cas9 gene editing system based onspCas9 has restriction on lactic acid bacteria at present. It is urgent to develop a suitable gene editing system for lactic acid bacteria. In this study, we analysed the CRISPR system ofin depth, and then predicted the PAM sequence to activate its Cas9 protein. Our study provided the experimental foundation for the development of the CRISPR/lcCas9 gene editing system for lactic acid bacteria 【Method】In this study, six strains of whole genome-sequencedwere used as research object. Bioinformatics was used to analyze the CRISPR system. Cas protein structure, CRISPR system and homology of space were analysed. At the end, the second structure of the CRISPR repeat and PAM sequence recognized by Cas9 protein were predicted. 【Result】The CRISPR system ofhad similar structures characteristic of Cas9 protein, and the Cas gene were conserved. It was predicted that the tracrRNA was located between Cas9 and Cas1, and the repeat sequence could form a secondary structure with a stem length of seven bases. According to the sequence characteristics of CRISPR spacers, sixcould be divided into three genotypes. Then the spacer sequences were blasted one by one. The results showed that the six spacers aligned 14 original source sequences with different origins, and these spacer sequences were all derived from different plasmids. The PAM sequence recognized bylcCas9 protein preferred T/C, A/C at the 1stand 3rdbases. The 2ndand 4thbases had greater preferences for G and A.【Conclusion】The CRISPR system of six strains ofwere all type-IIA. Consequently, thegene and repeat sequences were highly conserved. The DR sequence formed a stable secondary structure, while TGMA as a PAM sequence was effectively identified by the Cas9 proetein of..
; CRISPR system; spacer;gene; PAM
10.3864/j.issn.0578-1752.2019.03.012
2018-08-03;
2018-11-14
国家重点研发计划(2017YFC1600903)、国家自然科学基金面上项目(31871833)
杨兰,E-mail:poplarorchid@163.com。通信作者逄晓阳,E-mail:pangxiaoyang@163.com。通信作者吕加平,E-mail:lvjp586@vip.sina.com
(责任编辑 赵伶俐)
