APOBEC3基因多态性与乳腺癌易感性的关联研究*
2019-02-16张明龙王秀华赵大龙马洪星郑立红
张明龙,吕 莹,刘 丹,王秀华,董 静,赵大龙,马洪星,郑立红△
(1.齐齐哈尔医学院遗传学教研室,黑龙江齐齐哈尔 161006;2.齐齐哈尔医学院生物技术实验中心,黑龙江齐齐哈尔 161006;3.齐齐哈尔医学院细胞生物学教研室,黑龙江齐齐哈尔 161006; 4.黑龙江省齐齐哈尔市建华医院检验科 161006;5.大庆油田总医院普外科,黑龙江大庆 163411)
乳腺癌是威胁女性健康的常见肿瘤。最近的全基因组关联研究(GWAS)聚焦于分析单核苷酸多态性位点(SNP)与乳腺癌易感性的关联,目前已经确定了67个常见的乳腺癌易感基因位点[1-3]。人载脂蛋白B mRNA编辑酶催化多肽样(APOBEC3)基因簇编码的蛋白质家族含有胞嘧啶脱氨基的结构域,能够将胞嘧啶转化为尿嘧啶,因此又称为胞嘧啶脱氨酶基因。乳腺癌组织及APOBEC3A和APOBEC3B基因的功能学研究结果显示,APOBEC3基因多态性与肿瘤的发生、发展关系密切[4-6]。本研究对黑龙江汉族乳腺癌群体的APOBEC3基因多态性进行研究,以期发现APOBEC3基因与乳腺癌发病机制及临床病理特征等生物学特性的相关性,现报道如下。

表1 引物序列和扩增产物长度
-:无数据
1 资料与方法
1.1一般资料 收集2013-2016年齐齐哈尔医学院附属第二医院、附属第三医院、建华医院和大庆油田总医院的标本,其中病例组为282例乳腺癌标本,均为无亲缘关系的汉族女性,采集在化疗或放疗之前;对照组标本302例,均来自于体检中心,要求无肿瘤和癌前病变,无肝炎病毒、艾滋病毒等病毒感染史。所有标本的采集均符合医学研究伦理委员会的要求,所有对象签订知情同意书。本研究对基本信息进行采集,包括年龄、肿瘤家族史、初潮年龄、绝经与否;对乳腺癌组的生物学特性等信息进行了汇总,其中包括肿瘤大小(T1~T4)、组织学分级(G1~G3)、淋巴结转移与否、雌激素受体阳性与否、孕激素受体阳性与否及病理组织分型。
1.2方法 采用全血DNA快速提取试剂盒提取基因组DNA,并采用微量分光光度计对DNA进行浓度和纯度检测,所有DNA标本的浓度和纯度需满足后续检测的要求。PCR:采用聚合酶链式反应-限制性多态性内切酶技术(PCR-PFLP)对APOBEC3基因的3个SNP位点和1个基因拷贝数变异(CNV)位点进行检测,引物序列、限制性内切酶和产物片段见表1,引物合成委托中美泰和生物技术公司合成。基因分型:采用3%琼脂糖凝胶电泳对PCR和酶切产物进行扩增,根据片段长度进行判读。

2 结 果
2.1标本基本特征数据及均衡性检验 共搜集584例标本,两组年龄、初潮年龄、是否绝经、肿瘤家族史及临床生物学特征数据(肿瘤大小,肿瘤分期,淋巴结转移,雌激素受体和孕激素受体阳性)比较,差异无统计学意义(P>0.05),见表2。
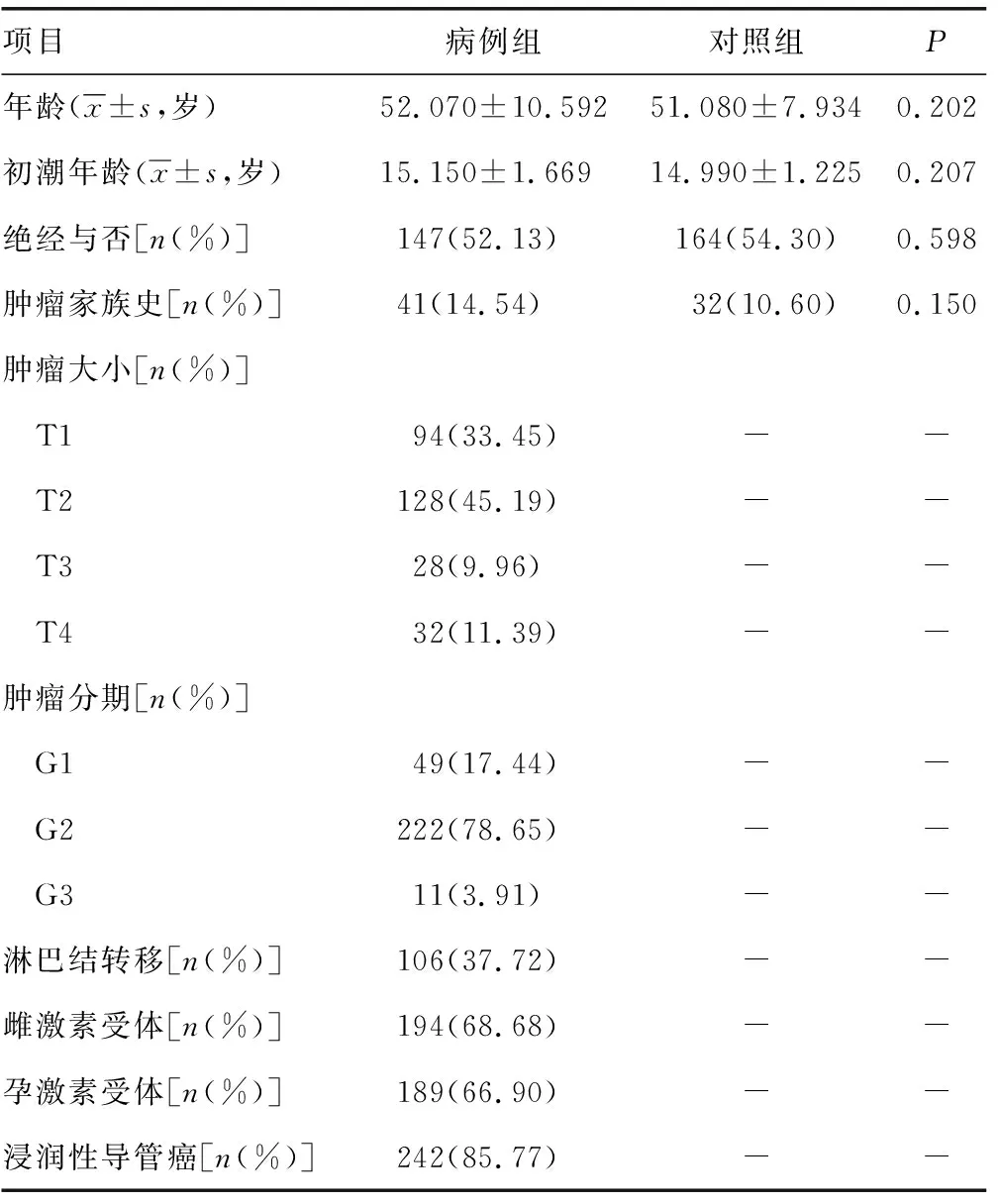
表2 两组基本特征数据信息
-:无数据
2.2平衡性校验 两组各位点的基因型分布观察值与期望值比较,差异无统计学意义(P>0.05),符合Hardy-Weinberg遗传平衡定律。LD检验结果显示,3个SNP位点(rs5757402、rs17370615和rs12157810)构成连锁,可以构成单倍型分析。
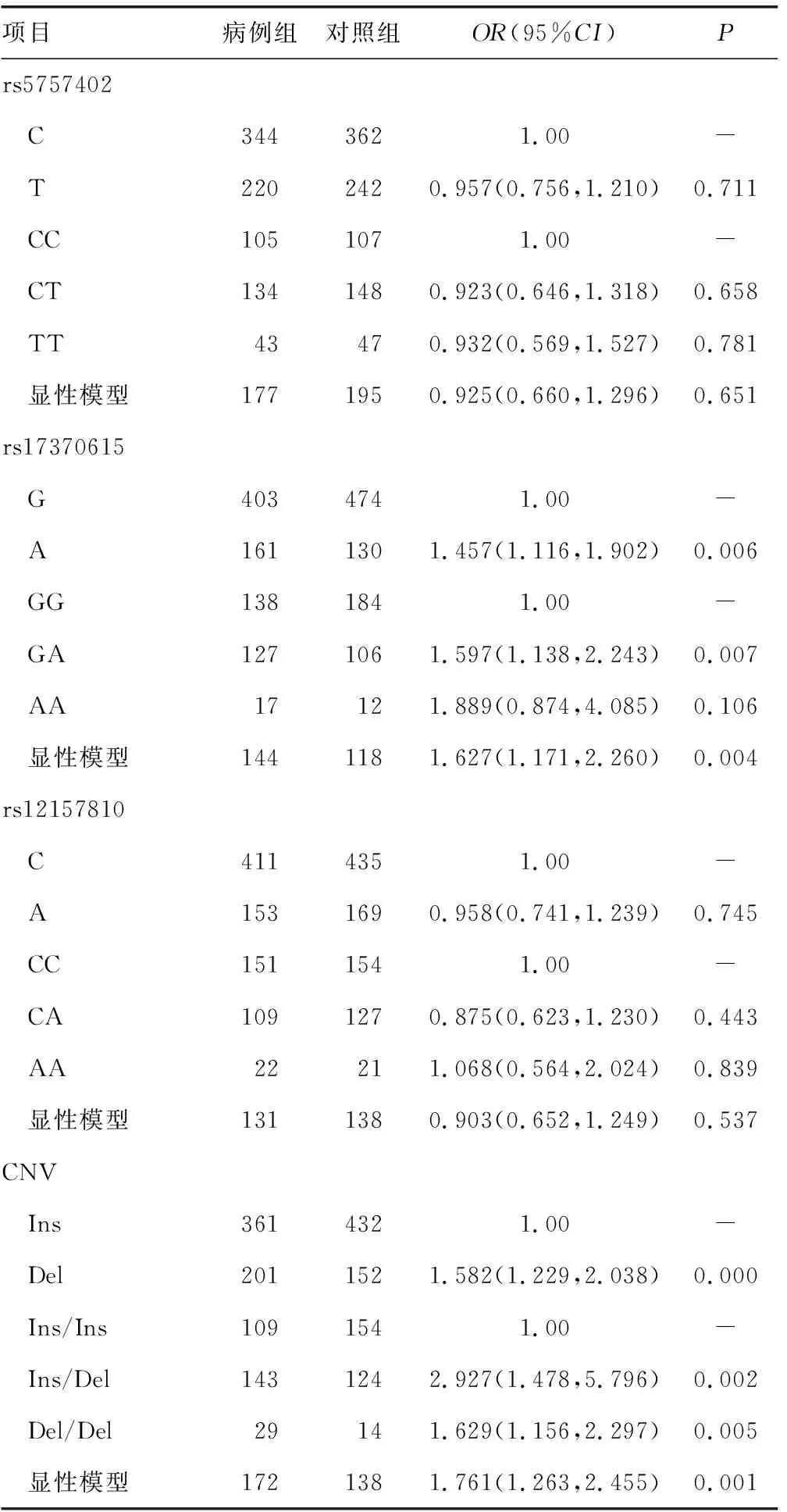
表3 基因型分布和等位基因频率分布及显性不同遗传模式下对易感性风险预测(n)
-:无数据
2.3APOBEC3基因多态性与乳腺癌易感性分析 通过对病例组和对照组的APOBEC3基因的3个SNP位点和CNV位点进行检测,两组间比较结果显示rs17370615和CNV位点的基因频率分布在组间比较差异有统计学意义(P<0.05);rs17370615的GA基因型频率在组间比较差异有统计学意义(P<0.05),对乳腺癌易感性OR值为1.597,CNV位点的Ins/Del和Del/Del基因型频率分布在组间比较差异有统计学意义(P<0.05),对乳腺癌易感性OR值分别为:2.927和1.629;显性模型下,rs17370615和CNV位点在两组间比较差异有统计学意义(P<0.05),对乳腺癌易感性的OR值分别为1.627和1.761;单倍型CGC和CAA的频率分布在组间比较差异有统计学意义(P<0.05),对乳腺癌易感性的OR值分别为0.346和4.587,见表3、4。
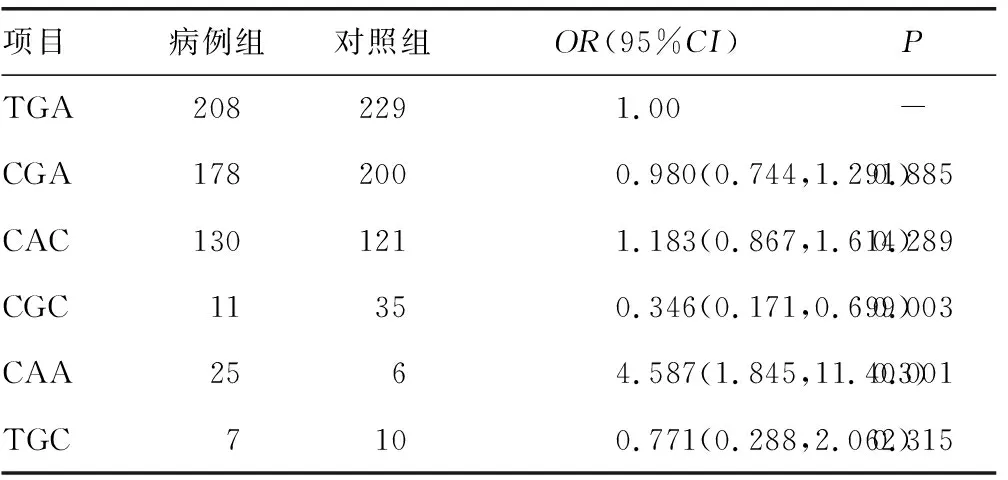
表4 单倍型频率分布及显性遗传模式下对易感性风险预测(n)
-:无数据
2.4APOBEC3基因多态性与乳腺癌生物学特性的关联分析 rs5757402、rs12157810和CNV位点的基因型和显性模型之间的乳腺癌的临床生物学特性(肿瘤大小、肿瘤分期、淋巴结转移、雌激素受体和孕激素受体阳性)比较,差异无统计学意义(P>0.05),rs17370615的基因型GA和显性模型多态性与肿瘤分期(是否大于G1)具有明显关联(P<0.05),见表5。
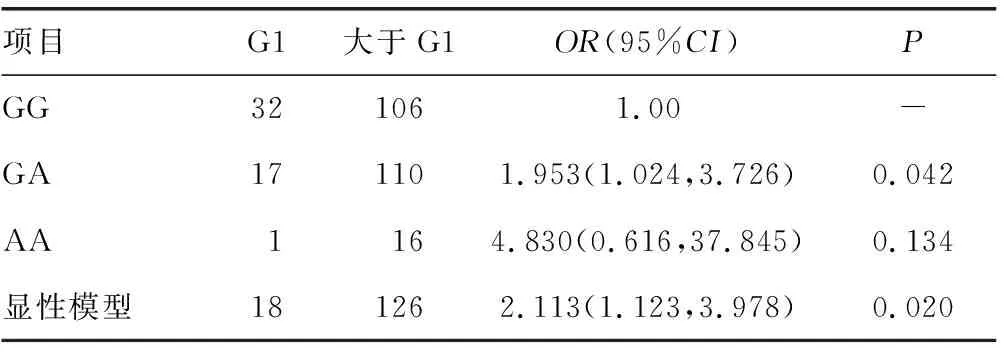
表5 病例组的APOBEC3基因多态性与临床生物学特性的关联
-:无数据
3 讨 论
APOBEC3基因簇编码的家族蛋白都含有1个胞嘧啶脱氨基结构域,一方面具有抑制反转录病毒的细胞免疫功能[7],另一方面还具有抑制内源性的反转座子(LINEs)的作用[8]。基于A3s的胞嘧啶脱氨基功能,可以通过对5-甲基胞嘧啶和5-羟甲基胞嘧啶的脱氨基作用,使错配的基因得以修复,亦使DNA发生去甲基化。同样基于脱氨基功能,A3s又能使DNA发生突变,通过触发DNA突变,APOBEC3基因可能在肿瘤的发生中具有重要作用。基因组测序技术的检测结果证实,由APOBEC3基因所介导的特异性基因突变C>T在多种肿瘤中广泛存在[9],NIK-ZAINAL等[10]对21个原发性乳腺癌组织的完整基因组进行测序,检测到多个与APOBEC3基因密切相关的高突变区,称为kataegis。基因突变对包括乳腺癌在内所有遗传性肿瘤的病因学机制是可以确定的[11],然而SNP位点对于乳腺癌遗传度的贡献仍然有限,最近的研究表明,CNV在基因组中经常出现, CNV可以解释一些复杂疾病的部分遗传度水平[12]。从不同基因突变类型所引起的遗传效应的差异角度解释,CNV较SNP位点对于基因组序列的影响更大。
本研究选取APOBEC3A基因的3个SNP位点(rs5757402,rs17370615和rs12157810)和位于APOBEC3A和APOBEC3B之间的一段缺失变异位点,通过检测和分析,结果显示,3个SNP位点中rs17370615位点的等位基因频率、GA基因型频率和显性模型下,在乳腺癌组和对照组之间具有明显差异,OR值分别为1.457、1.597和1.627。CNV位点的等位基因频率和基因型分布在两组之间差异有统计学意义(P<0.05),携带Del/Del和Ins/Del基因型的个体患乳腺癌的风险是携带Ins/Ins基因型的个体的1.629倍和2.927倍;两组间单倍型比较结果显示,CGC和CAA在两组间比较具有显著性差异,对乳腺癌患病OR值分别为0.346和4.587。以上结果表明APOBEC3基因多态性与乳腺癌发病风险具有一定关联性,特别是rs17370615和缺失型CNV位点,可以认为携带GA、Ins/Del和Del/Del基因型及单倍型CAA是患乳腺癌的危险因素,而单倍型CGC可以认为是保护因素。本研究中的缺失型CNV位点的检测结果与LONG等[13]对中国上海人群的分析结果相同,携带缺失型基因Del的个体较携带Ins个体患病风险显著增加,而欧美白人[14]、摩纳哥黑人[15]和中亚伊朗人[16]群体的检测结果也同样支持以上论断;而对于3个SNP位点的选取和检测结果,摩纳哥黑人群体中rs17370615位点的多态性与乳腺癌发病具有相关性。
本研究将基因多态性与乳腺癌组的临床生物学特性进行比较, rs17370615位点乳腺癌组与肿瘤分期存在关联,提示存在于APOBEC3A启动子区域的SNP位点可能与肿瘤的分化程度相关,而其余位点均与临床数据无关联,无论是从A3s家族蛋白的抑制病毒反转录和内转座子角度,还是从脱氨酶介导的高突变致瘤机制方面来阐明,APOBEC3基因的变异或缺失特别是APOBEC3B基因的整码缺失,与诸多肿瘤的发生存在一定关联性[17-18]。
综上所述,本研究通过对黑龙江汉族女性乳腺癌和对照组的APOBEC3基因多态性的检测和分析,rs17370615和CNV位点的多态性与乳腺癌易感性存在关联性,GA、Ins/Del和Del/Del基因型是乳腺癌发病的危险因素;除rs17370615位点与肿瘤分期存在关联以外,APOBEC3基因多态性与乳腺癌临床生物学特性无明显关联,尚不能认为缺失型等位基因作为单一因素影响乳腺癌的发生、发展和转移过程。本研究所取得的阶段性数据可以为本地区群体的乳腺癌易感基因的筛选、发病风险评估提供前期研究数据,为乳腺癌的个体化防治和诊断做出贡献。
