红曲黄酒来源芽孢杆菌普鲁兰酶基因克隆和生物信息学分析
2019-01-28徐友强孙宝国蒋玥凤许春艳王文华范光森李秀婷
徐友强,孙宝国,蒋玥凤,侯 洁,许春艳,王文华,滕 超,熊 科,范光森,李秀婷,
(1.北京食品营养与人类健康高精尖创新中心,北京工商大学,北京 100048;2.北京工商大学食品学院,北京 100048;3.北京市食品风味化学重点实验室,北京工商大学,北京 100048)
红曲黄酒是我国的代表性黄酒之一,其成品色红、味醇、香浓,独具特色[1]。红曲黄酒酿造以大米为原料,大米淀粉质量分数在70%以上,淀粉的水解,即液化和糖化,是黄酒酿造原料的主要转化过程之一,并为微生物生长提供碳源[2]。淀粉水解速度对于酿造体系微生物的生长及代谢具有显著影响,如果液化和糖化速度过慢,则微生物因缺乏碳源而生长代谢缓慢;如果液化和糖化速度过快,会造成发酵醪中糖浓度过高,进而抑制微生物的代谢[3]。因此,淀粉水解对于黄酒的酿造过程和产品品质影响极大,而决定酿造体系淀粉水解的内在主要源动力,即为微生物来源的淀粉酶[4]。

表1 红曲黄酒来源的主要微生物Table1 Major microorganisms originated from Hongqu glutinous rice wine
传统红曲黄酒酿造过程有多种微生物共同参与[5]。微生物可以产生淀粉酶,以降解原料大米中的淀粉,是酿造过程原料中淀粉转化的内在原动力[4]。已有研究通过现代微生物学和分子生物学技术结合传统微生物分离培养,对红曲黄酒酿造过程真菌和细菌区系变化规律进行分析,并从酒药和酿造过程样品中分离大量霉菌、酵母菌和细菌资源(表1)[6-13]。研究表明,传统红曲黄酒酿造过程是霉菌、酵母菌和细菌共同参与的结果[6-13],并且多种微生物具有产生淀粉酶的能力[14-19]。红曲黄酒酿造真菌来源的淀粉酶主要为α-淀粉酶和葡萄糖淀粉酶[18-19],主要作用于淀粉的内部不规则切开直链淀粉的α-1,4-糖苷键,但不水解支链淀粉的α-1,6-糖苷键,也不水解靠近α-1,6-糖苷键的α-1,4-糖苷键,而大米淀粉中支链淀粉的含量在75%以上,因而酶解产物主要是麦芽糖、少量葡萄糖以及一系列分子质量不等的低聚糖和糊精[18]。这两种酶均难以越过支链淀粉分子中的α-1,6-糖苷键分支点,导致红曲黄酒酿造周期相对较长,原料利用率普遍较低[20]。
与真菌所产淀粉酶种类不同,细菌除产生α-淀粉酶外,还可以产生β-淀粉酶和普鲁兰酶等[21]。其中,普鲁兰酶可以作用于支链淀粉的α-1,6-糖苷键,即可以特异性水解普鲁兰多糖,有助于淀粉的充分水解。基于作用底物和产物的差异,目前普鲁兰酶主要分为I型普鲁兰酶、II型普鲁兰酶、I型普鲁兰糖水解酶、II型普鲁兰糖水解酶和III型普鲁兰糖水解酶[22]。I型普鲁兰酶(EC 3.2.1.41)作用于支链低聚糖的α-1,6-糖苷键,产物为麦芽三糖;II型普鲁兰酶(EC 3.2.1.41)可作用于α-1,4-和α-1,6-糖苷键,水解产物包括葡萄糖、麦芽糖和麦芽三糖;I型普鲁兰糖水解酶(EC 3.2.1.135),又名Neopullulanase,主要在非还原端水解α-1,4-糖苷键,产物是潘糖;II型普鲁兰糖水解酶(EC 3.2.1.57),又名Isopullulanase,主要作用于α-1,4-糖苷键,水解产物为异葡糖基麦芽糖;III型普鲁兰糖水解酶(EC 3.2.1.-)可作用于α-1,4-和α-1,6-糖苷键,水解产物为潘糖、麦芽糖和麦芽三糖。基于这类酶的特性,细菌来源普鲁兰酶在黄酒酿造的淀粉水解过程中应具有重要的作用。但是到目前为止,红曲黄酒微生物淀粉酶的研究仍局限于真菌,特别是霉菌高产淀粉酶菌株的选育及应用[14-16],该过程细菌产普鲁兰酶的相关研究鲜见报道,不利于全面认知红曲黄酒酿造的淀粉水解过程,制约了酿造机制的深入解析,进而导致红曲黄酒酿造效率较低,并且不同批次产品品质稳定性差。前期研究发现,细菌中的芽孢杆菌属菌株存在于红曲黄酒整个酿造过程中,是红曲黄酒酿造的典型代表性微生物[5,23-24],且具有表达普鲁兰酶的能力[10]。基于上述研究现状,在前期从红曲黄酒酿造酒曲中分离具有淀粉水解能力的芽孢杆菌基础上,本实验对芽孢杆菌所携带普鲁兰酶的基本性质进行全面的生物信息学分析,以为后续的深入研究提供参考。
1 材料与方法
1.1 材料与试剂
芽孢杆菌BHQ03、BHQ04和BHQ06由前期工作分离获得,聚合酶链式反应(polymerase chain reaction,PCR)引物委托北京奥科鼎盛生物科技有限公司完成,基因测序委托华大基因完成。克隆载体pEASY-T1、Trans Taq-T DNA聚合酶 北京全式金生物技术有限公司;Escherichia coli DH5α感受态细胞、细菌基因组DNA提取试剂盒 天根生化科技(北京)有限公司;BIOMIGA Gel/PCR Extraction Kit 北京拜尔迪生物技术有限公司。
LB培养基:酵母粉5 g/L、NaCl 10 g/L、胰蛋白胨10 g/L,pH 7.0,LB固体培养基加入2%琼脂粉,121 ℃灭菌20 min。
1.2 仪器与设备
紫外透射反射分析仪 上海康华生化仪器制造厂;Microfuge 20R高速冷冻离心机 美国Beckman Coulter公司;T100TM基因热循环仪 美国Bio-Rad公司;DYY-III-8B稳压稳流电泳仪 北京市六一仪器厂。
1.3 方法
1.3.1 菌种鉴定
在前期工作中,研究通过芽孢杆菌培养基对红曲黄酒酒曲中的芽孢杆菌进行分离和纯化,继而利用淀粉平板对筛选的芽孢杆菌进行培养,依据平板水解圈的大小对菌株的淀粉水解性能进行评估,获得3 株具有显著淀粉水解性能的芽孢杆菌,编号BHQ03、BHQ04和BHQ06,分别接种培养12 h,离心收集菌体,按照细菌基因组DNA试剂盒操作步骤提取基因组DNA。利用细菌16S rRNA基因克隆引物27f和1492r进行PCR扩增获得芽孢杆菌的16S rRNA基因[25],扩增条件:94 ℃、5 min;94 ℃、30 s,55 ℃、30 s,72 ℃、90 s,循环30 次;72 ℃、10 min,4 ℃恒温。
1.3.2 普鲁兰酶基因克隆与测序
根据菌种鉴定结果,NCBI检索近源芽孢杆菌基因组信息,依据注释普鲁兰酶基因序列,设计简并引物(表2),进行普鲁兰酶基因的扩增,扩增条件:94 ℃、5 min;94 ℃、30 s,55 ℃、30 s,72 ℃、2.5 min,循环30 次;72 ℃、10 min,4 ℃恒温。
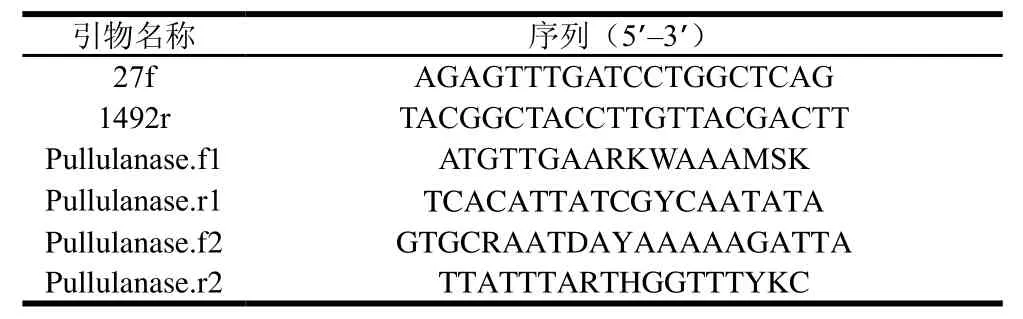
表2 菌种鉴定和普鲁兰酶基因扩增引物Table2 Primers used for strain identification and amplification of the pullulanase genes
扩增所得DNA条带,通过BIOMIGA胶回收试剂盒进行DNA条带纯化,然后按照pEASY-T1载体说明书要求进行普鲁兰酶基因的连接,转化E. coli DH5α感受态细胞,通过蓝白斑筛选挑取阳性克隆子,送至华大基因进行普鲁兰酶基因序列测定。
1.3.3 系统发育分析
菌种鉴定系统发育树构建:将测序所得16S rRNA基因序列提交EzbioCloud细菌专业分类鉴定在线网站(https://www.ezbiocloud.net/taxonomy),将序列相似性大于等于97%的序列下载到本地,按照系统发育分析软件MEGA 5.0要求制作序列比对文件,通过Neighbour-Joining算法构建系统发育树,Bootstrap values为1 000。
蛋白质系统发育树构建:选取代表性的不同普鲁兰酶亚家族蛋白序列,参照菌种鉴定系统发育树构建步骤构建蛋白质系统发育树。
1.3.4 普鲁兰酶序列和基本性质分析
序列拼接及比对选用DNAMAN 7.0软件完成、信号肽的预测通过SignalP 4.1网站进行(http://www.cbs.dtu.dk/services/SignalP/)、理化性质分析采用ExPASy-ProtParam tool完成(http://web.expasy.org/protparam/)。
1.3.5 普鲁兰酶亲/疏水性及跨膜区分析
蛋白亲水/疏水性分析提交预测软件ExPASy-ProtScale完成(http://web.expasy.org/protscale/),利用在线软件TMHMM 2.0进行蛋白跨膜区的预测(http://www.cbs.dtu.dk/services/TMHMM)。
1.3.6 普鲁兰酶二级、三维结构预测和催化机制分析
蛋白二级结构利用软件SOPMA Secondary Structure Prediction Method(https://npsa-prabi.ibcp.fr/ cgi-bin/npsa_automat.pl?page=npsa_sopma.html)进行分析。蛋白三维结构预测通过SWISS-MODEL(http://swissmodel.expasy.org/)完成。分子对接通过DISCOVERY STUDIO 2.5软件完成。
2 结果与分析
2.1 菌种鉴定
16S rRNA基因系统发育分析表明,3 株菌在系统发育树上与B. wiedmannii、B. thuringiensis、B. toyonensis、B. pseudomycoiders和B. cereus聚于同一分支,表明亲缘关系较近(图1),BHQ03与B. cereus、B. wiedmannii、B. thuringiensis、B. toyonensis和B. pseudomycoides的同源性分别为100.00%、99.82%、99.45%、99.45%和99.45%;BHQ04与B. wiedmannii、B. cereus、B. thuringiensis、B. toyonensis和B. pseudomycoides的同源性分别为100.00%、99.83%、99.66%、99.66%和99.31%;BHQ06与B. wiedmannii、B. cereus、B. thuringiensis、B. toyonensis和B. pseudomycoides的同源性分别为100.00%、99.85%、99.55%、99.55%和99.41%。进一步序列分析发现与BHQ03相比,BHQ04与BHQ06亲缘关系更近。因此,后续以BHQ03和BHQ06为研究对象,选取16S rRNA基因序列相似性最高的B. wiedmannii、B. toyonensis、B. thuringiensis和B. cereus共4 株菌基因组注释普鲁兰酶基因序列设计引物。由于B. cereus基因组注释普鲁兰酶基因序列与其余3 株菌差异较大,为降低引物的简并性,以B. cereus基因组注释普鲁兰酶基因序列单独设计引物,然而PCR扩增并未获得预期目标基因。以B. wiedmannii、B. toyonensis和B. thuringiensis基因组注释普鲁兰酶基因序列设计引物,其中,Pullulanase.f1/Pullulanase.r1的参考基因序列NCBI登录号:AT260_RS18700、BTOYO_RS09760和CAB88_RS24225;Pullulanase.f2/Pullulanase.r2的参考基因序列NCBI登录号:AT260_RS06110、BTOYO_RS00120和CAB88_RS13920。
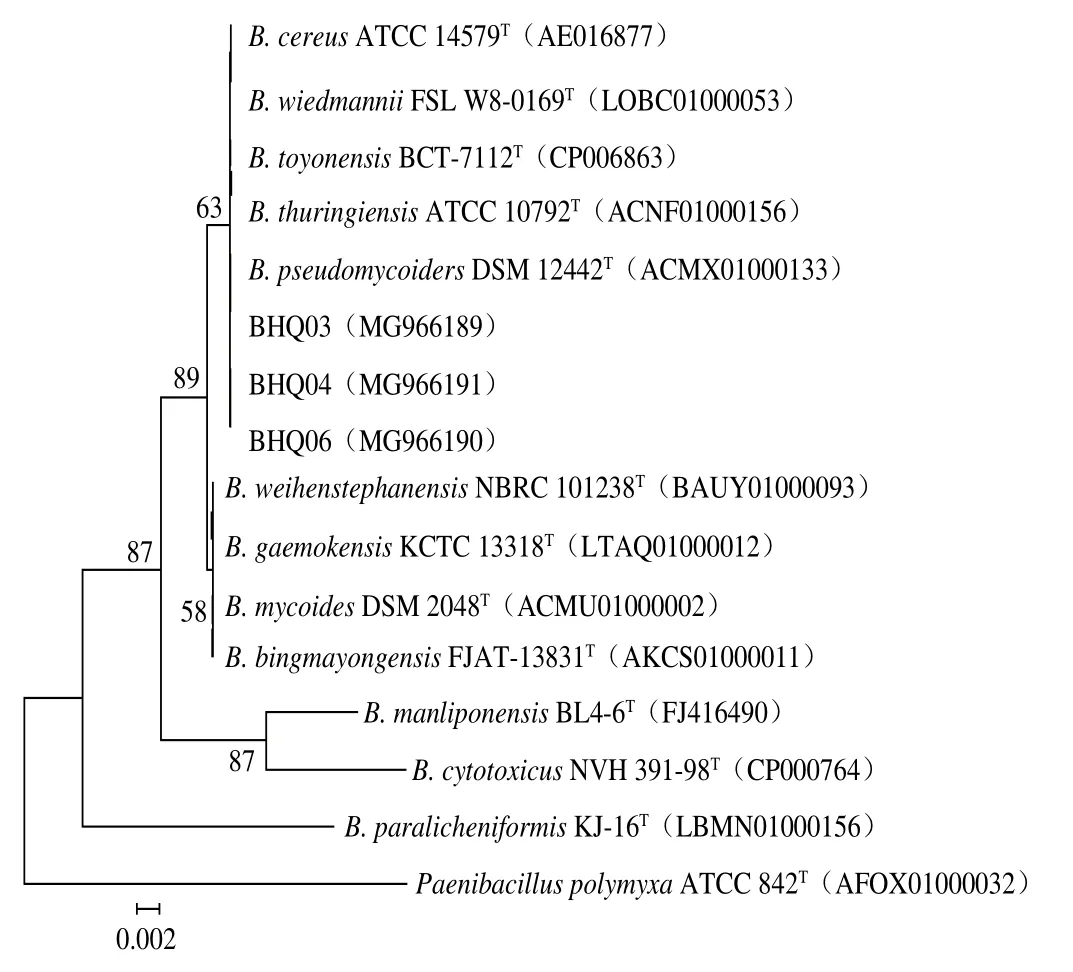
图1 芽孢杆菌16S rRNA基因系统发育分析Fig.1 Phylogenetic analysis of Bacillus strains based on 16S rRNA gene sequences
2.2 普鲁兰酶基因克隆和序列解析
上述分离的2 株Bacillus sp.均携带2 个普鲁兰酶基因,且同一菌株来源的2 个基因在序列上差别较大,比如菌株BHQ03的2 个普鲁兰酶基因pulL1和pulL2序列相似度仅有19.72%,但是2 株菌对应的普鲁兰酶基因序列相似度较高,测序和比对结果表明,pulL1和pulL3的长度均为2 142 bp,共有79 个碱基存在差异,序列相似度达96.31%,pulL2和pulL4的长度均为2 559 bp,仅有6 个碱基存在差别,序列相似度达99.77%。从基因进化角度讲,pulL2和pulL4较pulL1和pulL3在进化上更加保守。将上述4 个普鲁兰酶编码基因提交到NCBI,登录号分别为:pulL1(MG971224)、pulL2(MG971225)、pulL3(MG971226)和pulL4(MG971227)。


图2 普鲁兰酶基因克隆和序列测定结果Fig.2 Cloning and sequencing of the pullulanase genes
2.3 普鲁兰酶基本性质和序列比对分析
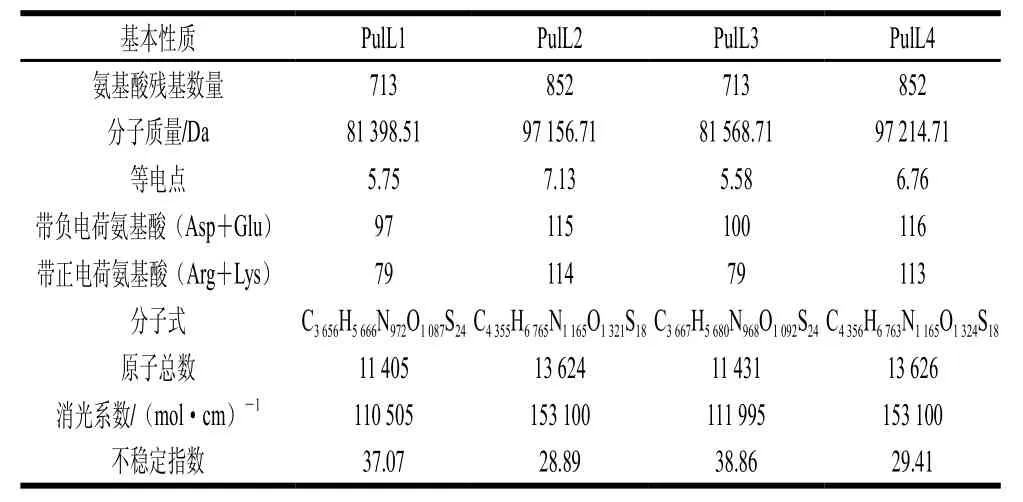
表3 普鲁兰酶基本性质分析Table3 Analysis of the basic characteristics of the pullulanases
对普鲁兰酶的基本性质包括氨基酸残基数量、分子质量、等电点、带负电荷氨基酸、带正电荷氨基酸、分子式、原子总数、消光系数和不稳定指数进行分析,结果见表3。上述基本酶学性质的分析,有助于后续蛋白质的分离纯化和性质研究。其中,4 个酶的不稳定指数均小于40,说明蛋白本身在结构上是稳定的[26]。研究发现,蛋白稳定性的决定因素之一在于其序列中的二肽[26],PulL1、PulL2、PulL3和PulL4之间的氨基酸组成存在差异(表3),导致其序列中的二肽存在不同,是不同普鲁兰酶不稳定指数差异的主要原因。在基因序列测定和酶基本性质的信息解析基础上,研究进一步对酶的氨基酸序列进行系统发育和比对分析。

图3 普鲁兰酶系统发育分析Fig.3 Phylogenetic analysis of the pullulanases
选取代表性普鲁兰酶进行系统发育分析,发现PulL1、PulL2、PulL3和PulL4均属于I型普鲁兰酶(图3),隶属于GH13家族。PulL1和PulL3与B. thuringiensis来源的I型普鲁兰酶进化关系最近,序列相似性分别为99.58%、96.35%,而PulL2和PulL4与B. bombysepticus来源I型普鲁兰酶进化关系最近,序列相似性分别为99.41%、99.77%。I型普鲁兰酶(EC 3.2.1.41)又被称为真普鲁兰酶、脱支酶,仅作用于α-1,6-糖苷键,特异性水解普鲁兰多糖及相关分支多糖,水解产物为麦芽三糖[22]。I型普鲁兰酶不具有水解α-1,4-糖苷键的能力,底物专一性好,在直链淀粉的生产中具有很好的应用。另外,在实际应用中往往将其与其他类型的淀粉水解酶搭配使用,以有效降解淀粉为小分子单糖或多糖[22]。
普鲁兰酶氨基酸序列比对结果表明,PulL1和PulL3存在25 个氨基酸的差别,序列相似度为96.49%;PulL2和PulL4仅有3 个氨基酸的差别,序列相似度为99.65%(图4)。序列比对表明,本研究的4 个普鲁兰酶具有I型普鲁兰酶的4 个特征性高度保守区域,虽然PulL2和PulL4的序列相似度非常高,但是在高度保守区域Region II的催化活性中心存在差别,PulL2的D(Asp)突变为G(Gly),说明普鲁兰酶PulL2和PulL4的催化特性可能存在显著差别(图4)。研究表明淀粉酶的催化机制虽然相似,但是不同种类淀粉酶底物特异性和产物存在差异,推测原因在于,虽然不同种类淀粉酶的活性中心相同,但高度保守区域特别是活性中心位点附近的氨基酸残基不同[21,27-28]。对于同种淀粉酶,由于序列,特别是活性中心附近氨基酸序列的差别,也会带来酶的特性,包括最适温度和pH值、催化反应动力学等的变化[21]。图4的比对结果显示,普鲁兰酶PulL1和PulL2的高度保守区催化活性中心附近的保守区域氨基酸残基也存在差别,可能影响酶的催化性质,有待后续研究。在上述4 个特征性高度保守区域之外,I型普鲁兰酶还具有1 个特征性的7氨基酸残基特征区域“YNWGYDP”(图4标示289~295区域)。文献研究表明该区域与酶的底物特异性密切相关,使酶特异的水解α-1,6-糖苷键[29]。PulL1和PulL3该特征区域存在单氨基酸残基的突变(D294N),该突变可能对酶催化底物的特异性和催化效率产生影响。
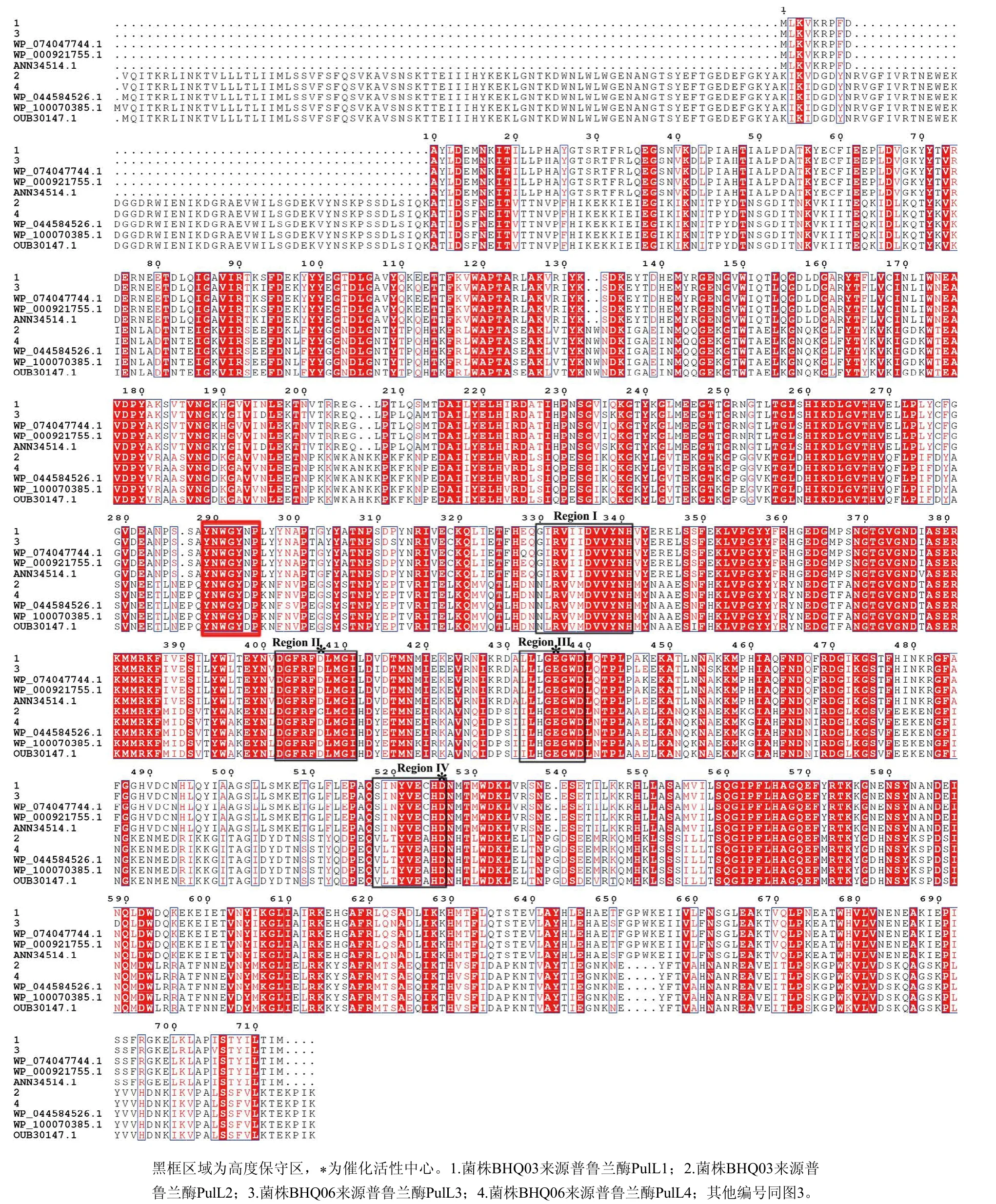
图4 普鲁兰酶氨基酸序列比对Fig.4 Amino acid sequence alignment of the pullulanases
2.4 普鲁兰酶疏水性分析、信号肽和跨膜区预测

图5 普鲁兰酶疏水性分析Fig.5 Hydrophobicity analysis of the pullulanases
采用Hphob/Kyte & Doolittle算法计算PulL1和PulL2的疏水性(图5)。PulL1疏水指数最小值为-3.411(78位),最大值为2.756(554位),总平均亲水性指数(grand average of hydropathicity,GRAVY)为-0.412。PulL2疏水指数最小值为-2.822(336位),最大值为3.289(16位),GRAVY为-0.675。特定位点的氨基酸亲水性指数越大,表明该位点的疏水性越高[30]。PulL1和PulL2虽然都是亲水性蛋白,但是在氨基酸残基亲水性指数和蛋白的总平均亲水性指数均存在差异,说明二者亲水性存在差别,但总平均亲水性指数均为负值,属于亲水性蛋白。氨基酸的疏水作用在很大程度上影响蛋白的稳定性[31],氨基酸疏水性分析对于通过定点突变或者区域替换提高蛋白的稳定性具有重要指导作用。

图6 普鲁兰酶信号肽和跨膜区预测Fig.6 Prediction of the signal peptide and transmembrane domain of the pullulanases
文献报道,不同I型普鲁兰酶有分泌到胞外[32-33]、结合在细胞膜上[34]和存在于细胞内[35]3 种形式。同一普鲁兰酶也可能形成不同的存在形式,例如,Bacillus sp. S-1来源碱性普鲁兰酶具有两种存在形式,前体蛋白主要存在于胞内;成熟蛋白可以结合在细胞膜上[36]。图6表明,PulL2的N-端具有信号肽序列,可以引导其由胞内分泌到胞外,而PulL1并不具有信号肽序列。研究一般认为,没有信号肽的蛋白不能转运,即翻译完毕的蛋白会滞留在合成的区域。因此,生物信息学分析结果说明Bacillus sp.BHQ03来源的2 个普鲁兰酶分别具有分泌到胞外和存在于胞内两种存在形式。
2.5 普鲁兰酶二级、三维结构预测和催化过程分析
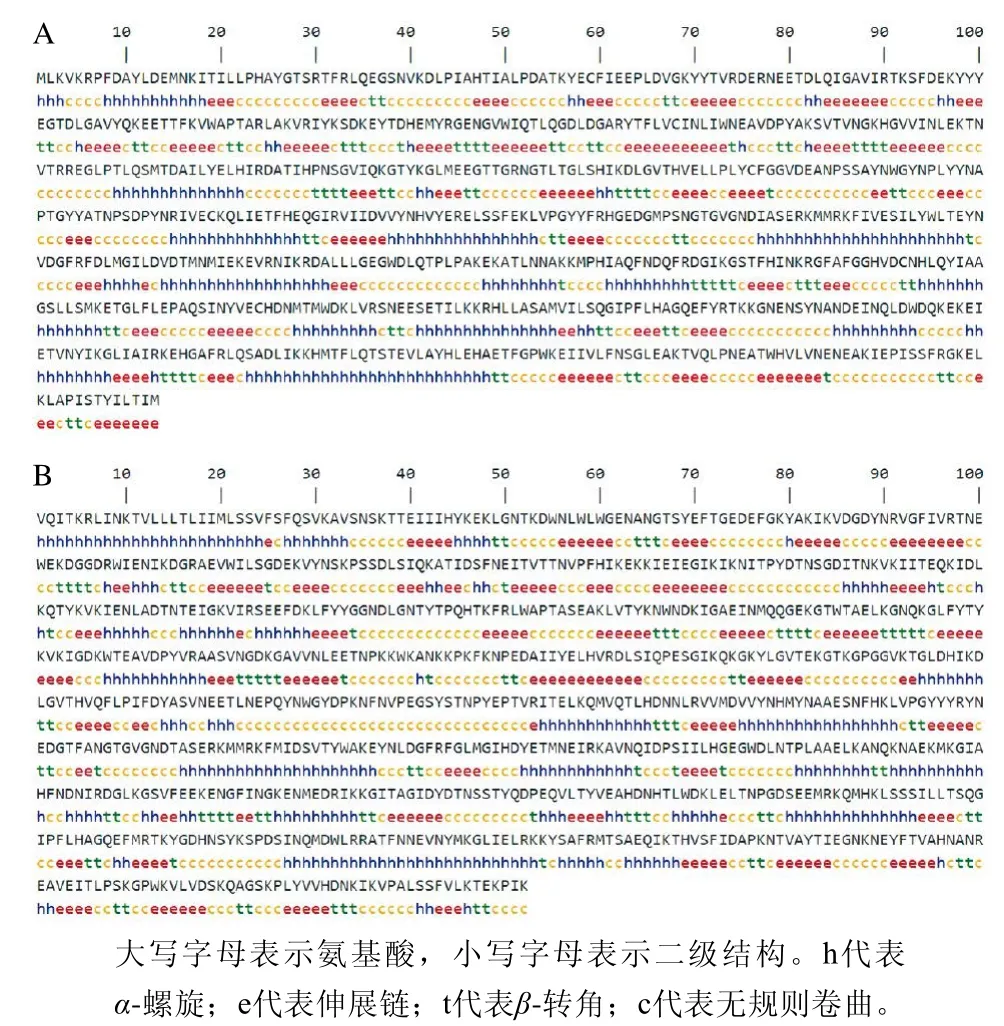
图7 普鲁兰酶PulL1(A)和PulL2(B)二级结构预测Fig.7 Prediction of the secondary structures of the pullulanases
二级结构预测表明,PulL1的α-螺旋占蛋白质的31.00%,伸展链占蛋白质的24.82%,β-折叠占蛋白质的11.64%,无规则卷曲占蛋白质的32.54%(图7A);PulL2的α-螺旋占蛋白质的30.75%,伸展链占蛋白质的25.94%,β-折叠占蛋白质的10.68%,无规则卷曲占蛋白质的32.63%(图7B)。可见,对于Bacillus sp. BHQ03来源普鲁兰酶,α-螺旋和无规则卷曲是蛋白结构的主要组成部分,伸展链和无规则卷曲分散其间。二级结构的预测对于蛋白的研究具有重要帮助,例如蛋白质二级结构统计分析得到的规则可用于全新蛋白质的设计;当序列同源性较低时,二级结构的预测有助于蛋白之间结构与功能的关系分析;在基于二级结构片段堆积的三级结构预测中首先需要正确的二级结构预测;另外,二级结构的预测有助于多维核磁共振中二级结构的确认和晶体结构的解析[37]。

图8 普鲁兰酶三维结构同源建模和催化机制分析Fig.8 Homology-based modeling of three dimensional structures and catalytic mechanism analysis of the pullulanases
与PulL1相比,PulL2的N-端(137~219)具有小的结构域(图8A和8B),文献表明普鲁兰酶N-端区域的截短有助于酶在异源表达时的可溶性,提高表达效率[38]。原因可能是普鲁兰酶蛋白分子较大,截短后降低了蛋白错误折叠的几率。与N-端相比,PulL1的C-端(624~713)与PulL2的C-端(763~852)在结构上差异较小。以PulL1为对象,对底物普鲁兰糖与酶进行分子对接,确定底物与酶催化活性中心可能成键的氨基酸残基(图8C、D),解析催化活性中心与底物的作用机制。文献研究表明,普鲁兰酶的活性中心一般是Asp-Glu-Asp催化三联体结构,两个Asp分别行使酶的亲核基团和稳定过渡态中间物的作用,Glu参与酸碱催化[21,39],催化过程共发生二次置换反应(图8E)。底物的糖残基首先与酶的活性部位结合,该糖基氧原子被充当质子供体的氨基酸残基Glu质子化(图8E II);活性部位的氨基酸残基Asp对糖残基的C1碳原子进行亲核攻击,与底物形成共价中间产物,同时断裂底物糖苷之间的糖苷键,置换出底物的糖基配基部分(图8E III);糖基配基离去之后,水分子被氨基酸残基Glu和另一氨基酸残基Asp激活,将Asp的亲核氧与糖残基C1之间的共价键C1-O-Asp水解掉,置换出酶分子的Asp残基,完成水解反应(图8E IV和V)。
3 结 论
本研究对红曲黄酒酿造酒曲分离的具有淀粉水解特性的芽孢杆菌进行分子生物学初步鉴定和普鲁兰酶基因克隆与生物信息学分析。鉴定结果表明,分离的两株芽孢杆菌BHQ03和BHQ06与B. thuringiensis、B. pseudomycoides、B. toyonensis、B. wiedmannii和B. cereus的亲缘关系较近。基因克隆和测序发现,2 株菌各携带2 个不同序列的普鲁兰酶基因。进一步序列分析显示,BHQ03来源普鲁兰酶PulL1和PulL2均为I型普鲁兰酶,结构稳定,属于亲水性蛋白。PulL2存在信号肽,可以分泌到胞外。两个普鲁兰酶均具有淀粉水解酶的典型高保守区域,且BHQ03来源的普鲁兰酶PulL2催化活性位点存在突变(D407G),表明该蛋白可能存在催化上的特殊性。基于已有研究和三维结构模拟,分析了普鲁兰酶催化的具体作用过程。
上述研究首次报道红曲黄酒酿造酒曲来源芽孢杆菌的普鲁兰酶序列和基本生物信息学特性。后续在基因克隆基础上,拟开展蛋白质的表达、分离纯化和性质研究,以进一步解析不同普鲁兰酶,特别是Bacillus sp. BHQ03来源PulL2的淀粉水解特性,为科学认识红曲黄酒酿造的淀粉水解过程提供依据。
