蛋白质力谱测试中生物分子链耦联技术
2019-01-14胡小唐胡春光何程智高晓晴韩梦柯马国腾李宏斌胡晓东
胡小唐,李 帅,胡春光,何程智, 3,高晓晴,韩梦柯,马国腾,李宏斌,,胡晓东
蛋白质力谱测试中生物分子链耦联技术
胡小唐1, 2,李 帅1, 2,胡春光1, 2,何程智1, 2, 3,高晓晴1, 2,韩梦柯1, 2,马国腾1, 2,李宏斌1, 2,4,胡晓东1, 2
(1. 精密测试技术及仪器国家重点实验室(天津大学),天津 300072;2. 天津大学南昌微技术研究院,天津 300072;3. 北京软物质科学与工程高精尖创新中心(北京化工大学),北京 100029;4. 加拿大英属哥伦比亚大学化学系,温哥华 V6T 1Z4)
蛋白质力谱测试是研究生物学的重要组成部分.蛋白质力谱测试的成功取决于两大技术:一是分子尺度的力谱测量能力,即皮牛量级的力学分辨力和纳米级别的空间分辨力;二是蛋白质样品制备能力,通过将微观生物分子链与介观微球/探针/基底耦联,实现对分子尺度蛋白质的有效测量.然而,蛋白质样品的耦联情况很难在视觉上直接观测,耦联效果的好坏决定测试的成败.因此,蛋白质样品相关制备方法、耦联工艺一直是单分子力谱测试中的研究重点.针对3种主要单分子力谱测试技术即光镊、磁镊及原子力显微术对测试蛋白质的需求特点,特别是对多分子链耦合样品的测试需求特点,介绍了多种基于基底修饰、蛋白质修饰和DNA链修饰的提高连接待测蛋白质与DNA链/微球/探针/玻片/云母片的方法,分析了各自的优缺点,并总结了典型的应用,为相关领域中样品的制备提供参考方案.
蛋白质力谱;光镊;磁镊;原子力显微术;耦联
生物学是研究生命现象和生物活动规律的科学,包括肌肉收缩、物质输运、细胞分裂、DNA复制等生命体的活动,都是与分子马达的推动息息相关的.而单分子力谱是研究生物学的重要技术手段之一,例如细胞操控、分子马达、DNA、蛋白质折叠/解折叠等.在研究蛋白质折叠/解折叠动力学特性过程中,具有代表性的单分子力谱技术中的光镊[1-3]、磁镊[4-5]以及原子力显微术[6-8]在各自研究领域中发挥着优势.然而,三大技术在进行蛋白质力谱测试时也面临一个共性问题,即蛋白质样品的耦联效率低.由于待测的目标蛋白通常都是纳米级别,一方面视觉上无法直接看到,另一方面由于其尺寸小,在与DNA分子链或玻片/云母片基底进行连接时难于判别其是否连接到分子链或基底上,使实验变得复杂化,增加了待测蛋白与测试系统连接的难度.
原子力显微术系统中,待测的生物样品通过探针以及玻片/云母片表面与蛋白的非特异性相互作用(物理吸附)进行连接;磁镊系统中,待测的目标蛋白是通过两条/一条DNA链间接连接或者经过生物素及地高辛修饰后直接连接到磁球及玻片表面;常见的光镊系统有两类:单光镊和双光镊.其中单光镊中的生物样品与系统的连接构型和磁镊系统相似,都是将生物样品一端连接在玻片上,另一端连接在微球表面;双光镊系统又分为光镊和微针组成的和两个光镊组成的两种,前者是通过微针先捕获一个微球,将其送到光镊中,然后微针再捕获一个微球,而后者是通过双光镊分别捕获一个微球,二者的共同点是均要捕获两个微球,待测的生物样品多数情况下是通过两条/一条DNA链间接地连接到双光镊系统中的两个聚苯乙烯/二氧化硅微球表面.然而在单分子力谱实验中,生物样品的耦联效果即待测目标蛋白质与DNA链/玻片/云母片/探针的连接牢固程度将对后续的实验效率起决定性的作用.
早期研究者们在利用单分子力谱技术研究DNA时,通过在DNA的两端分别修饰生物素和地高辛、借助生物素和地高辛分别与聚苯乙烯/二氧化硅/磁球表面修饰的链霉亲和素和地高辛抗体间的反应将DNA连接在测试系统中进行实验,耦联效果理想.随着单分子力谱技术的适用范围不断扩大,当应用其研究蛋白质折叠/解折叠时,多数借助待测蛋白与两条DNA链所形成的二硫键及DNA链与微球表面的链霉亲和素或地高辛抗体作用将待测蛋白连接在测试系统中,但是此方法产率较低,笔者小组的产率在5%左右,而且此方法通常需要室温下过夜反应,这对许多在室温下并不是特别稳定的蛋白质是非常不利的.其次,在反应过程中蛋白质上的巯基之间或DNA上的巯基之间也会发生氧化反应形成二硫键,生成蛋白-S-S-蛋白或者DNA-S-S-DNA这样的副产物.而且由于蛋白质尺寸通常很小,DNA-S-S-DNA与DNA-S-S-蛋白-S-S-DNA 在尺寸上很难区分,使后续实验变得更加复杂.再次,由于两种DNA分子(Biotin和Digoxigenin修饰)同时与蛋白质反应,通过此方法制备的蛋白-DNA嵌合体产物中除了理想产物Biotin-DNA-蛋白-DNA-Digoxigenin外,还会存在Biotin-DNA-蛋白-DNA-Biotin和Digoxigenin-DNA-蛋白-DNA-Digoxigenin两种副产物,无法用于光镊实验.为了提高样品产率,后期有研究者改为利用一条DNA链将待测蛋白连接在测试系统或者将待测蛋白不通过DNA链作用直接连接在测试系统中.但相比于利用两条DNA链的情况,这会出现激光辐射热会使待测蛋白变性的可能性增大的弊端.为进一步提高耦联效率,研究者们不断尝试新方法,如对待测蛋白进行各种标签修饰、巯基修饰等以更牢固地将其连接在测试系统中.总结研究者们使用的修饰方法,主要包含三大类:第一类是基底(二氧化硅/聚苯乙烯微球、磁球及玻片/云母片)修饰;第二类是蛋白质修饰;第三类是DNA链修饰.
1 修饰方法
1.1 基底修饰
在单分子力谱实验中,基底修饰可分为两种.一种是二氧化硅、聚苯乙烯微球以及磁球修饰,通常情况是在这3种材质微球表面包被地高辛抗体或者链霉亲和素,借助生物素-链霉亲和素间的特异性相互作用以及地高辛-地高辛抗体间的抗原-抗体反应将微球与待测蛋白质或DNA链连接.除此之外,也有通过在二氧化硅微球表面经过一系列反应修饰上巯基,借助微球表面的巯基与待测蛋白表面的巯基所形成的二硫键将微球与待测蛋白连接.另一种是对玻片/云母片基底修饰,可分为两大类:一类为硅烷氨基化处理,氨基化处理后的基底可通过双官能团化学试剂Maleimide-PEG-Biotin使得基底表面带有生物素,用于与表面修饰有链霉亲和素的待测蛋白反应进而将待测蛋白连接在基底表面;氨基化处理后的基底也可通过双官能团化学试剂Maleimide-PEG-NHS使得基底表面带有Maleimide基团,通过Maleimide将表面带有半胱氨酸残基的待测蛋白连接在基底表面;或者通过Maleimide与带有巯基的辅酶CoA反应,借助CoA与待测蛋白表面修饰的ybbR标签间的相互作用将待测蛋白连接在基底表面;也可通过Maleimide与双官能团试剂SH-PEG4-chloroalkane反应,进而将HaloTag Ligand连接在基底表面,用于与表面修饰有HaloTag标签的待测蛋白相连接.另一类为硅烷羧基化处理,羧基化处理后的基底可通过双官能团试剂BD-PEG-NH2将表面修饰有hAGT (SNAP)tag标签的待测蛋白连接在基底表面.
1.2 蛋白质修饰
为了研究蛋白质的特性,通常需要在待测蛋白表面做一些修饰进而将待测蛋白与测试系统相连接.修饰方法主要分为3种.
1) 蛋白修饰巯基
通常情况下,由于自身带有巯基的待测蛋白比较少,所以通过基因合成的手段在待测蛋白表面修饰半胱氨酸残基,借助半胱氨酸残基带有的巯基或待测蛋白自身带有的巯基与微球或基底表面或DNA链上带有的巯基所形成的二硫键将待测蛋白与微球或基底相连接.
2) 蛋白修饰标签蛋白
标签蛋白是指在表达待测蛋白时在其表面共同表达的特定蛋白用于与测试系统相连接,在使用的标签蛋白中,最常见的是Spytag、hAGT(SNAP)tag、ybbR tag及HaloTag 4种.Spytag是通过在待测蛋白表面修饰上Spytag标签,借助Spytag与SpyCatcher两者间反应形成的酰胺键将待测蛋白与DNA链或基底相连接;hAGT tag是通过在待测蛋白表面修饰上hAGT tag标签,借助hAGT tag与BG-PEG-amino间的相互作用将待测蛋白与基底相连接;ybbR tag是通过在待测蛋白表面修饰上ybbR tag标签,在SFP催化转移酶的作用下将待测蛋白与基底连接;HaloTag是通过在待测蛋白表面修饰上HaloTag标签,借助HaloTag与HaloTag Ligand间的相互作用将待测蛋白与基底连接.
3)蛋白修饰生物素或链霉亲和素
生物素与链霉亲和素间的相互作用是常见的最稳定的非共价键相互作用,表面修饰生物素是通过基因合成技术在待测蛋白表面修饰有生物素,借助生物素与微球或基底表面修饰的链霉亲和素间的特异性反应将待测蛋白与微球或基底连接;表面修饰链霉亲和素是通过基因合成技术在待测蛋白表面修饰有链霉亲和素,借助链霉亲和素与基底或探针表面修饰的生物素间的特异性反应将待测蛋白与基底或探针连接.
1.3 DNA链修饰
在单纯研究DNA链的特性时,仅需在DNA链的一端修饰生物素另一端修饰地高辛,借助生物素-链霉亲和素、地高辛-地高辛抗体间的相互作用将待测DNA链与微球或基底连接;在将DNA链作为待测蛋白的连接把手时,通常是在DNA链的一端修饰生物素或地高辛,另一端修饰巯基或氨基,借助巯基与待测蛋白表面的巯基所形成的二硫键、生物素或地高辛与微球表面的链霉亲和素或地高辛抗体间的相互作用将待测蛋白与微球连接;还有一种情况是通过在寡核苷酸一端修饰叠氮化物后,对经过DBCO激活后的待测蛋白进行定点敲击反应,通过寡核苷酸将待测蛋白与DNA链连接,进而间接地连接到微球表面.
总结以上3种修饰方法,做出示意图如图1所示.

图1 修饰方法
2 应 用
2.1 原子力显微术测试系统
早期在用原子力显微术系统研究蛋白质折叠/解折叠的实验中,是借助待测蛋白与探针和玻片基底的物理吸附力将待测蛋白连接在测试系统中,然而由于吸附力小,蛋白容易与探针或玻片脱落,极大地降低了实验效率.因此,在后续的实验中,研究者们不断尝试新方法来提高待测蛋白与测试系统的连接牢固度进而提高实验测试效率.
Los Georgyi等[9]于2008年首次提出借助HaloTag标签与不同HaloTag Ligand配体的相互结合作用研究细胞成像以及蛋白固化的方法,证明了HaloTag标签蛋白的有效性,为相关领域的研究者提供了一个新的方法.而后有研究者将该方法应用于原子力显微术系统中,即通过使用云母基底氨基化处理、待测蛋白HaloTag标签修饰及探针金修饰三步法,将待测蛋白(I27)4连接在云母基底表面,进而进行后续的蛋白质解折叠拉伸实验[10],其原理及部分实验数据如图2所示.
图2(a)和(b)介绍了HaloTag标签法的全过程,即首先用APTES对云母基底进行氨基化处理,再用一端带有NHS另一端带有巯基的SM(PEG)24将HaloTag Ligand连接在云母基底表面,通过待测蛋白(I27)4表面修饰的标签蛋白HaloTag与基底修饰的HaloTag Ligand间的相互作用将待测蛋白(I27)4连接在基底上;图2(c)对应的是蛋白质拉伸实验数据,力谱曲线中的拐点代表蛋白质的解折叠,通过蠕虫链模型,可以拟合得到每个拐点所对应的蛋白质的轮廓长度伸长量,进而得到解折叠力及对应的伸长量关系.
在使用HaloTag标签法实现提高待测蛋白与测试系统的连接牢固度的同时,也有不同的课题组结合自己研究需要进行不断的探索和尝试.Yin等[11]的ybbR标签法是借助SFP催化转移酶的催化作用将带有ybbR标签的待测蛋白与不同结构的小分子化合物连接,并通过实验验证了ybbR标签法的灵活性及高效性.Zakeri等[12]提出Spytag-SpyCatcher标签法,用于将两种蛋白质连接在一起,并设计实验验证了Spytag-SpyCatcher在不同温度、pH、液体环境下的稳定性,最后将该种方法应用于原子力显微术系统中,通过解折叠力的大小验证了Spytag-SpyCatcher结合的牢固度.Pippig等[13]通过在待测绿色荧光蛋白GFP表面分别修饰ybb Rtag标签及GCN4肽键链,在FP催化转移酶的作用下将待测蛋白与一端修饰有辅酶CoA的DNA单链借助CoA与ybbR所形成的共价键将蛋白与DNA单链连接,并将该方法用于原子力显微术系统中,验证了该方法可用于探针转移单分子实验,为DNA折纸技术及分子自组装提供有效手段,其原理[13]如图3所示.
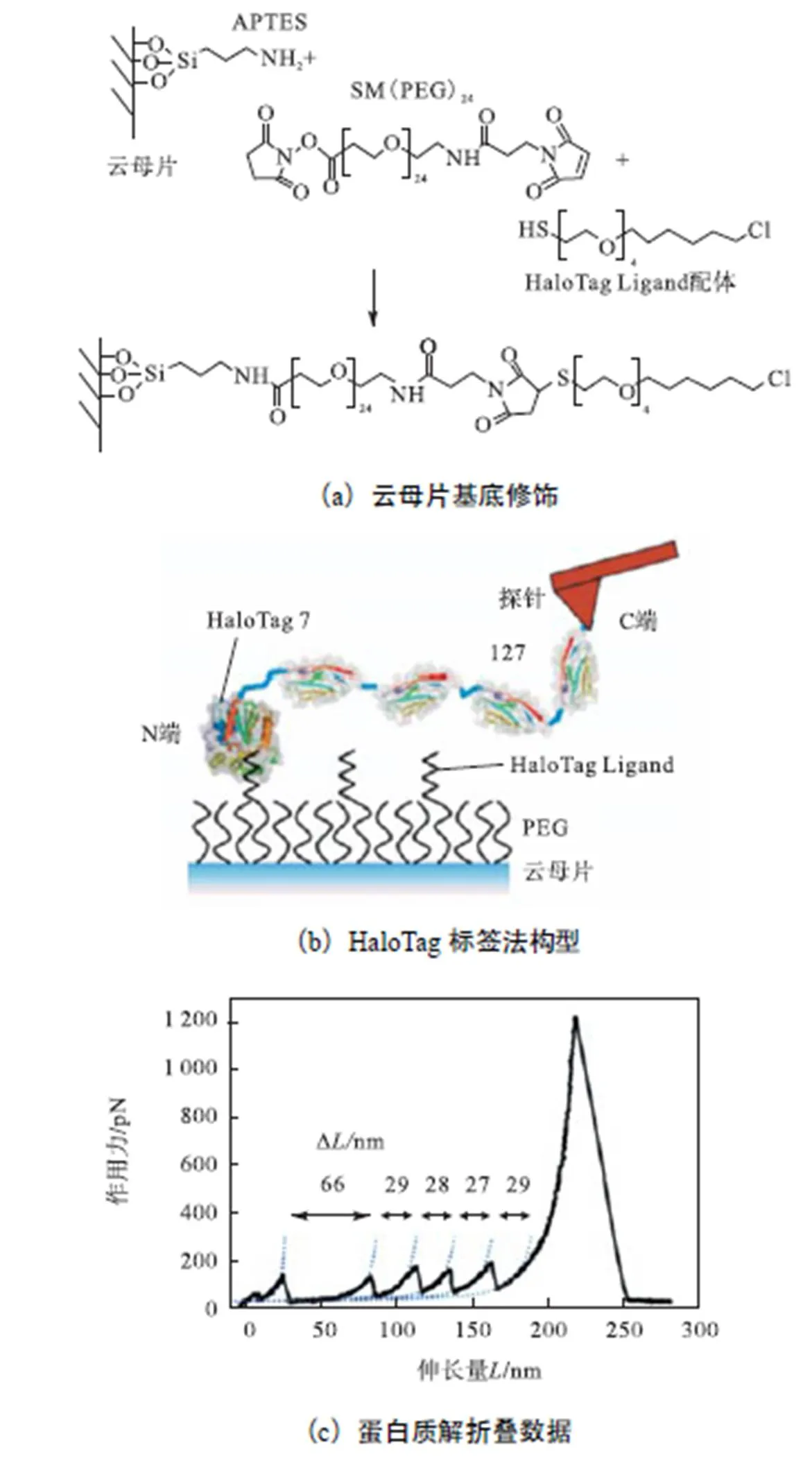
图2 HaloTag标签法原理及部分数据
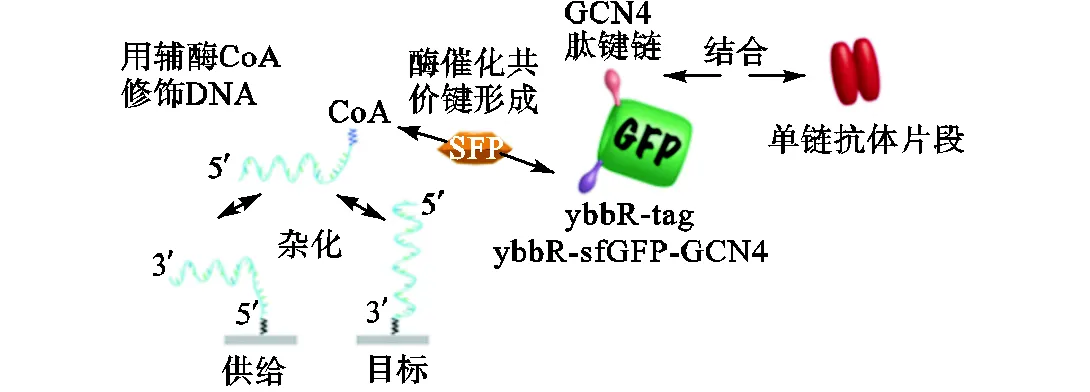
图3 ybbR标签法原理
通过在待测蛋白ddFLN4表面修饰链霉亲和素mcSA2及ybbR标签蛋白,相应地在探针表面通过PEG修饰辅酶CoA、在基底表面通过PEG修饰生物素,借助CoA与ybbR形成的共价键将待测蛋白与探针连接,借助待测蛋白表面的链霉亲和素与基底表面的生物素的特异性反应进行拉伸实验或者在探针表面通过FIVAR修饰上生物素、在基底表面通过修饰CoA进而将修饰有链霉亲和素的待测蛋白ddFLN4连接在基底表面,借助探针上的生物素与基底表面的链霉亲和素间的特异性反应进行拉伸实验,是应用于原子力显微术系统中通过对待测蛋白进行标签修饰进而对其进行拉伸实验的一种新方法[14],其原理及部分数据如图4所示.
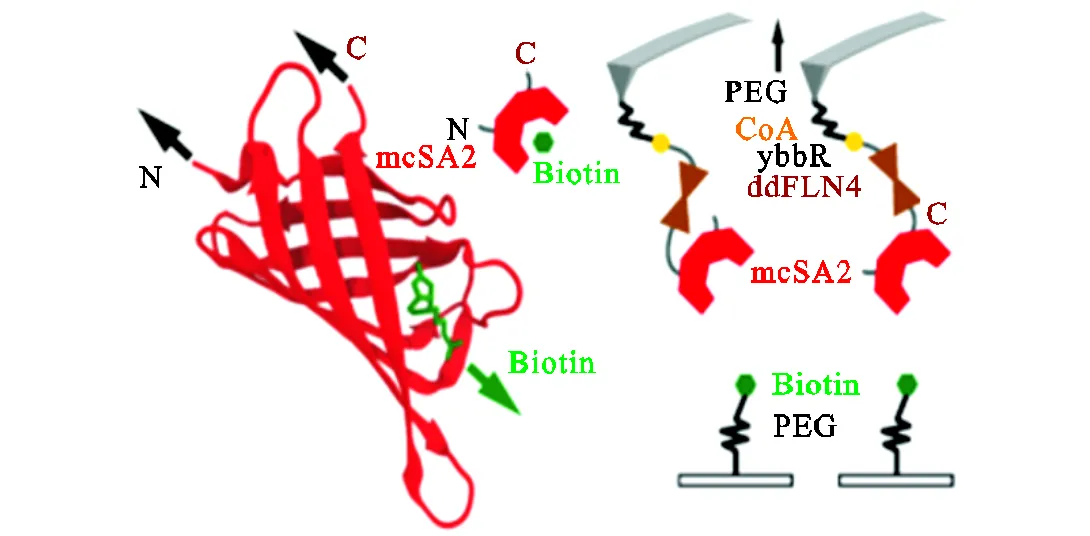
通过在待测蛋白表面修饰链霉亲和素、在基底表面通过PEG作用修饰生物素Biotin,借助生物素和链霉亲和素间的相互作用实现待测蛋白与系统的牢固连接.通过从链霉亲和素的N端及C端拉伸待测蛋白质ddFLN4所获取的不同力谱曲线.以上实验数据表明从链霉亲和素不同端点拉伸待测蛋白所需要的拉伸速度不同,进而所对应的解折叠力和折叠力有所不同,说明链霉亲和素不同端点与待测蛋白的相互作用力有所不同.此外,上述数据表明利用链霉亲和素标签法进行生物样品耦联时,不管是从N端还是C端进行连接,耦联效果均非常好,从而验证了该耦联方法的有效性.
综观应用于原子力显微术测试系统中的生物分子链耦联技术,为提高待测蛋白与系统的连接牢固度,研究者们多采用对待测蛋白进行标签修饰的方式,例如:HaloTag标签法,该方法可用于包含半胱氨酸的蛋白质及带有SNAP标签的蛋白质,而且在构建重组蛋白过程中,HaloTag可提高表达级别及在大肠杆菌中的溶解性;ybbR标签法,该方法操作简单,成本较低;链霉亲和素修饰法,该方法中的生物素链简单易制备,链霉亲和素链稳定可长时间保持活性,从而可长效用于反复的解折叠、折叠拉伸实验.以上各方法描述的都是对待测蛋白进行修饰,然而为提高连接牢固度,还需在对待测蛋白修饰的基础上,相应地对玻片/云母片/探针进行修饰以进行特定的连接,修饰方法相对蛋白修饰比较简单,这里不再赘述.
2.2 磁镊测试系统
在用磁镊系统研究蛋白质折叠/解折叠的实验中,通常情况下待测蛋白是通过一条或两条DNA链与玻片基底和磁球借助地高辛-地高辛抗体、生物素-链霉亲和素间相互作用实现其与测试系统的连接.然而由于此种方法中待测蛋白与DNA链的连接效率非常低,因此探究新的连接方法显得极为重要.
严洁课题组通过在待测蛋白的N端修饰HaloTag标签,C端修饰AviTag标签,N端通过HaloTag与玻片表面修饰的HaloTag 配体连接,C端通过AviTag与生物素反应后再通过生物素-链霉亲和素相互作用将待测蛋白间接地连接到表面修饰有链霉亲和素的磁球表面[15-17].Yoon课题组将二硫键连接法即借助待测蛋白与DNA链间形成的二硫键将待测蛋白连接在基底表面应用于自己课题组的磁镊系统中,先后研究了蛋白质SNARE、GlpG在折叠/解折叠过程中的动力学特性及能量改变信息[18-19].在磁镊系统中,使用Spytag-SpyCatcher标签法将待测蛋白GlpG表面修饰Spytag标签后与一端修饰生物素另一端通过核糖体修饰SpyCatcher及一端修饰地高辛、另一端通过核糖体修饰SpyCatcher的两条DNA链连接在一起进而间接将待测蛋白连在经PEG修饰的玻片及链霉亲和素包被的磁球表面进行拉伸实验[20],其原理如图5所示.
在磁镊系统中,使用二硫键连接法的操作简单但连接效率极低;HaloTag标签法通过在待测蛋白表面修饰HaloTag标签,对玻片基底修饰HaloTag 配体,借助二者间的相互作用实现待测蛋白与磁镊系统的连接,该方法的连接效率大为提高;Spytag标签法在节省反应时间及成本的同时,由于DNA链是连接在目标蛋白的两端(N端和C端),所以也存在不能改变拉伸目标蛋白的几何形状的问题.
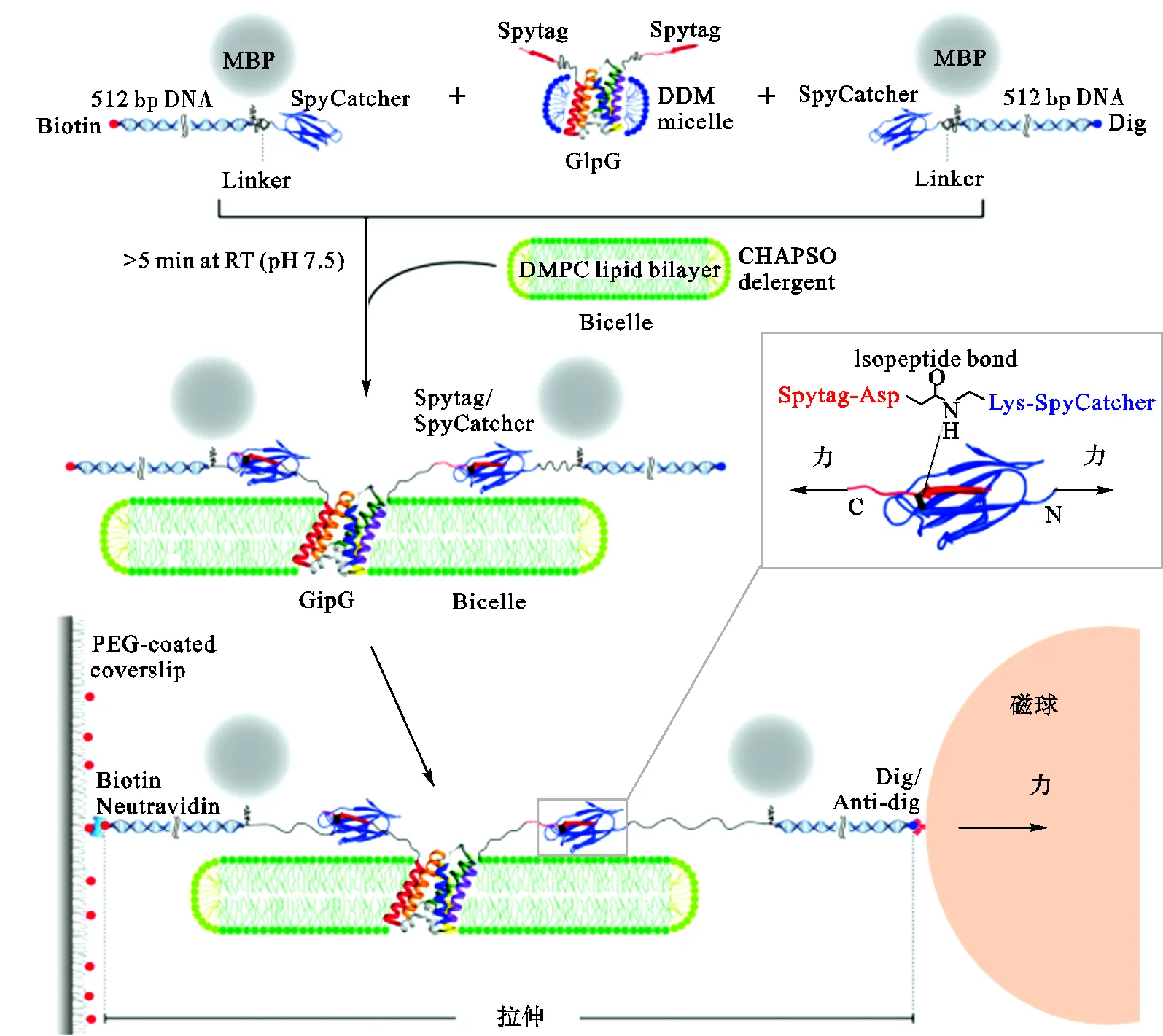
图5 Spytag-SpyCatcher标签法原理
2.3 光镊测试系统
在用双光镊系统研究蛋白质折叠/解折叠实验中,待测蛋白往往是通过两条或一条DNA链,借助待测蛋白与DNA链所形成的二硫键将待测蛋白间接连接到微球表面.
在待测蛋白表面修饰半胱氨酸残基,借助残基中的巯基与DNA链一端修饰的巯基所形成的二硫键将待测蛋白通过DNA链连接在微球表面,用于蛋白质RNase H单体及多聚体的解折叠/折叠研究[21].
Gebhardt等[22]在2010年将该种方法应用于自己小组的双光镊系统中生物样品的连接上,成功研究了包含有3个GCN4结合位点的蛋白质LZ26GCN4的折叠/解折叠动力学参数特性以及相应的能量图谱.
笔者小组也将该方法应用于自己搭建的双光镊系统中,用于研究蛋白质(NuG2)4的折叠/解折叠动力学特性.实验中,待测蛋白(NuG2)4通过两条不同长度即400bp和800bp的DNA链连接在双光阱所捕获的表面分别包被有地高辛抗体和链霉亲和素的聚苯乙烯微球上,通过在待测蛋白(NuG2)4两端分别
修饰半胱氨酸,借助半胱氨酸与DNA链一端修饰的巯基所形成的二硫键将待测蛋白与DNA链进行连接,借助DNA链另一端分别修饰的地高辛或生物素与微球连接.图6是该种方法的实验数据.理想情况下待测蛋白(NuG2)4与两条DNA链连接后的条带应该是在1200bp位置附近,然而图6(a)的琼脂糖凝胶电泳图中该位置处的条带特别浅,说明该种方法的连接效率低,导致获取图6(b)蛋白质(NuG2)4力谱曲线的效率特别低.
使用两条DNA链连接待测蛋白方法的优点是提高了空间分辨率,减少待测蛋白与微球间的非特异性相互作用,但缺点是连接效率低.
为了提高生物样品的连接效率,Rief课题组在该种方法的基础上进行了改变,即通过待测蛋白质表面所修饰的半胱氨酸残基与寡核苷酸链连接,然后借助蛋白质表面的寡核苷酸链与双链DNA一端所连接的寡核苷酸单链进行杂化反应,最终形成微球-双链DNA(连有寡核苷酸单链)-待测蛋白(连有寡核苷酸单链)-双链DNA(连有寡核苷酸单链)-微球结构,借助该种方法,Rief组成功研究了蛋白质Hsp70及Hsp90在折叠/解折叠过程中不同结合位点状态变化所对应的特性信息[23-24].
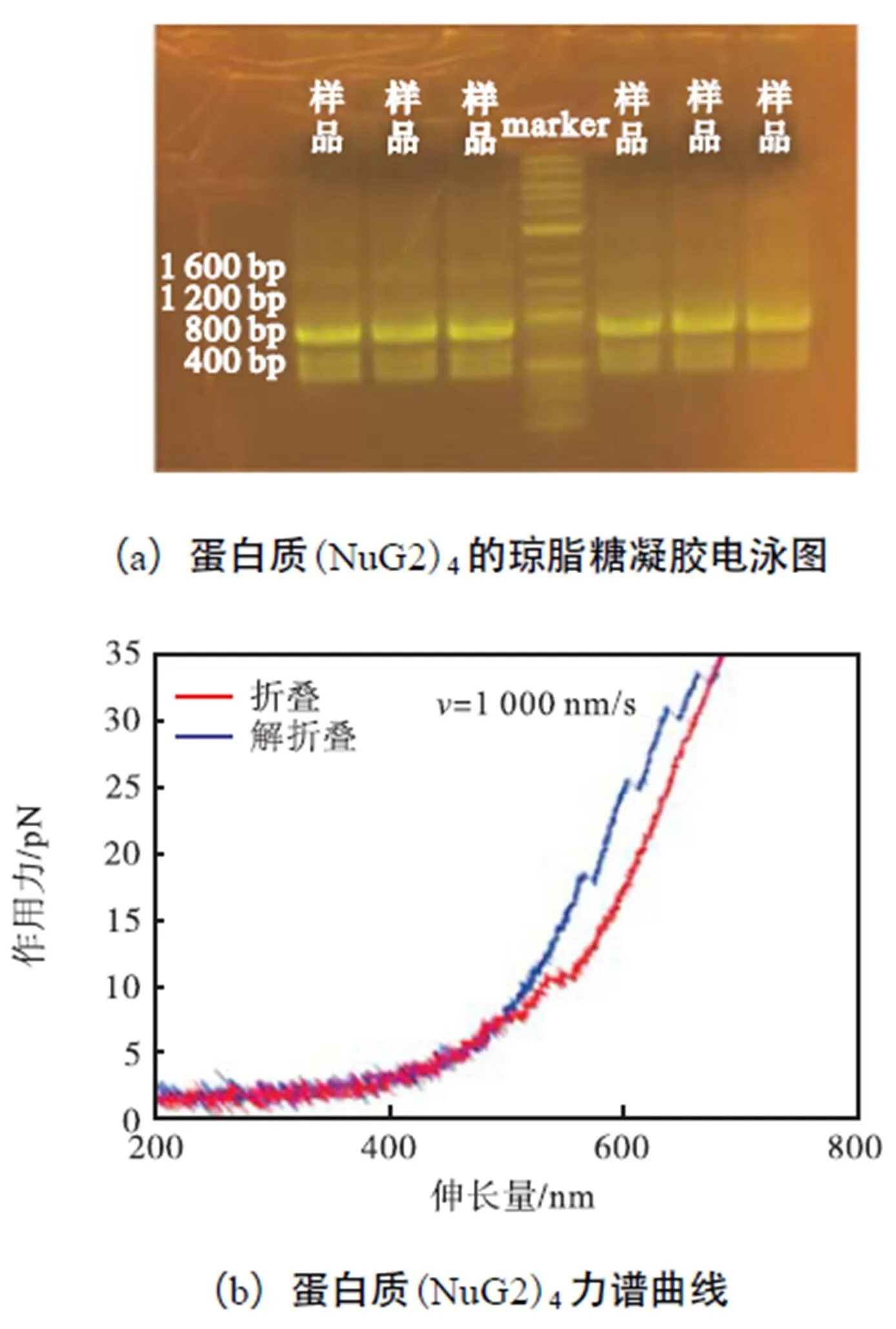
图6 二硫键连接法实验数据
在光镊系统中,使用Avi标签法的多为微球-待测蛋白-双链DNA-微球结构,通常可以通过两种方法来形成这种结构.方法一为将经过基因修饰表面带有AviTag标签并经过表达后表面连有生物素的待测蛋白直接与一端修饰有巯基另一端修饰有地高辛的双链DNA反应,通过待测蛋白与双链DNA形成的二硫键将待测蛋白与DNA链连接在一起、通过微球表面的链霉亲和素及地高辛抗体分别与待测蛋白表面的生物素及双链DNA一端的地高辛反应最终实现微球-待测蛋白-双链DNA-微球结构[25-27];方法二为将一端修饰有巯基的DNA寡核苷酸链经过DTDP活化后与经过基因工程修饰后表面带有生物素和巯基的待测蛋白反应形成待测蛋白-寡核苷酸链的结构,再将其与一端带有寡核苷酸链另一端修饰有双地高辛的双链DNA进行杂化反应,最终形成带有生物素的待测蛋白-带有双地高辛的双链DNA结构,通过生物素-链霉亲和素、地高辛-地高辛抗体反应将待测蛋白连在双光阱中的两个微球表面[28-30].
通过一条DNA链将待测蛋白连接在微球表面即在待测蛋白的一端修饰生物素另一端修饰半胱氨酸,借助半胱氨酸上的巯基与DNA链一端修饰的巯基所形成的二硫键将待测蛋白与DNA链连接起来,而后通过待测蛋白一端修饰的生物素与微球表面的链霉亲和素的特异性反应、DNA链另一端修饰的地高辛与微球表面的地高辛抗体的抗原抗体反应将待测蛋白连接在双光镊系统中,用于研究从不同方向拉伸卷曲蛋白GCN4的动力学特性[31].
综观光镊系统中常用的生物分子链耦联技术,使用两条DNA链将待测蛋白连接在系统中的方法虽然操作简单,但是连接效率非常低;使用一条DNA链相比于使用两条DNA链将待测蛋白连接在光镊系统中,连接效率会有所提高,但缺点是降低了信噪比,同时激光照射样品池所产生的热效应会使待测蛋白发生变性的可能性增大.
3 讨 论
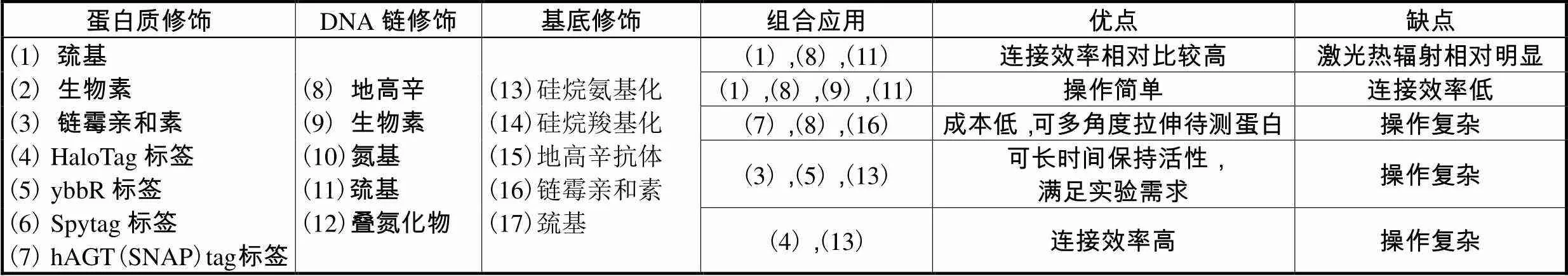
随着研究者们不断地深入研究,在用光镊系统研究蛋白质的折叠/解折叠问题时,系统逐渐由单光镊发展为双光镊,结合测试系统中样品的连接构型不同即单光镊是通过捕获一个微球借助DNA链的作用实现微球与待测蛋白一端的连接,待测蛋白的另一端通过DNA链连接在玻片表面;而双光镊系统是通过捕获两个微球,借助两条DNA链的作用实现待测蛋白与微球的连接.但由于使用两条DNA链连接待测蛋白的效率低,有研究者开始尝试仅用一条DNA链连接待测蛋白,而待测蛋白的另一端直接与其中的一个微球连接,即借助DNA链的修饰和对待测蛋白进行标签修饰等多种修饰方法相结合的方式实现生物样品的耦联,连接效率有所提高.应用在光镊系统中的生物样品耦联方法同样适用于磁镊系统中.在原子力显微术系统中,结合不同待测蛋白的结构特点也不断地探索出新的玻片/云母片基底修饰及待测蛋白标签修饰方法,如链霉亲和素修饰法,通过在待测蛋白表面修饰链霉亲和素,在玻片基底修饰生物素,借助链霉亲和素和生物素间的相互作用实现待测蛋白与玻片的连接,有效地改善了生物样品与测试系统连接效率低的问题.本文介绍了应用于单分子力谱技术中常见的3种修饰方法并分别列举、总结了应用于原子力显微术、磁镊及光镊三大单分子力谱测试系统中的生物分子链耦联的方法及各自的优缺点.对于不同的测试系统,为了提高生物样品的连接效率,多采取多种修饰方法相结合的方式,如表1所示.
表1 修饰方法对比
Tab.1 Comparison of the modification methods
4 结 语
在用光镊、磁镊及原子力显微术三大技术研究蛋白质折叠/解折叠实验中,待测蛋白与DNA链/探针/玻片/云母片/微球的连接牢固度直接影响着实验的成功率,因此探究一种高效的生物分子链耦联技术是每一位相关领域研究者的目标.本文提供了一个现有研究方法的基础总结,希望在今后的科研中,可以探索更多更丰富更高效的生物样品耦联方法.
致 谢
在此特别感谢天津大学药物科学与技术学院尉迟之光老师在实验及论文撰写过程中提供的帮助.
[1] Jagannathan B,Marqusee S. Protein folding and unfolding under force[J]. Biopolymers,2013,99(11):860-869.
[2] Lei Hai,He Chengzhi,Hu Chunguang,et al. Single-molecule force spectroscopy trajectories of a single protein and its polyproteins are equivalent:A direct experimental validation based on a small protein NuG2[J]. Angewandte Chemie International Edition,2017,56(22):6117-6121.
[3] Ma Lu,Cai Yiying,Li Yanghui,et al. Single-molecule force spectroscopy of protein-membrane interactions [J].ELife,2017,6:e30493.
[4] Chen Hu,Fu Hongxia,Zhu Xiaoying,et al. Improved high-force magnetic tweezers for stretching and refolding of proteins and short DNA[J]. Biophysical Journal,2011,100(2):517-523.
[5] Long Xi,Parks Joseph W,Bagshaw Clive R,et al. Mechanical unfolding of human telomere G-quadruplex DNA probed by integrated fluorescence and magnetic tweezers spectroscopy[J]. Nucleic Acids Research,2013,41(4):2746-2755.
[6] Lee Whasil,Zeng Xiancheng,Zhou Huanxiang,et al. Full reconstruction of a vectorial protein folding pathway by atomic force microscopy and molecular dynamics simulations[J]. Journal of Biological Chemistry,2010,285(49):38167-38172.
[7] Li Hongbin,Oberhauser Andres F,Fowler Susan B,et al. Atomic force microscopy reveals the mechanical design of a modular protein[J]. Proceedings of the National Academy of Sciences,2000,97(12):6527-6531.
[8] Marszalek Piotr E,Dufrêne Yves F. Stretching single polysaccharides and proteins using atomic force microscopy[J]. Chemical Society Reviews,2012,41(9):3523-3534.
[9] Los Georgyi V,Encell Lance P,McDougall Mark G,et al. HaloTag:A novel protein labeling technology for cell imaging and protein analysis[J]. ACS Chemical Biology,2008,3(6):373-382.
[10] Taniguchi Yukinori,Kawakami Masaru. Application of HaloTag protein to covalent immobilization of recombinant proteins for single molecule force spectroscopy[J]. Langmuir,2010,26(13):10433-10436.
[11] Yin Jun,Straight Paul D,McLoughlin Shaun M,et al. Genetically encoded short peptide tag for versatile protein labeling by Sfp phosphopantetheinyl transferase [J]. Proceedings of the National Academy of Sciences of the United States of America,2005,102(44):15815-15820.
[12] Zakeri Bijan,Fierer Jacob O,Celik Emrah,et al. Peptide tag forming a rapid covalent bond to a protein,through engineering a bacterial adhesin[J]. Proceedings of the National Academy of Sciences,2012,109(12):4347-4348.
[13] Pippig Diana A,Baumann Fabian,Strackharn Mathias,et al. Protein-DNA chimeras for nano assembly[J]. ACS Nano,2014,8(7):6551-6555.
[14] Bauer Magnus S,Milles Lukas F,Sedlak Steffen M,et al. Monomeric streptavidin:A versatile regenerative handle for force spectroscopy [EB/OL]. https:// www. biorxiv.org/content/early/2018/03/08/276444.fill. pd,2018.
[15] Chen Hu,Chandrasekar Saranya,Sheetz Michael P,et al. Mechanical perturbation of filamin A immu-noglobulin repeats 20-21 reveals potential non-equilibrium mechanochemical partner binding function [J]. Scientific Reports,2013,3:1642.
[16] Chen Hu,Yuan Guohua,Winardhi Ricksen S,et al. Dynamics of equilibrium folding and unfolding transitions of titin immunoglobulin domain under constant forces[J]. Journal of the American Chemical Society,2015,137(10):3540-3546.
[17] Chen Hu,Zhu Xiaoying,Cong Peiwen,et al. Differential mechanical stability of filamin A rod segments[J]. Biophysical Journal,2011,101(5):1231-1237.
[18] Min Duyoung,Jefferson Robert E,Bowie James U,et al. Mapping the energy landscape for second-stage folding of a single membrane protein[J]. Nature Chemical Biology,2015,11(12):981.
[19] Min Duyoung,Kim Kipom,Hyeon Changbong,et al. Mechanical unzipping and rezipping of a single SNARE complex reveals hysteresis as a force-generating mechanism[J]. Nature Communications,2013,4:1705.
[20] Min Duyoung,Arbing Mark A,Jefferson Robert E,et al. A simple DNA handle attachment method for single molecule mechanical manipulation experiments[J]. Protein Science,2016,25(8):1535-1544.
[21] Cecconi Ciro,Shank Elizabeth A,Dahlquist Frederick W,et al. Protein-DNA chimeras for single molecule mechanical folding studies with the optical tweezers[J]. European Biophysics Journal,2008,37(6):729-738.
[22] Gebhardt J C M,Bornschlögl T,Rief M,et al. Full distance-resolved folding energy landscape of one single protein molecule[J]. Proceedings of the National Academy of Sciences,2010,107(5):2013-2018.
[23] Bauer Daniela,Merz Dale R,Pelz Benjamin,et al. Nucleotides regulate the mechanical hierarchy between subdomains of the nucleotide binding domain of the Hsp70 chaperone DnaK[J]. Proceedings of the National Academy of Sciences,2015,112(33):10389-10394.
[24] Jahn Markus,Buchner Johannes,Hugel Thorsten,et al. Folding and assembly of the large molecular machine Hsp90 studied in single-molecule experiments [J].Proceedings of the National Academy of Sciences,2016,113(5):1232-1237.
[25] Gao Ying,Zorman Sylvain,Gundersen Gregory,et al. Single reconstituted neuronal SNARE complexes zipper in three distinct stages[J].Science,2012,337(6100),1340-1343.
[26] Jiao Junyi,Rebane Aleksander A,Ma Lu,et al. Kinetically coupled folding of a single HIV-1 glycoprotein 41 complex in viral membrane fusion and inhibition[J].Proceedings of the National Academy of Sciences,2015,112(22):E2855-E2864.
[27] Zhang Yongli,Xi Zhiqun,Gao Ying,et al. Direct observation of helix staggering,sliding,and coiled coil misfolding[J]. Biophysical Journal,2012,102(3):175a.
[28] Stigler Johannes,Ziegler Fabian,Gieseke Anja,et al. The complex folding network of single calmodulin molecules[J].Science,2011,334(6055):512-516.
[29] Woodside Michael T,Anthony Peter C,Behnke-Parks William M,et al. Direct measurement of the full,sequence-dependent folding landscape of a nucleic acid[J]. Science,2006,314(5801):1001-1004.
[30] Žoldák Gabriel,Stigler Johannes,Pelz Benjamin,et al. Ultrafast folding kinetics and cooperativity of villin headpiece in single-molecule force spectroscopy[J]. Proceedings of the National Academy of Sciences,2013,110(45):18156-18161.
[31] Gao Ying,Sirinakis George,Zhang Yongli. Highly anisotropic stability and folding kinetics of a single coiled coil protein under mechanical tension[J]. Journal of the American Chemical Society,2011,133(32):12749-12757.
(责任编辑:田 军)
Coupling Technologies of Biological Molecular Chains in Protein Force Spectroscopy Testing
Hu Xiaotang1, 2,Li Shuai1, 2,Hu Chunguang1, 2,He Chengzhi1, 2, 3,Gao Xiaoqing1, 2,Han Mengke1, 2,Ma Guoteng1, 2,Li Hongbin1, 2, 4,Hu Xiaodong1, 2
(1. State Key Laboratory of Precision Measuring Technology and Instruments(Tianjin University),Tianjin 300072,China;2. Nanchang Institute for Microtechnology of Tianjin University,Tianjin 300072,China;3. Beijing Advanced Innovation Center for Soft Matter Science and Engineering(Beijing University of Chemical Technology),Beijing 100029,China; 4. Department of Chemistry, University of British Columbia,Vancouver V6T 1Z4,Canada)
Protein force spectroscopy testing is one of the important components of the study of biology. The success of protein force spectroscopy testing depends on the following factors:the capability of force spectroscopy measure-ment at the molecular scale,namely piconewton-scale force resolution and nanometer-scale spatial resolution,and the protein sample preparation technique,which achieves effective measurement of protein samples at the molecular scale through the coupling of microscopic biomolecular chains with mesoscopic microspheres/probes/substrates. However,the coupling of protein samples is invisible and its coupling effect seriously affects the test efficiency. Therefore,the preparation method and coupling technology of protein samples are the key points in the measurement of single-molecule force spectroscopy. According to the various demands of three single-molecule force spectroscopy testing techniques on the samples,particularly the test requirement of multi-molecular chain-coupled samples,the authors introduce different methods to improve the connection efficiency between protein and DNA handles/micro-spheres/probes/glass slides/mica in optical tweezers,magnetic tweezers,and atomic force microscopy based on substrate,protein,and DNA modification,and discuss their advantages and disadvantages. The typical applications are summarized to provide a reference for the preparation of biological samples in related fields.
protein force spectroscopy;optical tweezer;magnetic tweezer;atomic force microscopy;coupling
the National Natural Science Foundation of China(No. 61223008),the Key Program of the Natural Science Foundation of Tianjin,China (No. 15JCZDJC31600).
10.11784/tdxbz201807038
Q71
A
0493-2137(2019)02-0113-09
2018-07-27;
2018-09-07.
胡小唐(1952— ),男,教授,xthu@tju.edu.cn.
胡春光,cghu@tju.edu.cn.
国家自然科学基金资助项目(61223008);天津市自然科学基金重点资助项目(15JCZDJC31600).
