高光谱技术融合平板菌落法同步计数酸奶中益生菌
2019-01-07石吉勇吴胜斌邹小波李文亭
石吉勇,吴胜斌,邹小波*,张 芳,赵 号,李文亭
(江苏大学食品与生物工程学院,江苏 镇江 212013)
益生菌是指宿主摄入一定量后对宿主产生保健作用的一类活性有益微生物[1-2]。研究表明,酸奶中的益生菌进入肠道后,具有改善肠道正常菌群、抗癌抗肿瘤、增强人体自身免疫力等重要功效[3-5]。目前市面上常见的功能性酸奶中含有的菌种主要有保加利亚乳杆菌、嗜热链球菌、嗜酸乳杆菌、干酪乳杆菌和植物乳杆菌等。其中,干酪乳杆菌、嗜酸乳杆菌、植物乳杆菌为常见的益生菌种,而保加利亚乳杆菌和嗜热链球菌在酸奶中主要起发酵作用[6-7],属于发酵菌种,难以通过胃部进入肠道正常的生长繁殖,不能发挥其保健功能。因此开发一种酸奶中益生菌的简单、快速、高效的鉴别计数方法对于维护消费者权益和保障食品安全具有重要意义。
传统鉴别计数食品中微生物的方法主要有形态学鉴别计数法[8-9]、分子生物学定量法[10-12]、机器视觉法[13-16]等。形态学鉴别计数法主要是通过染色、镜检等方法获取特定微生物的培养形态及特征进行检测,具有直观、准确等优点,但该方法费时费力、操作繁琐。分子生物学定量法是从微生物的基因水平上进行鉴别,其优点是精确、高效,但该方法局限性较大,如操作复杂、费时费力、试剂昂贵等。机器视觉技术是近些年来兴起的一种基于视觉算法的新兴计数,该方法主要是通过微生物纹理、颜色等特征信息,从形态学角度进行一系列的处理以达到分割计数的效果,具有简单、快速、高效等优点,但仍然存在较大的缺陷,如对于光照、相机、算法要求较高等。
高光成像技术结合传统的光谱分析技术与图像分析技术于一体,能够同时捕获样本的光谱信息与空间位置信息,是近些年来在食品品质安全监测应用中新兴的一门快速无损检测技术[17]。菌落在生长繁殖过程中,外部结构(形状、纹理)和内部结构(结构、成分)均会发生一定的改变[18]。高光谱对菌落内部含氢基团较为敏感[19],由于不同菌种具有不同内部组分含量,因此可通过提取图像中像素点的光谱信息以达到鉴别及计数的目的,然而利用高光谱技术实现对益生菌酸奶中益生菌的鉴别计数研究鲜有报道。因此本实验采用高光谱技术对益生菌酸奶中各类益生菌进行鉴别及计数,解决混合发酵益生菌酸奶中多种益生菌同时存在的情况下对每种益生菌数量计数的难题,为快速、无损判断各类益生菌酸奶益生功能活性提供依据。
1 材料与方法
1.1 菌种与培养基
菌种:实验所用菌株保加利亚乳杆菌(Lactobacillus bulgaricus)、嗜热链球菌(Streptococcus thermophiles)、嗜酸乳杆菌(Lactobacillus acidophilus)、干酪乳杆菌(Lactobacillu caseii)、植物乳杆菌(Lactobacillus plantarum)均购自中国工业微生物菌种保藏管理中心。
培养基:实验所用MRS培养基参照GB 4789.35—2016《食品微生物检验 乳酸菌检验》附录A规定制备,MC培养基配方参照GB 4789.35—2016,LC培养基的配制参考文献[20]方法,麦芽糖-MRS培养基和山梨醇-MRS培养基的制备参照文献[21],MRS和AC培养基的配制按照文献[22]方法。
1.2 仪器与设备
高光谱图像采集系统由江苏大学组装搭建[23],主要零部件包括高光谱摄像机(ImSpector,V10E,芬兰)、150 W的光纤卤素灯(Fiber-Lite DC950Illuminator,DolanJenner Industries Inc,MA,美国)、精密电控移动平台(Zolix,SC30021A,北京)、软件系统为SpectralCube软件(芬兰Spectral Imaging有限公司提供)、光谱提取软件为ENVI 4.5(V.4.5,Research System Inc boulder CO. USA)。
LD-2A离心机 北京京力离心机有限公司;HI 2214 ORP Meter 2011 pH计 哈纳仪器有限公司;GHP-9050隔水式培养箱 上海合恒仪器设备有限公司;HYL-C小型组合式摇床 太仓市强乐实验设备有限公司;SW-CJ-1D单人净化工作台 浙江苏净净化设备有限公司;BCD-216SDN冰箱 海尔集团;AB123电子天平上海豪晟科学仪器有限公司;DSX-280B手提式压力蒸汽灭菌器 上海珂淮仪器有限公司;XTL-165-VT数码光学显微镜 凤凰光学集团有限公司。
1.3 方法
1.3.1 菌落高光谱数据的采集及标定
1.3.1.1 菌种的培养
将需要灭菌处理的相关材料进行高压灭菌处理(121 ℃,20 min),并将其转移至超净工作台中。然后将经过扩增培养的各菌液用0.85%生理盐水进行梯度稀释,选择2 种合适的稀释梯度(生长出的菌落数量在30~300 CFU之间)进行涂布,每个涂布浓度平行4 次,得到5 种菌总共40 个平板,5 个批次培养后得到5 种菌总共200 个培养皿。此类培养皿中生长出的菌落供后期提取菌落光谱信息使用,传统方法计数的培养按照参考文献[20-22]方法进行培养和计数。
1.3.1.2 高光谱的采集与标定
高光谱摄像头的分辨率为775×1 628,高光谱的波长范围为432~963 nm,最终得到一个大小为775×1 628×618的高光谱数据块。在进行光谱采集时可能会出现由于打光不均匀导致光照强度在各个波段的不同以及传感器中暗电流的存在,所获取的高光谱图像在光照强度较弱的波段出现较大的噪声,而且会在不同波长下光谱图像出现亮度差异较大等情况[24]。因此在图像采集完成后需要对光谱图像进行黑白板校正[25],校正公式如下式所示:
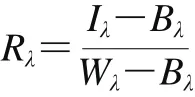
式中:Iλ为样品原始高光谱图像中λ波段对应的反射强度;Bλ为高光谱黑板校正图像中λ波段对应的反射强度;Wλ为高光谱白板校正图像中λ波段对应的反射强度;Rλ为样品校正后高光谱图像中λ波段对应的反射强度。
1.3.2 高光谱信息的提取与数据处理
1.3.2.1 高光谱信息的提取
图像采集并标定完后,采用ENVI软件进行特征光谱信息的提取[26]。所选对象菌落生长较好、大而厚实,定义一个圆形感兴趣区域,提取此感兴趣区域在不同波长下的光谱信息。采集培养皿中5 个单独菌落的光谱数据并取平均值得到一条原始光谱,以此方式得到200 个培养皿对应的200 条光谱数据。
1.3.2.2 高光谱信息的预处理
菌落的感兴趣区域除包含一些可进行样本表征的有利信息外,通常受环境、机器运行状况等影响,会携带一些样本信息以外的光谱信息,如基线漂移、高频噪声、背景信息等,都会严重干扰实验结果,因此有必要对所获的光谱区域进行光谱预处理,除去光谱中无用光谱信息、降低噪声干扰、减少基线漂移提高建立模型的准确性和稳定性[27]。本研究采用标准正态变量变换(standard normal variate transformation,SNV)、一阶导数、二阶导数、多元散射校正(multiplicative scatter correction,MSC)4 种方法进行光谱预处理[28],通过比较4 种方法处理后的效果,选出最优方法作为本实验的预处理方法。
1.3.2.3 主成分分析(principal component analysis,PCA)
高光谱信息中除了含有大量的噪声以外,还存在着大量的无关变量或重叠信息。为降低众多信息中共存、相互重叠的信息,本研究采用PCA法对光谱数据进行降维处理。
1.3.3 菌落计数模型的建立
本实验通过建立K-最近邻法(K-nearest neighbors,KNN)、误差反向传播神经网络(back propagationartificial neural network,BP-ANN)、最小二乘支持向量机(least squares support vector machines,LS-SVM)3 种模型,对比3 种模型识别率判断最佳的计数模型。由于主成分数对KNN模型的识别结果具有较大影响,本实验选取前10 个主成分和10 个K值对模型进行优化。BPANN模型的输出层单元设为5 个(菌落种类),传递函数为双曲线正切函数,初始权重为0.95,学习因子和动量因子均为0.1,收敛误差设置为0.000 2,训练迭代次数为1 500 次。采用交叉验证和网格搜索法对LS-SVM模型的最佳参数进行选择,以校正集交叉验证均方根误差最小为指标确定最优的径向基核正规划参数γ和基于径向基核函数的参数σ2,经过优化得到,最佳主成分为4时,最佳参数γ为2.358 5,σ2为1.302 1。将经过预处理后的200 条光谱数据集按照K-S(Kennard-Stone)法分成125 个校正集和75 个预测集分别建立KNN模型、BP-ANN模型和LS-SVM模型,校正集数据用来建立模型,预测集用来检测模型的准确性,通过比较3 种模型的识别率和最佳主成分数优选出最佳计数模型。模型的识别结果将5 种菌落分别分为1、2、3、4、5 类,每条光谱对应着一个模式识别编号。编号1为保加利亚乳杆菌,编号2为干酪乳杆菌,编号3为嗜热链球菌,编号4为嗜酸乳杆菌,编号5为植物乳杆菌。统计编号1、2、3、4、5的数量,即对应着不同菌落的数量,具体流程详见图1。
1.3.4 益生菌酸奶中各种菌落数量及总数的确定
1.3.4.1 益生菌酸奶的制备
益生菌酸奶的制备参照文献[8]方法进行。
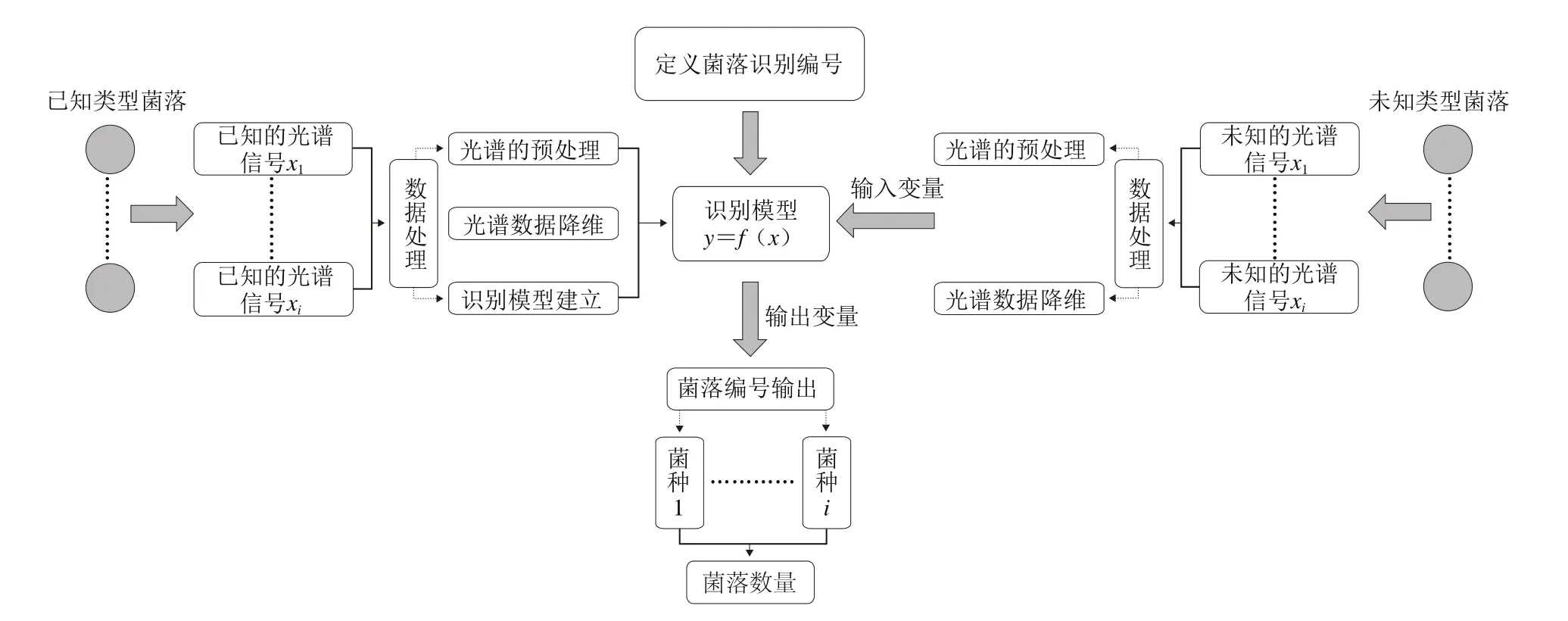
图1 高光谱菌落计数流程Fig. 1 Flow chart showing the process of colony counting using hyperspectral imaging
1.3.4.2 基于高光谱技术的模式识别法同时计数各种菌落及总数
取出部分自制酸奶样品,用0.85%生理盐水稀释至10-8,经过大量预实验表明10-6、10-7两个浓度梯度生长的菌落数量在30~300 CFU之间,因此选择10-6、10-7两个浓度梯度作为涂布浓度。吸取该稀释浓度的菌液0.1 mL均匀涂布在MRS培养基上,置于(36±1)℃恒温培养箱内培养48 h后取出。提取混合菌落中每一株菌落的光谱信息,按照1.3.1、1.3.2节方法进行光谱的提取和预处理操作,最后将处理后的光谱数据集代入1.3.3节中已建立好的光谱模型中,根据菌落的识别结果统计每一种菌的数量。识别结果中5 种编号的总和为酸奶中菌落总和,编号2、4、5的和为自制酸奶中益生菌的总和。
1.3.4.3 传统培养计数方法计数
采用上述相同的浓度梯度进行不同菌落的传统方法计数。嗜热链球菌的计数按照GB 4789.35—2016中规定的MC培养基计数法计数,将稀释后的菌液加入到无菌培养皿中,每个培养皿中加入15 mL MC培养基,(36±1)℃培养(72±3)h,将菌落中等偏小、边缘整齐平滑的红色菌落计数为嗜热链球菌;干酪乳杆菌的计数按照文献[20]规定的选择性培养基-LC培养基计数,具体为取稀释菌液于LC培养基中涂布,然后在(25±1)℃培养(72±3)h,计数培养基上生长出来的菌落记为干酪乳杆菌数量;嗜酸乳杆菌和植物乳杆菌的数量按照文献[21]所描述的方法进行计数,采用山梨醇-MRS培养基计数植物乳杆菌的数量,麦芽糖-MRS培养基计数植物乳杆菌和嗜酸乳杆菌的数量,然后两者的差值即为嗜酸乳杆菌的数量;按照文献[22]中的酸化MRS培养基在厌氧的培养环境下计数保加利亚乳杆菌的数量。
实验过程中2 种方法在涂布时保证为同一批稀释度样品,且涂布量相同,每份样品平行10 次,记录各菌落的数量,将2种方法进行显著性分析。
2 结果与分析
2.1 光谱预处理及PCA结果
2.1.1 光谱预处理结果
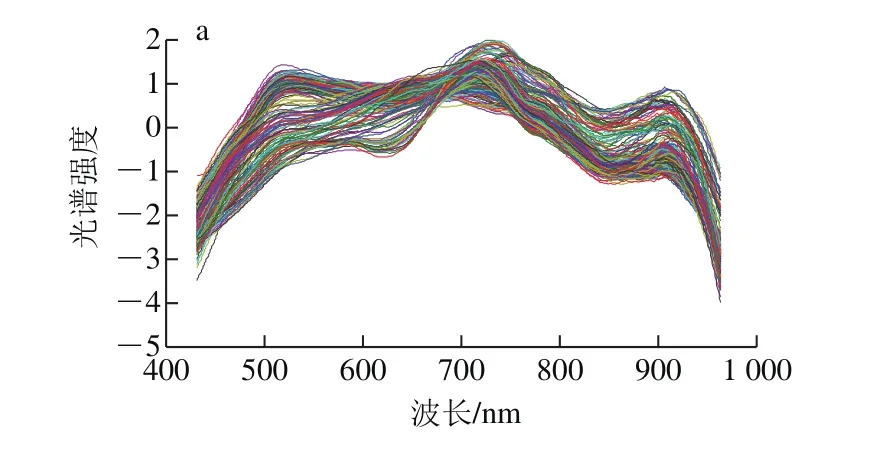
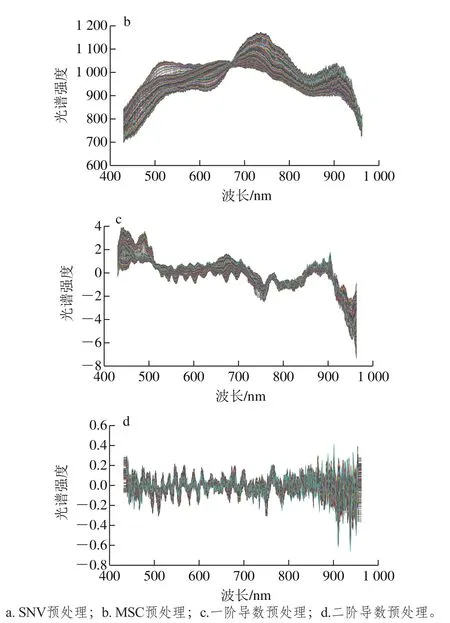
图2 不同预处理方法后的菌落样本的光谱曲线Fig. 2 Hyperspectral curves of colony samples with different pretreatments
采用SNV、MSC、一阶导数、二阶导数4 种不同的预处理方法对618 个波长下的全光谱数据集进行预处理。预处理后的各菌落光谱曲线如图2所示。根据4 种预处理得到的光谱曲线去噪效果来看,SNV预处理后的光谱曲线明显较其他几种预处理的光谱曲线平滑,其原因可能在于SNV预处理在消除样本颗粒大小、散射或光程引起的样本间光谱误差效果较好。虽然MSC在处理校正散射方面较强,但在其他噪声方面不足,求导处理后的光谱曲线更注重的是确定吸收峰和肩峰的位置,提高光谱的灵敏度和光谱细节,而在降噪方面略显不足[29-30]。但是合适预处理方法的判定仍需要综合模型的识别率进行选择。
由表1可以看出,SNV预处理后的光谱模型识别效果优于其他预处理后的模型,可以有效对光谱中的噪声和其他干扰进行校正。因此本研究在后续的实验中均采用SNV预处理方法。
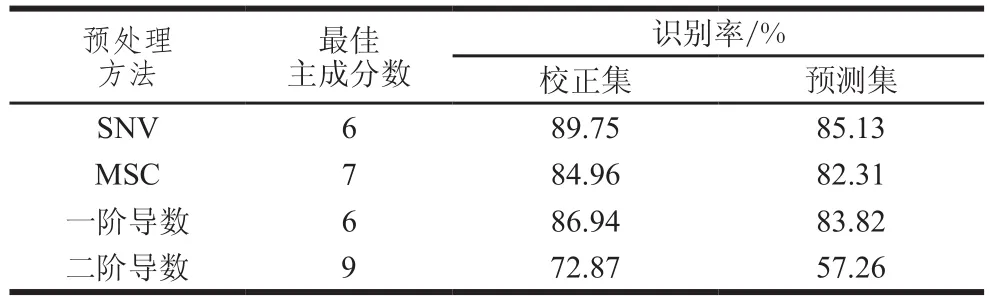
表1 不同预处理方法对模型效果的结果分析Table 1 Comparison of different spectral preprocessing methods
2.1.2 PCA结果
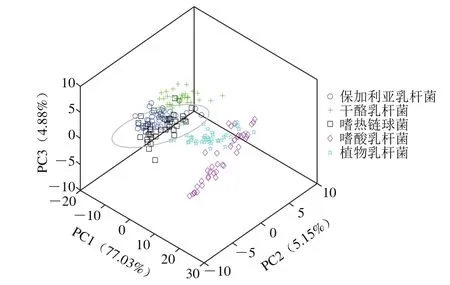
图3 三维主成分空间分布图Fig. 3 Three-dimensional projection from principal component analysis
由图3可知,第1主成分代表了77.03%的光谱信息,第2主成分代表了5.15%的光谱信息,第3主成分代表了4.88%的光谱信息,前3 主成分的累计贡献率达到85%以上,能够代表光谱中的大多数信息。根据各菌落在三维主成分中的分布来看,几种菌落均具备明显的聚类特征,暗示了菌落与菌落之间具备一定的类别特征。其中干酪乳杆菌、嗜酸乳杆菌和植物乳杆菌3 种菌落的区别特征较为明显,能很好地与其他菌落进行区分。保加利亚乳杆菌和嗜热链球菌2 种菌落重叠较为严重,其原因可能是这2 种菌落均为发酵菌种,且两者之间存在共生作用,具有相似的代谢物,因此需要通过进一步建立模型进行鉴别区分。
2.2 最优计数模型选择结果
将200 条光谱数据集按照K-S方法随机分成125 条校正集光谱和75 条预测集光谱,分别建立KNN、BPANN、LS-SVM模型,模型的识别结果如表2所示。当主成分为9时,LS-SVM模型的识别率高于另外2 种模型,模型的综合识别率中校正集识别率为99.20%,预测集识别率为93.33%,为最优计数模型。KNN模型识别率相对于LS-SVM识别率低的原因可能在于内部算法方面不足。KNN模型采用的是“表决”方法,按照同类样本相互靠近和少数服从多数的原则进行表决,在解决不同种属的样本分类问题上效果较好,而在解决同种属、不同类问题上效果较差[31]。在需要进行分类鉴定的几种菌落中,保加利亚乳杆菌、嗜酸乳杆菌、干酪乳杆菌、植物乳杆菌属于相同的属(乳杆菌属)不同的种,因而区分率较低。尽管BP-ANN校正集的识别率达到了100%,但是在进行预测集验证的时候识别率仍然很低,其原因可能是BP-ANN算法是一种按照误差逆传播算法训练的多层前馈网络,当输出不等于期望输出时则进入反向传播过程,直到网络的全局误差小于给定的值后学习终止,实际上预测能力并没有达到相应的水平,因此导致较低的识别率。LS-SVM是在经典SVM算法上进行改进,代替经典SVM中复杂的二次优化问题获得支持向量,降低了计算的复杂性,加快求解速度,并且能够在少量的训练样本中进行高维特征空间学习。从最佳主成分数量上来看,KNN模型具有最小的主成分数7,所建模型相对简单,LS-SVM所建模型具有最大的主成分数,相对KNN和BPANN模型较复杂,权衡考虑计数模型的准确性,故本研究选取了识别率最高的LS-SVM模型作为最佳计数模型。
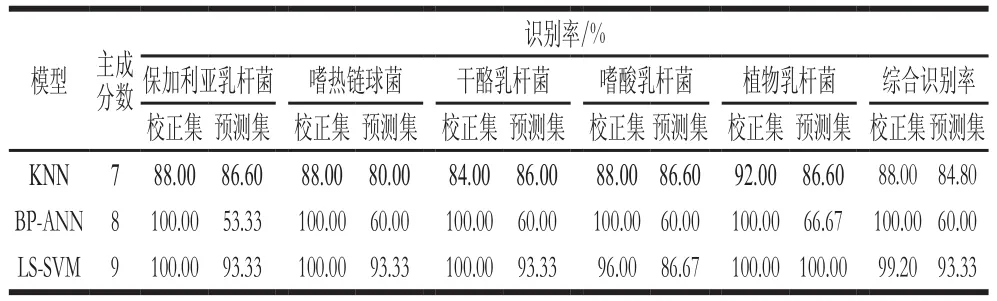
表2 不同模型的识别结果比较Table 2 Comparison of recognition results with different models
2.3 高光谱菌落计数法与传统培养基计数法的比较

表3 2 种计数方法结果的比较Table 3 Comparison of two counting methods
由表3可知,2 种不同的计数方法结果较为接近,其中相同的菌落采用不同的计数方法得到的结果均未达到显著性差异(P>0.05),但高光谱计数法未能与传统方法达到一致,其原因可能为:1)传统的微生物计数法本身就是一个大约近似的方法,选择性培养基并不是完全的具备选择性;2)在实验过程中存在着样品均匀性、偶然性问题;3)高光谱数据在进行处理的过程中仍然存在较大的噪声,导致在建立模型时,由于模型的精度不够而导致的模型错误识别等。但就总体而言,2 种方法在益生菌总数的计数中没有表现出较大的偏差,可以表明2 种方法在理论计数上无明显差异。因此可以表明,高光谱计数法可以对酸奶中活性益生菌快速、无损、准确地进行计数,避免了传统计数法对酸奶中活性益生菌技术需要不同培养基的繁琐过程,满足了实际生产和检测工作中的需要。
3 结 论
本实验运用高光谱技术实现对混合发酵酸奶中的发酵菌种(保加利亚乳杆菌、嗜热链球菌)和益生菌种(干酪乳杆菌、嗜酸乳杆菌和植物乳杆菌)的鉴别与计数。首先采集经过48 h培养的5 种菌种的高光谱信息,将得到的光谱信息分别采用不同的预处理方式(SNV、MSC、一阶导数、二阶导数)进行预处理降噪,采用PCA法对预处理后的光谱数据集进行降维,采用不同的模式识别方法建立5 种菌落的快速计数模型,根据几种模型的识别率确定最佳的计数模型。最后培养自制酸奶样品,对比高光谱菌落计数与传统培养计数法,判断高光谱菌落计数的可行性。结果表明,采用SNV预处理后的光谱在提高模型精度效果上最佳。当主成分数为9时,LS-SVM模型所对应的校正集识别率为99.20%,预测集识别率为93.33%,模型的识别率和稳定性为最佳计数模型。对比最佳模型菌落计数和传统计数法对自制酸奶中各种益生菌的计数结果,二者并无显著性差异(P>0.05),验证了高光谱技术对混合发酵酸奶中多种菌种同时存在情况下对每种益生菌同时计数方法的可行性。
