基于癌症基因组图谱构建胃癌预后评估模型
2018-12-27王举窦忠霞姜洪伟王永强高小平张勇
王举,窦忠霞,姜洪伟,王永强,高小平,张勇
胃癌是严重威胁人类健康的常见消化道恶性肿瘤之一,发病率居恶性肿瘤第四位,仅次于肺癌、乳腺癌和结直肠癌,死亡率居第三位。由于发病隐匿,早期症状不明显,就诊时大多属晚期,5年总生存率(overall survival,OS)仅为 28.3%[1]。然而影响胃癌发生、发展及预后的因素尚不明确,因此,从基因分子水平挖掘与胃癌预后相关的基因,对胃癌治疗及预后评估具有重要意义。由美国国家癌症研究所(National Cancer Institute,NCI)和国家人类基因组研究所(National Human Genome Research Institute,NHGRI)于2006年启动的癌症基因组谱图(The Caner Genome Atlas,TCGA)计划,试图通过应用基因组分析技术,特别是采用大规模的基因组测序,将人类全部癌症的基因组变异图谱绘制出来,筛选癌基因和抑癌基因,进行系统分析,了解肿瘤发生发展的机制,发展新的诊断和治疗方法。TCGA数据库包含了丰富的肿瘤和数据类型,截止2018年9月27日,TCGA共收录人类69个部位、43种类型的肿瘤,病例总数达33 096例。数据类型包括基因测序(gene sequencing)、DNA 拷贝数分析(DNA copy number analysis)、转 录 谱 分 析(transcriptome analysis)、甲基化分析(methylation analysis)、小分子非编码RNA分析(miRNA analysis)和临床信息。其中,临床资料涵盖患者性别、年龄、种族、族群、肿瘤分化程度、体积、TNM分期和随访时间等。基于大数据库的生物信息学分析,为肿瘤研究指明了方向,通过差异分析、功能及通路富集分析、生存分析等,可以筛选影响肿瘤发生发展的核心基因和信号通路。本研究首先从TCGA网站下载胃癌及临床相关数据,筛选胃癌及癌旁组织的差异表达基因(differential expressed genes,DEGs),再进行Cox比例风险回归模型分析,构建胃癌预后评估模型。
1 资料与方法
1.1 数据下载 登录TCGA网站(https://portal.gdc.cancer.gov/repository),病例选择胃癌(Stomach),文件-数据分类选择转录谱(Transcriptome profiling),数据类型选择基因表达定量(Gene Expression Quantification),工作流程类型(workflow type)选择原始数据HTSeq-Counts,共407个文件,每个文件对应1个样本,样本包括胃癌及癌旁正常组织,将选择的文件加入Cart,以命令行形式下载。同法下载胃癌临床样本文件。
1.2 数据处理 采用perl脚本将下载的407个文件合并、转化成一个矩阵(Matrix),行名为基因名/EnsembleID/基因功能注释,列名为样本名,其中功能注释包括蛋白编码基因(Protein coding gene)、长链非编码RNA(lincRNA)、假基因(pseudogene)等。本研究目的是构建mRNA胃癌预后评估模型,因此根据基因功能注释只从矩阵提取蛋白编码基因进行后续分析。同样,胃癌临床文件合并成一个行名为样本名,列名为各临床病理特征的矩阵。
1.3 筛选DEGs 采用R语言“edgeR”包从蛋白编码基因矩阵中筛选胃癌及癌旁正常组织的DEGs,用于后续Cox比例风险回归模型分析。“edgeR”包通过命令首先剔除本底表达水平低的基因,分别计算癌症组和正常组的组间差异、组内差异,将组间差异大于组内差异的基因再进行配对t检验。筛选标准,校正后P值(FDR,False Discovery Rate)<0.01,差异倍数(Fold change,FC)>2,即FC取2的对数(log2FC)>1,或log2FC<-1。将DEGs数据可视化,以R“ggplot”包绘制火山图,并选择前20个上调、下调最显著的DEGs,采用R“pheatmap”包绘制热图。
1.4 Cox比例风险回归模型分析 本研究首先从TCGA下载临床文件,提取生存数据,将DEGs与生存数据合并生成一个行名为样本名,列名为生存时间、生存状态以及DEGs的矩阵,再采用R“survival”包Cox函数对DEGs做单因素回归分析,得到与胃癌预后有关的DEGs。在此基础上进行Cox多因素分析,得到胃癌预后评估模型,Risk Score=β1X1+β2X2+…+βnXn,β表示基因相关系数,X表示基因表达量。根据公式计算样本风险值(Risk Score),取中位数作为界值,将胃癌患者分为高、低风险组,并以R“pheatmap”包将模型数据可视化。同时,采用“survival”包做高、低风险Kaplan-Meier生存曲线,以“survival ROC”包绘制ROC曲线验证该模型的准确性。
1.5 Cox回归分析影响胃癌预后的因素 从胃癌临床文件提取相关信息、与生存文件及Risk Score文件合并成一个矩阵,包含生存时间、生存状态、性别、年龄、分化程度、TNM分期以及Risk Score(Lowvs.High)。先采用Cox单因素回归分析,再将分析结果中与胃癌预后相关的因素纳入Cox多因素回归分析,筛选影响胃癌预后的独立危险因素。
2 结果
2.1 TCGA胃癌数据DEGs分析结果 合并后的矩阵包含胃癌样本375例,癌旁正常样本32例,基因56 863个,按基因功能注释,提取出编码基因19 660个,差异表达分析共筛选出4 332个DEGs(FDR<0.01,|log2FC|>1),其中 2 145个 DEGs呈高表达,2 187个DEGs呈低表达。选取前20个上下调的DEGs,分别绘制热图及火山图,见图1。
2.2 Cox比例风险回归模型分析结果 采用R“survival”包的Cox函数对4 332个DEGs做单因素回归分析,结果显示,710个DEGs与胃癌OS有关(P<0.05)。选择P<0.001的25个DEGs纳入Cox多因素分析,得到包含8个DEGs胃癌预后评估模型,按公式计算每个样本的风险值,Risk Score=BCHE×0.071+INPP5J×(-0.151)+VCAN×0.166+IGFBP1×0.065+CGB5×0.123+HP×0.067+PSG9×0.154+MEI14×0.149,根据中位数将样本分为高、低风险组,并以可视化高低风险热图呈现,见图2。同时,采用R语言“survival”包的survdiff函数对8个DEGs进行批量Kaplan-Meier生存曲线分析,结果与Cox单因素分析结果相似,7个DEGs与胃癌预后相关,见图3。
2.3 Cox生存分析及ROC曲线 采用R“survival”包对预后评估模型呈高、低风险组进行Cox生存分析,结果显示,高风险组5年OS为56.20%,低风险组5年OS为17.27%,差异有统计学意义(χ2=39.232,P<0.001),见图4A。采用R“survival ROC”包绘制5年ROC曲线,判断风险评估模型的准确性,结果曲线下面积(AUC)=0.781,该风险评估模型预测胃癌预后有一定的准确性,见图4B。
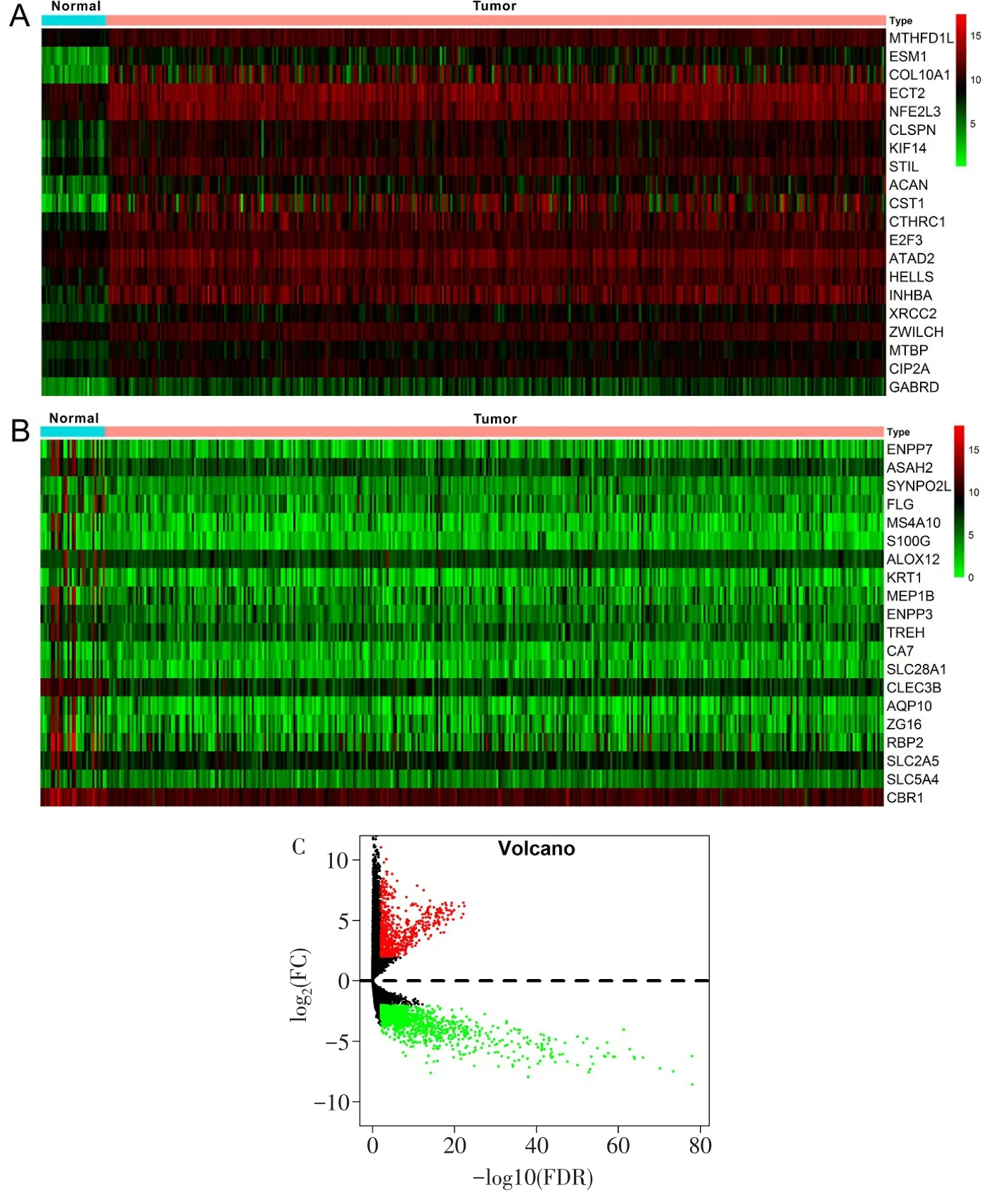
Fig.1 The heatmap and volcano map of DEGs in gastric cancer and normal tissues of TCGA图1 TCGA胃癌及正常组织DEGs热图和火山图

Fig.2 The heatmap of high-and low-risk score based on the predictive prognosis model of TCGA gastric cancer cases图2 TCGA胃癌预后评估模型高低风险热图
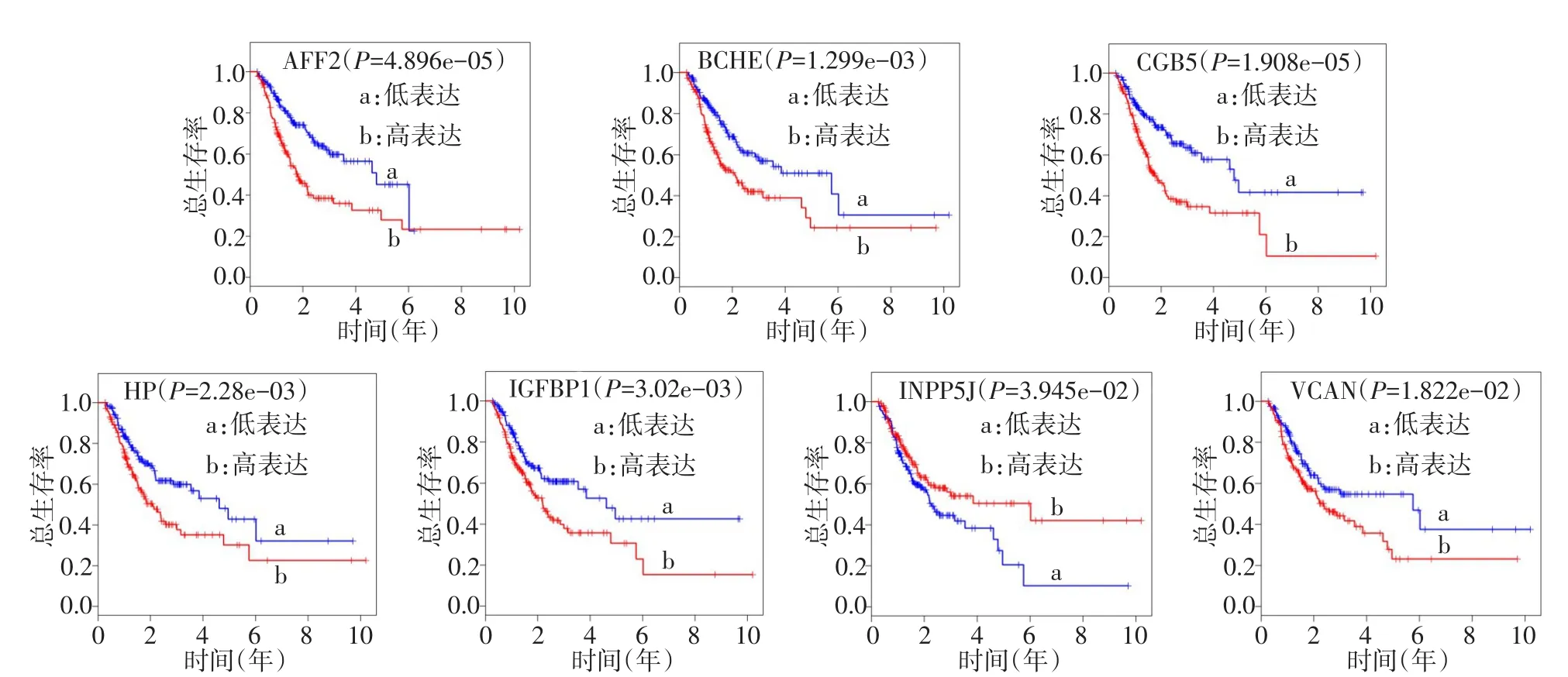
Fig.3 The Kaplan-Meier survival curves of each gene in the predictive prognosis model of gastric cancer图3 胃癌预后评估模型中各基因的Kaplan-Meier生存曲线图

Fig.4 The Kaplan-Meier survival curve(A)and ROC curve(B)of high-and low-risk of TCGA gastric cancer cases图4 TCGA胃癌高、低风险Kaplan-Meier生存(A)及ROC曲线图(B)
2.4 筛选影响胃癌预后的独立危险因素 以表1中的因素为自变量并给予赋值,以生存时间为因变量行Cox单因素回归分析,结果显示,年龄>67岁、肿瘤TNM分期晚(Ⅲ+Ⅳ期)、淋巴结转移、远处转移、风险模型评分为高风险是影响TCGA胃癌患者预后的危险因素(表2)。以表2中Cox单因素分析有统计学意义的因素为自变量,生存时间为因变量进一步行Cox多因素回归分析,结果显示高龄和风险评估模型评分为高风险是影响TCGA胃癌患者预后的独立危险因素,见表3。

Tab.1 Independent variable assignment of prognosis related factor in TCGA gastric cancer cases表1 TCGA胃癌患者预后相关因素赋值表
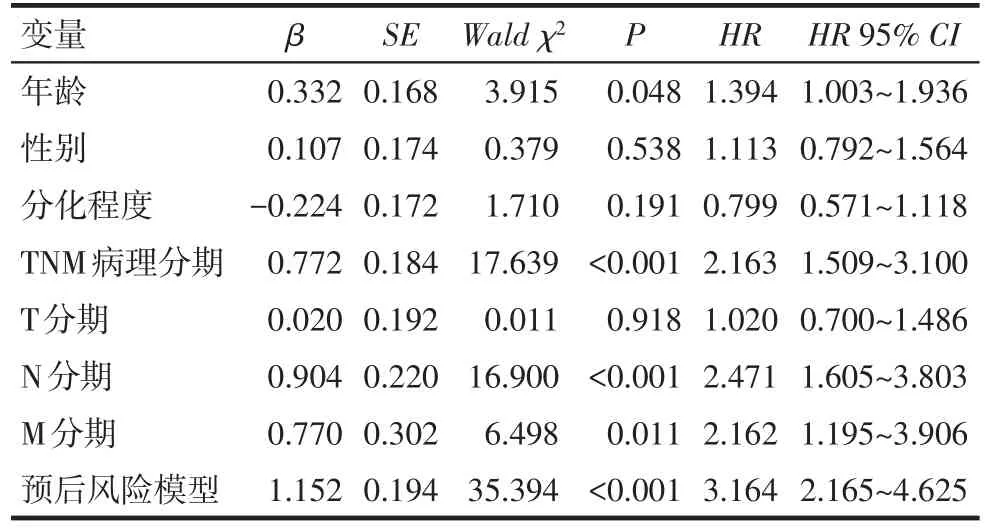
Tab.2 The Cox univariate regression of prognosis related factors for TCGA gastric cancer cases表2 TCGA胃癌患者预后相关因素的Cox单因素回归分析
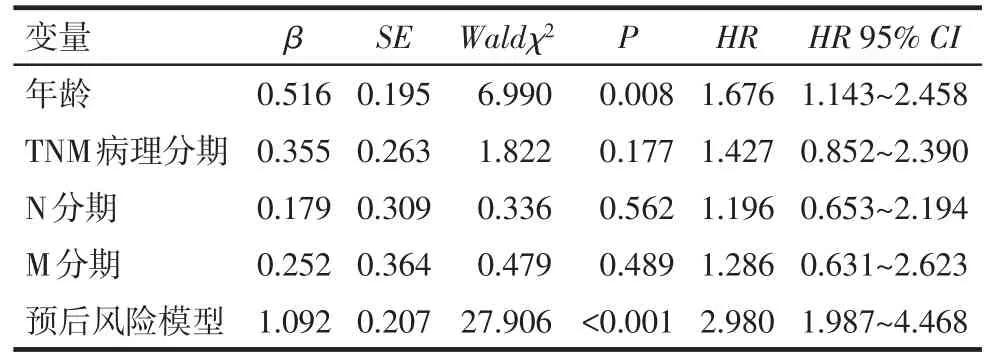
Tab.3 The Cox multivariate regression of prognosis related risk factors for TCGA gastric cancer cases表3 TCGA胃癌预后相关因素的Cox多因素回归分析
3 讨论
胃癌遗传学背景复杂,按分子生物学特征,可分为EB病毒(EBV)阳性、微卫星灶不稳定型(MSI)、基因组稳定型(GS)和染色体不稳定型(CIN)。TCGA数据库包含胃癌RNA-Seq样本375例,临床信息完整,为胃癌临床基础研究提供了重要的资源。本研究采用生物信息学方法,首先筛选出差异表达基因,纳入Cox比例风险回归分析,构建了包含8个DEGs(BCHE、INPP5J、VCAN、IGFBP1、CGB5、HP、PSG9、AFF2)的胃癌预后评估模型,通过Kaplan-Meier和ROC曲线验证了该模型的有效性和准确性,而且证实该模型可以用于预测胃癌的预后。BCHE、VCAN、IGFBP1、CGB5、HP、PSG9和AFF2可能是癌基因,而INPP5J是抑癌基因。鉴于在胃癌预后评估中的重要作用,这些基因可能在胃癌发生发展过程中发挥重要作用,可能成为胃癌精准治疗的新靶点,值得深入研究。
目前该预后评估模型的8个DEGs在胃癌及其他恶性肿瘤中的报道较少。Koie等[2-3]报道,血清BCHE水平是膀胱癌、前列腺癌的独立预后因素,而且血清BCHE增高的膀胱癌、前列腺癌患者5年OS和DFS均高于BCHE降低的患者,这似乎与本研究分析的结果相反,其原因在于肿瘤的异质性,另外,TCGA数据标本来源于肿瘤及正常组织,BCHE在肿瘤组织及血液中的表达可能不一致。Chida等[4]采用基因芯片技术筛选了与Ⅱ/Ⅲ期结肠癌复发相关的基因,包括VCAN,进一步免疫组化研究证实基质VCAN是判断结肠癌复发的重要指标。Li等[5]采用R语言包分析GEO数据库,筛选了与胃癌预后相关的12个节点基因,其中包括VCAN。IGFBP1是一种分泌性蛋白,调节细胞增殖、迁移、侵袭及黏附等生物学活性。Luo等[6]报道IGFBP1抑制胃癌细胞BGC-823的迁移活性,在幽门螺旋杆菌诱发的胃癌发生发展过程中起保护作用。Geis等[7]证实HIF-2α通过上调IGFBP1抑制肝癌细胞的淋巴结转移。这与本研究分析结果相反,肿瘤细胞的异质性可能是重要原因。Yang等[8]分析TCGA胃癌数据,同样证实CGB5是影响胃癌总生存率和无进展生存率的独立危险因素。结合珠蛋白(haptoglobin,HP)属急性期反应蛋白,也是一种分泌性蛋白,其分子结构类似免疫球蛋白,主要由肝脏合成,研究发现HP在肺癌[9]、胰腺癌[10]、肝癌[11]等多种肿瘤患者血清中增高。近来报道PSG9促进肿瘤新生血管生成,与结直肠癌、肝癌的发生发展密切相关[12-13]。Ooms等[14]报道抑癌基因INPP5J通过PI3K/AKT信号通路影响乳腺癌的发生发展,细胞、动物实验均证实,INPP5J敲减一方面促进肿瘤细胞增长,另一方面却抑制肿瘤细胞的迁移、侵袭。
综上文献所述,该预后评估模型中基因与恶性肿瘤发生发展密切相关,可以从不同角度去深入研究,有些基因可作为肿瘤早期诊断的分子标志物,如分泌性蛋白HP,有些基因可作为预后评估的指标,如BCHE、CGB5、VCAN,有些基因可作为精准治疗的新靶点,如PSG9。然而本研究还存在不足之处,第一,胃癌预后评估模型基于TCGA数据库RNA测序分析的结果,缺少临床及细胞、动物功能学实验。第二,某些基因,如BCHE、IGFBP1的TCGA分析结果与文献报道相反,肿瘤异质性是重要原因,数据分析的可靠性也需要考虑。
