基于TCGA数据挖掘筛选肺鳞癌预后相关lncRNA分子标签
2018-12-07何杨婷肖金荣王唤卓李旸凯
刘 颖,王 可,何杨婷,肖金荣,王唤卓,李旸凯,魏 晟,*
(1.华中科技大学同济医学院公共卫生学院流行病与卫生统计学系环境与健康教育部重点实验室,湖北 武汉 430030;2. 华 中科技大学同济医学院附属同济医院胸外科,湖北 武汉 430030)
肺癌是全世界发病率和死亡率最高的恶性肿瘤之一,且发病人数逐年上升[1-2]。作为肺癌的主要类型,非小细胞肺癌(non-small cell lung cancer,NSCLC) 占肺癌病例的85%~90%,肺鳞状细胞癌(肺鳞癌)是NSCLC的主要类型[3]。全球每年约40万人死于肺鳞癌。由于肺鳞癌易转移和缺乏用于诊断和预后的生物标志物,肺鳞癌患者5年生存率不足15%[4]。改善肺癌患者的预后情况是提高肺癌病人生存率的关键因素[5-6]。因此,寻找与肺鳞癌预后相关的生物标志物,通过生物标志物对肺鳞癌患者的预后情况进行早期评估,对提高肺鳞癌病人的预后及生存率非常重要。
长链非编码RNA(long non-coding RNA,lncRNA)是近年来才认识到的一种具有重要调控功能的分子标志物,其长度大于200 nt且不具备编码功能。当前lncRNA的功能仍在研究之中,但初步的研究结果发现其可通过促癌或抑癌作用,影响多种肿瘤细胞的增殖扩散或凋亡过程[7-9]。分子标签(signature)是指将一定数量的分子标记物进行组合,形成一个新的变量,并用此变量来判断或定义某些生物学特性。与单个标记物的分子模式不同,分子标签不单以单基因功能为研究基础,而且更加注重基因之间的共同协调作用,从整体和系统水平上对某种特定的生物学特性进行描述[10]。肿瘤基因组图谱数据库(the Cancer Genome Atlas,TCGA)数据库作为目前全球范围内最大的肿瘤公共数据库,对常见肿瘤提供了多种组学数据,是当前研究肿瘤组学数据的重要来源[11]。本研究通过对TCGA数据库中肺鳞癌转录组测序数据的挖掘,评估lncRNA表达水平对肺鳞癌病人进行预后预测的价值。
1 资料与方法
1.1 研究对象
2017年12月从TCGA网站(https://cancergenome.nih.gov/)下载肺鳞癌病人癌和癌旁组织的RNA测序数据及临床数据。RNA测序数据由肺鳞癌组织样本通过Illumina高通量测序平台进行测序得到。下载的RNA测序数据包含肺鳞癌病人496例癌组织的lncRNA表达数据以及48例癌旁组织的14 165个lncRNA表达数据。肺鳞癌患者的临床数据包括466例肺鳞癌的相关临床信息,如性别、年龄、种族、病理分期、吸烟状态、放疗、化疗情况及存活状态等。
1.2 研究方法
1.2.1 数据预处理 去除在肺鳞癌病人中生存时间显示为0的患者以及表达值缺失超过5% 的lncRNA。最终有48例肺鳞癌病人癌组织和癌旁组织的3 921个lncRNA位点用于差异表达分析;用于预后分析的数据集包括443例肺鳞癌病人的lncRNA表达数据和临床数据。
1.2.2 差异表达分析和热图绘制 对48例肺鳞癌病人癌组织和癌旁组织的3 921个lncRNA进行配对t检验,根据t检验对应的P值,采用BH法(Benjamini and Hochberg,1995)计算每个lncRNA的错误发现率(false discorvery rate,FDR)值[12]。同时计算癌和癌旁组织中lncRNA表达水平的倍数变化(fold change)。本次研究确定lncRNA差异表达的标准为FDR<0.05且倍数变化绝对值≥3[13]。其中,倍数变化≥3为lncRNA在肺鳞癌组织中表达水平上调,倍数变化≤1/3为lncRNA在肺鳞癌组织中表达水平下调。对筛选出的差异表达的lncRNA进行非监督聚类分析,并绘制热图(heatmap)。
1.2.3 lncRNA标志物筛选 采用LASSO(Least Absolute Shrinkage and Selector Operation)Cox回归进行肺鳞癌预后相关lncRNA筛选。并通过LASSO Cox的系数构建lncRNA分子标签。lncRNA分子标签=β1×EXPlncRNA1+β2×EXPlncRNA2+...+βn× EXPlncRNAn。 (EXP:lncRNA的表达水平,β:回归相关系数)。代入相关数据后,计算分子标签得分。分别通过lncRNA分子标签得分的中位数将肺鳞癌病人划分为高表达组和低表达组;lncRNA分子标签得分的四分位数将肺鳞癌病人划分为4组。计算不同风险组病人发生死亡的风险比(hazard ratio,HR)及95%置信区间(confidence interval,CI)。
1.2.4 构建预测模型 在lncRNA分子标签的基础上加入相关临床变量,如年龄、性别、种族、吸烟情况、放疗情况、化疗情况和临床病理分期,构建关于肺鳞癌预后的预测模型,并用Harrell’s C统计量对预测模型的预测效果进行评估,C统计量的范围在0.50~0.70时说明预测模型的准确性一般,当C统计量大于0.70时说明预测模型的准确性较优[14-15]。
1.3 统计学分析
应用SAS 9.4软件对数据进行统计分析。运用R 3.3.0软件和GraPad- Prism 5软件进行相关图形制作。连续性资料表示为x±s。运用t检验的方法比较两样本之间均数的差异情况。计数资料之间的差异性分析釆用χ2检验或确切概率计算法。
2 结 果
2.1 纳入对象基本情况
符合条件的肺鳞癌患者443人,其中男性患者死亡220人(73.33%),未死亡108人(75.52%);女性患者死亡80人(26.67%),未死亡35人(24.48%);肺鳞癌死亡患者平均年龄(66.9±8.5)岁,未死亡患者平均年龄(68.9±7.8)岁。种族、吸烟、放疗、化疗、生存时间等情况见表1。
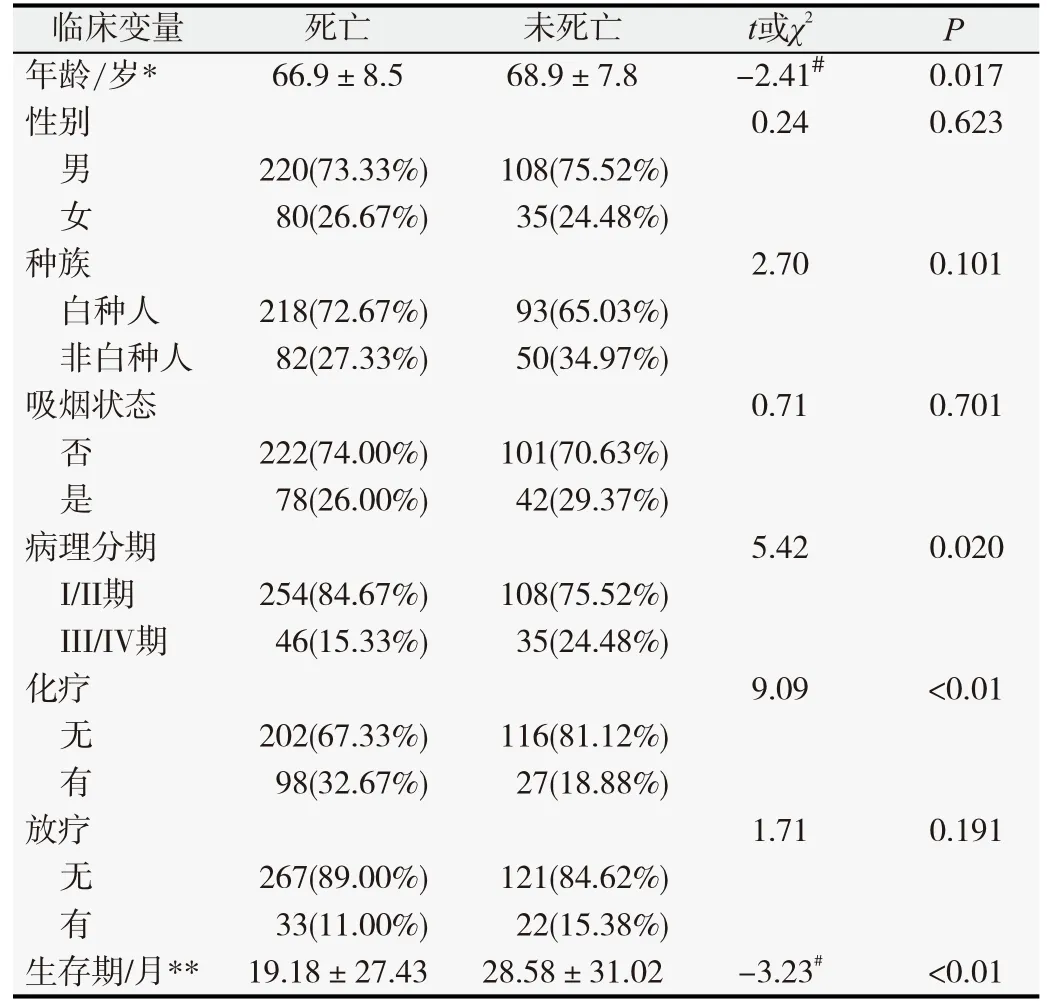
表1 从TCGA数据库中纳入分析的肺鳞状细胞癌患者的基本特征(n=443)
2.2 癌组织和癌旁组织中差异表达的lncRNA
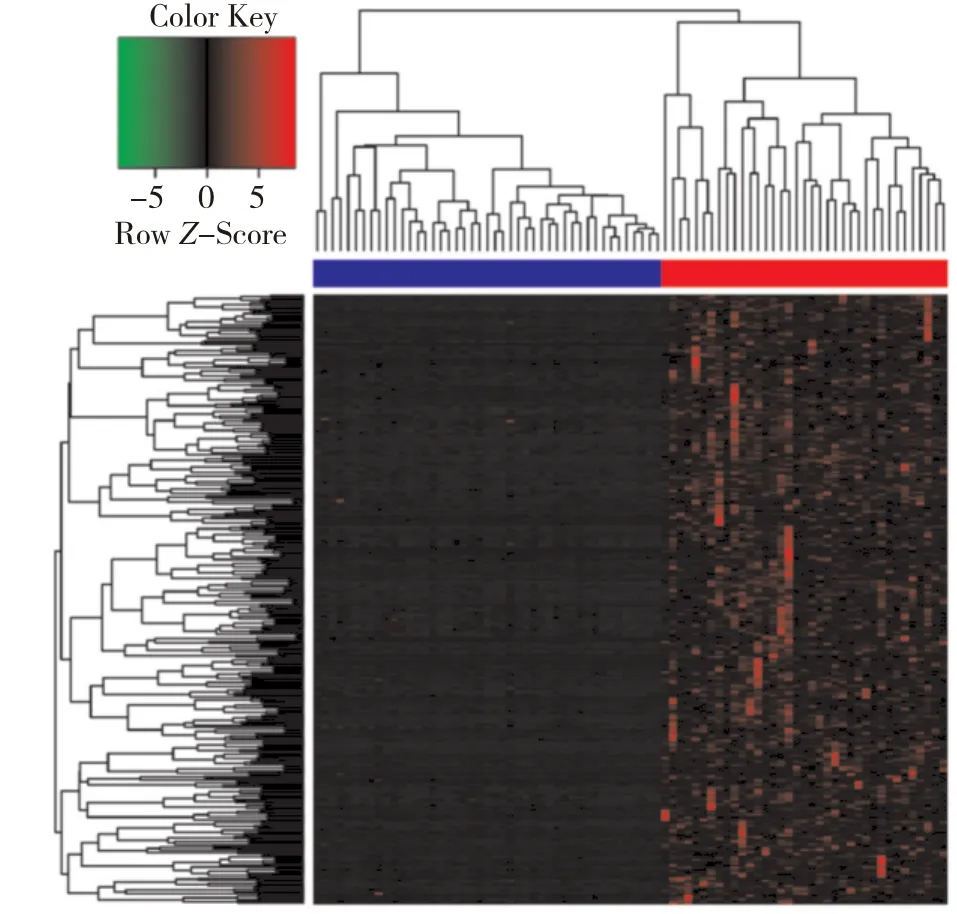
对48例肺鳞癌病人癌和癌旁组织的lncRNA位点进行配对t检验,经分析满足条件FDR<0.05且倍数变化绝对值≥3的lncRNA共有322个,所有lncRNA在癌组织中均表达上调。对差异表达的lncRNA进行无监督聚类分析,结果以热图的形式呈现,图中红色表示癌组织,蓝色表示癌旁组织,见图1。
2.3 LASSO Cox回归筛选与肺鳞癌预后相关的lncRNA
在322个具有差异的lncRNA位点中,筛选出6个lncRNA进入模型,分别为ENSG00000186615.9(KTN1-AS1)、 ENSG00000204949.7(FAM83A-AS1)、 ENSG000 00232855.5(AF131217.1)、ENSG00000258592.1(RP11-108M12.3)、 ENSG00000259230.1(CTD-2555C10.3)、ENSG00000278514.1(AC068831.16)。lncRNA在TCGA数据库中的基因注释信息以及经LASSO Cox回归分析得到的回归系数和lncRNA在癌和癌旁组织中的倍数变化及FDR值情况见表2。肺鳞癌和癌旁组织中这些lncRNA表达情况见图2。6个lncRNA均在癌组织中出现表达上调,差异有统计学意义(FDR<0.01)。
对LASSO Cox回归筛选出的6个lncRNA及其系数构建lncRNA分子标签,分子标签值计算公式为:
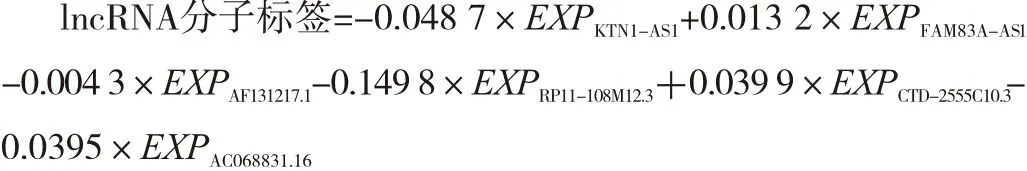
(EXP:基因表达水平)
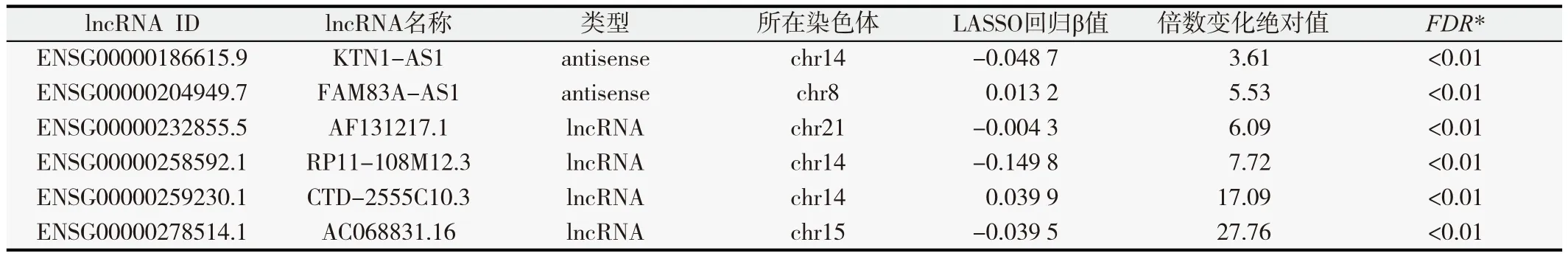
表2 从TCGA数据库中筛选出6个lncRNA的注释信息及其在癌组织和癌旁组织中的表达情况比较
2.4 lncRNA分子标签与肺鳞癌预后的关系
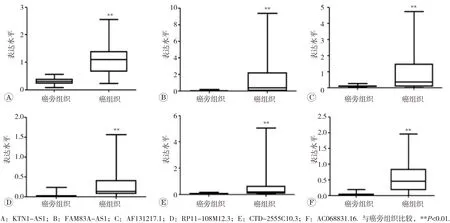
图2 LASSO Cox回归筛选出6个lncRNA在肺鳞癌癌旁组织和癌组织中的表达水平
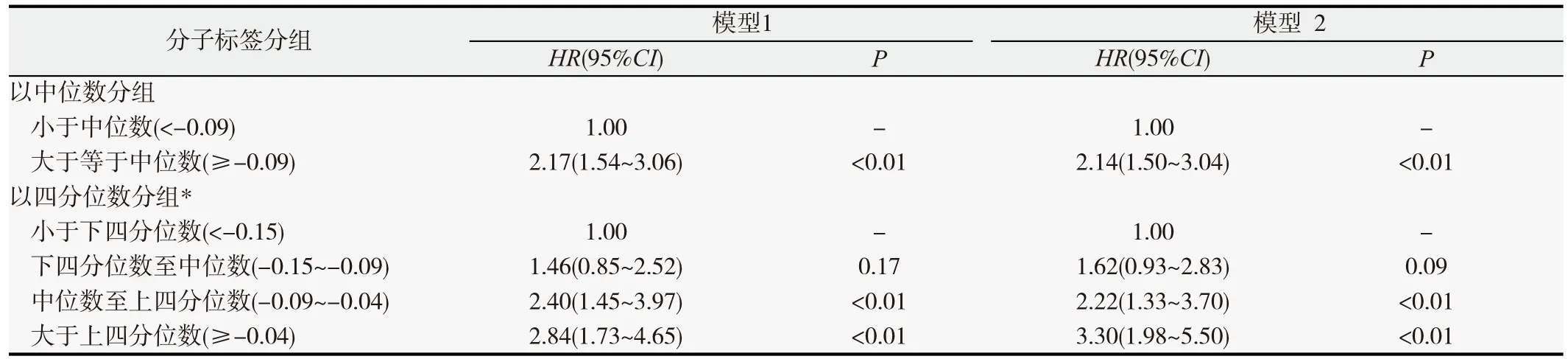
表3 lncRNA分子标签与肺鳞癌预后之间的关系
采用Cox回归分析方法,建立2个基于lncRNA分子标签的肺鳞癌预后预测模型。模型1未调整变量,模型2调整年龄、性别、种族、吸烟状态、化疗情况、放疗情况和病理分期变量。结果见表3。调整相应临床变量后,高表达组肺鳞癌病人的死亡风险是低表达组病人的2.14倍,且差异具有统计学意义(HR=2.14,95%CI: 1.50~3.04,P<0.01)。当根据分子标签表达水平的四等分点-0.15、-0.09、-0.04将肺鳞癌患者划分为4组时,分子标签值位于下四分位数和中位数之间时,HR为1.62(95%CI:0.93~2.83,P=0.09);分子标签值位于中位数与上四分位数之间时,HR为2.22(95%CI:1.33~3.70,P<0.01);分子标签值大于上四分位 数 时 , HR为 3.30(95%CI: 1.98~5.50, P<0.01);lncRNA分子标签的Kaplan Meier生存曲线如图3所示,调整变量后随着分子标签水平的升高,肺鳞癌病人发生死亡的风险逐渐增加(P<0.01)。
2.5 lncRNA分子标签的预测模型对肺鳞癌病人预后的预测价值
Cox生存分析结果显示,单独使用lncRNA分子标签进行肺鳞癌病人预后状况预测时,C统计量的结果为0.63(95%CI:0.58~0.69)。当加入临床变量年龄、性别、种族、吸烟、放疗、化疗、病理分期后,其预测模型的C统计量为0.69(95%CI:0.64~0.75)。两者相比,差异具有统计学意义(P=0.015)。通过模型效果评价标准可以看出,当在lncRNA分子标签的基础上加入临床变量构建预后预测模型时,其预测效果进一步优化,表明包含lncRNA分子标签和临床变量的预测模型可以对肺鳞癌病人的预后状况进行有效预测。
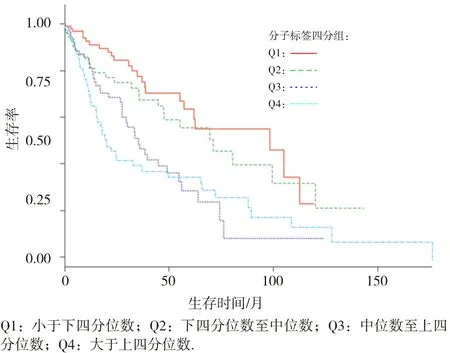
图3 lncRNA分子标签表达水平不同的肺鳞癌病人的生存曲线比较
3 讨 论
本研究采用TCGA数据库中lncRNA表达数据探讨与肺鳞癌预后相关的lncRNA标志物。经过差异表达分析及LASSO Cox回归发现6个与肺鳞癌预后相关的lncRNA位点并组成lncRNA分子标签,进一步进行临床指标的相关分析。结果显示,随着分子标签水平的升高,肺鳞癌病人发生死亡的风险逐渐增加。当根据lncRNA分子标签的中位数将人群分为高表达组和低表达组时,高表达组病人的死亡风险是低表达组肺鳞癌病人2.14倍。当调整了性别、年龄、吸烟状态、种族、病理分期、化疗情况、放疗情况构建预后预测模型后,其C统计量的结果增加至0.69。由以上结果可见,基于lncRNA分子标签的预测模型具有较为良好的效能,可以对肺鳞癌病人的预后状况进行有效预测。
本研究发现的6个lncRNA位点在包括肺鳞癌在内的多种肿瘤发生发展中的作用,在以往的研究中已经有报道。lncRNA KTN1-AS1的高表达是头颈部鳞状细胞癌预后的危险因素,且其参与构建的3-lncRNA 标志物能较好预测患者的生存情况[16]。lncRNA FAM83AAS1可通过促进MAPK信号传导通路发挥致癌作用,促使乳腺上皮等细胞癌变,也可通过激活EGFR下游传导信号促进肿瘤细胞增殖侵袭和EGFR酪氨酸激酶抑制剂耐药,高表达预示乳腺癌预后不良[17-18]。基于TCGA数据库的研究中,lncRNA RP11-108M12.3在肺鳞癌组是不良预后的保护因素,与本研究结果一致,且其参与构建的5-lncRNA标志物也具有独立的预后价值[19]。LncRNA CTD-2555C10.3的高表达是肺腺癌的预后危险因素,其参与构建的7维转录组分子标签对肺腺癌预后有较好的指示作用[20]。
本次研究采用TCGA数据库在全基因组范围内挖掘肺鳞癌预后相关lncRNA,检测方法一致,样本量较大,可信度较高。首先,本研究采用LASSO Cox回归的方法筛选肺鳞癌预后相关的lncRNA标志物,避免了样本量远小于自变量带来的多重共线性问题和减小了II类错误。第二,本研究将单个lncRNA构建分子标签,进一步构建lncRNA预测方程比单个lncRNA位点具有更好的预后预测效果。
当然,本研究也存在一定的局限性:第一,本研究构建的预测模型仅考虑了lncRNA的表达,未考虑其他水平的生物标志物对肺鳞癌病人预后的影响,如甲基化、以及DNA序列变异等,因此其应用价值还有待进一步的研究;第二,本研究中预测模型建立在美国肺鳞癌人群,但其在中国肺鳞癌人群中的预测效果如何仍需要进一步验证。
综上所述,经TCGA转录组测序数据库的挖掘,筛选出lncRNA 分子标签及其和临床变量构建的预后预测模型对肺鳞癌预后有较好的预测价值,为肺鳞癌的临床预后判断提供了更多可供选择的生物标志物。
